pAPN基因敲除的IPEC-J2介導的TGEV感染特征分析

摘 要: 旨在探究豬氨基肽酶N(porcine aminopeptidase N,pAPN)基因敲除的豬空腸上皮細胞系(intestinal porcine epithelial cell line J2,IPEC-J2)介導豬傳染性胃腸炎病毒(transmissible gastroenteritis virus,TGEV)感染的特征,為深入了解pAPN基因在TGEV感染過程中的作用機制提供理論依據。研究分為pAPN基因敲除IPEC-J2組(IPEC-J2-KO組)、野生型IPEC-J2組(IPEC-J2-WT組)和未接種TGEV的野生型IPEC-J2組(Mock組)3組,每組設置3個重復。首先通過實時熒光定量PCR(quantitative real-time PCR,qPCR)確定IPEC-J2接種TGEV毒株后收取細胞樣品的最佳時間節點;其次,對IPEC-J2-KO進行了脫靶效應檢測;然后,通過qPCR、蛋白免疫印跡(western blot,WB)、間接免疫熒光分析(indirect immunofluorescence assay,IFA)和50%組織細胞感染量(50% tissue culture infective dose,TCID50)對接種TGEV的IPEC-J2-KO、IPEC-J2-WT及Mock進行感染特征分析;最后,通過WB檢測IPEC-J2-KO、IPEC-J2-WT和Mock中NF-κB p65及其磷酸化蛋白pp65的表達情況。qPCR結果顯示,接毒24 h是收集細胞樣本以評估TGEV對IPEC-J2影響的最佳時間節點;脫靶分析結果顯示,在IPEC-J2-KO中未檢測到脫靶效應;病毒感染特征分析結果顯示,與IPEC-J2-WT相比,IPEC-J2-KO內病毒拷貝數、病毒滴度均極顯著降低(Plt;0.001),與Mock相比,IPEC-J2-KO內病毒拷貝數、病毒滴度均無顯著差異(Pgt;0.05),且IPEC-J2-KO內未檢測到TGEV-N蛋白的表達;此外,與Mock相比,接種TGEV后,IPEC-J2-WT組NF-κB p65的磷酸化水平極顯著上調(Plt;0.001),而IPEC-J2-KO組無顯著差異(Pgt;0.05)。本研究表明,IPEC-J2-KO可有效抵抗TGEV的感染,接種TGEV未影響IPEC-J2-KO中先天免疫相關信號通路中轉錄因子NF-κB的活性。該研究為IPEC-J2作為TGEV感染特征研究的細胞模型提供了理論依據,為闡明pAPN基因在TGEV入侵宿主細胞的機制及抗病豬新品種的研究奠定了基礎。
關鍵詞: 豬氨基肽酶N;IPEC-J2;TGEV;NF-κB
中圖分類號:S813.1
文獻標志碼:A
文章編號:0366-6964(2024)08-3395-13
收稿日期:2024-01-23
基金項目:深圳市科技計劃項目(CJGJZD20210408092402006);中國農業科學院科技創新工程(ASTIP-IAS05)
作者簡介:夏振濤(1999-),男,河南鹿邑人,碩士生,主要從事動物遺傳育種與繁殖的研究,E-mail:17093646235@163.com
通信作者:牟玉蓮,主要從事動物遺傳育種與繁殖的研究,E-mail:mouyulian@caas.cn
Characteristics Analysis of TGEV Infection Mediated by IPEC-J2 with Knockout of
pAPN Gene
XIA" Zhentao1, WANG" Nan1, WANG" Wanjie1, ZHOU" Qilü1, HUANG" Lei2, MU" Yulian1*
(1.Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193," China;
2.Agricultural Genomics Institute at Shenzhen, Chinese Academy of Agricultural Sciences, Shenzhen
518120," China)
Abstract:" The aim of this study was to explore the characteristics of porcine transmissible gastroenteritis virus (TGEV) infection mediated by porcine aminopeptidase N (pAPN) gene knockout intestinal porcine epithelial cell line J2 (IPEC-J2) and to provide theoretical basis for further understanding the mechanism of pAPN gene in the process of TGEV infection. The study was divided into 3 groups, pAPN gene knockout IPEC-J2 group (IPEC-J2-KO group), wild-type IPEC-J2 group (IPEC-J2-WT group), and wild-type IPEC-J2 group not inoculated with TGEV (Mock group), with 3 replicates set up in each group. Firstly, quantitative real-time PCR (qPCR) was used to determine the best time point for collecting cell samples following inoculation with TGEV strain. Secondly, the off-target effect of IPEC-J2-KO was detected. Then, the infection characteristics of IPEC-J2-KO, IPEC-J2-WT inoculated with TGEV and Mock were analyzed by qPCR, western blot (WB), indirect immunofluorescence assay (IFA), and 50% tissue culture infective dose (TCID50). Finally, the expression of NF-κB p65 and its phosphorylated protein pp65 in Mock, IPEC-J2-WT and IPEC-J2-KO were detected by WB. The results of qPCR showed that 24 hours after exposure was the best time point to collect cell samples to evaluate the impact of TGEV on IPEC-J2; The off-target analysis results showed that no off-target effects were detected in IPEC-J2-KO; The results of virus infection characteristic analysis showed that the virus copy number and virus titer in IPEC-J2-KO were significantly lower than those in IPEC-J2-WT (Plt;0.001). Compared with the Mock, there was no significant difference in virus copy number and virus titer in IPEC-J2-KO (Pgt;0.05), and no expression of TGEV-N protein was detected in IPEC-J2-KO. In addition, compared with the Mock, after inoculation with TGEV, the phosphorylation level of NF-κB p65 was significantly increased in the IPEC-J2-WT group (Plt;0.001), while there was no significant difference in IPEC-J2-KO group (Pgt;0.05). This study results showed that IPEC-J2-KO can effectively resist TGEV infection, and TGEV did not affect the activity of the transcription factor NF-κB in the innate immunity-related signaling pathway in IPEC-J2-KO. This study provided the evidence that IPEC-J2 could serve as a cell model for the study of TGEV infection characteristics, and laid the foundation for elucidating the mechanism of pAPN gene in TGEV invading host cells and researching new disease-resistant pig varieties.
Key words: porcine aminopeptidase N; IPEC-J2; TGEV; NF-κB
*Corresponding author:MU Yulian, E-mail; mouyulian@caas.cn
豬傳染性胃腸炎(transmissible gastroenteritis,TGE)是由豬傳染性胃腸炎病毒(transmissible gastroenteritis virus,TGEV)誘發的高度接觸性消化道傳染病,主要導致豬的急性腹瀉、脫水、嘔吐和死亡,2周齡內的仔豬感染TGEV的病死率高達100%,給全球生豬養殖產業帶來了巨大經濟損失[1]。TGEV首次報道于1946年[2],隨后TGE在歐洲、亞洲、非洲和南美等許多國家爆發,中國自1958年以來開始有TGE的相關報道,TGE在我國豬群中的傳播呈現明顯的季節性、散發性和區域性特點[3]。TGEV是RNA病毒,其基因組具有較高重組性和突變率[4],在中國不同地區分離了TGEV HN-2012[5]、TGEV HB-1[6]、TGEV NMG[7]和TGEV-FJ[8]等不同株系的突變毒株。同時,流行現狀分析表明,TGEV的隱性感染以及與豬輪狀病毒(porcine rotavirus,PoRV)、豬德爾塔冠狀病毒(porcine deltacoronavirus,PDCoV)等腸道病毒的混合感染現象普遍存在[9-10],使TGE的防治更為困難。研究發現,一種可引發人類肺炎的新型犬冠狀病毒(CCoV-HuPn-2018)與TGEV Purdue株的全基因組有90%以上的相似性,提示TGE可能存在成為人畜共患病的潛在風險[11-12]。因此,探索TGEV的發病機制對于控制TGE的流行具有重要意義。
TGEV屬于冠狀病毒科、α冠狀病毒屬的一員,是一種單股正鏈RNA病毒,其基因組長約28.5 kb[13],病毒基因組結構包括9個開放閱讀框,編碼了4種不同的結構蛋白,包括核衣殼(N)蛋白、膜(M)蛋白、刺突(S)蛋白和包膜(E)蛋白以及3種輔助蛋白[14]。其中,S蛋白作為病毒表面重要的標志蛋白可特異性結合宿主細胞表面受體,決定了TGEV的組織嗜性和毒力[15]。氨基肽酶N(aminopeptidase N,APN)是一種II型整合性膜蛋白,又稱白細胞分化抗原13(cluster of differentiation 13,CD13),以胞外酶形式存在于細胞膜上[16],是多種冠狀病毒的受體,介導如人巨細胞病毒(human cytomegalovirus,HCMV)和人冠狀病毒229E(human coronavirus 229E,HCoV-229E)等多種病原體的感染[17-19]。1992年Delmas等[20]通過TGEV中和試驗確認了pAPN(porcine aminopeptidase N,pAPN)是TGEV入侵宿主的關鍵受體。研究表明,TGEV的S蛋白可與pAPN相互作用引發細胞感染[15]。pAPN基因在非易感細胞系犬腎細胞(madin darby canine kidney,MDCK)、非洲綠猴腎細胞(verda reno,Vero)和倉鼠腎成纖維細胞(baby hamster kidney-21,BHK-21)中過表達可使這些細胞對TGEV易感[21-22]。另有研究表明,利用成簇規律間隔短回文重復序列/CRISPR相關蛋白9(clustered regularly interspaced short palindromic repeats/CRISPR associated protein 9,CRISPR/Cas9)技術在豬睪丸細胞(swine testis,ST)與豬回腸上皮細胞(immortal pig intestinal-2I,IPI-2I)中敲除pAPN基因可顯著抑制TGEV感染[21,23]。Xu等[24]利用CRISPR/Cas9技術和體細胞克隆技術制備的分化抗原163(cluster of differentiation 163,CD163)和pAPN雙基因編輯豬可以抵抗TGEV感染,同時豬的繁殖和生產性能保持正常。上述研究證實pAPN是TGEV感染的必需受體。
冠狀病毒感染后,機體會產生與控制病毒感染及啟動適應性免疫反應相關的細胞因子,參與對外界刺激的響應,核因子κB(nuclear factor kappa-B,NF-κB)通路被認為是一種典型的促炎信號通路[25]。有研究表明,TGEV感染后NF-κB信號通路在多種易感細胞系,如豬空腸上皮細胞系(intestinal porcine epithelial cell line J2,IPEC-J2)[26]、ST細胞[27]和豬腎細胞(porcine kidney 15,PK15)[28]中被激活。Wang等[27]的研究結果顯示,TGEV-Nsp2(non-stuctural protein2,nsp2)是參與炎癥調節的關鍵病毒蛋白;Zhou等[28]的研究發現,TGEV-Nsp14(non-stuctural protein14,nsp14)以NF-κB依賴性方式來誘導干擾素β(interferon-β,IFN-β)的產生。Guo等[26]研究發現,TGEV的輔助蛋白ORF3b(open reading frame 3b,ORF3b)可能在感染早期通過抑制NF-κB途徑的激活來保護宿主細胞免于死亡,從而有利于TGEV的復制。上述研究表明,深入研究NF-κB活性在TGEV感染中的作用機制可為理解TGEV感染的病理生理過程提供思路。
目前,pAPN已被證實是TGEV的關鍵受體,且來源于豬空腸的IPEC-J2是了解豬腸道冠狀病毒感染致病機制的重要細胞模型[29-31],但pAPN基因敲除的IPEC-J2介導的TGEV感染的特征還未見報道。本研究利用王曉朋等[32]制備的pAPN基因敲除IPEC-J2,研究其在TGEV感染中的特征,為深入了解pAPN基因在TGEV感染過程中的作用機制和培育基因編輯抗病豬提供理論依據。
1 材料與方法
1.1 細胞與毒株
本研究中野生型IPEC-J2細胞系(IPEC-J2-WT)由華中農業大學何啟蓋老師惠贈,pAPN基因敲除的IPEC-J2細胞系(IPEC-J2-KO)[32]和豬腎細胞系(LLC-PK1)為北京畜牧獸醫研究所動物基因工程與種質創新團隊提供,TGEV-WH1毒株(GenBank:HQ462571)由華中農業大學肖少波老師惠贈。
1.2 主要試劑
M-PERTM哺乳動物蛋白抽提試劑(78501)、胎牛血清(10099-141)、青霉素/鏈霉素(5140-122)、DMEM(12100-046)、0.25% Trypsin-EDTA(25200-072)以及PBS(20012027)均購自賽默飛世爾科技(中國)有限公司;CD13/ANPEP Rabbit mAb(A5662)購自武漢愛博泰克生物科技有限公司;TGEV-N抗體由華中農業大學肖少波老師惠贈;GAPDH抗體(2118S)與熒光染料DAPI(sc-3598)購自Santa Cruz Biotechnology;Anti-NF-κB p65 Rabbit pAb(GB 11997—100)與Anti-Phospho-NF-κB p65(S536)Rabbit pAb(GB 113882—100)購自武漢賽維爾生物科技有限公司;RNA-easy Isolation Reagent(R701-01)購自南京諾唯贊生物科技有限公司;反轉錄試劑盒(RR047A)購自寶日醫生物技術(北京)有限公司;2×RealStar Fast染料法qPCR預混液(A304-10)購自北京康潤誠業生物科技有限公司;DyLight 488-山羊抗小鼠IgG(BA1126)購自武漢博士德生物工程有限公司。
1.3 細胞的培養
LLC-PK1細胞、IPEC-J2-WT與IPEC-J2-KO細胞均接種于含有15%胎牛血清、1%青霉素/鏈霉素的DMEM完全培養基中,在37℃和5% CO2的條件下培養,當細胞生長約80%的匯合度,使用濃度為0.1%的Trypsin-EDTA消化進行細胞傳代或凍存。
1.4 細胞接毒試驗
本研究中,為確定IPEC-J2接種TGEV最佳時間節點,設置接毒0、6、12、24、36與48 h不同時間處理組,每組設置3個重復。為探究IPEC-J2-KO的感染特征及對NF-κB活性的影響,設置未接種TGEV的野生型IPEC-J2組(Mock組)、接種TGEV的IPEC-J2-WT組和接種TGEV的IPEC-J2-KO組3組,每組設置3個重復。接毒步驟如下:待細胞的匯合度達到約80%,接種TGEV-WH1毒株,感染復數(multiplicity of infection,MOI)為1,接毒后于4℃吸附1 h,隨后使用PBS洗去未吸附的TGEV-WH1病毒粒子,在37℃、5% CO2的細胞培養箱中繼續培養,并于不同時間點收集細胞樣品。
1.5 IPEC-J2-KO的脫靶效應檢測
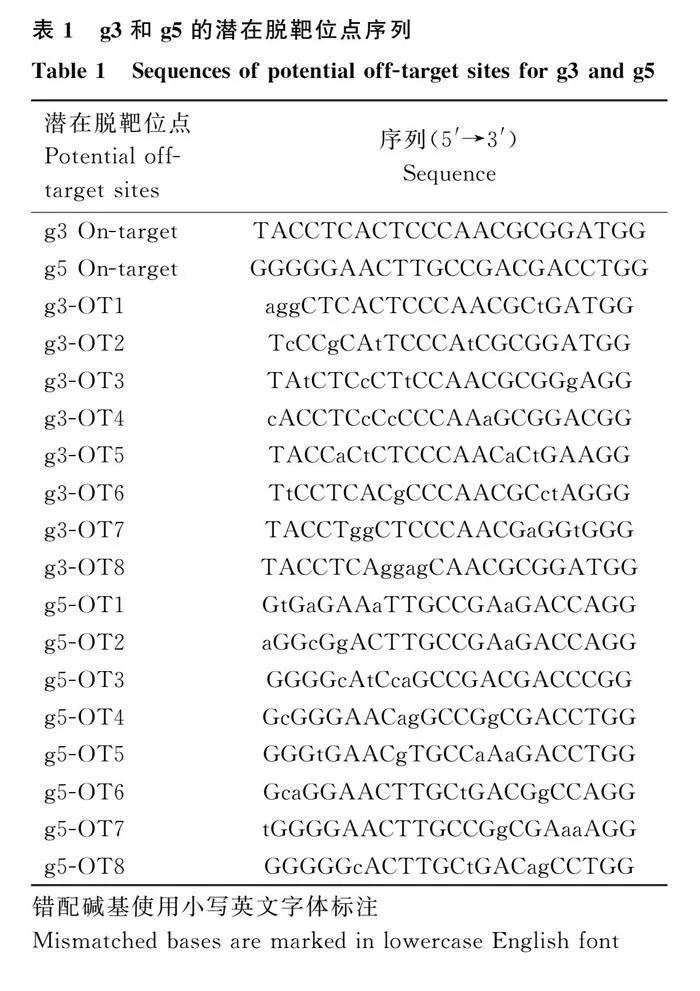
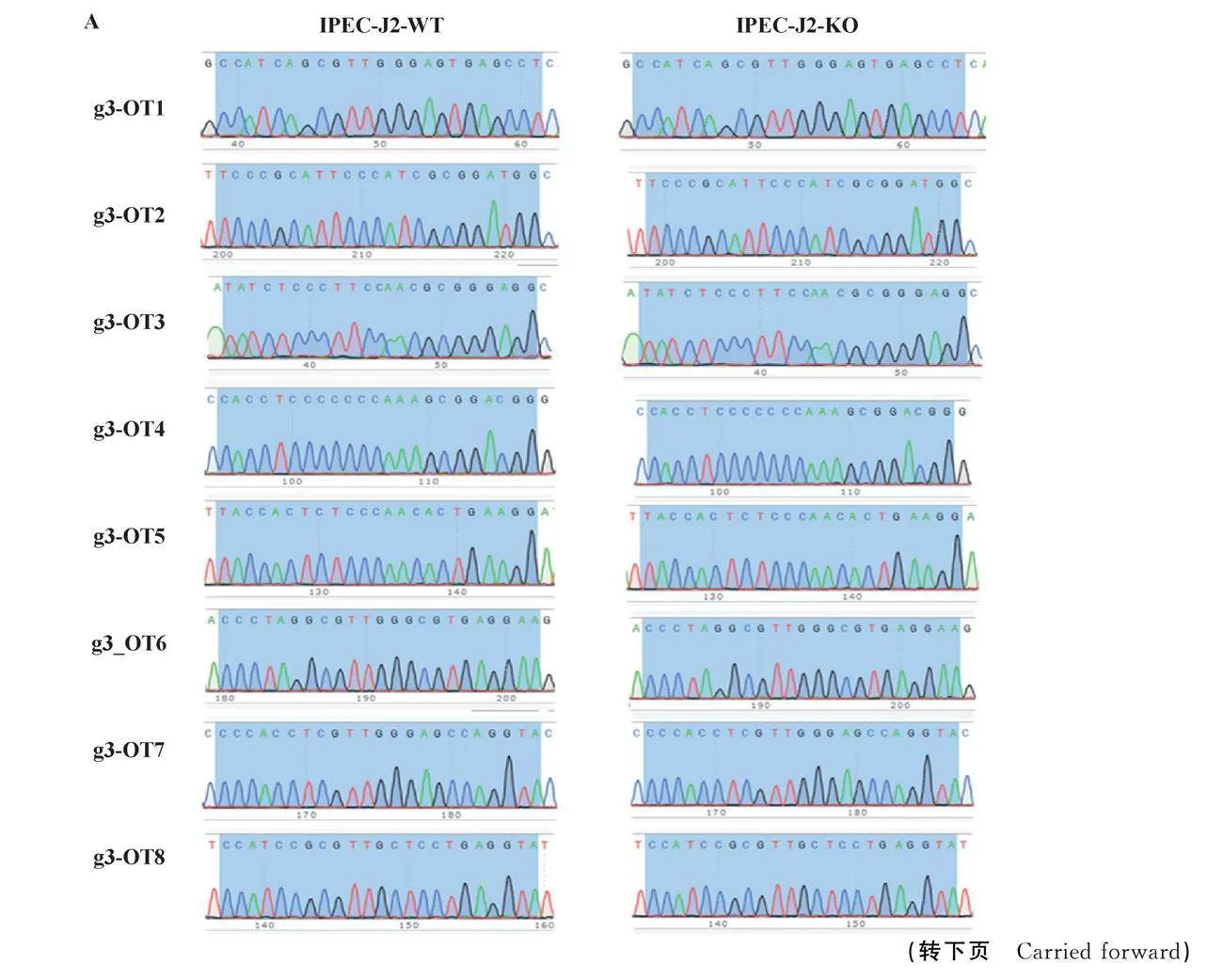
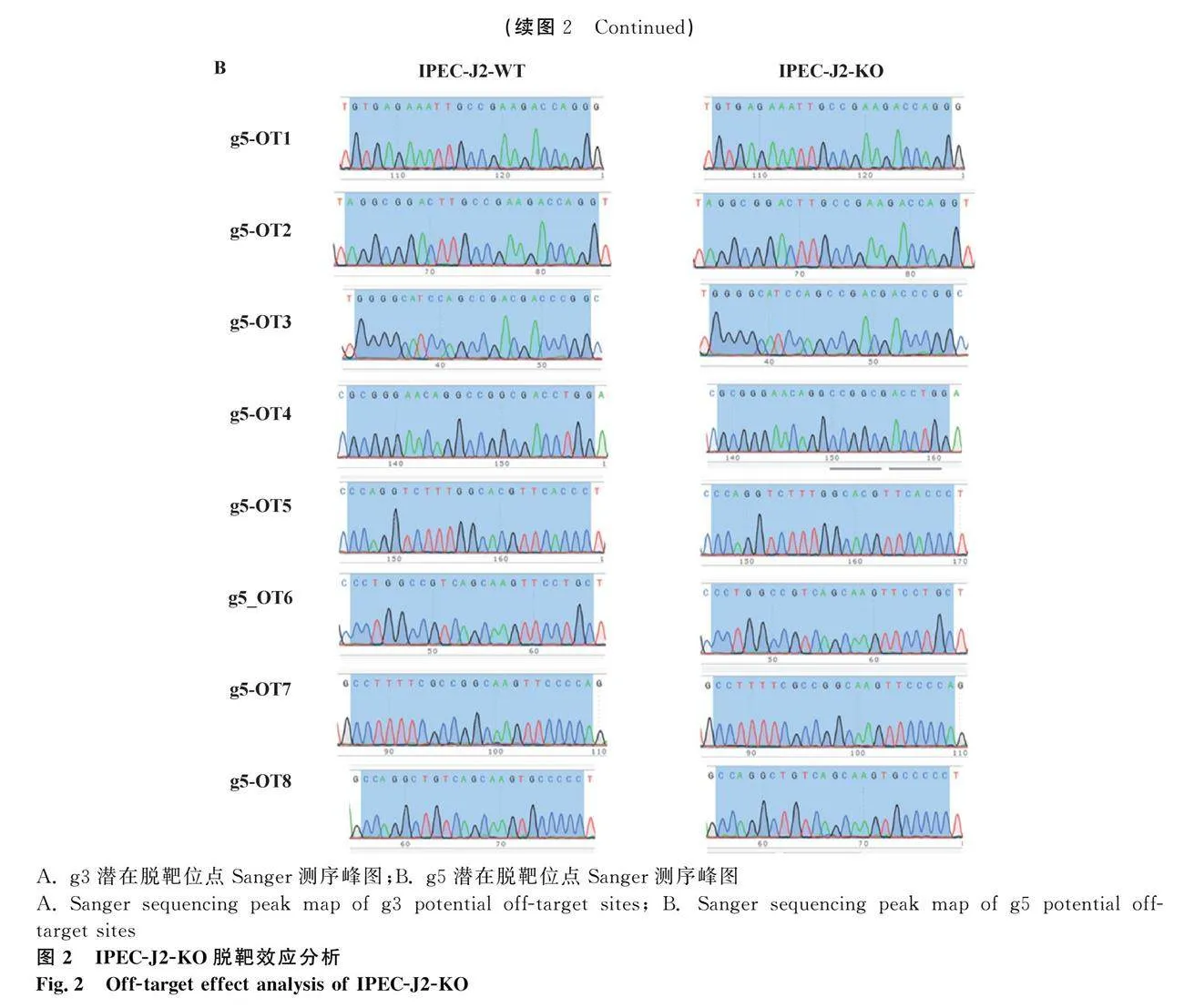
為評估IPEC-J2-KO是否存在脫靶,本試驗利用CRISPOR(http:∥crispor.tefor.net/)網址對王曉朋等[32]構建的IPEC-J2-KO中所使用的向導RNA(guide RNA,gRNA)g3與g5的潛在脫靶位點進行預測,每條gRNA各選取8個潛在脫靶位點進行脫靶效應檢測(表1),針對上述潛在的脫靶位點設計相應的擴增引物(表2),并對IPEC-J2-WT與IPEC-J2-KO的DNA樣品進行PCR擴增,PCR產物進行Sanger測序,通過對比IPEC-J2-WT與IPEC-J2-KO的DNA序列判斷g3與g5是否存在脫靶效應。
1.6 TGEV病毒基因組RNA拷貝數檢測
實時熒光定量PCR(quantitative real-time PCR,qPCR)檢測接毒后IPEC-J2-WT與IPEC-J2-KO中TGEV基因組拷貝數。提取IPEC-J2-WT與IPEC-J2-KO的RNA,反轉錄后得到cDNA,使用檢測引物(TGEV-N-F:5'-GAGTTGTCTGGGTTGCCAAG-3';TGEV-N-R:5'-TGGATTGTTGCCTGCCTCTA-3')進行qPCR檢測;反應體系為:qPCR預混液10 μL、ddH2O 7.2 μL、TGEV-N-F 0.4 μL、TGEV-N-R 0.4 μL、cDNA 2 μL;反應程序為:95℃預變性2 min;95℃變性5 s,60℃退火34 s,共40個循環;95℃ 15 s,60℃ 1 min,95℃ 15 s。將含有TGEV-N端基因的質粒作為標準品并按照梯度進行稀釋,質粒稀釋后拷貝數分別為:1×109、1×108、1×107、1×106、1×105、1×104(copies·μL-1),根據稀釋梯度做出相應的標準曲線,并以此計算細胞樣品中TGEV病毒基因組拷貝數。
1.7 蛋白免疫印跡(western blot,WB)檢測
提取接毒后IPEC-J2-WT與IPEC-J2-KO的蛋白質,以GAPDH為內參,檢測pAPN、TGEV-N、NF-κB p65與pp65的相對表達情況。使用BCA法測定細胞裂解液中蛋白濃度,將細胞中的蛋白樣品于100℃溫度條件下變性,IPEC-J2蛋白質樣品經12.5%的聚丙烯酰胺凝膠電泳分離后轉至0.45 μm的PVDF膜,使用Western快速封閉液室溫封閉10 min,隨后一抗4℃過夜孵育,二抗室溫孵育45 min,最后使用全自動化學發光圖像分析系統顯色并拍照。
1.8 間接免疫熒光分析(indirect immunofluorescence assay,IFA)檢測
接種TGEV毒株24 h后,棄去IPEC-J2-WT與IPEC-J2-KO細胞維持培養液,細胞經固定、透化、封閉后,室溫條件下孵育TGEV-N抗體(1∶500稀釋)1 h,PBS清洗3次后,室溫避光孵育二抗(1∶500稀釋)1 h,PBS清洗3次;室溫避光孵育DAPI(1∶2 000稀釋)15 min,再次使用PBS清洗3次后,于倒置熒光顯微鏡下進行觀察,并拍攝圖像。
1.9 50%組織細胞感染量(50% tissue culture infective dose,TCID50)檢測
待IPEC-J2長至80%的匯合度時,按“1.4”的接毒步驟接種TGEV,24 h后收取細胞樣品,將樣品置于-80℃低溫下凍結,隨后轉移至室溫迅速解凍,反復凍融3次后離心并過濾,收集TGEV濾液。將收集的TGEV濾液以10倍進行梯度稀釋(10-1~10-8),每個稀釋度設置8個重復,稀釋后的病毒濾液接種到培養有LLC-PK1的96孔細胞培養板中,將細胞培養物繼續在37℃、5% CO2的培養箱中培養,每隔12 h觀察并記錄96孔細胞培養板中LLC-PK1的病變孔數,采用Reed-Muench兩氏法計算病毒滴度,單位為毫升(mL)。
1.10 統計分析
本研究每個試驗至少進行3次重復,結果均以“平均值±標準誤”表示,試驗數據采用GraphPad Prism 8.3.0統計軟件進行顯著性差異分析并作圖,用單因素方差分析(One-Way ANOVA)來判斷不同處理間的差異及顯著性,Plt;0.05為差異顯著,Plt;0.01為差異極顯著。
2 結 果
2.1 TGEV感染IPEC-J2-WT最佳時間節點的選擇
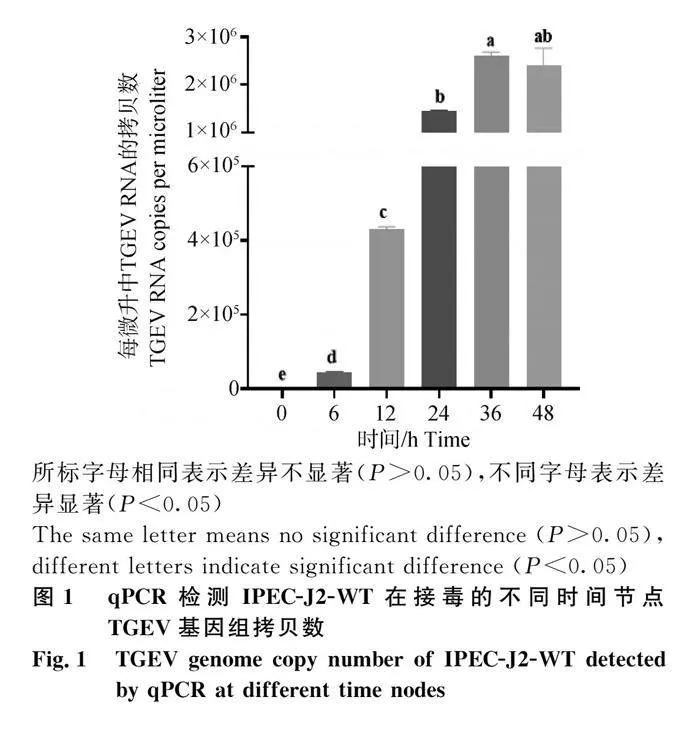
為確定TGEV感染IPEC-J2-WT后收取細胞樣品合適時間,將IPEC-J2-WT接種TGEV,分別于接毒0、6、12、24、36與48 h時收集細胞并提取RNA樣品,qPCR檢測IPEC-J2-WT中TGEV基因組拷貝數。結果顯示,TGEV-N基因拷貝數和Ct值之間的線性關系為:y=-3.433x+37.958,R2=0.996。接毒后不同時間收取的細胞樣品均能檢測到TGEV病毒核酸,與接毒6、12 h細胞相比,接種TGEV 24 h細胞的病毒基因組拷貝數顯著升高(Plt;0.05),接毒24、36、48 h細胞內病毒基因組拷貝數均處于相同數量級(106,圖1),表明接毒24 h IPEC-J2-WT中TGEV擴增速度較快且基因組拷貝數較高,故選取接毒24 h作為收取細胞樣品的最佳時間節點。
2.2 IPEC-J2-KO的脫靶檢測分析
本研究以王曉朋等[32]利用CRISPR/Cas9基因編輯技術制備的IPEC-J2-KO作為試驗材料。為確定其是否存在脫靶效應,針對所使用的g3與g5分別篩選了8個潛在脫靶位點并設計相應引物,PCR產物經凝膠電泳后進行測序。結果表明,g3與g5各8個潛在脫靶序列中,IPEC-J2-KO與IPEC-J2-WT的DNA序列均一致,表明IPEC-J2-KO中未出現脫靶效應(圖2)。
2.3 IPEC-J2-KO感染特征分析
為了探究IPEC-J2-KO與IPEC-J2-WT的感染特征差異,本研究設置Mock組、IPEC-J2-WT接毒組和IPEC-J2-KO接毒組進行試驗,從TGEV-N基因拷貝數、TGEV-N蛋白表達情況以及TCID50試驗3個方面進行驗證。
2.3.1 qPCR檢測IPEC-J2-KO中TGEV基因組拷貝數
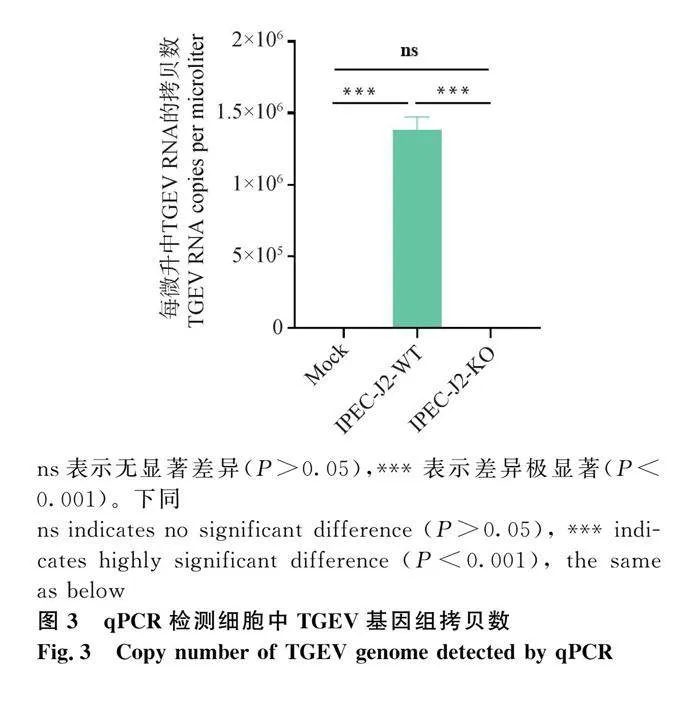
為了檢測IPEC-J2-KO對TGEV的抵抗力,qPCR檢測Mock組、IPEC-J2-WT接毒組和IPEC-J2-KO接毒組3組細胞中TGEV-N基因拷貝數。結果顯示,TGEV-N基因拷貝數和Ct值之間的線性關系為:y=-3.415x+38.086;R2=0.996。接毒后的IPEC-J2-WT中TGEV-N基因拷貝數極顯著高于IPEC-J2-KO組(Plt;0.001);與Mock組相比,IPEC-J2-KO中TGEV-N基因拷貝數無顯著差異(Pgt;0.05,圖3)。結果表明,接毒后的IPEC-J2-KO可抵抗TGEV感染。
2.3.2 WB檢測IPEC-J2-KO中TGEV-N蛋白表達
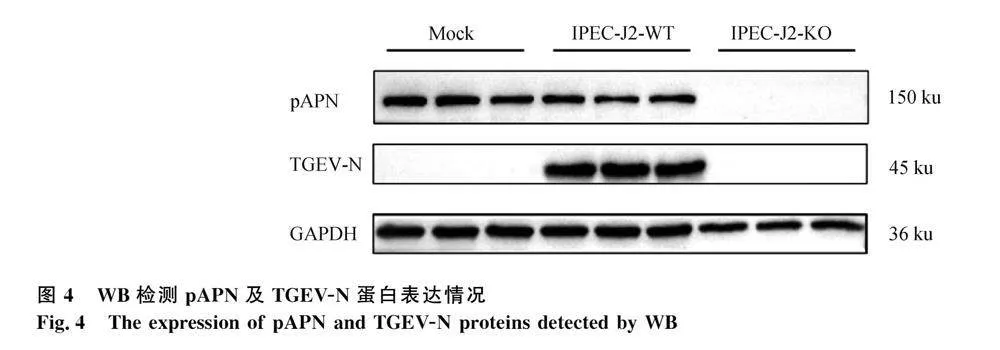
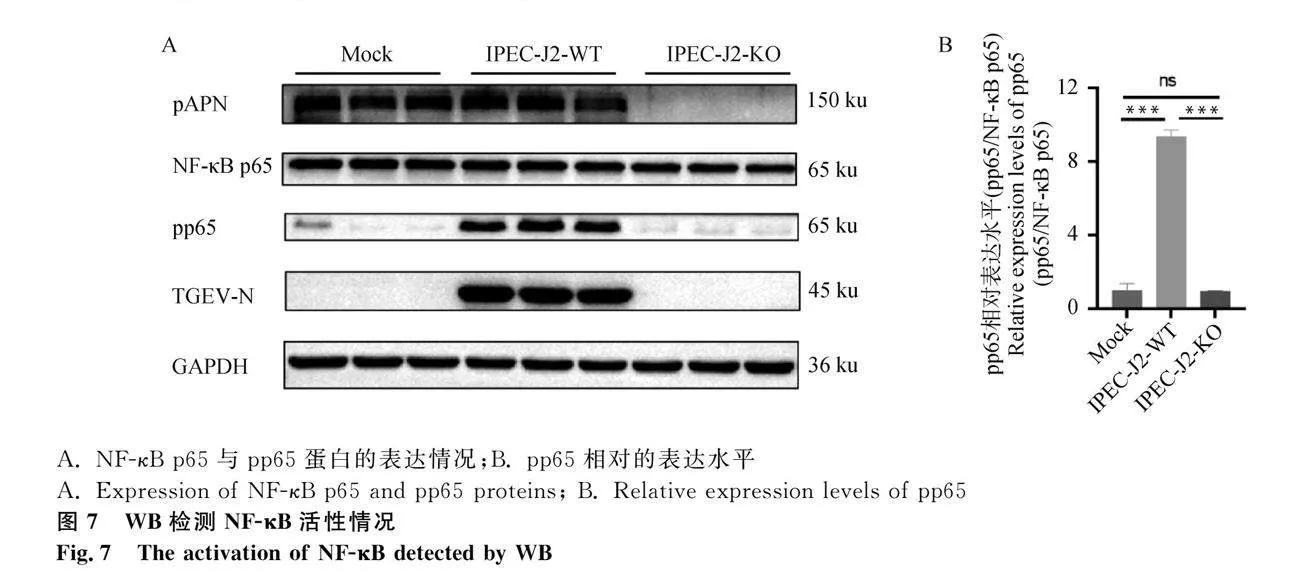
為了評估IPEC-J2-KO對TGEV的抗性,提取Mock組、接毒后的IPEC-J2-WT組和IPEC-J2-KO組中細胞的蛋白質,通過WB檢測pAPN及TGEV-N蛋白的表達情況。結果顯示,在Mock組和IPEC-J2-WT組均能檢測到pAPN蛋白表達,在IPEC-J2-KO中未檢測到pAPN蛋白,且接毒后的IPEC-J2-WT中可檢測到TGEV-N蛋白表達,而IPEC-J2-KO組中未檢測到TGEV-N蛋白表達(圖4)。表明IPEC-J2-KO細胞可抵抗TGEV感染。
2.3.3 IFA檢測TGEV-N蛋白表達情況
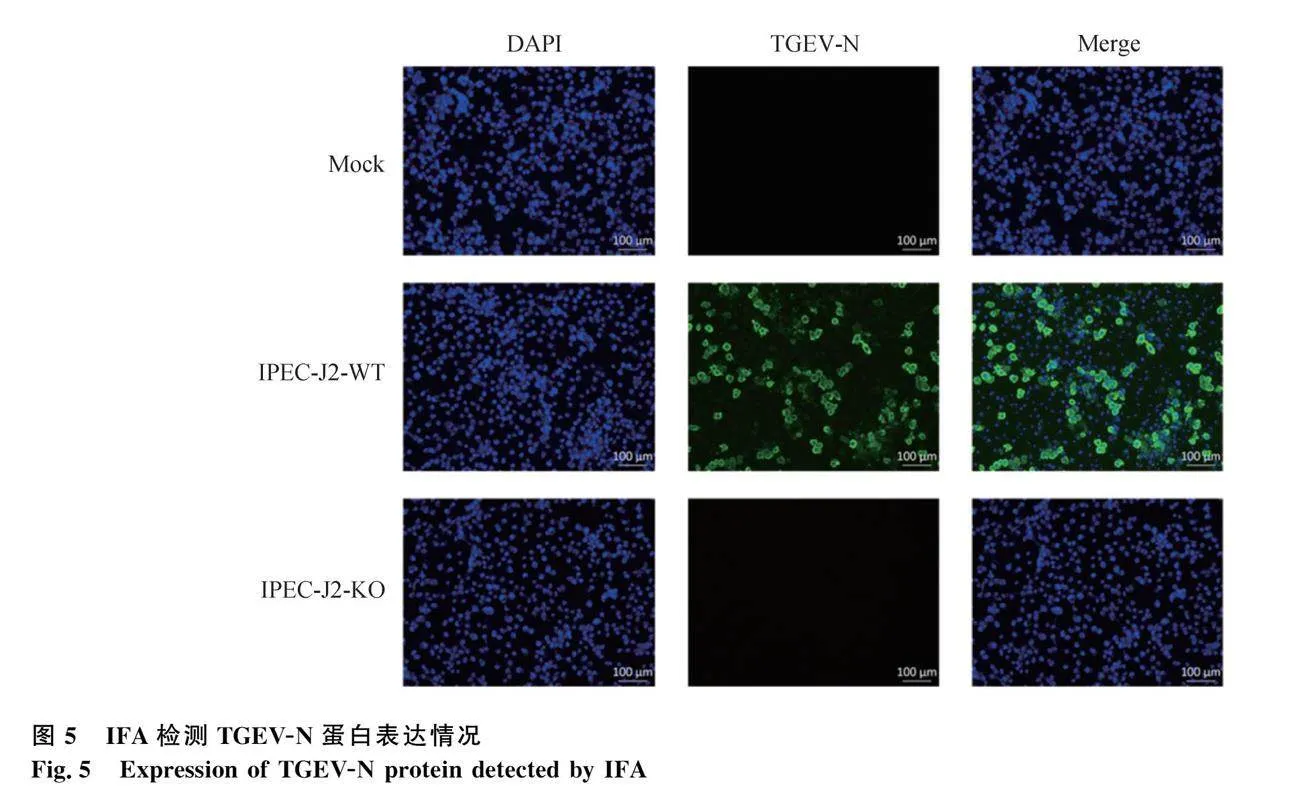
為了進一步確認IPEC-J2-KO對TGEV的感染特征,對Mock組、IPEC-J2-WT接毒組和IPEC-J2-KO接毒組的細胞進行IFA檢測。結果顯示,IPEC-J2-WT組中存在大量TGEV-N蛋白陽性細胞,在Mock組和IPEC-J2-KO組中未檢測到TGEV-N蛋白陽性細胞(圖5)。表明接毒后的IPEC-J2-KO不存在TGEV-N蛋白的表達,與WB結果一致。
2.3.4 TCID50檢測IPEC-J2-KO的病毒滴度
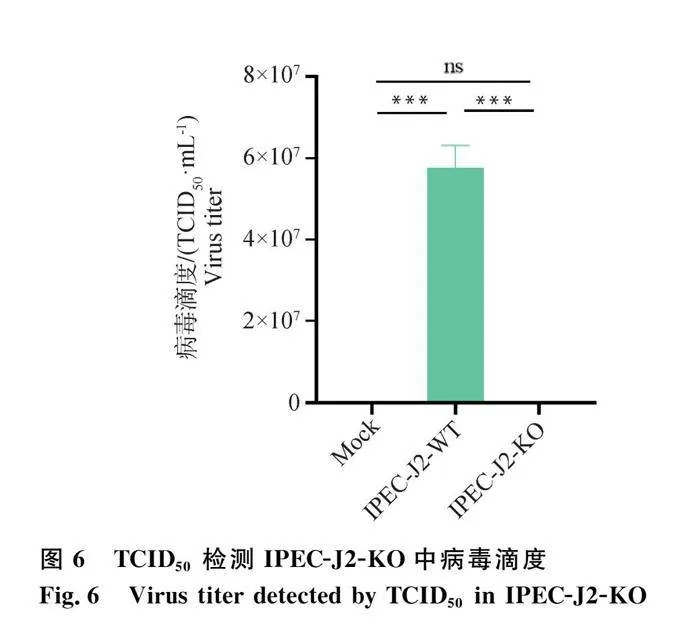
為評估TGEV在IPEC-J2-KO中的感染情況,利用LLC-PK1檢測Mock組、接毒后的IPEC-J2-WT組和IPEC-J2-KO組中細胞的病毒滴度,TCID50測定結果顯示,IPEC-J2-WT組細胞中的病毒滴度極顯著高于IPEC-J2-KO組(Plt;0.001);與Mock組相比,IPEC-J2-KO組中的病毒滴度無顯著差異(Pgt;0.05,圖6),這與qPCR、WB及IFA結果趨勢相符。表明IPEC-J2-KO可有效抵抗TGEV的感染。
2.4 TGEV感染對IPEC-J2中轉錄因子NF-κB活性的影響
為了評估TGEV對IPEC-J2中轉錄因子NF-κB活性的影響,收取Mock組、IPEC-J2-WT接毒組和IPEC-J2-KO接毒組的細胞蛋白樣品,通過WB檢測NF-κB p65及其磷酸化蛋白pp65表達情況。結果顯示,在Mock組、IPEC-J2-WT組和IPEC-J2-KO組均能檢測到NF-κB p65的表達(圖7A)。同時,與Mock組相比,IPEC-J2-WT組中NF-κB p65磷酸化水平極顯著的升高(Plt;0.001),而IPEC-J2-KO組無顯著差異(Pgt;0.05,圖7B)。表明接種TGEV可激活IPEC-J2-WT中NF-κB活性,而不影響IPEC-J2-KO的NF-κB活性。
3 討 論
TGEV是一種引起仔豬嚴重水樣腹瀉的豬腸道冠狀病毒[1],經過長期廣泛的傳播,TGEV進化出了毒力、傳染性和致病性均較強的突變株[33],并常與多種腸道病毒混合感染[10],嚴重威脅全球養豬業的發展。因此,探究TGEV的致病機理,闡明該病毒的感染機制至關重要。TGEV主要通過消化道和呼吸道入侵機體,感染會導致豬空腸損傷,免疫功能受損,并增加致病菌載量[34]。豬空腸IPEC-J2是TGEV易感細胞系之一,利用IPEC-J2建立的TGEV體外感染模型被廣泛應用于豬腸道疾病和感染機制的研究[31,35-36]。本研究測定了IPEC-J2-WT在接毒后不同時間點TGEV基因組拷貝數,結果顯示,接毒后不同時間點均能檢測到TGEV病毒核酸,其中,與接毒后6和12 h相比,24 h的病毒基因組拷貝數顯著升高,且24 h后病毒基因組拷貝數均處于相同數量級,表明接毒24 h是收集細胞樣本以評估TGEV對IPEC-J2影響的最佳時間節點。
APN是一種鋅離子依賴性Ⅱ型跨膜金屬糖蛋白,隸屬于金屬蛋白酶M1家族,1963年首次由Pfleiderer和Celliers從豬腎中分離出來[37],廣泛分布于哺乳動物不同器官和組織中,具有廣泛的生物學功能。pAPN在豬小腸粘膜細胞、腎臟刷狀緣膜和肝臟中高度表達[38]。研究發現,pAPN是TGEV的關鍵細胞受體,TGEV通過其S蛋白附著到宿主細胞表面的pAPN上進入宿主細胞[39]。敲除病毒宿主受體可能是研究宿主細胞反應的分子機制和加速家畜抗病育種進程的有效策略[40]。基因編輯技術可刪減、插入或替換基因序列,實現對目標基因的精準編輯。CRISPR/Cas9系統因具有組成較為簡單、編輯效率高等優點,已經成為目前應用最多的基因編輯技術,被廣泛應用于提高家畜繁殖效率[41-42]、生產性能[43-44]、抗病性以及動物模型構建等研究[24,40,45-46]。其中,gRNA作為該系統的核心組成,能夠幫助Cas蛋白準確定位到需要編輯的基因組區域,在基因編輯中起引導作用,同時gRNA的精確性與基因編輯的效率和安全性密切相關[47]。因此,設計高效且脫靶效應低的gRNA對于后續的試驗是至關重要的。本研究首先對王曉朋等[32]利用CRISPR/Cas9技術構建pAPN基因敲除IPEC-J2的gRNA進行脫靶位點預測,并對其潛在的脫靶位點附近DNA序列進行PCR擴增及測序。結果表明,IPEC-J2-KO與IPEC-J2-WT的DNA序列完全一致,未發現脫靶效應,證明構建IPEC-J2-KO所用gRNA準確度和安全性較高。
病毒的感染特征分析對于理解宿主-病原體相互作用機制具有重要意義[48]。TGEV-N蛋白編碼基因高度保守,針對TGEV-N基因保守序列設計特異性引物建立的qPCR檢測方法,不僅可用于表征感染細胞中TGEV的拷貝數,也可用于臨床快速診斷和實驗室檢測[49]。同時,TGEV-N蛋白是感染細胞中含量最多的病毒結構蛋白之一,可作為早期檢測及診斷TGEV感染的理想靶抗原[50]。近些年,利用CRISPR/Cas9系統構建的pAPN基因敲除動物模型在活體中被證實均可抵抗TGEV的感染[24,51],進一步證實了pAPN在TGEV感染中的重要作用。因此,為深入了解pAPN基因在IPEC-J2介導TGEV感染過程中的作用,本研究對IPEC-J2-KO介導的TGEV感染特征進行了分析。結果表明,與IPEC-J2-WT組相比,接毒后的IPEC-J2-KO中TGEV基因組拷貝數極顯著降低,且未檢測到TGEV-N蛋白的表達,證明IPEC-J2-KO可抵抗TGEV的感染。除此之外,TCID50試驗是一種可用于測定病毒在細胞培養物中感染能力和活性水平的方法。TCID50試驗中常用的細胞是LLC-PK1。LLC-PK1是豬腎上皮細胞,對所有腸道冠狀病毒均具有易感性,是常用的腸道冠狀病毒體外感染的細胞模型[52],TGEV在該細胞中增殖和感染效率更高。LLC-PK1在感染TGEV后會出現明顯的體積增大、變圓、成簇等細胞病變,易于試驗觀察[52]。本研究也利用TCID50試驗分析了IPEC-J2-WT和IPEC-J2-KO在接種TGEV后的病毒滴度變化。結果顯示,IPEC-J2-KO中病毒滴度與IPEC-J2-WT相比極顯著下降,這與之前的結果趨勢相符合一致。綜上所述,pAPN基因敲除的IPEC-J2可有效抵抗TGEV的感染。
小腸炎癥被認為是TGEV感染發病機制中的核心事件,IPEC-J2在TGEV感染24 h內會發生快速而廣泛的轉錄和代謝變化,炎癥相關通路和防御相關途徑也會被激活[53]。之前研究發現,TGEV可通過NF-κB、Toll樣受體(toll-like receptors, TLR)、腫瘤壞死因子(tumor necrosis factor,TNF)等信號通路誘導炎癥反應[54]。其中,NF-κB是炎癥反應的關鍵轉錄因子,在多種炎癥性疾病中扮演重要角色[55]。NF-κB通路在TGEV感染的ST細胞和IPEC-J2細胞中均能被激活,并導致促炎細胞因子表達上調[27]。此外,研究顯示TGEV誘導的NF-κB活性受相關基因的表達調控,如沉默早幼粒細胞白血病(promyelocytic leukemia,PML)基因的表達可以挽救TGEV誘導的NF-κB活性[56]。NF-κB p65磷酸化是NF-κB通路的一個重要指標,可以反映該通路的活性水平[57-58]。為了評估TGEV感染對IPEC-J2中先天免疫相關信號通路中轉錄因子NF-κB活性的影響,本研究檢測了NF-κB p65及其磷酸化蛋白pp65的表達情況。結果顯示,與Mock組相比,接毒后,IPEC-J2-WT中NF-κB p65磷酸化水平極顯著的升高,而IPEC-J2-KO中無顯著變化,表明接種TGEV未影響IPEC-J2-KO中先天免疫相關信號通路中轉錄因子NF-κB的活性,推測可能是因為pAPN基因的敲除導致TGEV無法進入細胞,從而未引起NF-κB活性的變化。
4 結 論
本研究在IPEC-J2上進行了TGEV感染特征分析,證實了IPEC-J2-KO在接毒后可有效抵抗TGEV的感染,且未影響NF-κB p65磷酸化水平,為闡明pAPN基因在TGEV入侵宿主細胞的機制提供理論依據,同時為研究抗病豬新品種奠定了堅實基礎。
參考文獻(References):
[1] LIU Q, WANG H Y. Porcine enteric coronaviruses: an updated overview of the pathogenesis, prevalence, and diagnosis[J]. Vet Res Commun, 2021, 45(2-3): 75-86.
[2] DOYLE L P, HUTCHINGS L M. A transmissible gastroenteritis in pigs[J]. J Am Vet Med Assoc, 1946, 108: 257-259.
[3] CHEN S Y, ZHANG H Y, CHU M F, et al. Prevalence of transmissible gastroenteritis among swine populations in China during 1983-2022: A systematic review and meta-analysis[J]. Microb Pathog, 2023, 183: 106320.
[4] TURLEWICZ-PODBIELSKA H, POMORSKA-MóL M. Porcine Coronaviruses: Overview of the State of the Art[J]. Virol Sin, 2021, 36(5): 833-851.
[5] 曹貝貝, 蘭培英, 韓 麗, 等. 豬TGEV HN-2012株ORF3a和ORF3b基因遺傳變異分析及其真核表達研究[J]. 西北農林科技大學學報(自然科學版), 2016, 44(4): 10-16.
CAO B B, LAN P Y, HAN L, et al. Genetic variation and eukaryotic expression of ORF3a and ORF3b from porcine TGEV HN-2012 strain[J]. Journal of Northwest Aamp;F University (Natural Science Edition), 2016, 44(4): 10-16. (in Chinese)
[6] 鄭賓賓, 宋 濤, 季裕婷, 等. 豬傳染性胃腸炎病毒HB-1株的分離鑒定和全基因組序列分析[J]. 中國獸醫雜志, 2023, 59(5): 36-41.
ZHENG B B, SONG T, JI Y T, et al. Isolation, identification and whole genome sequence analysis of porcine transmissible gastroenteritis virus HB-1 strain[J]. Chinese Journal of Veterinary Medicine, 2023, 59(5): 36-41. (in Chinese)
[7] 李嘉琛, 吳華偉, 陳曉春, 等. 豬傳染性胃腸炎病毒的分離與鑒定[J]. 中國獸藥雜志, 2018, 52(3): 25-30.
LI J C, WU H W, CHEN X C, et al. Isolation and identification of porcine transmissible gastroenteritis virus[J]. Chinese Journal of Veterinary Drug, 2018, 52(3): 25-30. (in Chinese)
[8] 俞伏松, 王 劭, 陳仕龍, 等. 豬傳染性胃腸炎病毒福建株的分離鑒定[J]. 福建農業學報, 2012, 27(11): 1160-1164.
YU F S, WANG S, CHEN S L, et al. Isolation and identification of porcine transmissible gastroenteritis virus FJ strain[J]. Fujian Journal of Agricultural Sciences, 2012, 27(11): 1160-1164. (in Chinese)
[9] 祖立闖, 王 芳, 劉愛華, 等. 我國豬傳染性胃腸炎的流行現狀及實驗室診斷技術研究進展[J]. 養豬, 2017(5): 123-128.
ZU L C, WANG F, LIU A H, et al. Research progress of epidemic status and laboratory diagnostic technology of porcine transmissible gastroenteritis in China[J]. Swine Production, 2017(5): 123-128. (in Chinese)
[10] YIN L, LIU X, HU D M, et al. Swine enteric coronaviruses (PEDV, TGEV, and PDCoV) induce divergent interferon-stimulated gene responses and antigen presentation in porcine intestinal enteroids[J]. Front Immunol, 2021, 12: 826882.
[11] VLASOVA A N, DIAZ A, DAMTIE D, et al. Novel canine coronavirus isolated from a hospitalized patient with pneumonia in east malaysia[J]. Clin Infect Dis, 2022, 74(3): 446-454.
[12] CHEN Y W, ZHANG Y Z, WANG X, et al. Transmissible gastroenteritis virus: An update review and perspective[J]. Viruses, 2023, 15(2): 359.
[13] ALONSO S, IZETA A, SOLA I, et al. Transcription regulatory sequences and mRNA expression levels in the coronavirus transmissible gastroenteritis virus[J]. J Virol, 2002, 76(3): 1293-1308.
[14] GALáN C, SOLA I, NOGALES A, et al. Host cell proteins interacting with the 3′ end of TGEV coronavirus genome influence virus replication[J]. Virology, 2009, 391(2): 304-314.
[15] REN X F, LIU B Q, YIN J C, et al. Phage displayed peptides recognizing porcine aminopeptidase N inhibit transmissible gastroenteritis coronavirus infection in vitro[J]. Virology, 2011, 410(2): 299-306.
[16] RIEMANN D, KEHLEN A, LANGNER J. CD13-not just a marker in leukemia typing[J]. Immunol Today, 1999, 20(2): 83-88.
[17] DELMAS B, GELFI J, KUT E, et al. Determinants essential for the transmissible gastroenteritis virus-receptor interaction reside within a domain of aminopeptidase-N that is distinct from the enzymatic site[J]. J Virol, 1994, 68(8): 5216-5224.
[18] YEAGER C L, ASHMUN R A, WILLIAMS R K, et al. Human aminopeptidase N is a receptor for human coronavirus 229E[J]. Nature, 1992, 357(6377): 420-422.
[19] SODERBERG C, GIUGNI T D, ZAIA J A, et al. CD13 (human aminopeptidase N) mediates human cytomegalovirus infection[J]. J Virol, 1993, 67(11): 6576-6585.
[20] DELMAS B, GELFI J, L′HARIDON R, et al. Aminopeptidase N is a major receptor for the enteropathogenic coronavirus TGEV[J]. Nature, 1992, 357(6377): 417-420.
[21] LI W T, LUO R, HE Q G, et al. Aminopeptidase N is not required for porcine epidemic diarrhea virus cell entry[J]. Virus Res, 2017, 235: 6-13.
[22] WANG B, LIU Y, JI C M, et al. Porcine deltacoronavirus engages the transmissible gastroenteritis virus functional receptor porcine aminopeptidase N for infectious cellular entry[J]. J Virol, 2018, 92(12): e00318-18.
[23] ZHU X Y, LIU S D, WANG X L, et al. Contribution of porcine aminopeptidase N to porcine deltacoronavirus infection[J]. Emerg Microbes Infect, 2018, 7(1): 65.
[24] XU K, ZHOU Y R, MU Y L, et al. CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance[J]. Elife, 2020, 9: e57132.
[25] DEDIEGO M L, NIETO-TORRES J L, JIMENEZ-GUARDENO J M, et al. Coronavirus virulence genes with main focus on SARS-CoV envelope gene[J]. Virus Res, 2014, 194: 124-137.
[26] GUO J X, ZHAO X M, LIU Z H, et al. Transmissible gastroenteritis virus ORF3b up-regulates miR-885-3p to counteract TNF-alpha production via inhibiting NF-kappaB pathway[J]. Vet Microbiol, 2021, 261: 109189.
[27] WANG L, QIAO X Y, ZHANG S J, et al. Porcine transmissible gastroenteritis virus nonstructural protein 2 contributes to inflammation via NF-kappaB activation[J]. Virulence, 2018, 9(1): 1685-1698.
[28] ZHOU Y R, WU W, XIE L L, et al. Cellular RNA helicase DDX1 is involved in transmissible gastroenteritis virus nsp14-induced interferon-beta production[J]. Front Immunol, 2017, 8: 940.
[29] WU Y, LI M W, TIAN J, et al. Broad antagonism of coronaviruses nsp5 to evade the host antiviral responses by cleaving POLDIP3[J]. PLoS Pathog, 2023, 19(10): e1011702.
[30] DU J, CHEN D W, YU B, et al. L-Leucine promotes STAT1 and ISGs expression in TGEV-infected IPEC-J2 cells via mTOR activation[J]. Front Immunol, 2021, 12: 656573.
[31] GUO Z Z, ZHANG C X, DONG J J, et al. Persistence infection of TGEV promotes enterococcus faecalis infection on IPEC-J2 cells[J]. Int J Mol Sci, 2022, 24(1): 450.
[32] 王曉朋, 徐 奎, 魏迎輝, 等. CRISPR/Cas9介導的豬IPEC-J2細胞CD13基因敲除細胞系的建立[J]. 畜牧獸醫學報, 2019, 50(7): 1319-1327.
WANG X P, XU K, WEI Y H, et al. Establishment of CD13 gene knockout IPEC-J2 cell lines mediated by CRISPR/Cas9 system[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(7): 1319-1327. (in Chinese)
[33] GUO R L, FAN B C, CHANG X J, et al. Characterization and evaluation of the pathogenicity of a natural recombinant transmissible gastroenteritis virus in China[J]. Virology, 2020, 545: 24-32.
[34] XIA L, YANG Y H, WANG J L, et al. Impact of TGEV infection on the pig small intestine[J]. Virol J, 2018, 15(1): 102.
[35] GUO J Y, LI F, QIAN S J, et al. TGEV infection up-regulates FcRn expression via activation of NF-kappaB signaling[J]. Sci Rep, 2016, 6: 32154.
[36] ZHAO X M, MA X L, GUO J X, et al. Circular RNA CircEZH2 suppresses transmissible gastroenteritis coronavirus-induced opening of mitochondrial permeability transition pore via targeting MiR-22 in IPEC-J2[J]. Int J Biol Sci, 2019, 15(10): 2051-2064.
[37] PFLEIDERER G, CELLIERS P G. Isolation of an aminopetidase from kidney particles[J]. Biochem Z, 1963, 339: 186-189.
[38] KUMARAVEL S, LUO G R, HUANG S T, et al. Development of a novel latent electrochemical molecular substrate for the real-time monitoring of the tumor marker aminopeptidase N in live cells, whole blood and urine[J]. Biosens Bioelectron, 2022, 203: 114049.
[39] CHEN L, LIN Y L, PENG G Q, et al. Structural basis for multifunctional roles of mammalian aminopeptidase N[J]. Proc Natl Acad Sci U S A, 2012, 109(44): 17966-17971.
[40] WHITWORTH K M, ROWLAND R R, EWEN C L, et al. Gene-edited pigs are protected from porcine reproductive and respiratory syndrome virus[J]. Nat Biotechnol, 2016, 34(1): 20-22.
[41] SHI X, TANG T, LIN Q Y, et al. Efficient generation of bone morphogenetic protein 15-edited Yorkshire pigs using CRISPR/Cas9[J]. Biol Reprod, 2020, 103(5): 1054-1068.
[42] 許美娜, 朱奕舟, 林思遠, 等. CRISPR/Cas9基因編輯技術在豬育種中的研究進展[J]. 廣東農業科學, 2022, 49(8): 87-96.
XU M N, ZHU Y Z, LIN S Y, et al. Progress of the application of CRISPR/Cas9 gene editing technology in pig breeding[J]. Guangdong Agricultural Sciences, 2022, 49(8): 87-96. (in Chinese)
[43] XIANG G H, REN J L, HAI T, et al. Editing porcine IGF2 regulatory element improved meat production in Chinese Bama pigs[J]. Cell Mol Life Sci, 2018, 75(24): 4619-4628.
[44] ZHENG Q T, LIN J, HUANG J J, et al. Reconstitution of UCP1 using CRISPR/Cas9 in the white adipose tissue of pigs decreases fat deposition and improves thermogenic capacity[J]. Proc Natl Acad Sci U S A, 2017, 114(45): E9474-E9482.
[45] XU K, ZHOU Y R, SHANG H T, et al. Pig macrophages with site-specific edited CD163 decrease the susceptibility to infection with porcine reproductive and respiratory syndrome virus[J]. J. Integr. Agric, 2023, 22(7): 2188-2199.
[46] NIU D, WEI H J, LIN L, et al. Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9[J]. Science, 2017, 357(6357): 1303-1307.
[47] MOTOCHE-MONAR C, ORDONEZ J E, CHANG O, et al. gRNA Design: How its evolution impacted on CRISPR/Cas9 systems refinement[J]. Biomolecules, 2023, 13(12): 1698.
[48] ZHANG S J, HUANG W Z, REN L L, et al. Comparison of viral RNA-host protein interactomes across pathogenic RNA viruses informs rapid antiviral drug discovery for SARS-CoV-2[J]. Cell Res, 2022, 32(1): 9-23.
[49] 張羽欣, 王樹茂, 段宏勇, 等. 豬傳染性胃腸炎病毒TaqMan實時熒光定量PCR檢測方法的建立與應用[J]. 中國獸醫科學, 2024(4): 1-8.
ZHANG Y X, WANG S M, DUAN H Y, et al. Establishment and application of TaqMan real-time quantitative PCR for detection of porcine transmissible gastroenteritis virus[J]. Chinese Veterinary Science, 2024(4): 1-8. (in Chinese)
[50] 董珮玲. 豬傳染性胃腸炎病毒N蛋白單克隆抗體的制備及其初步應用[D]. 武漢:華中農業大學, 2023.
DONG P L. Preparation of monoclonal antibody to Transmissiblegastroenteritis virus N protein and its initial application[D]. Wuhan: Huazhong Agricultural University, 2023. (in Chinese)
[51] WHITWORTH K M, ROWLAND R, PETROVAN V, et al. Resistance to coronavirus infection in amino peptidase N-deficient pigs[J]. Transgenic Res, 2019, 28(1): 21-32.
[52] XIAO W W, WANG X L, WANG J, et al. Replicative capacity of four porcine enteric coronaviruses in LLC-PK1 cells[J]. Arch Virol, 2021, 166(3): 935-941.
[53] ZHANG S S, CAO Y N, XU C, et al. Integrated metabolomics and transcriptomics analyses reveal metabolic responses to TGEV infection in porcine" intestinal epithelial cells[J]. J Gen Virol, 2023, 104(12): 10.
[54] MA X L, ZHAO X M, ZHANG Z C, et al. Differentially expressed non-coding RNAs induced by transmissible gastroenteritis virus potentially regulate inflammation and NF-κB pathway in porcine intestinal epithelial cell line[J]. BMC Genomics, 2018, 19(1): 747.
[55] 李宇航, 羅仍卓么, 王興平, 等. NF-κB信號通路調控奶牛乳腺炎的分子作用機制[J]. 畜牧獸醫學報, 2021, 52(10): 2740-2752.
LI Y H, LUORENG Z M, WANG X P, et al. Molecular regulatory mechanism of NF-κB signaling pathway regulating mastitis in dairy Cows[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(10): 2740-2752. (in Chinese)
[56] MA X L, ZHAO X M, WANG K L, et al. Identification and analysis of long non-coding RNAs that are involved in inflammatory process in response to transmissible gastroenteritis virus infection[J]. BMC Genomics, 2019, 20(1): 806.
[57] PAN W Z, DU J, ZHANG L Y, et al. The roles of NF-κB in the development of lung injury after one-lung ventilation[J]. Eur Rev Med Pharmacol Sci, 2018, 22(21): 7414-7422.
[58] LAWRENCE T. The nuclear factor NF-kappaB pathway in inflammation[J]. Cold Spring Harb Perspect Biol, 2009, 1(6): a001651.
(編輯 郭云雁)

