板栗PAT基因家族成員鑒定及不同脅迫響應分析
李金梅 聶興華 葛婧怡 褚世慧 劉陽 秦嶺 邢宇
DOI:10.13925/j.cnki.gsxb.20230520
摘??? 要:【目的】鑒定和分析板栗PAT基因家族及其對不同脅迫的響應,探究板栗PAT基因家族的抗逆功能。【方法】在板栗全基因組水平上進行搜索和鑒定板栗PAT基因家族成員,利用生物信息學方法研究其系統發育進化樹、基因結構和motif、蛋白理化性質、染色體定位、共線性和啟動子順式元件等。以燕山紅栗為試驗材料,分析鹽脅迫、抗病脅迫和干旱脅迫處理對板栗PAT基因家族表達模式的影響。【結果】在板栗基因組中共鑒定出包含DHHC結構域的21個PAT基因家族成員,他們與24個AtPAT基因家族成員共聚集為6個亞組;大多數CmPAT家族成員為具有親水性的堿性穩定蛋白;21個PAT基因家族成員不均勻地分布在板栗的9條染色體上;在CmPAT基因啟動子區域鑒定到多種非生物脅迫及激素響應元件;表達模式分析表明,多個CmPAT基因不同程度地參與抗病、干旱、鹽脅迫響應。【結論】共鑒定了21個板栗PAT基因家族成員,同時篩選到CmPAT24、CmPAT7、CmPAT14可能共同參與了鹽脅迫和干旱脅迫的調控,CmPAT7可能共同參與鹽脅迫、干旱脅迫、抗病脅迫的調控。
關鍵詞:板栗;棕櫚酰基轉移酶(PAT);脅迫處理
中圖分類號:S664.2?????????? 文獻標志碼:A??????????? 文章編號:1009-9980(2024)05-0847-14
收稿日期:2023-12-13??????? 接受日期:2024-02-16
基金項目:國家自然科學基金項目(32271929)
作者簡介:李金梅,女,在讀碩士研究生,研究方向為果樹分子生物學。E-mail:vmz0411@163.com
*通信作者 Author for correspondence. E-mail:xingyu@bua.edu.cn
果 樹 學 報 2024,41(5): 847-860
Journal of Fruit Science
Identification of PAT gene family members and analysis of their response to different stresses in Chinese chestnut
LI Jinmei, NIE Xinghua, GE Jingyi, CHU Shihui, LIU Yang, QIN Ling, XING Yu*
(College of Plant Science and Technology, Beijing University of Agriculture, Beijing 102206, China)
Abstract: 【Objective】 The reversibility of protein palmitoylation is a key mechanism for regulating cellular function. Palmitoyl transferase is an important part of this mechanism, which can connect palmitic acid to the cysteine residue of the target protein. Protein palmitoylation is a post-transcriptional modification that plays a critical role in protein transport and function. Palmitate transferase (PAT) catalyzes the occurrence of acylation. Acylated PAT proteins may be involved in a variety of stress responses in material transport and signal transduction. Palmitate transferase plays an important role in growth, development and response to harmful stresses in plants. However, understanding of PAT genes in chestnut is limited so far. In the experiment, we performed a genome-wide identification of the PAT gene family in chestnut and subsequently analyzed how it responded to various stresses in Chinese chestnut. 【Methods】 HMMER, NCBI-CDD and SMART searches were performed to identify PAT genes throughout the chestnut genome. The domain information of the candidate gene family members was obtained through preliminary screening, and the protein sequences without the DHHC domain were eliminated. Ultimately, 21 members of the chestnut PAT gene family were identified. The bioinformatics methods were used to study the phylogenetic tree, gene structure and gene motif, protein physicochemical properties, subcellular and chromosomal localization, collinearity and promoter cis-elements of the PAT genes. To check the stress-resistant function of PAT gene family, Yanshanhong cultivar was used as experimental material. For salt treatment, the seedling roots were submerged in a solution containing 200 mmol·L-1 NaCl and samples were collected on the 7th, 10th and 14th days. Drought treatment was also carried out. Samples were collected and tested at different water loss in fresh leaves, specifically at 0%, 5%, 30% and 50%. To treat disease resistance, Chestnut Blight Mycelia Blocks were cultured on PAD medium. After 3 days, the PAD agar blocks containing chestnut blight mycelia were placed on perforated branches. Samples were collected at 0 h, 12 h, 24 h and 3 days after inoculation. Fresh branches that were only treated with punching served as controls. All samples were then harvested, immediately frozen in liquid nitrogen, and stored at -80 ℃. The influence of salt, disease resistance and drought stress on the expression pattern of the PAT gene family was examined using the real-time quantitative fluorescence analysis. 【Results】 Through a comprehensive genome search and identification, we acquired 21 members of the PAT gene family in the chestnut genome that possessed the DHHC domain. In the phylogenetic evolutionary tree, they grouped into six branches with 24 members of the AtPAT gene family. By analyzing the physical and chemical properties of the protein, it was found that most members of the CmPAT family were hydrophilic, basic and stable proteins, and that their sequence lengths varied significantly. Subcellular localization showed that CmPAT7 was localized in the chloroplast, CmPAT4 in the nucleus, CmPAT23 in the cytoplasm, and the remaining PAT proteins in the plasma membrane. Gene structure and motif visualization showed 10 conserved motifs, and 21 PAT family members all had the DHHC domain, which was the most conserved. Their gene lengths also varied widely, ranging from 3750 to 24 300 bp, and each family member contained 7.28 introns and 8.57 exons. Members of the PAT gene family were unevenly distributed across the nine chromosomes. The CmPAT gene family was more conserved in the process of species differentiation. There were many abiotic stress and hormone response elements in the promoter region of the CmPATs gene. These included basic cis-acting elements, light-response elements, hormone-response elements and many stress-response elements, of which cis-acting and light response elements accounted for the largest proportion. An examination of the transcription factor prediction word cloud identified up to 39 transcription factors related to the control of 21 CmPATs gene. Of these, the Dof protein family was the most abundant at 22%, followed by the AP2/ERF protein family and the BBR-BPC protein family at 17% and 10%, respectively. These protein families may play a crucial role in regulating PAT genes in chestnut. Analysis of the expression pattern revealed that several CmPAT genes were involved to varying degrees in responses to disease resistance, and drought and salt stress. 【Conclusion】 A total of 21 CmPATs gene were identified. The study found that the expression levels of CmPAT24, CmPAT7 and CmPAT14 significantly increased in response to salt and drought stresses, demonstrating their involvement in the regulation of these stressors. Notably, the CmPAT7 gene showed significant upregulation under salt, drought and disease resistance stresses, indicating its role in regulating all three types of stresses.
Key words: Chinese chestnut; Palmitate transferase (PAT); Stress treatment
生物體正常行使功能必須依賴蛋白質等大分子物質,經復雜翻譯修飾作用(post-translatioanal modification,PTM)的蛋白質可以發揮正常功能及相互之間的調節作用[1]。蛋白質翻譯修飾的類型眾多[2],其中,脂鏈修飾根據連接脂肪酸鏈的類型不同,分為豆蔻酰化(myristoylation)、酰基化(acylation)等類型[3-5]。棕櫚酰基轉移酶(palmitate transferase,PAT)催化的酰基化是將棕櫚酰基以硫脂鍵的方式轉移到蛋白質的半胱氨酸殘基上,從而影響蛋白質生物合成過程[6-8]。催化這個反應的是一類活性中心含有Asp-His-His-Cys (DHHC)基序的酶,因此它們又被稱為DHHC家族[9]。棕櫚酰基轉移酶最先被發現于酵母中[10-11],并廣泛存在于哺乳動物與植物中[12-13]。在楊樹的相關研究中,利用酰基-生物素交換法和質譜法鑒定出的酰基化蛋白參與物質運輸、信號轉導和脅迫的響應[14]。其中DHHC類棕櫚酰基轉移酶是目前研究最多的家族。前人研究表明,在許多高等植物基因組中均鑒定出了數目不等的PAT家族成員,例如水稻(Oryza sativa)有30個,玉米(Zea Mays)有40個,擬南芥(Arabidopsis thaliana)有24個等[15-16]。人們對擬南芥中PAT家族成員研究最為透徹,其中AtPAT4[17]、AtPAT10[18]、AtPAT13、AtPAT14[19-20]、AtPAT15[21]、AtPAT21[22]、AtPAT24[23]等在生長發育、衰老、非生物脅迫反應等過程中具有重要作用,并且根據演化關系將擬南芥中24個PATs聚為3個分支,PAT1~9屬于分支一,PAT11~16屬于分支二,PAT18~22屬于分支三,其余成員由于演化關系較遠并未進行聚類分析[24]。
板栗(Castanea mollissima)原產于我國,營養物質豐富,是中國重要的經濟作物之一。中國的板栗種植總面積約為186.6萬hm?,年產量高達194.7萬t,占世界板栗年總產量一半以上,穩居世界首位[25]。板栗種植主要位于淺山地帶,灌溉條件有限,往往靠自然降水補充水分。同時,當栽植地區的土壤pH超過7.5、含水量低于田間最大持水量的26.6%時,其生長階段易受到多種環境脅迫的影響,包括鹽脅迫、干旱脅迫等[26]。板栗生產中也會遭受多種病蟲害侵襲,其中栗疫病最為嚴重[27]。因此,提高板栗的抗逆性,對提高板栗產業極為重要。
筆者通過比對PAT保守結構域序列,在板栗全基因組中鑒定出了21個PAT基因家族成員,并分析其系統發育樹、基因結構、理化性質、染色體定位、共線性和啟動子順式作用元件等信息,同時分析板栗PAT家族在不同脅迫下的基因表達模式,為板栗PAT基因家族抗逆功能的分析提供理論研究基礎。
1 材料和方法
1.1 試驗材料
材料為在北京市懷柔區板栗試驗站采集的燕山紅栗,選取大小、質量一致的燕山紅栗種子,放置在溫度24 ℃、濕度60%的恒溫培養箱中催芽,催芽后利用1/2 Hogland營養液進行水培,置于人工氣候室(室溫23~25 ℃,光周期:16 h光照/8 h黑暗)培養,獲得具有根系的板栗實生苗,選取生長一致的幼苗進行后續試驗。
1.2 板栗PAT基因家族成員的鑒定及多序列比對和系統進化樹構建
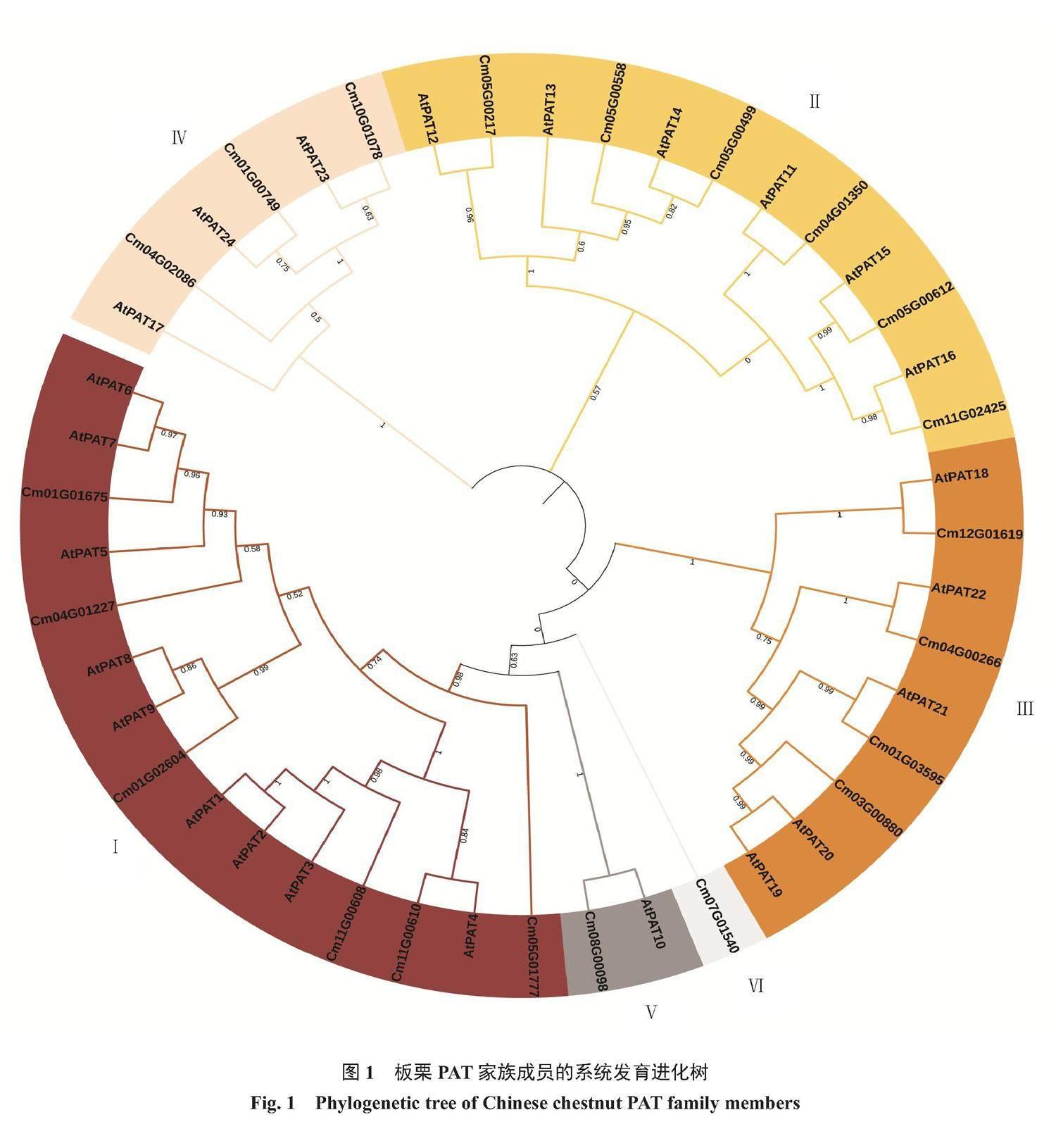
從板栗基因組網站(http://castaneadb.net/)下載板栗基因組數據,包括基因數據文件,蛋白數據文件、GFF注釋文件等。利用pfam(http://pfam.xfam.org/)查找PAT家族基因隱馬爾科夫模型(PF01529),并下載所需家族hmm數據信息。使用HMMER3.0搜索得到24個PAT蛋白序列,利用NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)Conserved Domain Search和SMART對初篩得到的候選基因家族成員進行結構域信息查對,對比剔除無DHHC結構域的蛋白序列,最終確定21個板栗PAT基因家族成員。將選取的21個板栗PAT蛋白序列和24個擬南芥PAT蛋白序列使用ClustalW進行多序列比對,通過軟件MEGA11.0[28]中的最大似然法(maximum likelihood,ML)構建進化樹,Bootstrap檢驗設定1000次重復,其他參數設置為默認參數。使用在線網站itol(https://itol.embl.de/)對系統發育進化樹進行美化。并根據與擬南芥的進化關系,對板栗進行命名,例如Cm08G00098命名為CmPAT10、Cm05G00499命名為CmPAT14、Cm01G01675命名為CmPAT7、Cm04G01227命名為CmPAT5、Cm05G00558命名為CmPAT13、Cm01G02604命名為CmPAT8、Cm11G00608命名為CmPAT3、Cm03G00880命名為CmPAT19、Cm04G02086命名為CmPAT17等,進化關系較遠或無進化關系的Cm07G01540命名為CmPAT40、Cm05G01777命名為CmPAT77。
1.3 板栗PAT基因結構與Motif可視化分析
利用MEME(http://meme-suite.org/tools/meme)進行CmPATs保守結構域分析,使用TBtools中的Gene Structure View對基因結構和保守結構域進行可視化分析。
1.4 板栗PAT基因家族的蛋白質理化性質分析和亞細胞定位
利用ExPASy(https://www.expasy.org/)對板栗PAT基因家族成員進行蛋白質理化性質分析,預測 PATs 氨基酸數目、蛋白分子質量、等電點、親水性平均系數、脂肪系數等,通過WoLF PSORT(https://www.genscript.com/wolf-psort.html)預測PAT蛋白的亞細胞定位。
1.5 板栗PAT基因家族染色體定位與共線性分析
使用MCScanX(The Multiple Collinearity Scan toolkit)工具進行染色體定位分析并作可視化處理[29],并對板栗與擬南芥PAT基因的復制事件進行共線性分析[30],使用TBtools作可視化圖。
1.6 板栗PAT基因家族的啟動子順式作用元件分析
在TBtools軟件中,提取PAT基因上游的啟動子前1.5 kb序列,順式作用元件的預測利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcar-e/)進行[31],可視化結果使用TBtools繪圖顯示。
1.7 板栗PAT基因轉錄因子的預測
CmPATs基因的轉錄因子由在線網站http://plantregmap.gao-lab.org/預測得到[32],其詞云由在線網站https://www.genescloud.cn/制作而成。
1.8 不同的脅迫處理
選取生長狀況一致的水培實生苗進行土培,對土培材料進行200 mmol·L-1 NaCl的脅迫處理,以不加NaCl為對照,每個處理3盆(1盆1株),并分別在7、10和14 d時采取同樣位置0和200 mmol·L-1 NaCl處理的葉片。
在進行干旱處理時,選取長勢一致的水培轉土培的板栗實生苗30株,采摘新鮮葉片,分別在新鮮葉片失水量為0%、5%、30%和50%時取樣。
在進行抗病處理時,選取新鮮板栗樹枝條,用打孔器打孔,深度以達到形成層未到木質部為準,將培養3 d的栗疫病菌絲塊接種于枝條的打孔處。以只進行打孔處理的新鮮枝條為對照。取樣時間分別為接種后的0、12、24 h和3 d。
以上樣品均用液氮進行速凍后放置-80 ℃超低溫冰箱保存待測,每個處理均為3次重復。
1.9 RNA提取、cDNA合成及實時熒光定量PCR分析
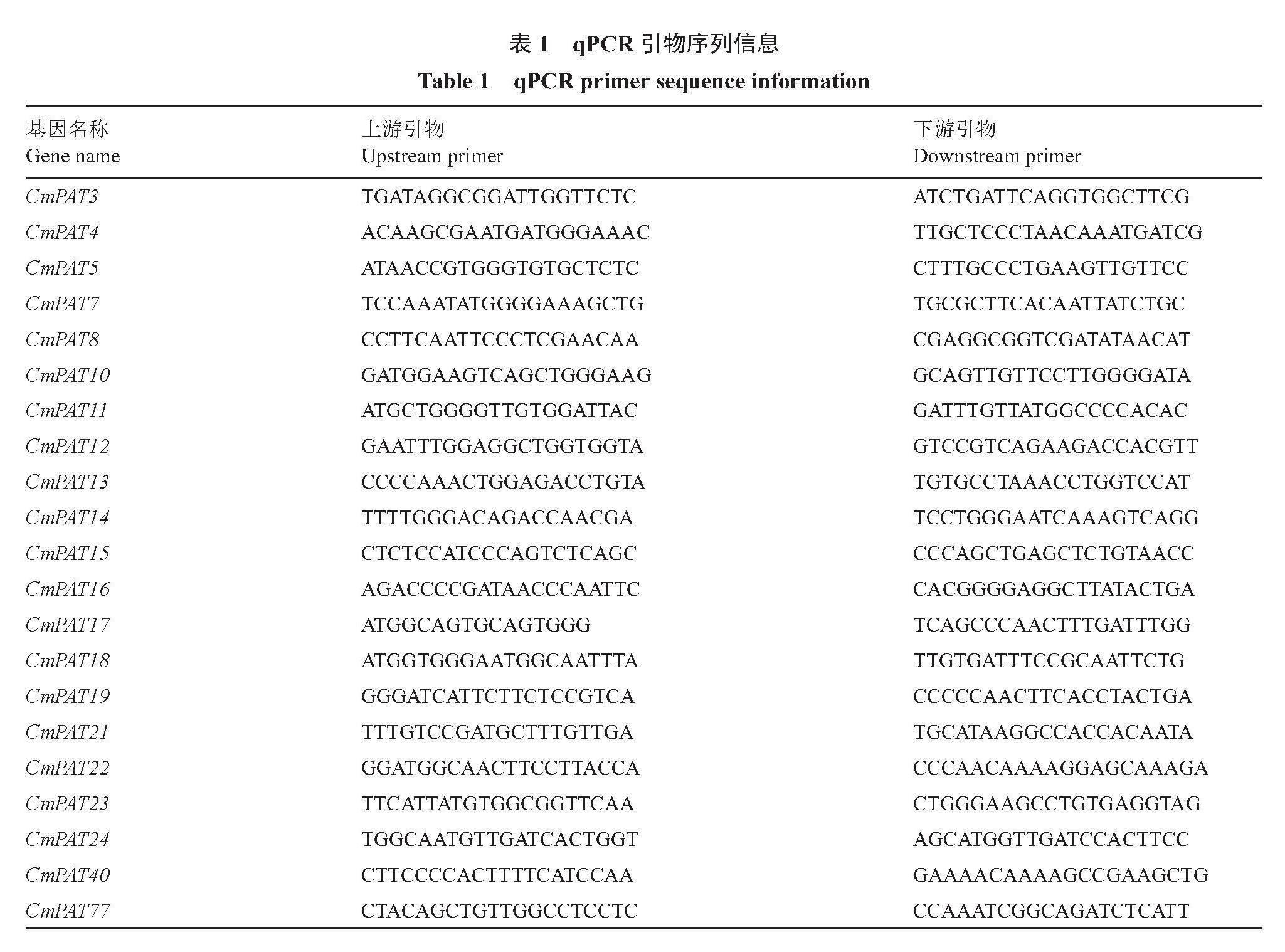
使用E.Z.N.A. Plant RNA Kit(Omega)試劑盒提取RNA。cDNA的合成使用Reverse Transcriptase M-MLV(RNase H-)試劑盒(TaKaRa,大連)。選擇Cmactin作為內參基因[20],利用Primer 3.0設計21個CmPAT基因的qPCR引物(表1),并由生工生物工程股份有限公司(上海)合成。使用Super Real Pre MixPlus (SYBR Green)試劑盒(Takala,大連),在CFX96(BIO-RAD,USA)上進行反應,每個樣品3次重復。qPCR反應程序為:預變性95 ℃ 2 min,變性95 ℃ 15 s,退火58 ℃ 30 s,延伸72 ℃ 30 s,共40個循環。利用2-△△Ct法計算CmPATs相對表達量。
2 結果與分析
2.1 板栗PAT基因的系統進化樹分析
利用HMMER、NCBI-CDD和SMART在板栗全基因組水平上進行搜索,并對獲得的CmPAT基因進行Blast,最終鑒定了21個CmPAT基因。為了解CmPAT蛋白的功能及進化關系,將21個CmPAT蛋白序列與擬南芥的24個AtPAT蛋白進行蛋白多序列比對,構建系統發育進化樹(圖1)。根據擬南芥分組情況,將系統進化樹分成6個亞組(Ⅰ~Ⅵ)。在6個亞組中,Ⅴ組和Ⅵ組為最小分支組,分別僅有一個成員:Cm08G0098和Cm07G01540,最大分支組為Ⅰ組(AtPAT1~9)和Ⅱ組(AtPAT11~16),均有6個成員。在多序列比對中,21個CmPAT成員均具有DHHC結構域。
2.2 CmPATs的基因結構和Motif可視化分析
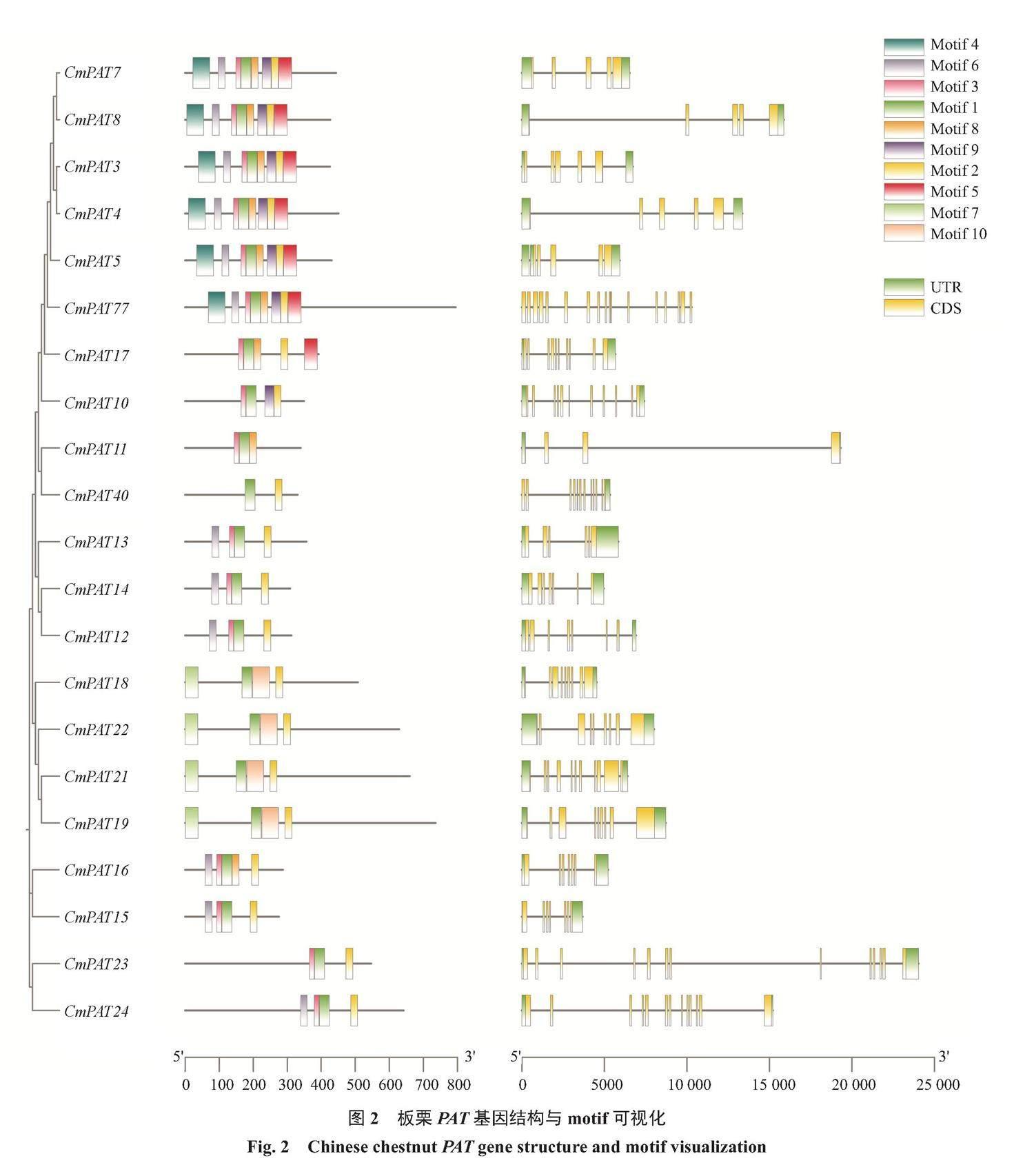
為了深入研究CmPATs的結構異同,通過在線網站MEME,在系統發育樹的基礎上預測到了PAT序列中的10個保守基序和基因結構圖(圖2)。在21個PAT基因中,發現每個PAT基因中包含不同種類和數目的Motifs。其中Motif4存在于6個CmPAT基因中(CmPAT7、CmPAT3、CmPAT4、CmPAT5、CmPAT77、CmPAT8),Motif5存在7個CmPAT基因中(CmPAT7、CmPAT3、CmPAT4、CmPAT5、CmPAT77、CmPAT8、CmPAT17)、Motif7和Motif10共同存在4個CmPAT基因中(CmPAT22、CmPAT21、CmPAT18、CmPAT19),說明它們具有相似的功能,在進化關系上也比較近。各個CmPAT的基因長度差異較大,范圍為3750~24 300 bp。所有的PAT基因都含有Motif1,則Motif1應為DHHC保守結構域。CmPAT77不存在UTR區,CmPAT40、CmPAT15只有1個UTR區,其他基因都具有2個以上的UTR區。
2.3 板栗PAT基因家族的蛋白理化性質分析和亞細胞定位
利用pfam數據庫與板栗V4基因組進行比對分析,獲得了板栗基因組中包含一個DHHC結構域的21個基因。隨后對其蛋白序列進行蛋白質理化性質分析,發現序列長度差異較大,在274~794個氨基酸之間,氨基酸長度最長的是CmPAT77,最短的是CmPAT15。平均分子質量為51.27 kDa,等電點為5.91~9.82,其中只有CmPAT3、CmPAT14、CmPAT23、CmPAT24、CmPAT77這5個蛋白的PI小于7,說明多數板栗PAT蛋白表現為堿性,少數為酸性。脂肪指數為73.60~104.69。蛋白的平均親水性(GRAVY)為-0.279~0.419,其中GRAVY為正值的有10個CmPAT蛋白,負值的有11個CmPAT蛋白。通過蛋白疏、親水性分析發現,僅有CmPAT12、CmPAT13、CmPAT16、CmPAT17、CmPAT40、CmPAT77這6個PAT蛋白脂肪系數大于100,為疏水性蛋白,其余均為親水性蛋白。同時,有12個PAT蛋白的不穩定系數小于40,9個PAT蛋白不穩定系數大于40,表明板栗PAT家族中大多為穩定蛋白。其中CmPAT77除了上述PAT家族共有的DHHC保守結構域外,還具有特異的泛素蛋白激酶序列。通過WoLF PSORT Prediction進行亞細胞定位,發現CmPAT7定位在葉綠體,CmPAT4定位在細胞核,CmPAT23定位在細胞質,剩余18個PAT蛋白定位在質膜中(表2)。
2.4 板栗PAT基因的染色體定位和共線性分析
繪制CmPAT基因在染色體上的分布圖,并利用1~12號染色體表示基因在染色體上的分布(圖3)。21個基因家族成員不均勻分布在1號、3號、4號、5號、7號、8號、10號、11號和12號染色體上,2號、6號和9號染色體上沒有CmPAT基因的分布。CmPAT24、CmPAT7、CmPAT8、CmPAT21分布于1號染色體上,CmPAT19分布在3號染色體上,CmPAT17、CmPAT11、CmPAT5和CmPAT22分布在4號染色體上,CmPAT77、CmPAT15、CmPAT13、CmPAT14、CmPAT12分布于5號染色體上,CmPAT40、CmPAT10、CmPAT23分別分布在7號、8號和10號染色體上,CmPAT3、CmPAT4及CmPAT16分布在11號染色體上,CmPAT18分布在12號染色體上。對板栗和擬南芥PAT基因進行共線性分析發現,在板栗與擬南芥之間存在10個具有共線性關系的PATs,在板栗間無共線性關系的PATs。板栗和擬南芥的PAT數目相差不大,因此CmPAT基因家族在物種間分化過程中比較保守(圖4)。
2.5 CmPATs基因啟動子順式作用元件分析
為探索CmPAT基因的功能,將21個板栗PAT基因上游1.5 kb的啟動子序列提交到PlantCARE網站進行順式作用元件預測。如圖5所示,在板栗PAT基因的啟動子區域中預測到基本順式作用元件、光響應元件,激素響應元件,多種逆境脅迫響應元件,植物生長發育相關元件等。基本順式作用元件數量最多,其中有943個TATA-box、623個CAAT-box 和152個AT~TATA-box等啟動子元件;其次為光響應元件,包括42個Box4、24個G-box、16個TCT-motif、13個GATA-motif、11個MRE、9個GT 1-motif等;在植物激素響應元件中,含有45個生長素響應元件(ARE、TGA-element)、19個赤霉素響應元件(P-box、TATC-box、GARE-motif、F-box)、24個乙烯響應元件ERE、60個脫落酸響應元件(ABRE、AAGAA-motif、ABRE4、ABRE3a)、42個水楊酸響應元件(as-1、TCA、TCA-element)等;在逆境脅迫響應元件中,含有7個干旱誘導元件(MBS、MBSI)、12個低溫響應元件LTR、20個損傷誘導元件(WUN-motif、WRE3)、12個抗病響應元件W-box等;植物生長發育相關元件包括12個參與玉米醇溶蛋白代謝調節元件O 2-site、4個與植物胚乳發育相關元件(GCN 4_motif、AACA_motif)、1個參與種子特異調控元件RY-element等;此外還預測到66個MYB和61個MYC參與環境適應性的響應元件等。推測CmPAT基因在板栗生長發育、激素調控、逆境脅迫及光調控中發揮著重要作用。
2.6 板栗PAT轉錄因子對應的詞云分析
通過轉錄因子預測發現,與21個板栗PAT基因調控相關的轉錄因子多達39種,其中以Dof蛋白家族的數量最多,BBR-BPC、AP2/ERF、MYB蛋白家族的數量在300~400之間,C2H2、GRAS、NAC、WRKY、TCP、HD-ZIP蛋白家族的數量在100~250之間,bHLH、TALE、bZIP、Trihelix、MYB_related、LBD、CPP、GATA、G2-like、MIKC_MADS、B3、WOX、HSF、C3H、ARF、ZF-HD、SBP蛋白家族的數量在20~85之間。其中Dof占比22%,AP2/ERF占比17%,BBR-BPC占比10%。這些轉錄因子可能在板栗中PAT基因的轉錄過程中發揮著重要作用(圖6)。
2.7 板栗PAT基因參與不同脅迫響應
為進一步了解板栗PAT基因在不同脅迫條件下的作用,對板栗實生苗進行不同濃度鹽脅迫和干旱脅迫處理,進行板栗PAT基因表達量熱圖繪制。
如圖7-A所示,21個CmPAT基因在不同時期不同程度地參與了鹽脅迫響應。在7 d時,相對于對照,有2個基因表達上調,5個基因表達下調。在10 d時,有11個基因表達上調,其中有6個基因表達明顯上調,2個基因表達明顯下調。在14 d時,有6個基因表達上調,5個基因表達下調,其中4個基因表達明顯下調。以上結果說明CmPAT基因廣泛參與鹽脅迫的響應。在干旱脅迫處理中,失水量為5%時,有2個基因表達明顯上調,失水量為30%時,有7個基因表達明顯上調,失水量為50%時,僅有1個基因表達未上調,且無表達下調的基因,說明CmPAT基因亦廣泛參與干旱脅迫響應。CmPAT24、CmPAT7、CmPAT14共同正向參與響應鹽脅迫與干旱脅迫(圖7-B)。
在植物生長過程中,除了非生物脅迫以外,還面臨著病原菌的侵染等生物脅迫,抗病的作用機制也較為復雜。在CmPAT基因響應抗栗疫病的轉錄組數據中發現,整體基因表達量數值較低。在栗疫病病菌侵染12 h時,與對照相比,基因表達量明顯上調的有2個,為CmPAT11和CmPAT4,分別上調了1.11倍和1.39倍,僅有CmPAT17表達量明顯下調,下調了72.61%。在栗疫病病菌侵染24 h時,與對照相比,有6個基因表達量明顯上調,分別為CmPAT24、CmPAT7、CmPAT16、CmPAT17、CmPAT14、CmPAT77,其基因表達量上調在1.5倍以上,其中CmPAT14和CmPAT77的基因表達量分別上調了2.79倍和2.49倍;CmPAT21和CmPAT22基因表達量下調較為明顯,分別下調了31%和37%。在栗疫病病菌侵染3 d時,與對照相比,有6個基因表達量明顯上調,分別為CmPAT7、CmPAT10、CmPAT16、CmPAT23、CmPAT17、CmPAT4,其基因表達量上調在1.5倍以上,其中CmPAT7和CmPAT17表達量分別上調2.61倍和2.44倍;CmPAT21、CmPAT5、CmPAT77基因表達量下調較為顯著,分別為42%、44%、57%。CmPAT7、CmPAT16和CmPAT17基因表達量在24 h和3 d 2個時期里均明顯上調,CmPAT21和CmPAT77均明顯下調,說明他們可能參與了響應栗疫病脅迫的網絡(圖7-C)。不同的CmPAT基因在不同脅迫中具有不同程度的響應,其中CmPAT7正向參與了鹽脅迫、干旱脅迫和抗病脅迫的調控。
3 討 論
蛋白質棕櫚酰化是一種后轉錄修飾的可逆反應,棕櫚酰基轉移酶催化酰基化的發生,發生酰基化的蛋白可以參與多種物質運輸、信號轉導脅迫響應。因此PAT家族作為參與調控植物生長發育與逆境脅迫的相關蛋白逐漸進入人們的視野。
在本研究中,筆者共鑒定出板栗中21個PAT基因家族成員,其基因數目與擬南芥基因數目接近,但不同物種間的PAT基因家族成員數目差異說明該基因家族在植物進化過程中產生一定程度的分化和擴增[33]。將鑒定出的CmPAT家族成員通過系統發育進化樹、基因結構和motif、蛋白質理化性質、染色體定位、基因復制事件、共線性和不同脅迫下表達模式等生物信息學方法進行綜合分析。
在系統發育進化樹中發現21個CmPAT蛋白中有17個與擬南芥成員存在同源關系,在同一個亞組中的成員含有一致或相似的蛋白保守基序,支持了進化分析的結果,同時也表明在氨基酸水平上存在同源關系的CmPAT和AtPAT間可能具有相似的生物學功能。Zhou等[18]對定位在液泡的AtPAT10的功能進行研究,表明AtPAT10對擬南芥的發育及耐鹽性至關重要,因此CmPAT10可能也具有耐鹽性等功能。通過分析板栗PAT基因上游啟動子中順式作用元件的組成情況發現,其上游存在大量生長發育和各種脅迫應答相關的順式作用元件,他們在轉錄過程中可能受到多種轉錄因子的調控,以此提高板栗在生長發育過程中的抗逆性。這與其他物種通過對啟動子進行分析來研究PAT家族成員對不同非生物脅迫等的響應機制結果一致[34-36]。
板栗在生長過程中,易受到鹽害和干旱等非生物脅迫。張新業等[37]對鑒定的27個胡蘿卜PAT基因進行鹽脅迫處理,發現有3個DsPAT響應了鹽脅迫。本研究中鹽脅迫處理下發現CmPAT24、CmPAT7、CmPAT13、CmPAT14在10 d和14 d表達上調顯著。筆者推測這些基因在不同時期可能參與板栗對鹽脅迫的響應,與前人研究結果相似。Tian等[33]發現OsPAT30參與了水稻耐鹽性的調控。Qi等[38]研究表明,擬南芥中的一個棕櫚酰基轉移酶AtPAT10對鹽脅迫響應極為敏感。姜翰[39]在對蘋果PAT16的功能進行研究時發現MdPAT16能抵抗鹽脅迫并促進蘋果糖分的積累。但在擬南芥和蘋果中響應鹽脅迫的PAT10和PAT16在板栗實生苗中并沒有明顯的響應,這與前人研究結果不一致,同時Tian等[33]的結果也表明了PAT基因功能在不同物種中對鹽脅迫具有一定的保守性。此外,呂慧等[40]發現干辣椒果實在干旱和鹽脅迫的共同刺激下,促進了10個基因的表達。在本研究中,CmPAT24、CmPAT7、CmPAT14亦共同正向響應鹽脅迫和干旱脅迫,這與呂慧等[40]的研究結果一致,在鹽脅迫和干旱脅迫雙重脅迫下促進多個基因的表達。ZmPAT24(ZmTIP1)能夠調節玉米根毛的長度及參與干旱脅迫的調控[41]。在本研究中,干旱脅迫失水量50%時,大部分CmPAT基因的表達量均上調,僅有CmPAT21未表現出明顯的抗旱性。這與前人研究結果相似,不同之處在于大部分CmPAT基因均參與調控板栗響應干旱脅迫。據報道,Deng等[36]發現部分GhPAT基因在病原菌、干旱、鹽等非生物脅迫下參與響應,并驗證了GhPAT27參與陸地棉花黃萎病抗性響應。Gao等[42]發現AtPAT13和AtPAT16通過棕櫚酰化修飾NB-LRR蛋白R5L1調控擬南芥的抗病機制。在本研究中,根據抗病脅迫轉錄組測序的結果發現,CmPAT7、CmPAT16和CmPAT17基因表達量在24 h和3 d 2個時期均明顯上調,CmPAT21和CmPAT77均明顯下調。說明他們可能參與了響應栗疫病脅迫的網絡。推測這5個基因極有可能參與板栗抗栗疫病的響應,并在板栗抗栗疫病中特異表達。CmPAT24、CmPAT7、CmPAT14共同響應鹽脅迫和干旱脅迫的調控,CmPAT7共同響應鹽脅迫、干旱脅迫和抗病脅迫的調控,部分CmPAT基因共同參與各種脅迫,與前人的研究結果一致。
4 結 論
筆者在本研究中共鑒定出21個板栗PAT基因家族成員,他們在鹽脅迫、干旱脅迫和抗病脅迫等生物和非生物脅迫中發揮著重要作用。其中CmPAT24、CmPAT7、CmPAT14共同正向參與了鹽脅迫和干旱脅迫的調控,CmPAT7同時正向響應鹽脅迫、干旱脅迫和抗病脅迫。研究結果可為研究板栗PAT基因家族的抗逆功能提供理論基礎。
參考文獻References:
[1]?? LI Y H,ZHANG Y Z,FENG F J,LIANG D,CHENG L L,MA F W,SHI S G. Overexpression of a Malus vacuolar Na+/H+ antiporter gene (MdNHX1) in apple rootstock M. 26 and its influence on salt tolerance[J]. Plant Cell,Tissue and Organ Culture,2010,102(3):337-345.
[2]?? MANN M,JENSEN O N. Proteomic analysis of post-translational modifications[J]. Nature Biotechnology,2003,21:255-261.
[3]?? WALSH G,JEFFERIS R. Post-translational modifications in the context of therapeutic proteins[J]. Nature Biotechnology,2006,24(10):1241-1252.
[4]?? SEET B T,DIKIC I,ZHOU M M,PAWSON T. Reading protein modifications with interaction domains[J]. Nature Reviews Molecular Cell Biology,2006,7:473-483.
[5]?? SOREK N,BLOCH D,YALOVSKY S. Protein lipid modifications in signaling and subcellular targeting[J]. Current Opinion in Plant Biology,2009,12(6):714-720.
[6]?? LINDER M E,DESCHENES R J. Palmitoylation:Policing protein stability and traffic[J]. Nature Reviews Molecular Cell Biology,2007,8:74-84.
[7]?? BAEKKESKOV S,KANAANI J. Palmitoylation cycles and regulation of protein function (Review)[J]. Molecular Membrane Biology,2009,26(1):42-54.
[8]?? GREAVES J,CARMICHAEL J A,CHAMBERLAIN L H. The palmitoyl transferase DHHC2 targets a dynamic membrane cycling pathway:Regulation by a C-terminal domain[J]. Molecular Biology of the Cell,2011,22(11):1887-1895.
[9]?? RESH M D. Trafficking and signaling by fatty-acylated and prenylated proteins[J]. Nature Chemical Biology,2006,2:584-590.
[10] SRIVASTAVA V,WEBER J R,MALM E,FOUKE B W,BULONE V. Proteomic analysis of a poplar cell suspension culture suggests a major role of protein S-acylation in diverse cellular processes[J]. Frontiers in Plant Science,2016,7:477.
[11] OHNO Y,KIHARA A,SANO T,IGARASHI Y. Intracellular localization and tissue-specific distribution of human and yeast DHHC cysteine-rich domain-containing proteins[J]. Biochimica et Biophysica Acta-General Subjects,2006,1761(4):474-483.
[12] OHNO Y,KASHIO A,OGATA R,ISHITOMI A,YAMAZAKI Y,KIHARA A. Analysis of substrate specificity of human DHHC protein acyltransferases using a yeast expression system[J]. Molecular Biology of the Cell,2012,23(1):4543-4551.
[13] LI Y X,QI B X. Progress toward understanding protein S-acylation:Prospective in plants[J]. Frontiers in Plant Science,2017,8:346.
[14] SOREK N,PORATY L,STERNBERG H,BAR E,LEWINSOHN E,YALOVSKY S. Activation status-coupled transient S acylation determines membrane partitioning of a plant Rho-related GTPase[J]. Molecular and Cellular Biology,2007,27(6):2144-2154.
[15] BATISTI? O,KUDLA J. Analysis of calcium signaling pathways in plants[J]. Biochimica et Biophysica Acta-General Subjects,2012,1820(8):1283-1293.
[16] GAGNE J M,CLARK S E. The Arabidopsis stem cell factor POLTERGEIST is membrane localized and phospholipid stimulated[J]. The Plant Cell,2010,22(3):729-743.
[17] WAN Z Y,CHAI S,GE F R,FENG Q N,ZHANG Y,LI S. Arabidopsis PROTEIN S-ACYL TRANSFERASE4 mediates root hair growth[J]. The Plant Journal,2017,90(2):249-260.
[18] ZHOU L Z,LI S,FENG Q N,ZHANG Y L,ZHAO X Y,ZENG Y L,WANG H,JIANG L W,ZHANG Y. PROTEIN S-ACYL TRANSFERASE10 is critical for development and salt tolerance in Arabidopsis[J]. The Plant Cell,2013,25(3):1093-1107.
[19] LAI J B,YU B Y,CAO Z D,CHEN Y M,WU Q,HUANG J Y,YANG C W. Two homologous protein S-acyltransferases,PAT13 and PAT14,cooperatively regulate leaf senescence in Arabidopsis[J]. Journal of Experimental Botany,2015,66(20):6345-6353.
[20] LI Y X,SCOTT R,DOUGHTY J,GRANT M,QI B X. Protein S-acyltransferase 14:A specific role for palmitoylation in leaf senescence in Arabidopsis[J]. Plant Physiology,2015,170(1):415-428.
[21] LI Y X,XU J F,LI G,WAN S,BATISTI? O,SUN M H,ZHANG Y X,SCOTT R,QI B X. Protein S-acyl transferase 15 is involved in seed triacylglycerol catabolism during early seedling growth in Arabidopsis[J]. Journal of Experimental Botany,2019,70(19):5205-5216.
[22] LI Y X,LI H J,MORGAN C,BOMBLIES K,YANG W C,QI B X. Both male and female gametogenesis require a fully functional protein S-acyl transferase 21 in Arabidopsis thaliana[J]. The Plant Journal,2019,100(4):754-767.
[23] HEMSLEY P A,KEMP A C,GRIERSON C S. The TIP GROWTH DEFECTIVE1 S-acyl transferase regulates plant cell growth in Arabidopsis[J]. The Plant Cell,2005,17(9):2554-2563.
[24] BATISTI? O,REHERS M,AKERMAN A,SCHL?CKING K,STEINHORST L,YALOVSKY S,KUDLA J. S-acylation-dependent association of the calcium sensor CBL2 with the vacuolar membrane is essential for proper abscisic acid responses[J]. Cell Research,2012,22(7):1155-1168.
[25] 劉曉書,劉芳,張俊. 京津冀地區板栗產業布局及前景分析[J]. 中國果樹,2022(2):99-102.
LIU Xiaoshu,LIU Fang,ZHANG Jun. Chestnut industry layout and prospect analysis in Beijing-Tianjin-Hebei region[J]. China Fruits,2022(2):99-102.
[26] 戴永務,劉偉平. 中國板栗產業國際競爭力現狀及其提升策略[J]. 農業現代化研究,2012,33(4):456-460.
DAI Yongwu,LIU Weiping. Study on current status and promotion strategy of international competitiveness of Chinese chestnut industry[J]. Research of Agricultural Modernization,2012,33(4):456-460.
[27] 郝雅瓊,劉紅星,王澤華,聶興華,李伊然,陳旭,王維香,秦嶺,邢宇. 栗疫病菌侵染板栗枝條的顯微觀察[J]. 植物保護,2022,48(1):179-184.
HAO Yaqiong,LIU Hongxing,WANG Zehua,NIE Xinghua,LI Yiran,CHEN Xu,WANG Weixiang,QIN Ling,XING Yu. Microscopic observation on infection process of chestnut branches by Cryphonectria parasitica[J]. Plant Protection,2022,48(1):179-184.
[28] TAMURA K,STECHER G,KUMAR S. MEGA11:Molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution,2021,38(7):3022-3027.
[29] CHEN C J,CHEN H,ZHANG Y,THOMAS H R,FRANK M H,HE Y H,XIA R. TBtools:An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[30] WANG Y P,TANG H B,DEBARRY J D,TAN X,LI J P,WANG X Y,LEE T H,JIN H Z,MARLER B,GUO H,KISSINGER J C,PATERSON A H. MCScanX:A toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research,2012,40(7):e49.
[31] LESCOT M,D?HAIS P,THIJS G,MARCHAL K,MOREAU Y,VAN DE PEER Y,ROUZ? P,ROMBAUTS S. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.
[32] GINESTET C. ggplot2:Elegant graphics for data analysis[J]. Journal of the Royal Statistical Society Series A:Statistics in Society,2011,174(1):245-246.
[33] TIAN Y,ZENG H,WU J C,HUANG J,GAO Q,TANG D Y,CAI L P,LIAO Z Y,WANG Y,LIU X M,LIN J Z. Screening DHHCs of S-acylated proteins using an OsDHHC cDNA library and bimolecular fluorescence complementation in rice[J]. The Plant Journal,2022,110(6):1763-1780.
[34] ZHOU J M,ZHANG Y L. Plant immunity:Danger perception and signaling[J]. Cell,2020,181(5):978-989.
[35] 龐宏光. 梨棕櫚酰基轉移酶基因家族鑒定及PbPAT14功能研究[D]. 保定:河北農業大學,2020.
PANG Hongguang. Identification of pear palmitoyl transferases gene family and functional study of PbPAT14[D]. Baoding:Hebei Agricultural University,2020.
[36] DENG Y H,CHEN Q J,QU Y Y. Protein S-acyl transferase GhPAT27 was associated with Verticillium wilt resistance in cotton[J]. Plants,2022,11(20):2758.
[37] 張新業,李文靜,朱姝,孫艷香,王聰艷,閆訓友,周志國. 三種傘形科蔬菜作物棕櫚酰基轉移酶基因家族的鑒定與分析[J]. 浙江農業學報,2023,35(6):1315-1327.
ZHANG Xinye,LI Wenjing,ZHU Shu,SUN Yanxiang,WANG Congyan,YAN Xunyou,ZHOU Zhiguo. Identification and analysis of PAT gene family in three kinds of Apiaceae vegetable crops[J]. Acta Agriculturae Zhejiangensis,2023,35(6):1315-1327.
[38] QI B X,DOUGHTY J,HOOLEY R. A Golgi and tonoplast localized S-acyl transferase is involved in cell expansion,cell division,vascular patterning and fertility in Arabidopsis[J]. The New Phytologist,2013,200(2):444-456.
[39] 姜翰. 蘋果棕櫚酰轉移酶基因MdPAT16促進糖分轉運和增強耐鹽性機理研究[D]. 楊凌:西北農林科技大學,2021.
JIANG Han. Mechanism of apple palmitoyltransferase gene MdPAT16 in promoting sugar translocation and enhancing salt tolerance[D]. Yangling:Northwest A & F University,2021.
[40] 呂慧,吉雪花,張中榮,朱冉冉,王世寧,謝雪果,袁雷.制干辣椒果實辣椒素對干旱、鹽及其雙重脅迫的響應[J].中國瓜菜,2022,35(2):78-84.
L? Hui,JI Xuehua,ZHANG Zhongrong,ZHU Ranran,WANG Shining,XIE Xueguo,YUAN Lei. Capsaicin of dry pepper fruit grown under drought,salt and combined stress condition[J]. China Cucurbits and Vegetables,2022,35(2):78-84.
[41] ZHANG X M,MI Y,MAO H D,LIU S X,CHEN L M,QIN F. Genetic variation in ZmTIP1 contributes to root hair elongation and drought tolerance in maize[J]. Plant Biotechnology Journal,2020,18(5):1271-1283.
[42] GAO J,HUANG G,CHEN X,ZHU Y X. PROTEIN S-ACYL TRANSFERASE 13/16 modulate disease resistance by S-acylation of the nucleotide binding,leucine-rich repeat protein R5L1 in Arabidopsis[J]. Journal of Integrative Plant Biology,2022,64(9):1789-1802.

