糖尿病腎病核心基因的篩選與鑒定
吳曉俊,倪飛雪,徐玉雪
糖尿病腎病(diabetic kidney disease, DKD)是糖尿病發展的常見并發癥,主要特征是尿白蛋白、腎小球過濾功能障礙,是終末期腎病的主要原因,嚴重增加了糖尿病患者的死亡率[1]。腎小球的功能障礙在DKD的病程發展中起著重要的作用,高濃度的血糖可誘導氧化應激、細胞凋亡[2]、免疫反應[3]等。近年來,內皮素拮抗劑、腎素-血管緊張素-醛固酮系統阻滯劑、血管緊張素轉換酶抑制劑等的研究進展,為DKD的治療提供了方向[4]。目前仍缺乏對DKD病理機制和藥物靶點的探究,且臨床治療結果不理想[5]。然而,生物信息學的發展為疾病研究提供了新的視角[6]。基于對DKD腎小球基因表達矩陣進行的加權共表達網絡分析(weighted gene co-expression network analysis,WGCNA)、最小絕對收縮和選擇算法(least absolute shrinkage and selection operator,LASSO)、特征選擇之支持向量機遞歸特征消除(support vector machine-recursive feature elimination,SVM-RFE)等分析方法,有助于對DKD的機制進行深入研究,篩選核心生物學標志物與治療靶點,尋找與DKD相關的潛在治療基因。
1 材料與方法
1.1 數據資料來源使用基因表達數據庫(GEO)(https://www.ncbi.nlm.nih.gov/geo/),搜索關鍵詞“Diabetic kidney disease”和“Homo sapiens”,選擇“Expression profiling by array”以獲得DKD的mRNA微陣列基因表達矩陣。篩選得到了GSE47183和GSE30528及對應數據平臺文件(包括30個正常對照樣本,23個DKD腎小球樣本),運用R(4.2.1)將基因表達數據整合并進行生物信息學分析。GEO屬于公共數據庫,數據庫中涉及的患者已獲得倫理批準,因此本研究不涉及倫理問題及其他利益沖突。
1.2 生物信息學數據分析使用R中的limma包對數據歸一化處理,通過WGCNA分別對GSE47183和GSE30528數據聚類分析,篩選與疾病具有顯著相關性的核心基因集,并通過韋恩圖(Veen diagram,Veen)對基因集進行重疊,對共有的顯著核心基因使用LASSO和SVM-RFE算法進一步分析,篩選目標核心基因。同時,為了得到與目標核心基因相關的藥物靶點,運用基因藥物分析網絡分析(https://dgidb.genome.wustl.edu/)預測基因藥物作用靶點,并且使用基因集合富集分析(gene set enrichment analysis,GSEA)篩選目標核心基因可能參與調控的信號通路。
1.3 RNA提取及實時熒光定量PCR使用南京諾唯贊RNA抽提試劑盒(L/N 7E632K2)提取高脂飲食(high-fat diet,HFD)喂養糖尿病小鼠與同齡健康C57小鼠腎臟組織總mRNA,使用Takara試劑盒將RNA逆轉錄為模板cDNA,檢測樣本量為1 μg,依照SYBR Ex Tap Ⅱ試劑說明書進行擴增,β-actin為內參,驗證核心基因足細胞標記蛋白(NPHS1)、Ⅰ型膠原蛋白α2鏈(collagen type I alpha 2 chain,COL1A2)、轉化生長因子β誘導蛋白(transforming growth factor beta induced,TGFBI)、分化集群48(cluster of differentiation 48,CD48)、1號染色體開放閱讀框21(chromosome 1 open reading frame 21,C1orf21)在腎臟中的基因表達水平。
1.4 統計學處理差異表達水平使用R(4.2.1)及GraphPad Prime 8.0統計分析,使用t檢驗,以標準誤差(SEM)表示平均值的誤差,P<0.05表示差異有統計學意義。
2 結果
2.1 差異基因mRNA的篩選和分析提取GEO數據庫GSE30528和GSE47183中DKD腎小球的mRNA數據,作火山圖分析。結果表明,DKD數據GSE30528共有317個具有顯著性差異的基因(P.adj<0.05, |log2FC|>1),235個基因表達下調(綠色),82個基因表達上調(紅色)(圖1A)。GSE47183數據共有209個具有顯著表達的差異基因(P.adj<0.05, |log2FC|>1),100個基因表達下調(綠色),109個基因表達上調(紅色)(圖1B)。使用Veen圖重疊差異基因,62個差異基因在兩個數據庫均表達(圖1C)。對此62個差異基因進行KEGG分析,結果顯示這些差異基因與細胞外基質(extracellular matrix,ECM)受體相互作用,PI3K-Akt信號通路具有顯著相關性。GO結果表明,差異基因主要與細胞因子的正調節具有相關性,主要在細胞外間質結構成分中起作用(圖2B-2D)。
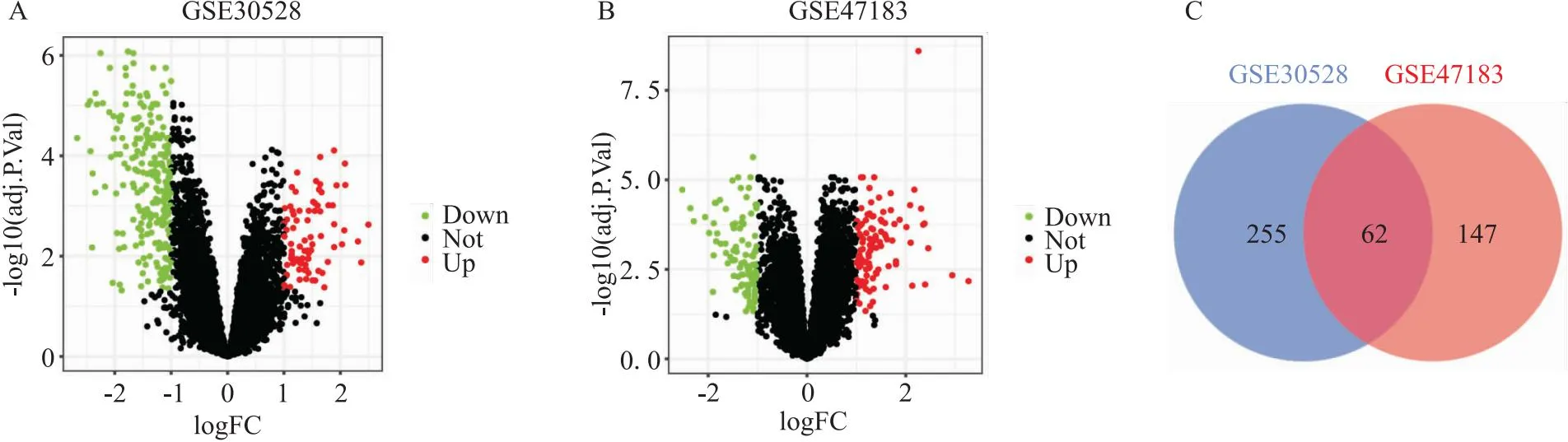
圖1 差異基因的提取
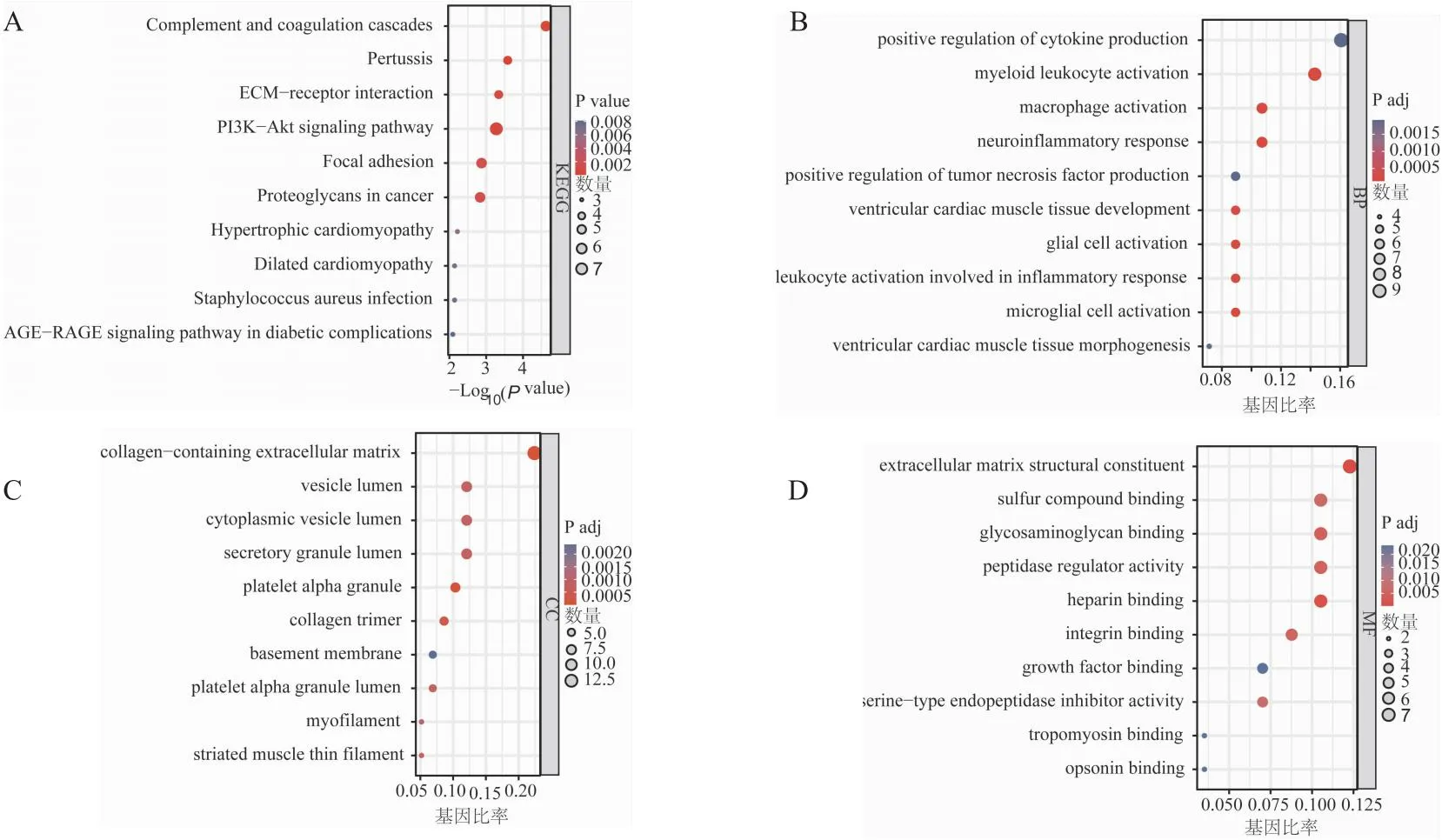
圖2 差異基因KEGG和GO分析
2.2 WGCNA篩選疾病核心基因將DKD表達矩陣GSE30528和GSE47183分別用R(limma包)對數據歸一化,皮爾遜相關系數對樣品聚類,進行WGCNA,篩選與疾病關系密切相關的核心基因。軟闕值分別為16和12時,滿足R2=0.9的無標度的網絡拓撲關系(圖3A、3B)。將數據集的鄰接矩陣轉換為TOM矩陣,對樣本聚類,構建WGCNA網絡(圖3C),篩選出不同顏色的模塊基因集。值得關注的模塊分別是GSE30528的ME turquoise(r=0.93,P=5e-10)和GSE47183的ME blue(r=0.72,P=5e-6),其與疾病狀態最具有關聯性(圖3D)。同時,證實了兩個模塊(turquoise模塊,blue模塊)中基因集的相關性和顯著性,turquoise模塊:Cor=0.92,P=3.8e-172(GSE30528),blue模塊:Cor=0.63,P=2.7e-21(GES47183),見圖3E、3F。分別將GSE30528、GSE47183的顯著性差異基因與WGCNA篩選的核心基因使用Veen圖重疊篩選,富集出37個核心基因(圖4A),對兩組數據進行合并,熱圖展現每個基因的表達水平,共有10個基因表達下調(藍色),27個基因表達上調(紅色)(圖4B)。

圖3 WGCNA篩選疾病核心基因
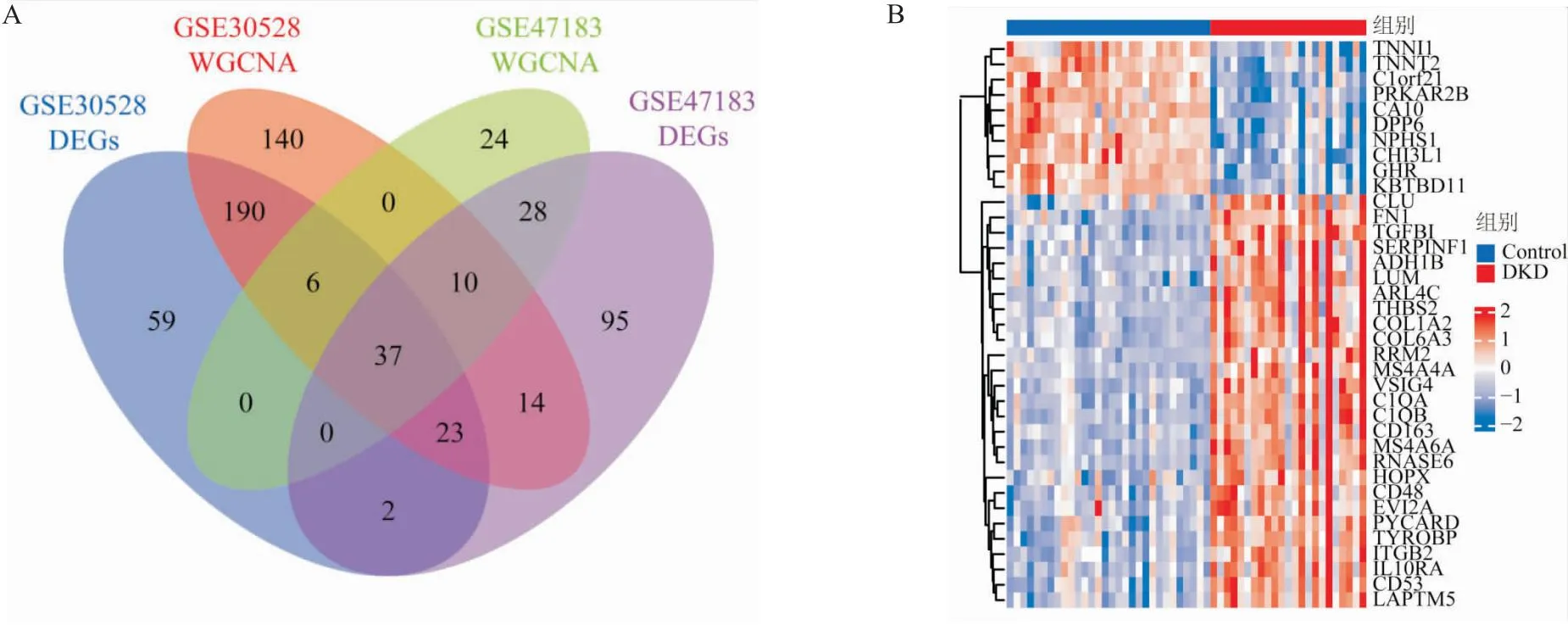
圖4 核心基因的篩選和展示
2.3 機器學習算法篩選特征基因對37個核心基因使用LASSO模型算法,繪制LASSO回歸圖形和交叉驗證圖形,篩選出6個特征基因(圖5A、5B)。同時,使用SVM-RFE算法對37個核心基因進行過濾,篩選特征基因的最優組合,10個特征基因被顯著富集(圖5C、5D)。Veen圖對兩種算法的核心基因取交集,共同篩選出5個目標核心基因,這5個重疊交叉的核心基因將是進一步研究的重點(圖5E)。

圖5 目標核心基因的篩選
2.4 目標核心基因的表達水平及相關性將GEO數據庫中的GSE30528和GSE47183數據合并,共有30個正常腎小球樣本,23個DKD腎小球樣本,分析5個核心基因在數據中的表達情況。結果表明,CD48、COL1A2、TGFBI的表達水平顯著升高,NPSH1、C1orf21的表達水平顯著降低(P. adj<0.05, |log2FC|>1)(圖6A-6E)。皮爾森相關性分析確定5個基因間的相關性(圖6F)。基于差異基因集構建的邏輯回歸模型,繪制的受試者工作特征曲線(receiver operating characteristic curve,ROC)顯示,5個核心基因的ROC曲線下面積(area under the receiver operating characteristic curve,AUC)均大于0.8(圖7A-7E),進一步表明這些核心基因可能與DKD疾病具有密切關系。
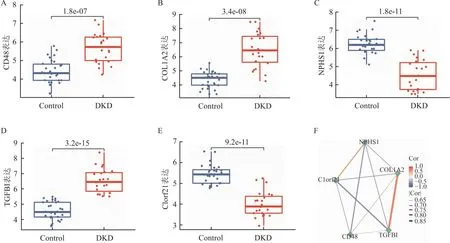
圖6 目標核心基因的表達水平及皮爾森相關性分析

圖7 目標核心基因的ROC曲線
2.5 核心基因的生物學功能和通路分析使用GSEA進行數據分析(c2.cp.all基因集作富集使用),結果顯示NPHS1在細胞外機制及細胞交流信號通路顯著富集(圖8A),COL1A2主要與整合素細胞表面互作相關(圖8B),CD48在自然殺傷細胞毒性和細胞表面相互作用中起作用(圖8C),而TGFBI與ECM糖蛋白和整合素途徑有關(圖8D)。然而C1orf21的研究目前匱乏,功能尚不明確,無法進行GSEA富集。

圖8 目標基因的GSEA分析
2.6 目標核心基因生物學驗證為進一步研究目標基因在DKD的表達,使用HFD(加小劑量鏈脲佐菌素)誘導的糖尿病小鼠腎臟組織,檢測5個核心基因(NPHS1、CD48、COL1A2、TGFBI、C1orf21)表達水平,如圖9所示,NPHS1表達降低(圖9A),CD48表達升高(圖9B),而COL1A2、TGFBI和C1orf21的表達則無顯著變化(圖9C-9E)。結果表明,NPHS1和CD48符合上述的生物信息學分析結果,它們可能與DKD的發生和發展具有密切關系。
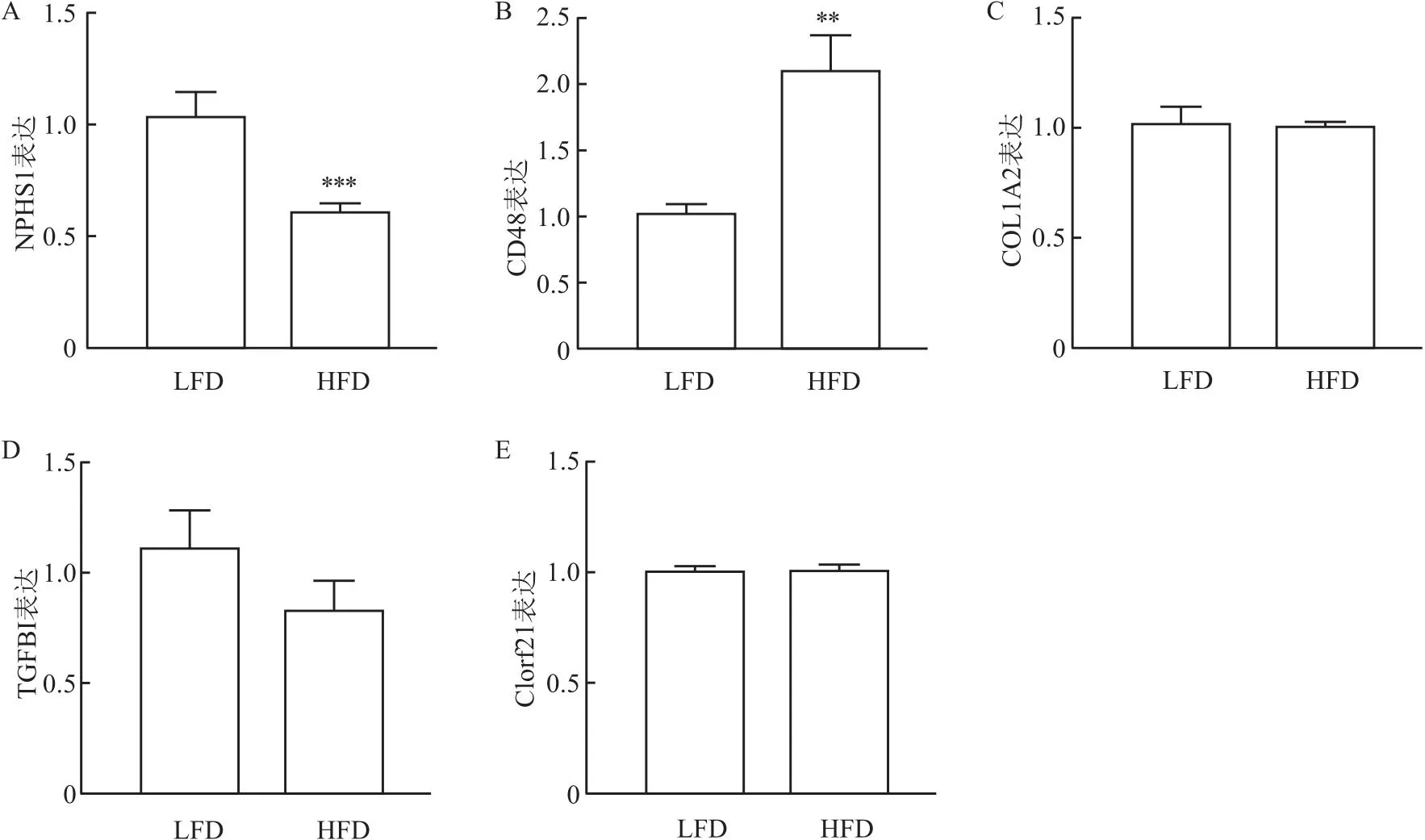
圖9 核心基因mRNA表達水平
2.7 核心基因的藥物治療與靶點預測為了研究核心基因對DKD的有效治療,進行了目標基因的基因-藥物的靶點預測,以及指導mRNA合成的有效miRNA,通過基因藥物相互作用網站預測基因靶點。僅預測出LOSARTAN以NPHS1為靶點發揮治療作用(圖10A)。使用3種軟件(miRanda、miRDB、TargetScan)分析NPHS1合成相關的miRNA,結果表明,多種miRNA直接指導NPHS1的合成(圖10B)。
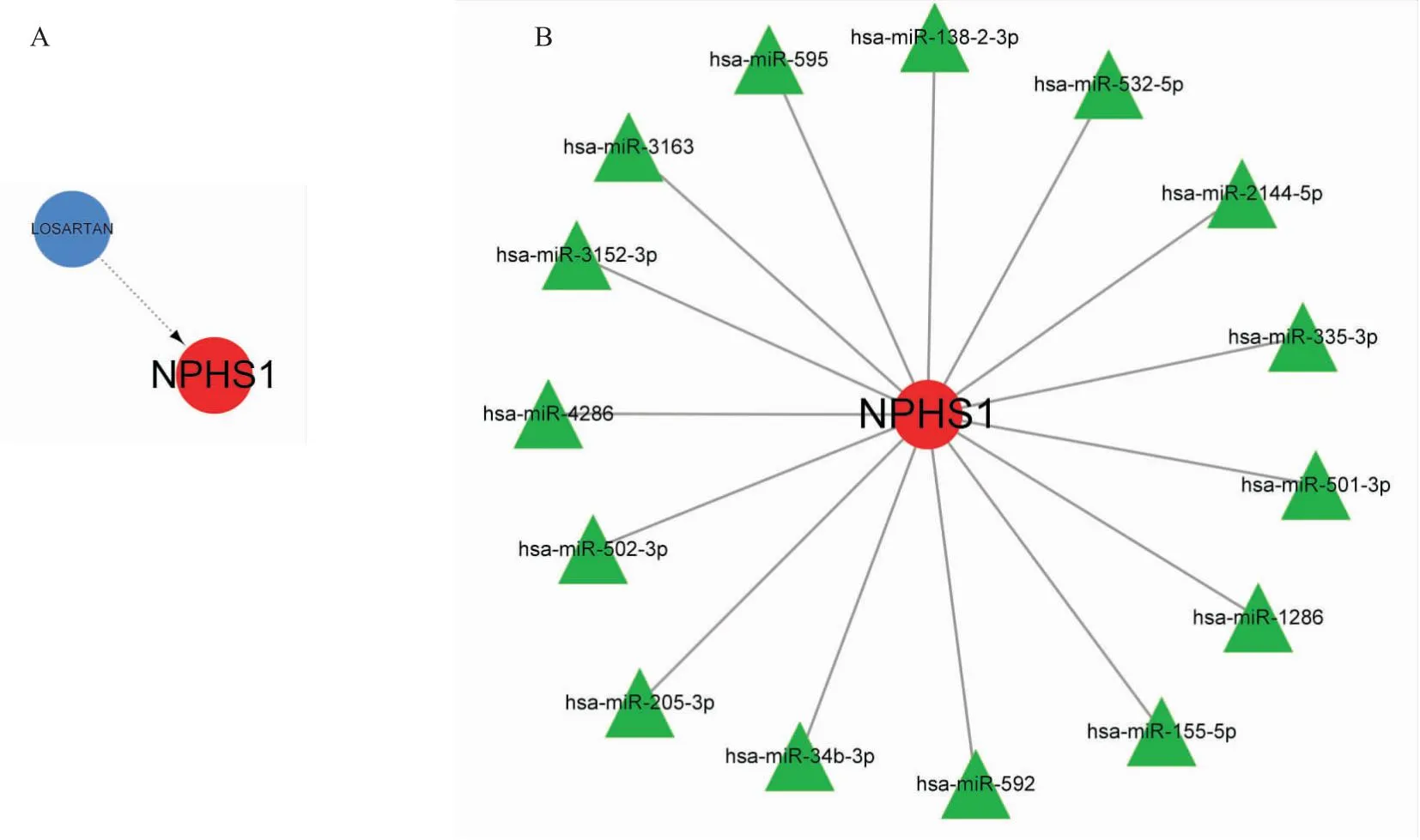
圖10 核心基因藥物及靶點預測
3 討論
DKD是導致腎衰竭的主要原因。盡管目前對于DKD疾病已有較清晰的了解,但是DKD的治療方法十分有限,具體病理機制仍不甚清楚[7]。近年來,基于mRNA微陣列分析技術的生物信息學迅速發展,為探究疾病發展的分子機制起到指導作用。在本研究中,通過GEO公共數據采用WGCNA對數據進行分類,篩選出37個與DKD具有密切相關性的顯著性差異基因,包括10個下調基因和27個上調基因。對此37個差異基因采用LASSO、SVM-RFE機器算法及皮爾森相關性分析進一步篩選出與DKD具有密切關聯性的5個核心基因(NPHS1、COL1A2、CD48、TGFBI、C1orf21),并通過HFD造模糖尿病小鼠腎臟組織的RNA檢測,進一步驗證了核心基因的表達情況。
對核心基因的GSEA分析結果發現,NPHS1與NEPH1信號通路及細胞交流呈負相關。有研究[8]表明,NEPH1是免疫球蛋白超家族中的一員,在結構上與腎素相關,誘導細胞黏附。NPHS1和NEPH1的基因產物Nephrin和NEPH1是Ig超家族的足細胞膜蛋白,NPHS1的缺乏會嚴重導致尿蛋白癥,使疾病迅速惡化[9]。在DKD中,分析結果表明,COL1A2的表達與DKD細胞外基質及細胞表面整合素相互作用顯著相關,COL1A2是I型膠原的成員,屬纖維化基因,因此COL1A2的高表達與DKD中的腎纖維化有關[10]。有證據指出COL1A2的表達與DKD中由高糖誘導的炎癥反應具有顯著相關性[11],針對COL1A2的治療,是DKD管理的潛在干預策略,然而,在糖尿病小鼠腎臟組織中,COL1A2卻沒有顯著變化。與此同時,越來越多的研究[12]發現,DKD的發展中存在著免疫機制的參與,T淋巴細胞、B淋巴細胞、巨噬細胞等與DKD密切相關,CD48主要參與自然殺傷細胞介導的細胞毒性及反應體細胞表面相互作用(圖8C)。CD48是信號淋巴細胞激活分子家族的成員,參與免疫細胞的黏附和激活[13],在DKD中的高表達激活了自然殺傷細胞的活力,引起了免疫細胞的激活。同樣的,TGFBI是轉化因子誘導蛋白,參與細胞的生長、分化以及細胞黏附等。在腫瘤研究中,TGFBI在很大程度上促進了腫瘤細胞的進展[14],在DKD中卻沒有報道。然而,TGFBI能夠調控ECM糖蛋白,而ECM蛋白與脂肪組織相關,并調節炎癥、纖維化、血管生成和代謝惡化[15]。因此推測TGFBI的表達調節DKD中的腎纖維化和免疫炎癥具有密切關系,但本研究顯示,TGFBI在HFD小鼠腎臟組織中并沒有發生顯著變化。除此之外,發現C1orf21同樣是被顯著富集出的基因,C1orf21是一個目前還未功能注釋的非特征性蛋白質編碼基因。針對C1orf21的研究十分匱乏,其表達差異意味著1號染色體的功能性障礙,這有待于進一步研究確認。綜上所述,NPHS1、CD48、COL1A2、TGFBI、C1orf21基因可能與DKD具有密切相關性,通過參與細胞表面相互作用,影響免疫細胞的激活、細胞黏附以及促進腎纖維化影響疾病的惡化。而基因靶點藥物和有效miRNA的預測為研究DKD的治療提供了重要指導。本研究表明,COL1A2、CD48與DKD的發展具有顯著相關性,而針對NPHS1的基因靶點藥物治療及miRNA的預測在DKD中有可能作為重要的突破點。

