赤琥珀螺線粒體全基因組測定與系統發育關系的研究
馬蕊 張衛紅 郭陽陽
摘 要 旨在通過高通量測序技術獲得赤琥珀螺(Succinea erythrophana)線粒體基因組全序列,對其結構及組成特征進行分析,并結合NCBI數據庫已公布的琥珀螺科及柄眼目物種序列,以最大似然法和貝葉斯法分別進行系統發育分析。結果顯示:①赤琥珀螺線粒體基因組全長? 14 023 bp(GenBank No.ON533899),由37個基因和一段富含AT的非編碼控制區組成,有20處基因間隔,13處基因重疊。A+T平均含量為77.3%,表現出明顯的AT偏向性。基因的排列順序、結構與組成、密碼子使用情況與琥珀螺科已報道種類相似。? ②除tRNA-Phe、tRNA-His、tRNA-Ser1、tRNA-Ser2外,其余? tRNAs呈典型三葉草結構。③Ka/Ks選擇壓力分析顯示其受到純化選擇作用。④系統發育研究揭示赤琥珀螺與同屬的腐敗琥珀螺(Succinea putris)親緣關系最近,然后與同科其他物種形成姐妹群關系。
關鍵詞 赤琥珀螺;琥珀螺科;線粒體基因組;系統發育
線粒體基因組是動物系統發育研究中廣泛使用的分子標記[1]。這主要得益于其分子結構相對簡單、具有較高的進化速率、母系遺傳、重組率低和拷貝數高等優點[2-3]。近年來,隨著基因組測序技術的發展和測序成本的下降,對線粒體基因組的研究報道大量增加,但不同動物類群的研究存在一定差異,對無脊椎動物的關注明顯少于脊椎動物。
琥珀螺科(Succineidae)是軟體動物門(Mollusca)、腹足綱(Gastropoda)、柄眼目(Stylommatophora)的主要類群[4]。該科物種形態多樣,大多數種類水陸兩棲,其中一些種類危害棉、麻、糧、蔬菜等農作物和園藝花卉,有些是家畜、家禽及野生動物寄生吸蟲、線蟲的中間宿主,而另有一些種類其軟體和貝殼均可入藥,因而琥珀螺科與人類關系十分密切[5]。但對琥珀螺科的研究多集中在形態方面,線粒體基因組研究十分有限,目前僅有腐敗琥珀螺(Succinea putris)[6]、五家渠尖緣螺(Oxyloma wujiaquensis)[7]和Omalonyx unguis[8]共3個物種的線粒體基因組被報道。
赤琥珀螺(Succinea erythrophana Ancey,1883)是琥珀螺科中的小型種類,為中國特有種,在吉林、北京、河北、山西、陜西、新疆和長江中下游及其以南的兩廣地區廣泛分布[9]。赤琥珀螺也是傳統中藥,中藥名為緣桑螺,主治小兒驚風,痔瘡,脫肛等癥(中藥大辭典,中華本草)。目前對赤琥珀螺的研究僅見貝殼形態和分布地的簡單記述[10],本試驗將對其進行線粒體全基因組測定以及系統發育關系研究,以期為該物種的系統進化及保護利用提供分子依據。
1 材料與方法
1.1 樣品采集
赤琥珀螺標本于2013年8月11日采自新疆伊犁察布查爾縣烏孫山公路邊及山坡松林地? (43°28′N,81°06′E),海拔2 106 m,采集人張衛紅等。標本鑒定由李成有和張衛紅依據其形態解剖特征完成[11],現保存于新疆大學生命科學與技術學院(標本號:XJU S13029)。
1.2 DNA提取
選取一只成體赤琥珀螺,剪取腹足肌肉約? 30 mg,使用天根公司DNA組織提取試劑盒提取總DNA。提取結果經檢測合格后(DNA質量濃度超過100 ng/μL)送上海天昊生物有限公司進行高通量測序。
1.3 線粒體基因組測序及組裝
利用二代高通量測序技術(Illumina Hiseq平臺)對赤琥珀螺線粒體全基因組測序。對下機后的數據進行質控,低質量讀段、N率較高序列及測序接頭序列等被過濾。質控后的contigs以同屬物種腐敗琥珀螺(GenBank No. JN627206)作為參考基因組進行序列比對,將雙端數據輸入MetaSPAdes中進行組裝。使用ORF finder在線分析確定蛋白質編碼基因的邊界,使用? tRNAscan預測tRNA的位置及其二級結構。通過MITOS在線服務[12]進行結構功能注釋,并在Geneious R11軟件[13]中對注釋結果手動校正。拼接組裝后的序列使用OGDRAW在線平臺[14]繪制基因結構圖。最后,將赤琥珀螺線粒體全基因組數據提交至NCBI數據庫,獲得GenBank登錄號為ON533899。
1.4 基因組序列分析
使用Geneious R11軟件統計堿基組成情況;在CodonW v1.4.2軟件中統計密碼子偏向性及使用情況;通過DnaSP v.5.10軟件[15]計算每個蛋白質編碼基因同義替換(Ks)和非同義替換(Ka)概率,Ka/Ks大于1、等于1和小于1分別表示受到正選擇、中性進化和純化選擇作用[16]。對線粒體基因組鏈的非對稱性(strand asymmetry),即兩條鏈的組成偏差利用公式AT shew=(A-T)/(A+T)和GC shew=(G-C)/(G+C)分別進行計算。
1.5 系統發育分析
基于柄眼目不同類群具代表性的14個物種(表1)的線粒體蛋白質編碼基因,以新腹足目塔螺科物種Lophiotoma cerithiformis作為外群,使用貝葉斯法(Bayesian analysis,BI)和最大似然法(Maximum likelihood methods,ML)進行系統發育分析(表1)。其中,蛋白質編碼基因使用PhyloSuite v1.2.2軟件[17]提取串聯。BI樹使用MrBayes v3.2.6軟件[18]生成,馬爾可夫鏈蒙特卡洛分析運行100萬代,每1 000代抽樣保存1個樣本,前25% burnin;ML樹使用IQ-TREE v.1.6.8軟件構建,建樹模型選用GTR+F+G,自舉檢驗次數設定為1 000次。使用iTOL在線程序[19]對系統發育樹進行美化。
2 結果與分析
2.1 線粒體基因組結構及組成
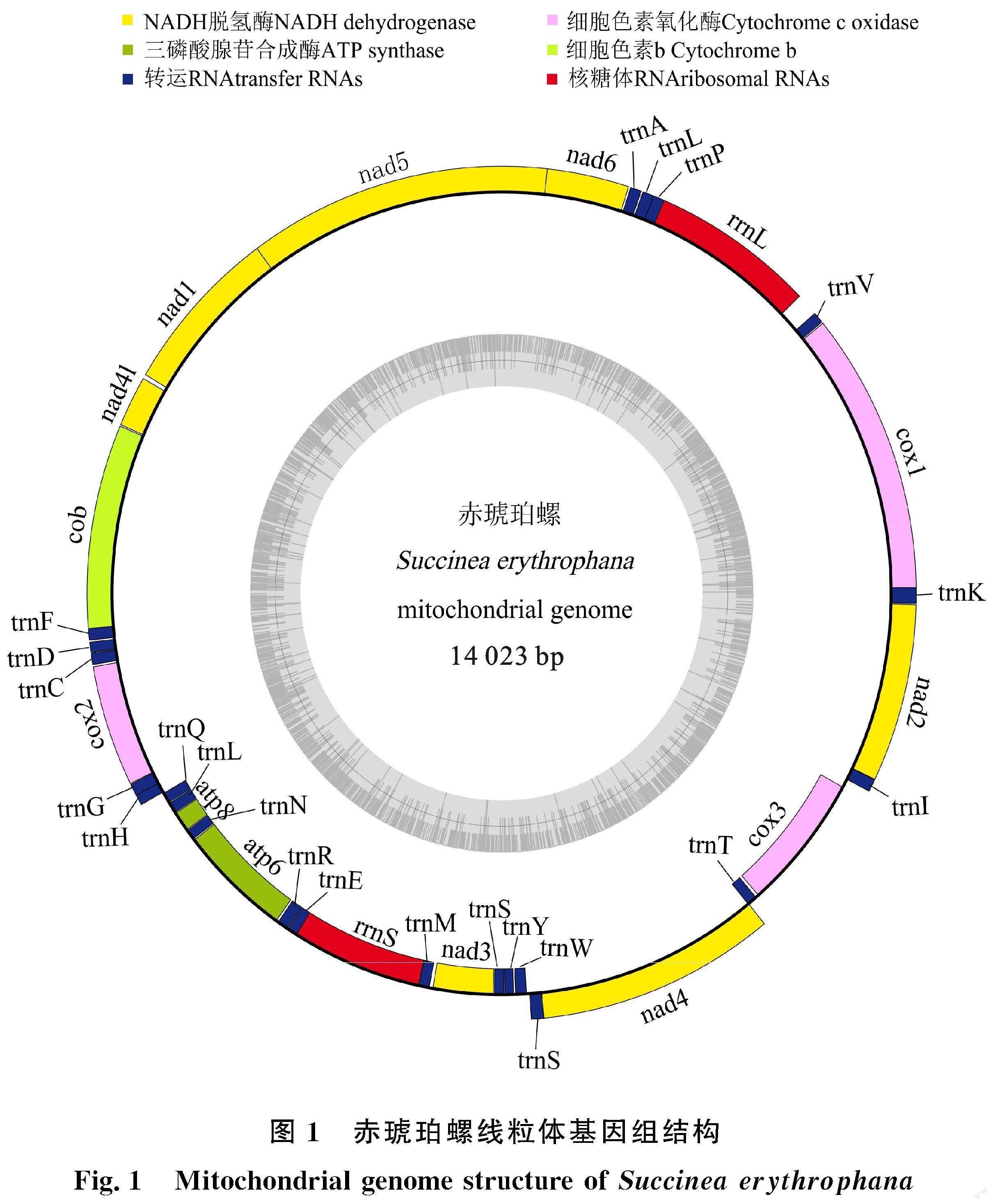
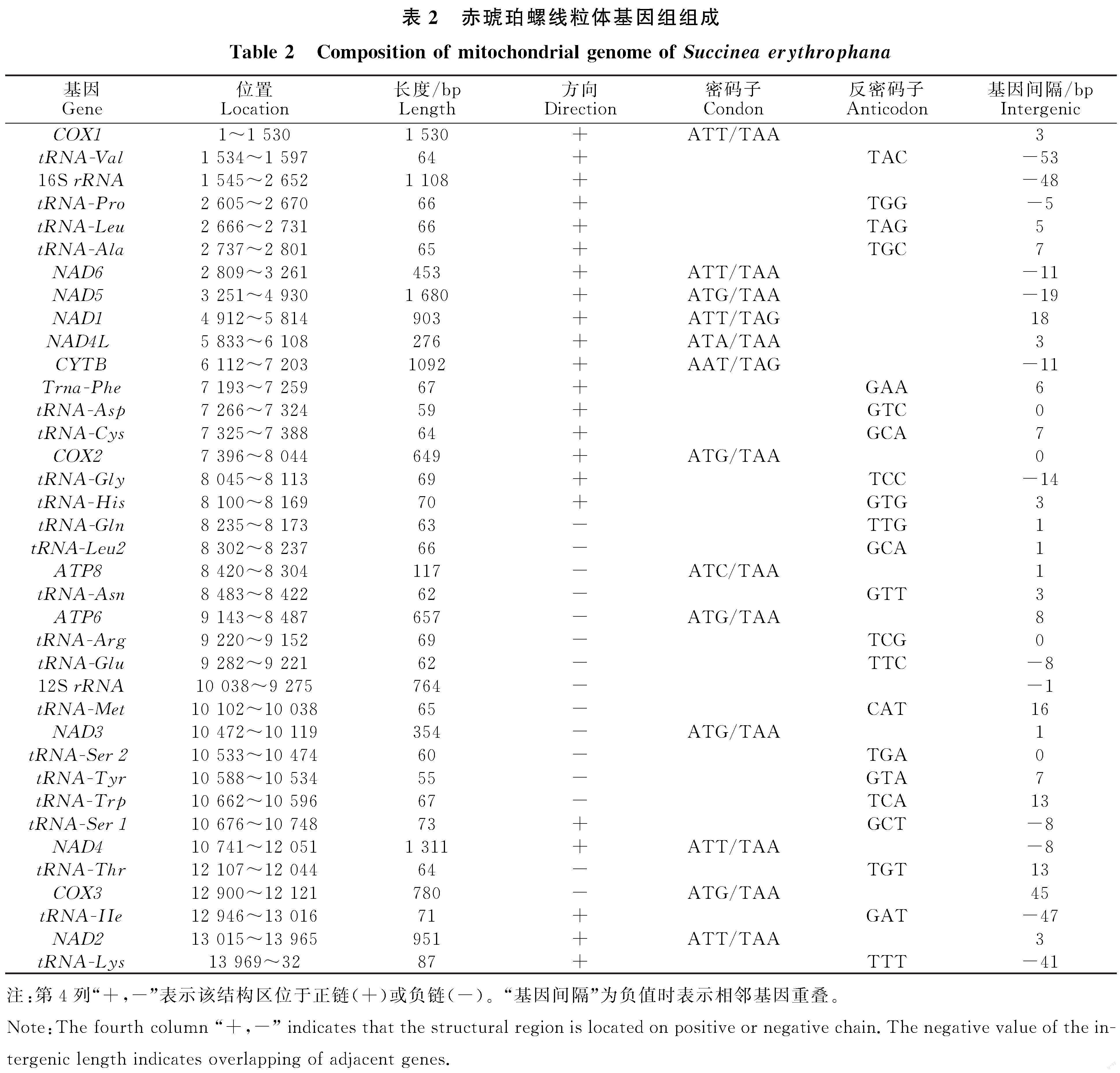
赤琥珀螺線粒體基因組為典型環狀閉合雙鏈結構(圖1),全長14 023 bp,由37個基因組成,包括13個蛋白質編碼(Protein coding genes,PCGs)基因、22個tRNA基因、2個rRNA基因以及1個富含AT的非編碼區。A、T、C、G平均含量為33.5%、43.8%、10.6%和12.0%,其中? A+T含量為77.3%,存在明顯的AT偏向性。37個基因中22個位于正鏈,包括9個PCGs,12個tRNAs和1個rRNA基因;15個位于負鏈,包括4個PCGs,10個tRNAs和1個rRNA基因。存在基因間隔20處,基因重疊13處(表2)。
2.2 蛋白質編碼基因
赤琥珀螺13個蛋白質編碼基因長度在117 bp( ATP8)到1 680 bp( NAD5),總長? 10 753 bp,AT含量為76.7%,AT偏斜(AT shew)為? -0.16,GC偏斜(CG shew)為0.07。13個PCGs中,12個基因以ATN作為起始密碼子,只有CYTB 基因以TTG起始;11個基因以TAA為終止密碼子,只有 NAD1和CYTB基因以TAG終止。
13個PCGs共編碼3 666個氨基酸,使用頻率較高的密碼子為UUU(Phe)、UUA(Leu)和AUU(Ile),總體偏向于使用第3位為A或T(U)的密碼子(圖2)。
2.3 選擇壓力分析
赤琥珀螺線粒體基因組中所有PCGs的Ka/Ks值均小于1(圖3),即非同義突變的頻率少于同義突變,表明整體受到純化選擇作用。在13個PCGs中,ATP8、NAD2基因的Ka/Ks值相對較高(分別為0.392 8和0.387 9),表明它們受到較小的選擇壓力,而COX1的Ka/Ks值最小? (0.053 4),表明其受到的選擇壓力最大。
2.4 tRNA和rRNA基因
赤琥珀螺tRNAs基因總長為1 422 bp。除tRNA-Phe、tRNA-His、tRNA-Ser1、tRNA-Ser2外,其他18個tRNAs的二級結構均為典型三葉草結構(圖4)。其中,tRNA-Phe和tRNA-His均缺少TΨC臂,額外環較長,氨基酸臂中出現了錯配;tRNA-Ser1和tRNA-Ser2均缺少DHU臂,其中,tRNA-Ser1基因在原本的DHU臂處出現了1個8 bp的小莖環結構,額外環較長,氨基酸臂中出現兩對錯配。
對于rRNA基因,16S rRNA位于tRNA-Val和tRNA-Pro之間,長度為931 bp;12S rRNA位于tRNA-Glu和tRNA-Met之間,長度為764 bp。
2.5 控制區
赤琥珀螺線粒體基因組控制區與腐敗琥珀螺及Omalonyx unguis位置一樣,均位于COX3與tRNA-Ile基因之間,其中,前者控制區長度為? 45 bp,后兩者均為50 bp,但與同在新疆分布的五家渠尖緣螺有所不同,五家渠尖緣螺的控制區位于COX1和tRNA-Val基因之間,長度為42 bp。
2.6 系統發育分析
基于線粒體基因組13個蛋白質編碼基因構建的ML和BI系統發育樹拓撲結構一致,故此處選取其中一種作為展示(圖5)。結果顯示柄眼目呈單系,“non-achatinoid”類群與“achatinoid”類群被分開,其中“achatinoid”類群僅由Achatina fulica作為代表。琥珀螺科(Succineidae)也為單系,與旋蝸牛總科(Helicoidea)和尾刺螺總科(Urocoptoidea)聚成的支系互為姐妹群關系。琥珀螺科中赤琥珀螺與同屬的腐敗琥珀螺親緣關系最近。
3 討論與結論
本研究測定了采集自新疆的陸生貝類物種赤琥珀螺的線粒體基因組序列,全長為14 023 bp,該長度處于琥珀螺科已發表物種的線粒體基因組大小范圍內(13 984~14 092? bp,見表1)。呈共價閉合環狀雙鏈結構,包含37個典型編碼基因和1個控制區。其線粒體全基因組展現明顯的AT偏向性,符合柄眼目動物類群相關特征[21]。鏈的非對稱性研究揭示赤琥珀螺在整個正鏈上有鏈的不對稱性逆轉,即T的含量高于A,G的含量高于C(AT skew為-0.16,GC skew為0.07),與腹足綱大部分種類相似[31]。
赤琥珀螺PCGs結構及排列與同科已報道物種一致[6-8]。其中,最長的基因為 NAD5(1 680 bp),最短的為 ATP8(117 bp),亦與腹足綱物種情況一致[32]。密碼子使用情況顯示偏向于使用第3位為A或T(U)的密碼子,與Guzmán等對Omalonyx unguis的研究結果一致[8]。對選擇壓力的研究發現,13個PCGs的Ka/Ks比值均小于1,表明受到純化選擇作用,但不同基因受選擇強度并不相同: ATP8和 NAD2基因受選擇壓力較小,因而有利于它們進化速率加快;相比較而言,COX1基因受選擇壓力較大,因而相對于其他基因可能更為保守,這與通常認為的COX1可作為DNA條形碼進行物種鑒定的觀點相一致[33]。
赤琥珀螺rRNAs的基因位置及長度與琥珀螺科已報道物種相似,但tRNAs基因的二級結構則有所不同。這與通常認為的線粒體基因組中基因排列順序和結構是可變的[34],且tRNA的位置變動要比rRNA以及蛋白質編碼基因更為常見的觀點相一致[35]。本研究中,赤琥珀螺的tRNA-Ser1基因在原本DHU臂處出現了1個8 bp的小莖環結構,而同科已報道的3個物種均缺乏這一結構。除此之外,赤琥珀螺tRNA-Phe和tRNA-His基因的二級結構缺失TΨC臂,亦與琥珀螺科已報道種不同[6-8]。盡管如此,由于陸生貝類許多重要類群的線粒體基因組當前未知,對于它們基因重排的機制、進化模式和變異程度至今尚不清楚。因此,對當前觀察到的基因結構差異在系統發育和進化研究中的意義有待更廣泛的? 探究。
腹足綱物種的系統發育分析一直備受關注,本研究中對高階層柄眼目的系統分類顯示其呈現單系,與Wade等的研究結果相一致[36]。柄眼目中對于琥珀螺科的分類問題亦備受重視,本研究對該科的研究結果與Guzmán等[8]的結果相一致,揭示琥珀螺科是Helicoidea + Urocoptoidea的姐妹群。對于赤琥珀螺的分類,本研究揭示其與同屬的腐敗琥珀螺關系最近,而與同在新疆分布的尖緣螺屬物種五家渠尖緣螺關系較遠,這與形態學研究進行的物種劃分結果一致。未來,期待有更多種類的線粒體基因組被測定研究,以便進行更為全面、深入的系統發育探討。
參考文獻 Reference:
[1] 薛 清,杜虹銳,薛會英,等.苜蓿滑刃線蟲線粒體基因組及其系統發育研究[J].生物技術通報,2021,37(7):98-106.
XUE Q,DU H R,XUE H Y,et al.Mitochondrial genome and phylogeny of Aphelenchoides medicagus[J].Biotechnology Bulletin,2021,37(7):98-106.
[2] 楊 婧,黃 原.線粒體基因組的高通量測序策略[J].生命科學,2016,28(1):112-117.
YANG J,HUANG Y.Strategy of high-throughput sequencing technology in the mitochondrial genome sequencing[J].Chinese Bulletin of Life Sciences,2016,28(1):112-117.
[3] 周 彤,周 娟,梁 爽,等.福建黃兔線粒體基因組全序列測定與分析[J].西北農業學報,2020,30(9):1295-1303.
ZHOU T,ZHOU J,LIANG SH,et al.Complete mitochondrial genome sequence and analysis of Fujian yellow rabbit[J].Acta Agriculturae Boreali-occidentalis Sinica,2020,30(9):1295-1303.
[4] PATTERSONC? M.Taxonomic studies of the land snail family Succineidae[J].Malacological Review,1971,4:131-202.
[5] 郭陽陽.新疆天山不同采集點琥珀螺屬形態和分子性狀的比較研究[D].烏魯木齊:新疆大學,2017.
GUO Y Y.Comparative research on morphological and molecular characteristics of? Succinea (Gastropoda,Succineidae) from different locations in Tianshan Mountains,Xinjiang[D].Urumqi:Xinjiang University,2017.
[6] WHITE T R,CONRAD M M,TSENG R,et al.Ten new complete mitochondrial genomes of pulmonates (Mollusca,Gastropoda) and their impact on phylogenetic relationships[J].BMC Evolutionary Biology,2011,11(1):1-15.
[7] 阿爾祖古麗·買買提吐爾遜,張子俊,張衛紅.五家渠尖緣螺線粒體基因組測序分析[J].華北農學報,2021,36(3):230-238.
AERZUGULI·M,ZHANG Z J,ZHANG W H.Complete mitochondrial genome sequence of Oxyloma wujiaquensis (Gastropoda,Succineidae) [J].Acta Agriculturae Boreali-Sinica,2021,36(3):230-238.
[8] GUZMN L B,VOGLER R E,BELTRAMINO A A.The mitochondrial genome of the semi-slug Omalonyx unguis (Gastropoda,Succineidae) and the phylogenetic relationships within Stylommatophora[J].PloS One,2021,16(6):e0253724.
[9] 錢周興,周衛川.中國常見陸生貝類圖鑒[M].杭州:浙江人民美術出版社,2014:36.
QIAN ZH X,ZHOU W CH.Illustrated Handbook of Common Terrestrial Mollusks in China[M].Hangzhou:Zhejiang Peoples? Art Press,2014:36.
[10] 陳德牛,高家祥.中國琥珀螺屬Succinea的研究[J].四川動物,1985(1):34-37.
CHEN D N,GAO J X.Research of Succinea in China[J].Sichuan Journal of Zoology,1985(1):34-37.
[11] 李成有.新疆濕生環境琥珀螺比較形態解剖學研究[D].烏魯木齊:新疆大學,2018.
LI CH Y.Comparative morphology and anatomy on Succineidae in wetland from Xinjiang[D].Urumqi:Xinjiang?? University,2018.
[12] BERNT M,DONATH A,JVHLING F,et al.MITOS:improved de novo metazoan mitochondrial genome annotation[J].Molecular Phylogenetics and Evolution,2013,69(2):313-319.
[13] KEARSE M,MOIR R,WILSON A,et al.Geneious basic:An integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics,2012,28(12):1647-1649.
[14] MARC L,OLIVER D,RALPH B.OrganellarGenomeDRAW (OGDRAW):a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes[J].Current Genetics,2007,52(5/6):267-274.
[15] LIBRAD P,ROZAS J.DnaSP v5:A software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
[16] YANG Z.Computational Molecular Evolution[M].New York:Oxford University Press,2006.
[17] ZHANG D F,GAO I,JAKOVLIC, et al.PhyloSuite:An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources,2020,20(1):348-355.
[18] HUELSENBECK J P,RONQUIST F.MRBAYES:Bayesian inference of phylogenetic trees[J]. Bioinformatics Applications Note,2001,17(8):754-755.
[19] LETUNIC I,BORK P.Interactive tree of life (iTOL) v3:An online tool for the display and annotation of phylogenetic and other trees[J].Nucleic Acids Research,2016,44(1):242-245.
[20] YANG X,XIE G L,WU X P,et al. The complete mitochondrial genome of Chinese land snail Aegista aubryana (Gastropoda:Pulmonata:Bradybaenidae)[J].Mitochondrial DNA Part A,2016,27(5):3538-3539.
[21] HUANG CH W,LIN S M,WU W L.Mitochondrial genome sequences of landsnails Aegista diversifamilia and Dolicheulota formosensis (Gastropoda,Pulmonata,Stylommatophora) [J].Mitochondrial DNA Part A,2016,27(4):2793-2795.
[22] KORBEK O,PETRUSEK A,ROVATSOS M.The complete mitogenome of Helix pomatia and the basal phylogeny of Helicinae (Gastropoda,Stylommatophora,Helicidae)[J].ZooKeys,2019,827:19-30.
[23] GAITN-ESPITIA J D,NESPOLO R F,OPAZO J C.The complete mitochondrial genome of the land snail Cornu aspersum (Helicidae:Mollusca):intraspecific divergence of protein-coding genes and phylogenetic considerations within Euthyneura[J].PloS One, 2013,8(6):e67299.
[24] WANG P,YANG H F,ZHOU W CH,et al.The mitochondrial genome of the land snail Camaena cicatricose(Muller,1774)(Stylommatophora,Cerionidae):the first complete sequence in the family Camaenidae[J]. ZooKeys,2014,451:33-48.
[25] HATZOGLOU E,RODAKIS G C,LECANIDOU R.Complete sequence and gene organization of the mitochondrial genome of the land snail Albinaria coerulea[J].Genetics,1995,140(4):1353-1366.
[26] HE ZH P,DAI X B,ZHANG SH,et al.Complete mitochondrial genome of the giant African snail,Achatina fulica (Mollusca,Achatinidae):a novel location of putative control regions (CR) in the mitogenome within Pulmonate species[J]. Mitochondrial DNA Part A,2016,27(2):1084-1085.
[27] GONZLEZ V L,KAYAL E,HALLORAN M,et al.The complete mitochondrial genome of the land snail Cerion incanum (Gastropoda,Stylommatophora) and the phylogenetic relationships of Cerionidae within Panpulmonata[J].Journal of Molluscan Studies,2016,82(4):525-533.
[28] MINTON R L,MARTINEZ C M,FARMAN M L,et al.Two complete mitochondrial genomes from Praticolella mexicana Perez,2011 (Polygyridae) and gene order evolution in Helicoidea (Mollusca,Gastropoda)[J]. ZooKeys,2016,626:137-154.
[29] LIN J H,ZHOU W C,DING H L,et al.The mitochondrial genome of the land snail Cernuella virgata (Da Costa,1778):the first complete sequence in the family Hygromiidae (Pulmonata,Stylommatophora)[J]. ZooKeys,2016,589:55-69.
[30] BANDYOPADHYAY P K,STEVENSON B J,CADY M T,et al.Complete mitochondrial DNA sequence of a Conoidean gastropod,Lophiotoma (Xenuroturris) cerithiformis:Gene order and gastropod phylogeny[J]. Toxicon,2006,48(1):29-43.
[31] SUN S? H,LI Q,KONG L F,et al.Multiple reversals of strand asymmetry in molluscs mitochondrial genomes,and consequences for phylogenetic inferences[J].Molecular Phylogenetics and Evolution,2018,118:222-231.
[32] PETRUSEK A,ROVATSOS M.The complete mitogenome of Helix pomatia and the basal phylogeny of Helicinae (Gastropoda,Stylommatophora,Helicidae) [J].ZooKeys,2019,827:19-30.
[33] 李曉葉,錢 路,陳茂華.DNA條形碼技術在幾種蘋果園鱗翅目害蟲鑒定中的應用[J].西北農業學報,2015,24(5):141-147.
LI X Y,QIAN L,CHEN M H.Application of DNA barcoding technology to identification of some Lepidoptera pests in apple orchards[J].Acta Agriculturae Boreali-occidentalis Sinica,2015,24(5):141-147.
[34] MINDELL D P,SORENSON M D,DIMCHEFF D E.Multiple independent origins of mitochondrial gene order in birds[J].Proceedings of the National Academy of Sciences,1998,95(18):10693-10697.
[35] XU X F,LI A ZH,CHENG SH M,et al.Reappraisal of phylogenetic status and genetic diversity analysis of Asian population of Lentinula edodes[J].Progress in Natural Science,2006,16(3):274-280.
[36] WADE C M,MORDAN P B,NAGGS F.Evolutionary relationships among the Pulmonate land snails and slugs (Pulmonata,Stylommatophora) [J].Biological Journal of the Linnean Society,2006,87(4):593-610.
Complete Mitochondrial Genome Sequencing and Phylogeny
Analysis of? Succinea erythrophana
Abstract In this study,we obtained the complete mitochondrial genome sequence of Succinea erythrophana by high-throughput sequencing,and analyzed its structural and compositional features,and performed phylogenetic analyses using maximum likelihood (ML) and Bayesian inference (BI) methods (mtDNA sequences of three species of Succineidae and some species of Stylommatophora used in this paper downloading from NCBI).The results showed that:①The complete mitochondrial genome of Succinea erythrophana was 14 023 bp in length (GenBank? No.ON533899),consisting of 37 genes and a segment of AT-rich non-coding control region,with 20 gene spacings and 13 gene overlaps.The average A+T was 77.3%,showing a significant AT bias.The order of gene arrangement,structure and composition,and codon usage were similar to those of the reported species in the Succineidae.? ②The secondary structure of most tRNAs in Succinea erythrophana showed a typical cloverleaf structure except for tRNA-Phe,tRNA-His,tRNA-Ser1,and tRNA-Ser2.③Ka/Ks selection pressure analysis of Succinea erythrophana showed that it was subject to purifying selection.④Phylogenetic studies showed the closest relatives of Succinea erythrophana and Succinea putris,which then formed sister groups with other species of the same family.
Key words Succinea erythrophana;Succineidae;Mitochondrial genome;Phylogeny

