具有局部和全局注意力機制的圖注意力網絡學習單樣本組學數據表征
周豐豐,張金楷
(1.吉林大學 計算機科學與技術學院,長春 130012;2.吉林大學 符號計算與知識工程教育部重點實驗室,長春 130012)
近年來,現代高通量生物醫學技術得到快速創新和發展,生物數據積累加速[1].這些數據極大促進了許多生物學過程的潛在機制研究,包括衰老過程和復雜的疾病發病機制[2].但大多數生物組學數據集具有高噪聲、多維度和多維異質性的特點.此外,生物組學的許多特征與表型無關,特征之間存在冗余.高通量技術生產的生物組學大多存在“大p小n”的維度災難問題,其中p指特征數量,n指樣本數量.因此,組學數據存在高維問題.特征選擇是克服高維組學數據維數災難的有效方法.特征選擇方法在生物信息學領域被廣泛應用于生物標記物識別和數據降維.而現有的應用于組學數據的特征選擇算法,基本都是使用傳統的分類學習算法對數據進行分類,即在組學數據降維的研究中,很少考慮深度特征選擇算法,導致組學數據分類精度較低.
目前基于深度學習的方法有許多嘗試采用基于圖的混合策略,在分析前將每個組學建模為一個單獨的圖,利用圖嵌入方法從每個網絡中學習節點及其周圍環境的低維表示.然后將新的基于圖的特征組合并輸入其他機器學習模型進行預測、分類等.在組學數據上構建網絡的常用方法包括蛋白質相互作用網絡、基于相關性的網絡、比值網絡等.Jonsson等[3]通過分析蛋白質相互作用網絡,發現與癌變相關的蛋白質特征常具有更密切的相關性,表明功能相似的特征或特征集通常以模塊化的形式反應有機體功能的表型.Wang等[4]通過分析蛋白質相互作用網絡揭示了肝臟特異性蛋白、肝臟疾病蛋白和重要信號通路分子之間的相互作用特征.Liu等[5]利用Pearson相關系數計算特征之間的相關性,建立相應的生物網絡,利用有特定樣本和無特定樣本時特征之間的相關性變化構建單個樣本網絡,為疾病的個性化治療提供了幫助.Netzer等[6]使用配對生物標記標識符(paired biomarker identifiers,PBI)作為指標,測量不同群體特征比的變化,并構建了相應的生物網絡.將單個特征作為網絡節點,通過特征之間比值關系的變化構建網絡的方法首先應用于代謝組學數據,之后推廣到基因組學數據[7].因此,選擇合適的網絡并找到高效的分類學習算法尤為重要.
本文在從組學數據的單樣本網絡中學習有用信息的基礎上,提出一種具有局部和全局注意力機制的圖注意力網絡(GATOr).首先,從每個樣本的組學數據中構建一個圖,以一個組學特征作為一個節點,兩兩特征之間的相關性作為邊的權值;由于構建圖的時間復雜度為平方,因此對無關特征進行預篩選,以減少單樣本圖中組學特征的數量.其次,提取圖注意力網絡中集成的局部和全局注意模塊的有用信息作為工程特征,并從該單樣本網絡中學習到的特征進行類預測任務.實驗結果表明,與現有的組學分類方法相比,GATOr具有更好的分類性能.
1 算法設計
1.1 單樣本網絡
單樣本網絡是一種基于參考數據集的利用單樣本數據構建的生物分子網絡,它是一種將復雜網絡的理論和方法應用于疾病研究和藥物開發的方法,可從系統的角度識別個體疾病所涉及的相互作用或功能失調[8].Liu等[5]提出了基于Pearson相關性的單樣本網絡,在疾病表征基因調控網絡的背景下獲得個體特異性或樣本特異性網絡.對于節點網絡,其構建需要多個樣本,但在臨床實踐中通常無法獲得.在單樣本[9]的基礎上對節點網絡進行表征或推斷是必要的.這種方法的優點是網絡只依賴于從每個模型中學習基于圖的變量,這些變量可用于其他機器學習模型的輸入,用于聚類、子類型發現或生存預測.
1.2 圖注意力網絡
圖神經網絡通過聚合網絡中多層鄰居節點對當前節點的影響,更新節點的嵌入式表示,然后用更新的嵌入式表示完成后續任務,如節點分類和鏈接預測等[10].Bruna等[11]提出了一種基于譜域的圖卷積神經網絡(GCN),譜域的卷積需要在Laplace矩陣上進行特征分解,每次都需進行節點的聚合,非常耗費算力.Defferrard等[12]對卷積核進行近似操作,提出了Chebyshev網絡,該網絡避免了Laplace矩陣的特征分解,降低了運算的復雜度.Kipf等[13]對其進行了進一步優化,提出了最初的圖卷積網絡模型,在譜域上的圖卷積網絡可以發揮其最大的效能.圖注意力網絡(GAT)先在各節點間采用消息傳遞的方式聚合鄰居節點[14],然后更新自身節點的信息,通過學習注意力權值,放大更重要的節點和邊的權重,使用注意力機制定義聚合函數,從而計算并更新節點的特征信息,得到節點局部結構新的特征.
GAT網絡由堆疊簡單的圖注意力層(graph attention layer)實現,每個注意力層對節點對(i,j),注意力系數計算方式為
(1)
其中aij為節點j到i的注意力系數,Ni表示節點i的鄰居節點.節點輸入特征為h={h1,h2,…,hN},hi∈F,節點特征的輸出為F,其中N,F分別表示節點個數和輸入特征維數;W∈F′×F表示在每個節點上應用的線性變換權重矩陣;a∈2F′為權重向量,可以將輸入映射到.最終使用Softmax進行歸一化并加入LeakyReLU以提供非線性.最終節點的特征輸出可表示為

(2)
其中σ表示非線性激活函數,如Sigmoid和ReLU.
1.3 GATOr網絡

圖1 基于局部和全局注意力機制學習組學特征表示的圖注意力網絡Fig.1 Graph attention network based on local andglobal attention mechanism to learn omicsfeature representation
本文提出的基于局部和全局注意力機制學習組學特征表示的圖注意力網絡(GATOr)整體結構如圖1所示.由圖1可見,其主要包含兩部分: 1) 單樣本網絡,將每個組學數據樣本建模為一個單樣本網絡,將特征作為節點,每對特征之間的相關性作為邊;2) 具有局部和全局注意力機制的圖注意力網絡,用于從單樣本網絡中學習表征特征向量進行分類任務.GATOr網絡的優化目標是評估中心節點附近某個鄰居節點的重要性,從而為其鄰居節點分配不同權重.中心節點的局部注意力只關注其一階鄰居,而全局注意力則關注圖中所有節點,局部與全局注意力機制的融合優化了特征提取能力,使下游的分類性能得到提高.
GATOr網絡引入了注意力機制,用于解決GCN對鄰居節點一視同仁的局限性,通過分配不同的權重給不同的鄰居,賦予模型更強的特征表示能力,將原始圖數據轉換到低維空間并保留關鍵信息,生成保留原始圖中某些重要信息的低維向量,同時也提高了節點分類等下游任務的分類性能.
1.3.1 構建單樣本網絡
本文將組學數據的樣本作為單樣本網絡訓練基于圖的GATOr模型.考慮到現實情況,僅利用一個樣本數據檢測復雜疾病惡性突變的臨界狀態和預警信號至關重要.雖然表達數據或測序數據在單個樣本的基礎上提供了關于分子譜的信息,但由于數據集每個病人只有一個樣本數據,無法利用傳統方法計算出基因的相似性網絡,因此需給出足量的參考樣本表征正常時期基因之間的相關性,通過對比單個樣本與參考樣本之間的差異反應單樣本特征[5,15].
首先基于基因共表達網絡構建出參考網絡,通常用無向圖表示,網絡中的節點表示特征,邊表示特征之間的相關性.給定n個參考樣本,參考樣本數據中任意一對特征x和y之間的相關性可使用Pearson相關系數(PCC)計算,用公式表示為

(3)

Yu等[16]檢索了查詢樣本中每個組學特征相對于參考樣本子集的方差,并計算了查詢樣本中兩個組學特征方差向量之間的PCC.在查詢樣本中,這兩個組學特征之間的PCC值被定義為基于參考的變異PCC(rvPCC).rvPCC取值范圍為-1~1,當rvPCC接近-1或1時,將兩個查詢特征定義為正相關或負相關[17].
組學數據集通常具有數千個甚至更多的特征,使得構建單樣本網絡的平方時間復雜性變得不切實際.本文使用t檢驗衡量每個特征與類標簽的關聯,并選擇排名靠前的k個特征(本文中k=800)[18]進行進一步分析.采用PCC測量特征間的冗余度.
至此已構建出一個完整的單樣本網絡,該網絡為一個加權無向圖,可用于各種基于圖的深度神經網絡,為在網絡層面表征個性化特征并分析生物系統開辟了新途徑.
1.3.2 局部與全局注意力機制
GAT使用特征向量a學習節點及其鄰居的相對重要性,可能無法捕獲分類任務的有用信息.假設與節點本身相似的鄰居節點可能更重要,則可通過直接計算兩個相連節點之間的相似度得到節點的相對重要性[19].節點的局部注意力只關注其鄰居,而節點的全局注意力從圖中所有節點中提取信息.基于雙重注意力機制的網絡,通過對低層詳細信息和高層語義信息的注意獲取高質量、獨特并可鑒別的特征[20].
局部注意力系數計算公式為
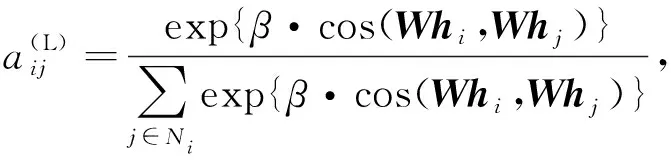
(4)
式中β表示標準偏差,cos(·)用于計算余弦相似度.為聚合來自節點鄰域的信息,式(2)可表示為
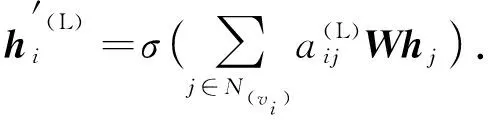
(5)
局部注意力模塊與圖注意力模塊的區別: 本文顯式地使用cos(·)計算節點之間的相似度作為相對重要性權重,而傳統方法使用可學習參數a學習節點之間的相對重要性.局部注意力是在圖上一個節點的鄰居上計算的,而本文在所有實體的集合上構造局部注意力.
本文還實現了全局注意力機制,其中節點可有選擇地聚合圖中任何其他節點的信息.擴展圖注意力層以進行全局操作.Mostafa等[21]提出了一種基于歐氏距離的注意力系數.全局注意力系數可表示為
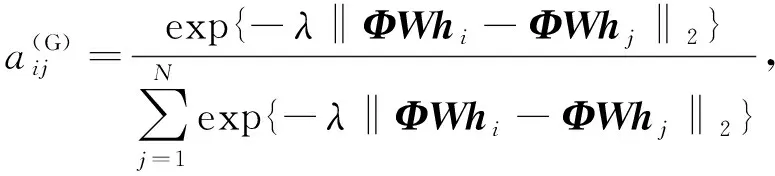
(6)
其中:Φ∈D×F′為嵌入矩陣,它將節點特征轉換到d維節點相似度空間;λ表示標準差的逆,‖·‖2表示2范數.
節點i的全局加權注意力為
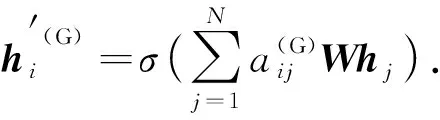
(7)


(8)
其中‖為串聯運算符.式(8)也可視為將不同注意力頭的輸出相連接.使用A個注意力頭,輸出特征向量的維數為2AF′,最終的特征向量也可表示為

(9)
其中G表示單樣本網絡的無向圖,注意力頭數A=2,g表示進行注意力操作的過程.
2 實 驗
2.1 數據集
使用4個數據集評估GATOr特征工程算法,這4個數據集均選自文獻[22]中整理的組學數據集: 數據集ROSMAP提供了阿爾茨海默病(AD)患者與正常對照組(NC)的組學數據;數據集LGG用于低級別膠質瘤(LGG)的分級分類;數據集KIPAN用于腎癌類型分類;數據集BRCA用于乳腺癌PAM50亞型的分型任務.每個數據集的預處理包括排除缺失值的特征以及隨機選擇參考樣本.
各數據集信息列于表1,其中第四列給出了每個數據集中兩個或多個類的詳細信息,最后一列給出了3種類型組學數據的特征數量,即mRNA表達(mRNA)、DNA甲基化(Methy)和miRNA表達(miRNA).數據缺失的特征被排除在進一步分析外.由于本文不討論多組學整合分析,因此3種類型組學數據混在一起進行計算.

表1 各數據集信息
2.2 評價指標
在進行構建單樣本網絡等時間復雜度較高的任務前,先通過特征預篩選降低特征維度.由表1可見,4個數據集的特征數量都遠大于樣本數量.考慮到構建單樣本網絡的平方時間復雜度,因此僅對有限數量的原始組學(OMIC)特征設計GATOr特征.
通過分層策略將每個數據集隨機分為80%的訓練數據集和20%的測試數據集,即保持訓練數據集和測試數據集的類分布.二分類任務的評價指標為分類精度(ACC)和ROC曲線下面積(AUC).對于多分類任務,只計算ACC.
2.3 實驗結果及分析
2.3.1 對比實驗
本文將GATOr的分類性能與以下7種組學數據基線方法進行比較.
1)k近鄰分類器(KNN): 基于查詢樣本的k個近鄰的類別實現投票策略.
2) 支持向量機分類器(SVM): 一種流行的基于最大間隔分割平面的分類器.
3)L1正則化訓練的線性回歸(Lasso): Lasso回歸是線性回歸模型的一種收縮和變量選擇方法,用于獲取定量響應變量的預測誤差最小的預測變量子集.
4) 隨機森林分類器(RF): 融合多棵隨機樹的決策.
5) 樸素Bayes分類器(NB): 基于Bayes定義和特征條件獨立假設的分類器方法.
6) 極限梯度提升算法(XGBoost): 提供了一種可擴展的快速梯度提升分類系統.
7) 全連接神經網絡分類器(NN): 使用具有交叉熵損失的全連接神經網絡作為基線神經網絡分類器.
GATOr算法與7種基線方法在4個數據集分類任務上的性能評估列于表2.由表2可見,GATOr框架在4個數據集上的ACC和AUC指標均優于其他基線分類器.與傳統的組學分類方法相比,GATOr還獲得了相對較小的標準差,具有更好的分類性能.

表2 GATOr算法與7種基線方法在4個數據集分類任務上的性能評估
2.3.2 消融實驗
首先,實驗評估了由單樣本網絡(SSN)學習到的嵌入特征的貢獻度.將沒有SSN模塊的GATOr過程表示為GATOr-SSN,即直接將預處理后的特征加載到下一個模塊中,而不使用SSN模塊.實驗結果列于表3.由表3可見,完整的GATOr過程在4個數據集上的兩個性能指標ACC和AUC都優于GATOr-SSN版本.因此,有必要將單樣本網絡引入到OMIC數據的特征工程任務中.
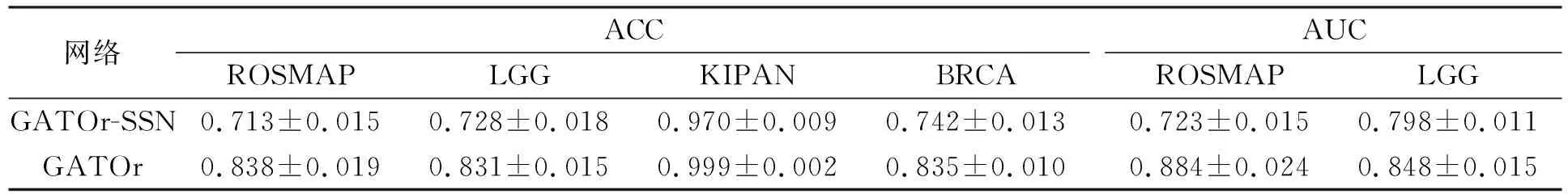
表3 單樣本網絡(SSN)嵌入特征的分類貢獻
其次,通過消融實驗評估GATOr主要模塊的貢獻.基線模型為圖注意力網絡GAT.將沒有局部和全局注意力機制的GATOr網絡分別表示為GATOr-Local和GATOr-Global.將這3種圖網絡與完整的GATOr網絡根據其提取的特征進行分類性能比較.
GATOr圖注意力網絡主要模塊的分類貢獻列于表4.由表4可見,移除任何一個模塊都會降低分類ACC和AUC值.去掉局部注意力機制導致的性能下降最大,表明在GAT網絡中僅包含全局注意力可能會使提取的特征分類性能惡化.而全局注意力機制和局部注意力機制的引入對基線GAT網絡具有積極貢獻,即使是基線GAT網絡也比表3中GATOr-SSN過程提取了有用的信息,以獲得更好的分類性能.

表4 GATOr圖注意力網絡主要模塊的分類貢獻
綜上所述,本文提出了一種結合局部和全局注意力機制的圖注意力網絡,用于從組學數據的單樣本網絡中學習有用信息.本文對組學數據所有的樣本構建其對應的單樣本網絡,通過具有局部和全局注意機制的圖注意力網絡從單樣本網絡中學習基于圖的組學特征表示進行類預測任務.實驗結果表明,即使是基線圖注意力網絡在分類任務上的性能也優于原始的組學特征,并且局部注意力和全局注意力的融合可以進一步提高數據分類性能.

