基于微衛星標記的克氏原螯蝦種群遺傳多樣性和遺傳結構分析
高楊 田燦 姜京京 唐永凱 張成鋒 馮文榮 李冰 陳銘 盧澤宇 蘇勝彥 朱健
摘要:為了解當前我國不同地區克氏原螯蝦種群的遺傳背景情況,以期為人工養殖、新品種選育和資源保護提供參考依據。選取江蘇洪澤湖(HZH)、湖北洪湖(HH)、湖南洞庭湖(DTH)、山東微山湖(WSH)、安徽巢湖(CH)、江西鄱陽湖(PYH)6個典型區域的克氏原螯蝦群體作為研究對象,采用14對微衛星引物對來自6個地區的168份樣品進行遺傳多樣性檢測。結果可知,各群體不同標記位點的等位基因數在3~14之間,平均期望雜合度為0.81,平均多態信息含量分布在0.42~0.59之間,各群體均處于中高度遺傳多樣性水平。6個群體均發生一定程度Hardy-Weinberg平衡偏離,僅有少數位點在單一群體滿足Hardy-Weinberg平衡,同時連鎖不平衡檢測發現9個連鎖座位對處于不平衡狀態。遺傳距離結果顯示,6個克氏原螯蝦群體的遺傳一致度(I)處于0.127 8~0.643 9之間,各群體間遺傳距離(D)處于0.440 2~2.057 3之間。群體間遺傳分化指數(Fst)接近0.5,反映出群體間的基因交流水平較弱,存在高度的遺傳分化。AMOVA分析發現,39.65%的遺傳變異來源于群體間,97.99%的遺傳變異來源于個體間。遺傳結構數據顯示,K=5時,6個群體間存在顯著的結構差異。上敘結果可知,6個地區的克氏原螯蝦群體具有豐富的遺傳多樣性,群體間出現了顯著分化,可通過隔離保種、雜交、選擇育種等方式保護利用克氏原螯蝦的種質資源,為我國克氏原鰲蝦產業的持續健康發展提供良種支持。
關鍵詞:克氏原螯蝦;遺傳多樣性:微衛星標記;遺傳結構
中圖分類號:S917;S966.12文獻標志碼:A
文章編號:1002-1302(2023)05-0191-09
克氏原螯蝦(Procambarus clarkii)別稱淡水小龍蝦,原產自北美洲,20世紀前中葉引入我國。該蝦具有肉質鮮美、生長速度快、環境適應力強的特點,進而深受漁民和消費者的喜愛[1-2]。21世紀以來,我國克氏原螯蝦養殖技術日益成熟完善,養殖方式愈發多元,養殖產量也日益增長。據中國漁業統計年鑒統計,我國克氏原螯蝦2018年產量達163.87萬t,2019年達208.96萬t,首次突破200萬t,2020年雖受疫情影響,產量仍達239.37萬t[3],創下歷史新高。當前,克氏原螯蝦已成為我國第六大淡水經濟養殖品種。然而在逆向選擇、捕大留小的生產模式下,養殖后代病害頻發、規格參差不齊,嚴重制約克氏原鰲蝦產業的持續健康發展,亟需通過良種選育來提高種質性能,促進產業持續健康發展。大量的水產養殖經驗告訴我們,優秀的種質來源決定了水產生物生產養殖的命脈,而在新品種選育過程中,對國內已存在的主要遺傳資源進行遺傳評估成為首先需要解決的問題。
遺傳多樣性常用來評估用于選育種質的選擇空間,因而受到國內外學者的重點關注。對于克氏原螯蝦遺傳多樣性的研究,當前主要集中在分子標記及區域群體的選擇上。關于分子標記,常用的檢測方法有Random Amplified Polymorphic DNA(RAPD)[4-5]、Amplified fragment length polymorphism (AFLP)[6]和Simple Sequence Repeats(SSR)[7-8]。其中,SSR以高的多態性和共顯性[9],成為研究克氏原螯蝦遺傳多樣性和遺傳關系的首選技術。關于區域群體的選擇則較為廣泛,遍及全國克氏原螯蝦養殖生產省份。邢智珺等采用8個微衛星標記分析了江蘇省境內8個地區克氏原螯蝦的遺傳多樣性[10];黃小芳等利用8對微衛星引物分析了廣西境內5個地區克氏原螯蝦遺傳多樣性[11]。研究結果均表明,選點區域內克氏原螯蝦群體遺傳多樣性豐富。以上研究選點分布集中于單一或少數的省份,尚未就全國范圍內典型水域的野生克氏原螯蝦群體開展研究。為此本研究進行了江蘇洪澤湖(HZH)、湖北洪湖(HH)、湖南洞庭湖(DTH)、山東微山湖(WSH)、安徽巢湖(CH)、江西鄱陽湖(PYH)6個典型水域克氏原螯蝦野生種質資源遺傳多樣性水平的差異研究,以期為克氏原螯蝦養殖提供重要的親本來源,為新品種的選育和野生種的資源保護工作提供參考。
1 材料與方法
1.1 試驗動物
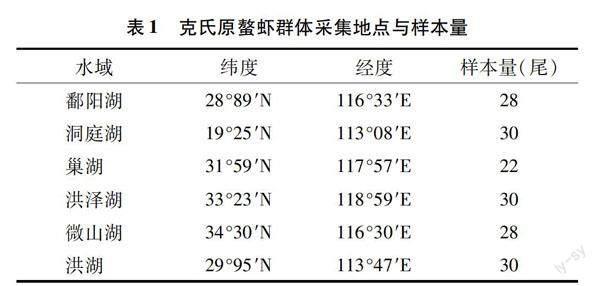
分別從江蘇洪澤湖(HZH)、湖北洪湖(HH)、湖南洞庭湖(DTH)、山東微山湖(WSH)、安徽巢湖(CH)、江西鄱陽湖(PYH)六大淡水湖泊中捕撈克氏原螯蝦群體,各群體取22~30尾個體。由表1可知,雌雄加以區分,剪去尾節,置于預先備好裝有乙醇的2 mL離心管中,-20 ℃保存。
1.2 基因組DNA提取
總基因組DNA提取采用苯酚/三氯甲烷法[12]提取尾節組織的總基因組DNA,DNA通過瓊脂糖凝膠電泳檢驗純度,[HJ1.5mm]紫外分光光度計測定D260 nm和D280 nm,合格的DNA稀釋濃度至0.05 mg/L,保存于 -20 ℃ 加以備用。
1.3 微衛星引物擴增
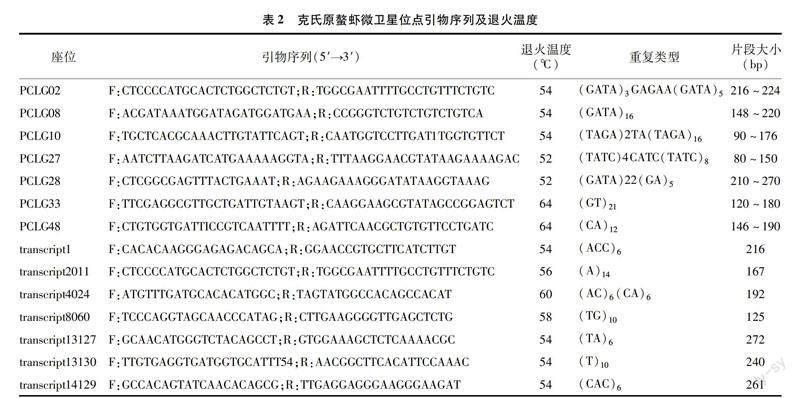
參考GenBank已發表的微衛星序列[13]及本實驗室自行開發的微衛星序列共14對(表2)。正向引物的5′端FAM標記,委托無錫天霖生物有限公司加以合成。PCR反應總體系:DNA模板 2.0 μL,Taq酶12.5 μL,上下游引物各1.0 μL,最后加滅菌的超純水至25.0 μL。
PCR反應程序為:94 ℃預變性5 min;94 ℃變性10 s,在最適退火溫度下退火10 s,72 ℃延伸 15 s,30個循環;最后72 ℃再延伸10 min,4 ℃保存。PCR產物通過ABI3730XL測序儀(美國應用生物系統公司)進行毛細管電泳檢測,通過GeneMarker軟件進行基因型分型。
1.4 數據分析
利用14對SSR標記鑒定到的6個克氏原螯蝦群體的基因型,使用Popgene32(v1.3.2)軟件[14]對6個克氏原螯蝦群體的等位基因數(Na)、有效等位基因數(Ne)、觀測雜合度(Ho)、期望雜合度(He)、似然比檢驗Hardy-Weinberg平衡、遺傳分化指數Fst(F-statistics,Fst)、Neis標準遺傳距離(Ds)和中性進行分析檢測。使用Genepop的列聯表方法進行連鎖不平衡分析[15]。使用Botstein等的方法計算每個位點的多態信息含量(PIC)[16]。遺傳結構分析采用Structue 2.3軟件[17],遺傳結構圖繪制采用CLUMPP1.1.2[18]和distruct軟件[19]。
2 結果與分析
2.1 克氏原螯蝦遺傳多樣性分析
通過毛細管電泳檢測14對微衛星引物在168份樣品中的基因型及其頻率分布,14個微衛星位點在6個克氏原螯蝦群體內共檢測到等位基因124個。由表3可知,這些標記在單個的克氏原螯蝦群體中的等位基因數在3~14之間,等位基因數最多的是PCLG08位點。各群體內的等位基因數在1~8之間不等,平均等位基因數最低的是巢湖群體,僅為1.93;最高的是洪澤湖群體,數值達4.14;其余群體的平均等位基因數均處于2~3之間不等。各群體的有效等位基因數在1.00~5.16之間,數目最多的同樣是PCLG08位點。從等位基因數和有效等位基因數的結果來看,洪澤湖群體分別有11個和9個位點的數值最高,數值最高的位點數量遠超其他群體。而在觀察雜合度和期望雜合度2項指標上,洪澤湖群體同樣多數位點指標數值顯著高于其他群體。14對微衛星的中性測試分析見表4,可知各個位點都處于95%置信區間范圍內,屬中性位點。
由表5可知,根據香農指數分析,洪澤湖克氏原螯蝦群體的遺傳多樣性最為豐富, 其余5組克氏原螯蝦群體的遺傳多樣性相差較小,群體間變異程度低。此外,各群體的多態信息含量介于0.26~0.73之間。當PIC值>0.5時,定義為高多態性;當PIC值處于0.25~0.50之間,定義為中度多態性。本調查分析中,洪澤湖群體在14個位點中均處于高度多態水平。巢湖群體有3個中度多態位點,11個高度多態;洞庭湖群體有6個中度多態位點,8個高度多態位點;洪湖群體有3個中度多態位點,11個高度多態位點;鄱陽湖群體有5個中度多態位點,9個高度多態位點;微山湖群體有2個中度多態位點,12個高度多態位點。綜合可知,洪澤湖群體多態性水平最高,遺傳多樣性也最為豐富,最適合作為養殖生產中的親本來源。
2.2 6個群體Hardy-Weinberg平衡分析和連鎖不平衡檢驗
Hardy-Weinberg平衡分析結果,由表6可知,沒有1對位點在6個群體中都符合哈代溫伯格平衡,各個位點在各群體中均存在明顯的哈代溫伯格平衡偏離現象。其中,僅有PCLG10位點在2個群體中存在哈代溫伯格平衡偏離,剩余的標記位點均在多個群體出現Hardy-Weinberg平衡偏離情況。
對6組克氏原螯蝦群體進行連鎖不平衡檢測,共檢測到59個基因座位對,其中,巢湖群體和洪澤湖群體均存在6個連鎖座位對的不平衡狀態,鄱陽湖群體是連鎖座位對處于不平衡狀態最少的群體,僅為3對,與此同時,各個群體處于不平衡狀態時相應的連鎖座位對并不相同。對于整個群體而言,一共檢測到9個連鎖座位對處于不平衡狀態。
2.3 6個群體遺傳距離分析
遺傳距離結果見表7,可知6個克氏原螯蝦群體的遺傳一致度(I)在0.127 8~0.643 9之間,遺傳距離(D)在0.440 2~2.057 3之間。遺傳一致度的數值結果同遺傳距離數值成反比關系,遺傳一致度數值越大,遺傳距離越小,反映出的親緣關系也越接近。6組不同區域的克氏原螯蝦群體中,微山湖和鄱陽湖群體間的遺傳一致度最高,數值達0.643 9,對應的遺傳距離則為最小的0.440 2,說明微山湖和鄱陽湖這2個群體親緣關系較近。親緣關系最遠的是洪湖和鄱陽湖群體,這2個群體的遺傳距離達2.057 3,數值相比其他群體差距明顯。長年遷徙下,群體間的遺傳距離結果偏大,各群體間交流受阻,群體為適應當地生活環境而出現分化現象。
2.4 克氏原螯蝦遺傳結構分析
Fis結果顯示,14個位點均出現負值,數值最小的是PCLG28位點,Fit結果顯示,8個位點出現負值,數值最大的是PCLG10位點,僅為0.3,尚未達0.5水平。Nm反映了群體間的交流程度,Nm越大,基因交流越為頻繁[20],在數值上同Fst值成反比,Fst是反映遺傳分化的指標。PCLG02位點下的群體交流程度較為頻繁,達4.81,同時,群體間的Fst數值大多接近0.5,6個群體僅存在微弱的基因交流,同時群體間已經出現高度分化現象。這可能與克氏原螯蝦自身習性密切相關,大部分時間棲息于自掘的洞穴內,且游泳運動水平不強引起相互間交流較少。AMOVA分析結果見表8,可知39.65%的變異來自于群體間,97.99%的變異來自于個體間(P<0.01),個體間的遺傳變異程度遠大于群體間。
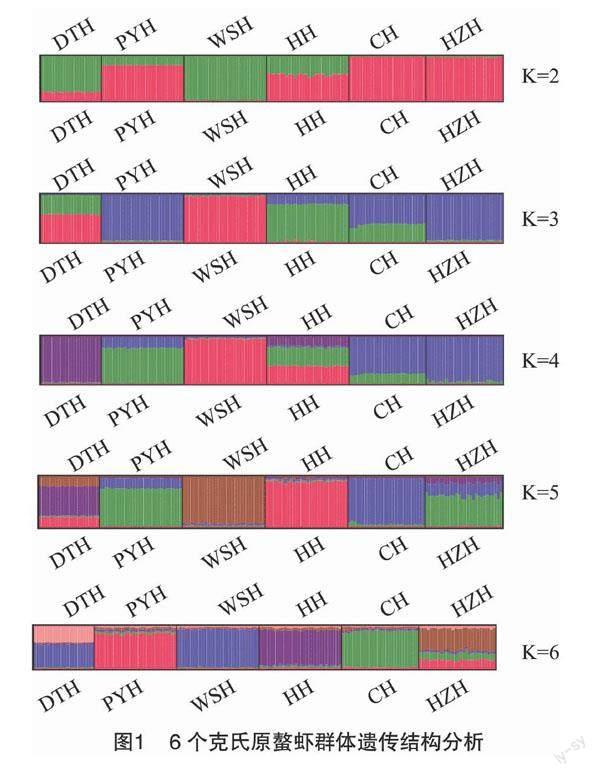
運用Structure軟件對6個克氏原螯蝦群體的遺傳結構進行分析,執行K=2~6的假設,設定10次重復,結果見圖1。K=2時,巢湖群體和鄱陽湖群體遺傳結構沒有差異;K=3時,鄱陽湖群體和洪澤湖群體遺傳結構沒有差異;K=4時,巢湖群體和洪澤湖群體遺傳結構差異不明顯; K=6時, 各群體間遺傳結構差異不如K=5時更為明顯。綜合來看,K=5時,結果最佳。
3 結論
3.1 我國大型淡水湖泊克氏原螯蝦遺傳多樣性豐富
遺傳多樣性作為生物多樣性的重要組成部分,是衡量生物攜帶遺傳信息程度的重要指標,其水平可直接反映物種的遺傳變異程度,而遺傳變異程度恰能反映物種的生長性能、繁育性能等育種指標情況。對克氏原螯蝦的溯源及入侵路線問題,國內的許多水產研究者已經進行過長足的研究,收集了多個水系多重地域的克氏原螯蝦種質群體進行遺傳分析,已經初步得出,克氏原螯蝦從我國江蘇省南京市引入,同時伴隨長江、淮河兩大水系逐漸向外擴展[21]。
相較以往的研究,本研究采樣點符合主要克氏原螯蝦群體養殖分布和遷移路線,涉及湖南、湖北、安徽、江西、江蘇、山東6省,且均來源于我國境內知名的大型淡水湖泊,研究對象更加富有代表性。其次,所選取的14個微衛星位點均為中高度多態性位點,結果更能精確反映這些群體的真實遺傳多樣性水平。運用14對微衛星引物測出6個不同克氏原螯蝦群體的平均期望雜合度0.81,平均多態信息含量0.42~0.59,綜合觀察發現,六大淡水湖泊的克氏原螯蝦具有較高的遺傳多樣性。遺傳多樣性大小依次為洪澤湖>洪湖>微山湖>巢湖>鄱陽湖>洞庭湖。造成這樣的原因可能是克氏原螯蝦從南京市引入,而洪澤湖隸屬淮河水系地理位置上同屬江蘇省,較好地保留了初始時的種質特征。另外很重要的一點是洪澤湖水域環境復雜,水生植物豐富,給個體間的交流提供了良好的環境條件。洪湖地處的湖北省近些年來一直都是我國克氏原螯蝦養殖產量最大的省份,圍繞水域湖泊更是興建了許多人工養殖基地,人為的引進種同野生種之間獲得了基因交流的機會,自身遺傳多樣性相對較高。此外,環境因素與生物遺傳多樣性息息相關,物種迫于環境壓力而引起自身的遺傳變異,同樣也是造成區域物種遺傳多樣性水平變化的重要原因。綜合發現,洪澤湖克氏原螯蝦群體遺傳多樣性最豐富,最適合作為人工選育的親本來源。除此外,還可通過建立適當規模的克氏原螯蝦種質資源保護區,進而有效地留住野生種資源。與之前研究相比,雖說存在采樣點經緯度上微弱的差異,但產生的結果卻是一致的,即我國大型淡水湖泊內克氏原螯蝦野生種群的遺傳多樣性豐富,可進一步為良種選育提供優質的種質資源。一般來說,外來引進物種常受瓶頸效應和遺傳漂變作用影響,生物多樣性會有所降低[22],外來引入的克氏原螯蝦可能經過多次雜交及人為的多次引入,造成自身遺傳多樣性不降反升,這一問題需要引起重點關注。
3.2 我國克氏原螯蝦群體分化現象嚴重
克氏原螯蝦的引入至今已有60~70年,喜攀爬使其很容易從當前水體跨越進入其他水體,進而同其他水體的相同物種進行長期雜交,加上受到不同的空間水文影響、山川河流阻隔,為群體間的分化創造了條件。除此,人為引種也會加劇原本親緣關系相近的物種在不同區域的分布[23]。
本研究采用14個多態位點對6個地域的克氏原螯蝦分析測試發現,各群體均存在顯著的雜合子不足現象。雜合子不足現象多由物種間非隨機交配或近親雜交引起,常見于多個物種間[24]。其中,近親雜交在克氏原螯蝦的群體內極為普遍。首先,幼年蝦體初期會附著于母體腹部尋求庇護,此期間內親緣關系相近的幼蝦會發生近交現象。更重要的是,受市場需求刺激,大規模的捕撈引起野生種群數量銳減,種群世代間基因比例波動大。非隨機交配和近交結果導致了所有檢測位點在所有群體中均偏離哈代溫伯格平衡,僅有少數位點在單一群體滿足哈代溫伯格平衡,這說明群體間已經出現基因型的分化差異。分化的原因可能源于為了適應環境變化的壓力。除此之外,克氏原螯蝦作為一種經濟水產作物,近些年養殖規模逐漸擴大,人工養殖區域和密度日益增加,而大型淡水湖泊恰好提供了適合進行克氏原螯蝦養殖的天然理想化場所,再經長期的地理隔離效應,已形成相對獨立的類群。
參考文獻:
[1]Senol R,Kilic S,Tasdelen K. Pulse timing control for LED plant growth unit and effects on carnation[J]. Computers and Electronics in Agriculture,2016,123:125-134.
[2]倪靜靜. 水溫、pH和飼料對克氏原螯蝦攝食行為及其肉質的影響[D]. 揚州:揚州大學,2016.
[3]農業農村部漁業漁政管理局. 2020年中國漁業統計年鑒[M]. 北京:中國農業出版社,2020.
[4]Barbaresi S,Gherardi F,Mengoni A,et al. Genetics and invasion biology in fresh waters:a pilot study of Procambarus clarkii in Europe[M]. Springer Netherlands,2007:381-400.
[5]Macaranas J,Mather P,Hoeben P,et al. Assessment of genetic variation in wild populations of the redclaw crayfish (Cherax quadricarinatus,von Martens 1868) by means of allozyme and RAPD-PCR markers[J]. Marine and Freshwater Research,1995,46(8):1217-1228.
[6]黃 羽,戴銀根,畢成武,等. 長江中下游地區6個克氏原螯蝦群體遺傳多樣性分析[J]. 南昌大學學報(工科版),2011,33(3):243-247.
[7]王長忠. 長江中下游地區克氏原螯蝦群體遺傳多樣性分析[D]. 武漢:華中農業大學,2009.
[8]邢智珺. 江蘇克氏原螯蝦典型群體遺傳多樣性微衛星分析[D]. 上海:上海海洋大學,2014.
[9]秦海峰,龍 寧,吳建國,等. 甜葉菊微衛星富集文庫的構建與多態性標記的篩選[J]. 作物學報,2014,40(3):447-456.
[10]邢智珺,姜虎成,陸 偉,等. 江蘇8個克氏原螯蝦群體遺傳多樣性微衛星分析[J]. 上海海洋大學學報,2014,23(5):656-662.
[11]黃小芳,唐章生,劉俊丹,等. 廣西不同地區克氏原螯蝦群體遺傳多樣性微衛星分析[J]. 南方農業學報,2020,51(2):437-444.
[12]Sambrook J,Fritsch E F,Maniatis T. Molecular cloning:a laboratory manual[M]. New York:Cold Spring Harbor Laboratory Press,1989.
[13]Belfiore N M,May B. Variable microsatellite loci in red swamp crayfish,Procambarus clarkii,and their characterization in other crayfish taxa[J]. Molecular Ecology,2000,9(12):2231-2234.
[14]Yeh F. Population genetic analysis of codominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany,1997,129:157.
[15]Rousset F. GENEPOP007:a complete re-implementation of the genepop software for Windows and Linux[J]. Molecular Ecology Resources,2008,8(1):103-106.
[16]Botstein D R,White R L,Skolnick M H,et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics,1980,32(3):314-331.
[17]Hubisz M J,Daniel F,Matthew S,et al. Inferring weak population structure with the assistance of sample group information[J]. Molecular Ecology Resources,2009,9(5):1322-1332.
[18]Jakobsson M,Rosenberg N A.CLUMPP:a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure[J]. Bioinformatics,2007,23(14):1801-1806.
[19]Rosenberg N A. Distruct:a program for the graphical display of population structure[J]. Molecular Ecology Notes,2004,4(1):137-138.
[20]劉倩倩,葉浩婷,李 放,等. 杭白芷種質資源遺傳多樣性的SSR分析[J]. 南方農業學報,2018,49(3):418-423.
[21]Yeh F C,Boy T J B. Population genetic analysis of codmminant and dominant markers and quantitave traits[J].Belgian Journal Botany,1997,129:157-163.
[22]Facon? B,Pointier? J? P,Glaubrecht? M,et? al.? A? molecular? phylogeography approach? to? biological? invasions? of? the? New? World? by? parthenogenetic? Thiarid snails[J]. Molecular Ecology,2003,12:3027-3039.
[23]黃 羽. 鄱陽湖流域克氏原螯蝦的資源狀況及長江中下游克氏原螯蝦遺傳多樣性研究[D]. 南昌:南昌大學,2012.
[24]Serrano M,Calvo J H,Martínez M,et al. Microsatellite based genetic diversity and population structure of the endangered Spanish Guadarrama goat breed[J]. BMC Genetics,2009,10:61.
收稿日期:2022-04-13
資助項目:中國水產科學研究院基本科研業務費(編號:2022XT01);江蘇省種業振興“揭榜掛帥”項目(編號:JBGS[2021]123);中國水產科學研究院淡水漁業研究中心基本科研業務費項目(編號:2021JBFM21)。
作者簡介:高 楊(1997—),男,安徽銅陵人,碩士研究生,研究方向為水產動物遺傳育種。E-mail:1463455739@qq.com。
通信作者:蘇勝彥,博士,副研究員,研究方向為蝦蟹遺傳育種,E-mail:ouhaicourse@hotmail.com;朱 健,博士,研究員,研究方向為水產動物遺傳育種和水產養殖,E-mail:zhuj@ffrc.cn。

