18種觀賞植物F3′5′H基因生物信息學分析
曾慧蘭 高陽 盧毅







摘要 采用生物信息學方法對18種觀賞植物類黃酮-3′5′-羥化酶基因(flavonoid-3′5′-hydroxylase,F3′5′H)的mRNA和氨基酸序列的理化性質、跨膜結構域、保守結構域、亞細胞定位、二級結構、三級結構和同源性進行預測與分析。結果表明,絕大多數觀賞植物的F3′5′H為親水性穩定蛋白質,以α 螺旋為主、無信號肽的跨膜蛋白質;大多數定位于內質網膜上;其三級結構模型為5ylw.1.A 鐵銹醇合成酶,為單鏈蛋白,屬于細胞色素P450基因家族;同源保守氨基酸序列為“LPPGP”“AGTDTS”和“PFGAGRRICAG”。
關鍵詞 生物信息學;觀賞植物;氨基酸序列;類黃酮-3′5′-羥化酶
中圖分類號 S 68? 文獻標識碼 A? 文章編號 0517-6611(2023)07-0103-07
doi:10.3969/j.issn.0517-6611.2023.07.026
Bioinformatical Analysis of Flavonoid-3′5′-Hydroxylase Genes Originated from 18 Ornamental Plants
ZENG Hui-lan1,2,GAO Yang1,LU Yi1
(1.College of Life Science and Resources and Environment,Yichun University,Yichun,Jiangxi 336000; 2. Key Laboratory of Crop Growth,Development and Regulation,Yichun University,Yichun,Jiangxi 336000)
Abstract Predictive analyses of the mRNA sequence or amino acid sequences of the flavonoid 3′5′-hydroxylase gene (F3′5′H) originated from 18 ornamental plants and their physical and chemical parameters,transmembrane domains,conserved domains,subcellular localizations,secondary structure,tertiary structure and evolutionary relationships were explored using bioinformatics method.The results suggested that most F3′5′H of the 18 ornamental plants are hydrophilic stable proteins and transmembrane proteins ,which are mainly made of alpha-helix and no signal peptides; they are mostly located on endoplasmic reticulum membrane and assigned to 5ylw.1.A,which belongs to cytochrome P450 superfamily and is a monomer named ferruginol synthase in tertiary structure analysis;the Homologous conserved amino acid sequences are “LPPGP” “AGTDTS” and “PFGAGRRICAG”.
Key words Bioinformatical;Ornamental plants;Amino acid sequence;Flavonoid-3′5′-hydroxylase
基金項目 江西省作物生長發育調控實驗室開放課題項目(KFJJ-201704);宜春學院博士科研啟動項目(2103360117013)。
作者簡介 曾慧蘭(1986—),女,江西宜春人,講師,博士,從事花色機理和園藝植物病害研究。
收稿日期 2022-04-12
花色是觀賞植物的重要性狀,植物的花色主要是由黃酮類化合物、類胡蘿卜素和生物堿3類物質含量決定的[1],而花色苷是黃酮類化合物的主要組成之一,能控制花的橙紅到藍紫等顏色[2]。類黃酮-3′5′-羥化酶 (flavonoid -3′5′ -hydroxylase,F3′5′H) 是花色苷代謝途徑中的關鍵酶之一[3]。F3′5′H催化花色素母體B環3′、5′位形成羥基集團,最終形成使花變藍的飛燕草色素,被稱為“藍色基因”[4],所以它對藍色花的育種具有非常重要的作用[5]。
目前,已從鶴望蘭(Strelitzia reginae)、矮牽牛 (Petunia hybrida)、非洲堇 (Saintpaulia spp.)、細莖石斛 (Dendrobium moniliforme) 等多種植物中分離出F3′5′H基因并對其功能進行了深入研究[6-9]。黃敏玲等[8]采用RT-PCR和RACE方法從鶴望蘭(Strelitzia reginae)黃色花萼中克隆到類黃酮生物合成途徑關鍵基因SrF3′5′H,并分析了其在鶴望蘭不同花期和花瓣中的表達含量。Qi 等[10]利用轉基因技術和qRT-PCR驗證了蝴蝶蘭屬(Phalaenopsis) PhF3′5′H在花色中的功能。嚴黎等[11]通過利用PCR方法擴增了丹參 (Salvia miltiorrhiza Bunge) 中的F3′5′H基因全長,并對該基因編碼蛋白質的理化定性、亞細胞定位、結構域等進行了分析。方穎等[12]以華麗龍膽 (Gentiana sino-ornata) 為材料利用RT-PCR技術克隆了GsF3′5′H全長,并進行了生物信息學分析。也有不少研究利用轉錄組測序技術關注不同花色植物中F3′5′H的表達與功能,并進行生物信息學分析[2,13-14]。雖然當前植物中F3′5′H基因功能和表達情況的研究不少,但目前對各植物中F3′5′H進行系統比較生物信息學相關的詳細分析鮮有報道。
該研究利用生物信息學原理和軟件,對NCBI GenBank 數據庫中已正式發表的18種觀賞植物的F3′5′H基因全長mRNA和氨基酸序列進行理化性質、亞細胞定位、跨膜結構、保守區域、二級結構、三級結構和同源性進行分析,旨在為藍色花卉的分子育種提供理論參考依據。
1 材料與方法
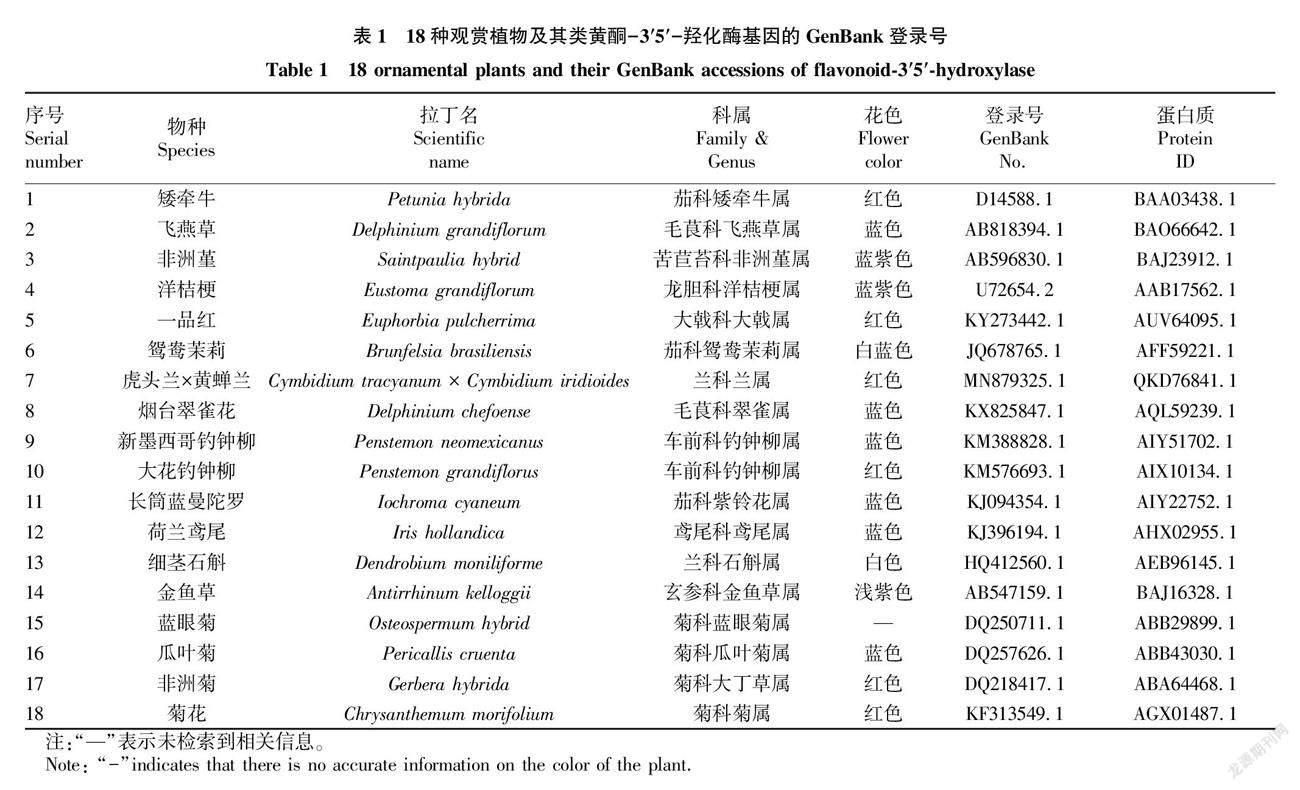
1.1 材料 從美國國家生物技術信息中心 (National Center for Biotechnology Information,NCBI) GenBank數據庫中檢索已正式注冊、物種來源明確的18種觀賞植物的F3′5′H基因全長mRNA核酸序列及其編碼的氨基酸序列 (表1)。在這18種觀賞植物中,藍紫色系花卉有10種,紅色系有6種,其他色系2種。
1.2 分析方法
運用ProtParam[15](https://web.expasy.org/protparam/) 對氨基酸的基本理化性質即氨基酸數、分子式、相對分子量、理論等電點、穩定性系數、帶負電殘基總數 (Asp+Glu)、帶正電殘基總數 (Arg+Lys)、親疏水性和脂肪族氨基酸指數進行在線分析;通過SignalP 4.1[16](https://services.healthtech.dtu.dk/service.php?SignalP-4.1) 和SignalP 5.0[17] (https://services.healthtech.dtu.dk/service.php?SignalP-5.0) 對各序列的信號肽進行預測;利用TMHMM 2.0[18] (https://services.healthtech.dtu.dk/service.php?TMHMM-2.0) 分析氨基酸序列的跨膜結構域,并通過SMART[19] (http://smart.embl.de/smart/change_mode.pl) 驗證跨膜區域;運用PSORT[20] (http://psort1.hgc.jp/form.html)在線軟件對氨基酸序列進行亞細胞定位分析;利用WebLogo 3[21] (http://weblogo.threeplusone.com/create.cgi) 進行保守區域分析;利用SOPMA[22] (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html) 進行氨基酸序列的二級結構分析;利用SWISS-MODEL[23] (https://swissmodel.expasy.org/) 進行氨基酸序列的三級結構分析;利用MEGA 11[24] (https://www.megasoftware.net/) 進行上述序列的多重比對,尋找保守區域,并采用鄰接法 (Neighbor-Joining,NJ) 迭代數為999次建立系統發育樹,分析其在遺傳學上的親緣關系。
2 結果與分析
2.1 F3′5′H理化性質分析
通過ProtParam 在線軟件對18種觀賞植物F3′5′H基因所編碼蛋白質的理化性質進行分析 (表2),結果表明,18種觀賞植物F3′5′H基因編碼的氨基酸數為502~535,平均為510.1;相對分子量為55 827.39~58 080.35,平均為56 737.91;理論等電點為6.52~9.31,平均為8.39;不穩定性系數為28.58~47.99,平均為38.365;帶負電殘基總數 (Asp+Glu)為49~60,平均為54.5;帶正電殘基總數 (Arg+Lys) 為52~66,平均為58.9;親水性平均系數為-0.146~0.085,平均為-0.041;脂肪族氨基酸指數為90.22~106.54,平均為96.42。從表2可知,有11種觀賞植物的F3′5′H為穩定的親水性蛋白質,占比61.11%;有4種觀賞植物的F3′5′H為不穩定的親水性蛋白質,占比22.22%;有2種觀賞植物的F3′5′H為穩定的疏水性蛋白質,占比11.11%;有1種觀賞植物的F3′5′H為不穩定的疏水性蛋白質,占比5.56%。
2.2 F3′5′H信號肽和跨膜區域分析
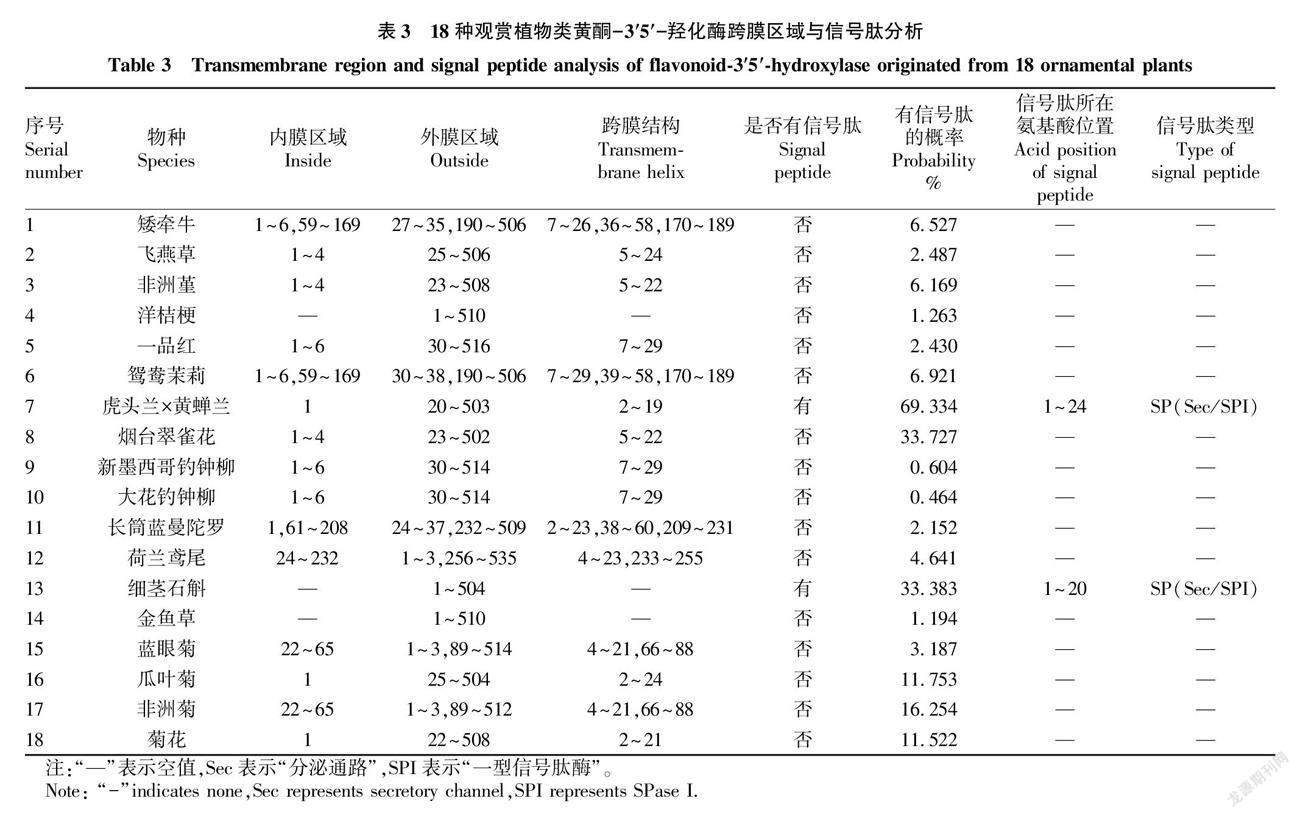
信號肽的主要作用是促進蛋白分泌到胞外,通常位于氨基酸序列的N端,是引導新合成的蛋白質向分泌通路轉移的短肽鏈[25],一般長度為5~30個氨基酸。通過SignalP 4.1和5.0對F3′5′H編碼的蛋白質進行信號肽分析 (表3),結果表明,所測的16種觀賞植物的F3′5′H均無信號肽,只有虎頭蘭×黃蟬蘭和細莖石斛具有信號肽,信號肽所在位置分別為1~24位和1~20位氨基酸,且都與分泌 (Sec/secretory) 通路相關的能被一型信號肽酶 (SPI) 切除的信號肽。從表3可知,有15種植物的F3′5′H為跨膜蛋白,具有內膜區域、外膜區域和跨膜區域;有3種植物的F3′5′H無跨膜區域,只有外膜區域。經SMART分析驗證,TMHMM 2.0所測的跨膜區域與SMART分析的結構域一致。
2.3 F3′5′H亞細胞定位分析
通過PSORT在線分析了8種觀賞植物F3′5′H的亞細胞定位 (表4),根據定位頻率和可靠性可知,該類蛋白位于內質網膜的可能性最大,為0.640~0.820,平均可靠性為0.786;其次為質膜、內質網腔、細胞核、過氧化物酶體、細胞外、高爾基體,最后為線粒體基質空間和葉綠體類囊體膜上。結合細胞生物學知識,可預測得F3′5′H可能有以下幾類運輸方式來行使催化功能:一類是F3′5′H蛋白在游離核糖體中合成后,通過信號肽錨定于內質網膜上,通過跨膜運輸和膜泡方式分選到過氧化物酶體等細胞器行使催化功能,如虎頭蘭×黃蟬蘭;第二類是在游離核糖體中合成后,通過信號肽錨定于內質網膜上,通過分泌方式分選到過氧化物酶體等細胞器行使催化功能,如細莖石斛;第三類是在游離核糖體中合成后轉移至內質網膜上,以跨膜運輸方式經內質網腔再直接運輸到胞外行使催化功能,如飛燕草、一品紅、煙臺翠雀花、新墨西哥釣鐘柳、大花釣鐘柳、荷蘭鳶尾;第四類是在游離核糖體中合成后轉移至內質網膜上,通過跨膜運輸方式分選到線粒體、葉綠體、高爾基體、過氧化物酶體等細胞器后行使催化功能,如非洲堇、長筒藍曼陀羅、藍眼菊、瓜葉菊、菊花;第五類是直接由細胞核內合成,不經跨膜方式,可能直接在細胞質中的線粒體、葉綠體等細胞器的特定部位中行使催化功能,如洋桔梗。
2.4 F3′5′H二級結構和三級結構分析
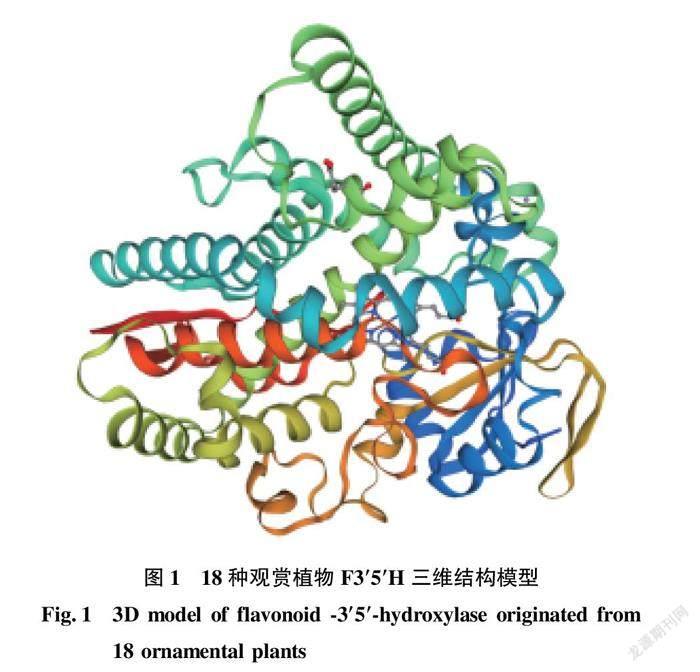
通過SOPMA軟件在線預測了18種觀賞植物F3′5′H的二級結構 (表5),表明F3′5′H主二級結構主要以 α 螺旋為主,其次為無規則卷曲、延伸主鏈,最少的二級結構是β轉角。通過SWISS-MODEL同源建模預測結果中可知 (表6),所測的所有蛋白質序列一致度均大于30%,適合用同源建模法進行三級結構預測,即符合SWISS-MODEL建模質量評估條件,同時質量評估系數 GMQE和QMEAND值均較大(取值范圍為0~1),建模可靠。從表6可知,所測的18種觀賞植物F3′5′H的三級結構均建模至同一蛋白模型 5ylw.1.A 鐵銹醇合成酶 (圖1),為單鏈蛋白,屬于細胞色素P450基因家族。
2.5 F3′5′H同源比對和系統發育分析
經MEGA 11中ClustalW比對分析,18種F3′5′H 編碼氨基酸的保守位點總計121個,明顯的保守序列有起始于48位的“LPPGP”序列、起始于334位的“AGTDTS”序列和起始于469位的“PFGAGRRICAG”序列。從WebLogo 3結果可知 (圖2),保守序列和ClustalW分析序列基本一致。
從構建的系統發育樹 (圖3) 可知,屬于同科植物的F3′5′H 其親緣關系較近,分屬茄科、菊科、蘭科、毛茛科、車前科的13種觀賞植物在科內F3′5′H親緣關系近。在不同科屬間,茄科與龍膽科的洋桔梗和大戟科的一品紅F3′5′H親緣關系近;車前科與玄參科的金魚草和苦苣苔科的非洲堇F3′5′H親緣關系較近;鳶尾科的荷蘭鳶尾與菊科F3′5′H親緣關系較近。
3 結論與討論
觀賞植物的類黃酮-3′5′-羥基化酶 (F3′5′H) 是花青苷代謝途徑中重要的關鍵酶,對藍紫色花色的形成有重要的作用。目前越來越多的研究關注F3′5′H的基因功能,并對其功能進行驗證[3,10,14],但國內外極少數研究從生物信息學角度關注各種觀賞植物F3′5′H基因和蛋白質序列的差異。該研究從GenBank中搜索已發表的F3′5′H全長mRNA序列及其編碼的蛋白質,共篩選出18種觀賞植物的核酸和蛋白質序列,從理化性質、亞細胞定位、保守序列、二級結構、三級結構、同源性等方面進行分析。
從理化性質和二級結構來看,雖然不同物種間的F3′5′H蛋白質理化性質略有差異,但大部分物種的不穩定性系數為小于40,親水性平均系數為負值,且二級結構均以α螺旋為主,表明該類酶為以α螺旋為主的穩定親水性單鏈蛋白,這與楊曉娜等[26]分析結果相一致。從三級結構來看,所測18種觀賞植物均建模至蛋白模型 5ylw.1.A 鐵銹醇合成酶,由此可推斷,在所有的分析指標中,三維結構可能是與其功能密切相關的一個指標,為有待進一步挖掘的功能指標。跨膜結構域、信號肽和亞細胞定位分析表明,15種植物的F3′5′H為P450基因家族跨膜蛋白,具有內膜區域、外膜區域和跨膜區域,也進一步說明了該蛋白的形成是經過了跨膜運輸加工后行使功能,且虎頭蘭×黃蟬蘭和細莖石斛有信號肽指引蛋白質通過分泌通路進行轉移,這與楊曉娜等[26]分析結果基本一致。從序列同源性特征來看,F3′5′H有3個較保守的序列,分別是起始于48位的“LPPGP”序列連接膜錨定位點和酶蛋白球體部分的結合域、起始于334位的“AGTDTS”序列氧分子形成結合域和起始于469位的“PFGAGRRICAG”序列血紅素結合域,這與楊曉娜等[26]分析結果一致。從系統發育樹來看,F3′5′H蛋白質序列可明顯表示出不同植物間的親緣關系,保守性較高,有利于區分不同物種間的親緣關系,該特性可能使其用于物種間的鑒定,提高物種分辨率。
近期雖然已有研究進行了高等植物F3′5′H基因cDNA及其氨基酸序列的生物學分析[26],但從該分析來看并未將所列植物的具體信息進行詳細剖析,且多數植物的信息不夠具體全面,甚至有些序列與該研究相同,但得出了不同的結果。原因可能為在線軟件的版本問題,或者雖然楊曉娜等[26]的研究列出23種高等植物的信息,但并未將該23種植物的全部信息詳細列出,而是進行了概述與總結。
生物信息學是采用數學、統計學和計算機等方法分析生物學、生物化學等數據的一門綜合性學科。當前基因組學、轉錄組學和蛋白質組學測序技術的發展,促進了生物信息學的發展,而生物信息學也從各方面更好地輔助了生物學和育種技術的發展。利用生物信息學分析了18種觀賞植物F3′5′H蛋白質序列的基本信息并做了詳細的剖析,可為后期藍色花的育種技術提供理論依據。
參考文獻
[1] TANAKA Y,BRUGLIERA F,CHANDLER S.Recent progress of flower colour modification by biotechnology[J].International journal of molecular sciences,2009,10(12):5350-5369.
[2] GUO L P,WANG Y J,DA SILVA J A T,et al.Transcriptome and chemical analysis reveal putative genes involved in flower color change in Paeonia ‘Coral Sunset’[J].Plant physiology and biochemistry,2019,138:130-139.
[3] HUANG H,HU K,HAN K T,et al.Flower colour modification of chrysanthemum by suppression of F3′H and overexpression of the exogenous Senecio cruentus F3′5′H gene[J].PLoS One,2013,8(11):1-12.
[4] TANAKA Y,BRUGLIERA F.Flower colour and cytochromes P450[J].Philosophical transactions of the royal society B:Biological sciences,2013,368:1-14.
[5] 張泰然,張和臣,武榮花.藍色花形成分子機理研究進展[J].植物學報,2020,55(2):216-227.
[6] SATO M,KAWABE T,HOSOKAWA M,et al.Tissue culture-induced flower-color changes in Saintpaulia caused by excision of the transposon inserted in the flavonoid 3′,5′ hydroxylase (F3′5′H) promoter[J].Plant cell reports,2011,30(5):929-939.
[7] 李莉,祁銀燕,解燕,等.矮牽牛編碼 F3′5′H 的藍色基因表達載體構建及轉化[J].西北植物學報,2011,31(6):1090-1096.
[8] 黃敏玲,樊榮輝.鶴望蘭類黃酮3′,5′-羥化酶基因 SrF3′5′H 的克隆及表達分析[J].中國細胞生物學學報,2012,34(11):1117-1122.
[9] WHANG S S,UM W S,SONG I J,et al.Molecular analysis of anthocyanin biosynthetic genes and control of flower coloration by flavonoid 3′,5′-hydroxylase (F3′5′H) in Dendrobium moniliforme[J].Journal of plant biology,2011,54(3):209-218.
[10] QI Y Y,LOU Q,QUAN Y H,et al.Flower-specific expression of the Phalaenopsis flavonoid 3′,5′-hydoxylase modifies flower color pigmentation in Petunia and Lilium[J].Plant cell,tissue and organ culture,2013,115(2):263-273.
[11] 嚴黎,劉琬菁,楊成民,等.丹參 F3′5′H 基因克隆及其序列分析[J].世界中醫藥,2020,15(5):689-695,701.
[12] 方穎,黃啟群,金雪花. 華麗龍膽GsF3′5′H和GsFNS基因的克隆及表達分析[J].西北植物學報. 2020,40(12): 2023-2030.
[13] LIANG C Y,RENGASAMY K P,HUANG L M,et al.Assessment of violet-blue color formation in Phalaenopsis orchids[J].BMC plant biology,2020,20(1):1-16.
[14] LU C F,LI Y J,CUI Y M,et al.Isolation and functional analysis of genes involved in polyacylated anthocyanin biosynthesis in blue Senecio cruentus[J].Frontiers in pant science,2021,12:1-20.
[15]
GASTEIGER E,HOOGLAND C,GATTIKER A.Protein identification and analysis tools on the ExPASy server[M]//WALKER J M.The proteomics protocols handbook.Totowa,NJ:Humana Press,2005:571-607.
[16] PETERSEN T N,BRUNAK S,VON HEIJNE G,et al.SignalP 4.0:Discriminating signal peptides from transmembrane regions[J].Nature methods,2011,8(10):785-786.
[17] ALMAGRO ARMENTEROS J J,TSIRIGOS K D,SNDERBY C K,et al.SignalP 5.0 improves signal peptide predictions using deep neural networks[J].Nature biotechnology,2019,37(4):420-423.
[18] MLLER S,CRONING M D,APWEILER R.Evaluation of methods for the prediction of membrane spanning regions[J].Bioinformatics,2001,17(7):646-653.
[19] LETUNIC I,KHEDKAR S,BORK P.SMART:Recent updates,new developments and status in 2020[J].Nucleic acids research,2021,49:D458-D460.
[20] NAKAI K,KANEHISA M.Expert system for predicting protein localization sites in gram-negative bacteria[J].Proteins-structure function and bioinformatics,1991,11(2):95-110.
[21] CROOKS G E,HON G,CHANDONIA J M,et al.WebLogo: A sequence logo generator[J].Genome research,2004,14(6):1188-1190.
[22] GEOURJON C,DELAGE G.SOPMA:Significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Computer applications in the biosciences,1995,11(6):681-684.
[23] WATERHOUSE A,BERTONI M,BIENERT S,et al.SWISS-MODEL:Homology modelling of protein structures and complexes[J].Nucleic acids research,2018,46(W1):W296-W303.
[24] TAMURA K,STECHER G,KUMAR S.MEGA11: Molecular evolutionary genetics analysis version 11[J].Molecular biology and evolution,2021,38(7):3022-3027.
[25] 鄭斌,詹希美.信號肽序列及其在蛋白質表達中的應用[J].生物技術通訊,2005,16(3):296-298.
[26] 楊曉娜,陳自宏,陳宏艷,等.高等植物類黃酮3′5′-羥化酶基因cDNA及其氨基酸序列的生物信息學分析[J].保山學院學報,2021,40(2):24-32.

