基于線粒體COI和D-loop序列的黑龍江流域蛇種群遺傳結構分析
胡宗云,楊培民,宋紅梅,牟希東
(1.遼寧省淡水水產科學研究院,遼寧省水生動物病害防治重點實驗室,遼寧遼陽 111000;2.中國水產科學研究院珠江水產研究所,農業農村部休閑漁業重點實驗室,廣州 510380)
線粒體DNA分子標記是核基因組分子標記的重要補充,已廣泛用于水產動物的系統發育、生物地理學和保護生物學研究[15]。細胞色素氧化還原酶I(COI) 基因位于mtDNA的蛋白質編碼區,進化速率適中,在物種分子系統演化和分類中應用較多;而D-loop序列屬于非編碼區,是mtDNA進化速度最快的區域,常用于魚類進化生物學和群體遺傳學研究。本研究測定、分析了黑龍江流域3個蛇群體的COI和D-loop序列,研究了其遺傳多樣性水平和群體間遺傳分化程度,以期為東北地區蛇種質資源保護提供理論依據。
1 材料與方法
1.1 材料
2020年4月至2021年5月,采用地籠網和掛網采集了3個群體88尾野生蛇樣本,其中撫遠群體30尾(黑龍江水系,標記為FY)、饒河群體29尾(烏蘇里江水系,標記為RH)、榆樹群體29尾(松花江水系,標記為YS)(表1)。樣本體長為13~23 cm,體重為16~51 g。每尾剪取鰭條1~2 g,保存于95%酒精內帶回實驗室。
表1 蛇樣品來源信息
Tab.1 The source information on samples of longnose gudgeon S.dabryi Bleeker

表1 蛇樣品來源信息
群體所屬河流取樣時間采集地經緯度樣品數FY黑龍江2020年4月黑龍江省佳木斯市撫遠市濃橋鎮 N48°11'58.36″;E134°17'1.28”30RH烏蘇里江2020年5月黑龍江省雙鴨山市饒河縣八五九農場 N47°20'13.50″; E134°9'10.42″29YS松花江2021年5月吉林省長春市榆樹市培英街道N44°50'13.35″ ;E126°37'30.50″29
1.2 方法
1.2.1 DNA提取、PCR擴增及測序
取鰭條0.1g左右,利用天根生化科技有限公司的基因組提取試劑盒(DP304),按照試劑盒說明書提取總DNA。COI序列擴增引物為C-F(5′-TC AACCAACCACAAAGACATTGGCAC-3′)和C-R(5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′)[16],D-loop序列擴增引物為D-F(5′-CTAACTCCCAAAGC TAGAATTCT-3′)和D-R(5′-ATCTTAGCATCTTCAGTG-3′)[17],上述引物均由生工生物(上海)有限公司合成。PCR反應體系為25 μL:含2.5 μL 10×PCR buffer(Mg2+plus,20 mmol/L)、2 μL dNTP mix(2.5 mmol/L)、0.25 μL Taq DNA polymerase(5 U/μL)、上下游引物各1 μL(10 μmol/L)、模板1 μL,雙蒸水補足至25 μL。PCR擴增條件為:94 ℃預變性5 min;35個循環,每個循環包括94 ℃變性1 min,56 ℃退火1 min,72 ℃延伸1 min 30 s;最后73 ℃總延伸10 min。本研究所有的PCR反應均在ABI 9700擴增儀上進行,擴增產物經1.2%瓊脂糖凝膠電泳檢測,獲得目的條帶的產物于-20 ℃冰箱內保存備用。PCR產物送至生工生物(上海)有限公司進行純化,并由ABI 3730全自動測序儀雙向測序,測序引物為上述PCR擴增引物。
1.2.2 數據處理
測得的序列經BioEdit編輯去除兩端低質量的堿基,然后在NCBI網站上進行比對,留取與蛇線粒體基因組(KF534790.1、KF612272.1)COI和D-loop序列同源性99%以上的序列。運用MEGA 6.0軟件包中的Alignment by ClustalW程序對留取的序列進行比對和剪切,并計算序列堿基組成、變異位點數和轉換與顛換值;采用鄰接法(Neighbor-Joining),基于Kimura雙參數法(Kimura 2-parameter,K2p)模型構建單倍型進化樹。利用DnaSP 6.0軟件統計兩個基因的單倍型數、單倍型多樣性(h)、平均核苷酸差異數(K)及核苷酸多樣性(π)。應用Arlequin 3.5 軟件進行中性檢驗,并用其分子方差分析(AMOVA)方法計算遺傳變異在群體內和群體間的分布及群體間遺傳分化系數(Fst),1 000次重復隨機抽樣重排后進行顯著性檢驗,以P<0.05作為差異顯著性水平。利用Popart 1.7軟件構建TCS單倍型網絡圖。
2 結果
2.1 序列組成與遺傳多樣性分析
COI和D-loop序列經比對剪齊后,分別獲得了長度為582~583 bpCOI和834~835 bp D-loop的同源序列(表2)。COI同源序列中共檢測出15個變異位點,其中簡約信息位點12個,單突變位點3個,一共定義了13個單倍型;平均堿基組成:A=28.7%、T=23.2%、G=28.9%、C=19.2%,A+T含量(52%)大于G+C含量(48%);平均轉換/顛換值為9.8。D-loop同源序列中共檢測到14個變異位點,包括9個簡約信息位點和5個單突變位點,定義單倍型15個,平均堿基組成:A=32%、T=31.9%、G=14.4%、C=21.8%,A+T含量(64%)大于G+C含量(36%);平均轉換/顛換值為4.6。COI和D-loop堿基組成均呈較明顯的AT偏好。
3個群體COI基因片段總體單倍型多樣性、核苷酸多樣性和核苷酸差異數分別為0.377 3、0.000 9和0.510 4;各群體的單倍型多樣性指數為0.330 7~0.406 2,單倍型多樣性指數大小順序為YS>RH>FY;核苷酸多樣性指數均為0.000 9(表2)。3個群體D-loop序列總體單倍型多樣性、核苷酸多樣性和核苷酸差異數分別為0.577 0、0.000 9和0.780 4;各群體的單倍型多樣性指數和核苷酸多樣性指數分別介于0.492 0~0.692 0和0.000 8~0.001 1;群體間D-loop單倍型多樣性指數、核苷酸多樣性指數大小順序一致,均為YS>RH>FY。3個群體基于COI和D-loop序列的遺傳多樣性指數相近,單倍型多樣性、核苷酸多樣性指數處于較低水平,樣本間兩個序列的核苷酸差異較小。
表2 蛇3個群體COI 和D-loop基因的遺傳多樣性參數
Tab.2 Genetic diversity parameters of COI and D-loop genes in three populations of longnose gudgeon S.dabryi Bleeker

表2 蛇3個群體COI 和D-loop基因的遺傳多樣性參數
遺傳多樣性參數COIFYRHYS總體D-loopFYRHYS總體序列長度582^583582^583582^583582^583835835834^835834^835變異位點數5771568914單一突變位點數46733565簡約突變位點數110123339單倍型數67513791015單倍型多樣性0.330 70.404 30.406 20.377 30.492 00.545 00.692 00.577 0核苷酸多樣性0.000 90.000 90.000 90.000 90.000 80.001 00.001 10.000 9平均核苷酸差異數k0.500 00.511 80.520 00.510 40.627 00.831 00.872 00.780 4
2.2 遺傳距離和遺傳分化
用Kimura雙參數法(Kimura 2-parameter,K2p)計算3個群體間遺傳距離和遺傳分化指數如表3所示。基于COI序列的分析結果顯示,3個群體的群體內和群體間遺傳距離相等,均為0.000 9;3個群體的群體間遺傳分化指數-0.004 9~0.007 6。基于D-loop序列的計算結果表明,3個群體的群體內遺傳距離為0.008 0~0.010 0,大小順序為RH =YS >FY;群體間遺傳距離為0.000 9~0.001 0,YS與FY群體間遺傳距離和FY與RH群體間遺傳距離相等(0.000 9),略小于YS與RH群體間的遺傳距離(0.001 0);3個群體間的遺傳分化指數介于-0.002 8~0.011 4。3個群體基于COI和D-loop序列的遺傳距離、遺傳分化指數較小,預示3個群體間遺傳差異較小,遺傳分化不明顯。
表3 蛇遺傳分化指數(對角線上)、群體間遺傳距離(對角線下)和群體內遺傳距離(對角線)
Tab.3 Pairwise Fst(above diagonal),genetic distance between(below diagonal) and within(diagonal) populations of longnose gudgeon S.dabryi Bleeker

表3 蛇遺傳分化指數(對角線上)、群體間遺傳距離(對角線下)和群體內遺傳距離(對角線)
群體FYRHYSFY0.000 9-0.004 9-0.003 4COIRH0.000 90.000 90.007 6YS0.000 90.000 90.000 9FY0.000 80.011 4-0.002 8 D-loopRH0.000 90.001 00.006 8YS0.000 90.001 00.001 0
基于COI序列的分子方差分析(AMOVA)顯示,群體內遺傳變異占遺傳變異的100.02%,遠大于群體間遺傳變異(-0.02%);基于D-loop序列AMOVA分析同樣顯示,群體內的遺傳變異占比(99.49%)也遠大于群體間遺傳變異(0.51%)。基于COI和D-loop序列的分子方差分析表明,3個蛇群體整體遺傳變異幾乎全部來自群體內,群體間遺傳變異占比極小(表4)。
表4 蛇3個群體COI和D-loop基因的AMOVA分析
Tab.4 The AMOVA analysis of COI and D-loop genes in three populations of longnose gudgeon S.dabryi Bleeker
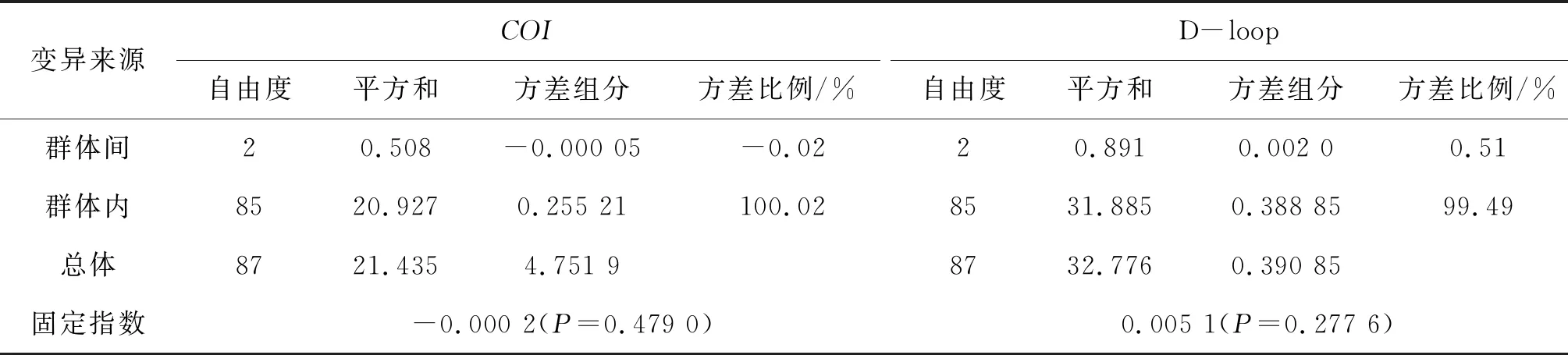
表4 蛇3個群體COI和D-loop基因的AMOVA分析
變異來源COI自由度平方和方差組分方差比例/%D-loop自由度平方和方差組分方差比例/%群體間20.508-0.000 05-0.0220.8910.002 00.51群體內8520.9270.255 21100.028531.8850.388 8599.49總體8721.4354.751 98732.7760.390 85固定指數-0.000 2(P=0.479 0)0.005 1(P=0.277 6)
2.3 單倍型聚類分析與分布
88個COI序列共檢測到13個單倍型(圖1),Hap1和Hap3為3個群體共享單倍型,在所測樣本中出現頻率分別為79.55%和4.55%,其中Hap1在每個群體占比均最高,屬于優勢單倍型;Hap9為FY和YS的共享單倍型,其余單倍型為3個群體特有的單倍型,FY、RH和YS群體特有單倍型的數量分別為3、5和2。88個D-Loop序列共定義了15個單倍型(圖2),Hap2、Hap7和Hap9為3個群體共享單倍型,其中Hap2數量達到58個,占樣本總數的65.91%,屬于優勢單倍型;Hap11、Hap12為FY與YS的共享單倍型,Hpa1、Hap8為RH與FY的共享單倍型,Hap6為FY和RH的共享單倍型,其余單倍型為各群體特有單倍型,特有單倍型數量1-3個不等。

圖1 蛇3個群體COI基因單倍型的NJ樹及在各群體中的分布
2.4 種群歷史動態


圖4 蛇COI(A)和D-loop序列的核苷酸錯配分布圖

表5 基于COI和D-loop序列的Tajima′D和檢驗
3 討論
3.1 蛇群體遺傳多樣性
單倍型多樣性(h)和核苷酸多樣性(π)是評價種群遺傳多樣性的重要指標,也是衡量物種或種群線粒體DNA變異程度的重要指標。本研究3個蛇群體基于D-Loop序列的單倍型多樣性和核苷酸多樣性分別為0.492 0~0.692 0、0.000 8~0.001 1,低于嘉陵江蛇種群(h:0.875~0.940;π:0.010 0~0.264 0)[4],也低于近緣種斑點蛇(S.punctatus)(h:0.703~0.960;π:0.021 7~0.048 2)[4]和長鰭吻(Rhinogobioventralis))(h:0.640~0.837;π:0.001 0~0.0.01 5)[18],這些差異除與物種的遺傳特性有關外,還與樣本量大小、序列長短、分析序列的位置有關。本研究88個蛇樣本基于COI序列的遺傳多樣性指數(h =0.377 3;π=0.000 9)略小于D-loop(h=0.577 0;π=0.000 9),這可能與COI和D-loop的進化速率不同有關:COI和D-loop分別位于線粒體的編碼區和非編碼區,COI受到的選擇壓力大于D-loop,進化速度相對較慢,發生變異的概率相對低一些,相似的結果在銀鯧(Pampusargenteus)[19]、強彈涂魚(Periophthalmuscantonensis)[20]、鯉(Cyprinuscarpio)[21]等魚類上亦有報道。根據GRANT等[22]設定h=0.5,π=0.005閾值,本研究3個蛇群體基于COI和D-loop序列的遺傳多樣性指數類型分別屬于低h低π型、高h低π型,考慮到核苷酸多樣性在一定程度上比單倍型多樣性更能反映群體的遺傳多樣性[22], 3個蛇群體遺傳多樣性處于較低水平,應加強該區域蛇的種質資源保護。
3.2 蛇群體遺傳分化
群體間遺傳距離、遺傳分化指數與群體分化程度為線性關系,是衡量群體分化程度的重要指標[23]。3個蛇群體基于兩個序列的群體間遺傳距離與群體內的遺傳距離均較小,大小范圍相互重疊且處于一個水平,群體間遺傳距離遠低于0.05種群這一標準[24],這說明3個群體間遺傳差異較小。群體間的遺傳分化指數小于0.05,表明群體間遺傳分化極小[25]。AMOVA分析表明,群體內的變異遠大于群體間變異,3個蛇群體總的遺傳變異主要來自群體內,預示3個蛇群體親緣關系較近。此外,本研究基于COI和D-loop序列的單倍型NJ系統發育樹和網絡圖顯示,3個蛇群體共享單倍型較多,特有單倍型交錯地聚集在一起或分布在祖先單倍型周圍,沒有形成明顯的地理譜系,也印證了群體間遺傳分化較小的觀點。上述結果可能與蛇棲息環境和繁殖習性有關:本研究中 3個群體的采樣點分布于黑龍江水系的支流,河流間處于連通狀態,采樣點之間未形成嚴格意義上的地理隔離;而蛇產漂流性卵,魚卵或魚苗隨水流進行擴散[1],利于群體擴散。換言之,敞開性水域和較強的擴散能力促進了3個蛇群體的基因交流,使群體間遺傳分化較小。相似的結果在蛇長江種群[26]、長鰭吻(R.ventralis)[18]、圓筒吻(Rhinogobiocylindricus)[27]等亞科魚類上亦有報道。鑒于3個群體遺傳距離較小、遺傳分化水平較低這一現狀,漁業資源管理實踐中可以將其設為一個管理單元(MU)。
3.3 蛇群體歷史動態


