玉米轉錄因子ZmEREB93負調控籽粒發育
龐浩婉,傅乾坤,楊青青,張元元,付鳳玲,于好強
玉米轉錄因子ZmEREB93負調控籽粒發育
龐浩婉1,傅乾坤1,楊青青1,張元元2,付鳳玲1,于好強1
1四川農業大學玉米研究所,成都 611130;2綿陽師范學院生命科學與技術學院,四川綿陽 621000
【目的】玉米作為重要的糧、經、飼多用作物,其產量的穩定對經濟發展和糧食安全意義重大。AP2/EREBP(APETALA2/ ethylene response element binding protein,AP2/EREBP)轉錄因子在植物生長發育及逆境應答中發揮重要作用。前期研究發現,玉米ZmBES1/BZR1-5轉錄因子靶基因可能參與調控種子大小。克隆,并對其表達特性及功能進行分析,為深入解析其調控玉米籽粒發育的功能與機制奠定基礎。【方法】從玉米自交系B73中克隆的全長序列,對其基因序列和編碼氨基酸序列特征進行生物信息學分析。隨后,通過實時熒光定量PCR(quantitative real-time PCR,qRT-PCR)分析其組織表達模式,分別構建植物和酵母表達載體,進行亞細胞定位和轉錄激活活性分析。經農桿菌介導法將轉入擬南芥,對轉基因株系的種子表型進行分析。最后,通過體外染色質免疫共沉淀測序(chromatin immunoprecipitation sequencing,Chip-seq)和共表達分析篩選ZmEREB93可能調控的候選靶基因,并通過酵母單雜交(yeast one hybrid,Y1H)驗證。【結果】成功克隆獲得,序列分析結果表明,無內含子,開放閱讀框長618 bp,編碼205個氨基酸,有一個高度保守的AP2結構域,屬于AP2家族的ERF亞類。qRT-PCR結果表明,在授粉后15和25 d的種子中表達量較高,其中,在25 d種子中表達量最高,約為15 d種子中表達量的11倍,在莖和根中有微量表達,在雄穗、花絲及苞葉中無表達。轉錄激活試驗結果表明,ZmEREB93蛋白在酵母細胞中不具有轉錄激活活性。亞細胞定位結果顯示,ZmEREB93蛋白定位于細胞核。與野生型株系相比,轉基因擬南芥株系種子的長和寬顯著變小且千粒重顯著降低。體外Chip-seq與共表達分析結果表明,、、及為ZmEREB93轉錄因子的候選靶基因。Y1H試驗表明,ZmEREB93蛋白可直接結合啟動子。【結論】玉米ZmEREB93作為轉錄因子在種子中特異性表達,負調控種子大小。
玉米;轉錄因子;AP2/EREBP;籽粒
0 引言
【研究意義】玉米作為中國重要的糧、經、飼多用作物,其產量的穩定對中國經濟發展和糧食安全意義重大。籽粒性狀是決定玉米產量與品質的重要因素,除環境外,轉錄因子、激素等內部因子在籽粒發育中也發揮重要作用[1]。因此,挖掘玉米籽粒發育相關基因,可用于培育高產玉米新品種。【前人研究進展】轉錄因子指能夠與基因啟動子區域的順式作用元件結合,進而激活或抑制靶基因轉錄的一類蛋白,在植物生長發育及逆境應答中起重要調控作用。植物中,轉錄因子被分為不同的類別,例如MYB、WRKY、bZIP以及AP2/EREBP等。其中,AP2/EREBP(APETALA2/ ethylene response element binding protein)作為植物中特有的一類轉錄因子,調控植物非生物脅迫響應、生長發育及免疫應答等途徑[2]。1998年,JOFUKU等[3]率先從模式植物擬南芥中分離了第一個與花發育有關的AP2基因,編碼蛋白含有2個AP2結構域。隨后的研究中,根據AP2/ERF結構域數量及序列差異,AP2/EREBP家族成員被劃分為AP2、RAV、ERF、Soloist等4個亞家族,但均有至少一個AP2/ERF結構域,由58—70個氨基酸殘基組成,可特異性結合GCC-box元件(AGCCGCC)或DRE/CRT元件(序列A/GCCGAC)[4]。其中,AP2亞類有2個AP2保守結構域,RAV亞類有1個AP2和B3結構域,ERF和Solosit亞類均只有1個AP2保守結構域[5]。根據ERF亞類成員的AP2結構域第14位和第19位氨基酸的不同,又可以分為ERF(第14位丙氨酸,第19位天冬氨酸)和DREB/CBF(第14位纈氨酸,第19位谷氨酸)兩類[6]。ERF亞類成員通過結合下游基因啟動子區的GCC-box或DRE/CRT core順式作用元件,調控靶基因表達,進而調控植物細胞內生理生化過程[7-9]。研究表明,AP2亞類主要參與調控開花、果實與種子發育等生長發育和逆境應答過程[10]。如,大麥HvAP2轉錄因子與MADS29互作,控制小花密度[11],過表達大豆可以促進開花[12],但過表達鳳梨后則延遲開花[13]。而且,擬南芥和水稻個別AP2成員基因突變后,種子變大,重量增加[14-15]。過表達鳳梨和萵苣會促使種子長度縮短,千粒重降低[13, 16]。但是,大豆GmAP2通過影響種子的長度,寬度和面積,進而正調控種子的大小[12]。目前,ERF亞類研究報道也較為深入,其功能涉及多方面,參與調控初級次生代謝、激素信號轉導、生長發育及逆境應答等過程[17-20]。如菊花負調控胚的發育,干涉可促進胚胎發育和結實率[20]。番茄沉默株系促進葉片衰老,而過表達可延遲葉片衰老[17]。RAV亞類主要參與調控葉片衰老及生物與非生物脅迫,Soloist亞類的研究則鮮有報道[21]。玉米AP2家族共有214個成員,其中,AP2亞族成員IDS1和ANT調控維管和花發育,IDS1決定穗狀花序分生組織的發育及成花數量,突變導致光合效率和生長率降低,以致產量降低[22-24]。此外,玉米AP2還調控節間和根系發育,其突變體節間變短和葉片起皺[25],過表達植株的支撐根數量增加,突變體的支撐根數量減少[26]。同時,研究發現玉米ERF亞類有166個成員,包括105個ERF成員和61個DREB成員,則主要參與逆境脅迫應答過程[22]。如表達受鹽脅迫誘導,轉化擬南芥的株系耐鹽性顯著提高[27],還可增強大斑病抗性,其突變體則易感大斑病[28],ZmEREB58通過促進表達,進而促進倍半萜的合成與積累[29]。【本研究切入點】目前,關于玉米ERF亞類參與調控種子發育的研究仍鮮見報道。前期研究中,發現ZmBES1/BZR1-5轉錄因子靶基因可能參與調控種子大小[30]。【擬解決的關鍵問題】本研究從B73克隆,并對其序列特征、表達模式、轉錄激活活性、亞細胞定位、調控籽粒發育的功能及調控靶基因進行分析,以期為后續深入研究的功能與調控機制提供參考依據。
1 材料與方法
1.1 材料準備
試驗材料為玉米自交系B73,由四川農業大學玉米研究所保存。選取飽滿的B73種子直接播種至大田,常規管理。待其進入生殖生長階段,進行人工授粉自交,并取授粉前的雄穗、花絲、苞葉、莖、根,以及灌漿后10、15和25 d的籽粒,經液氮速凍并快速研磨至粉末狀,裝于1.5 mL離心管中,-80℃保存備用,每個組織設置3個生物學重復。
1.2 生物信息學分析
從MaizeGDB(https://maizegdb.org/)網站獲取(Zm00001d010676)序列、編碼序列及編碼的氨基酸序列,利用在線工具GSDS(http://gsds. gao-lab.org/)進行結構可視化分析。同時,從NCBI(https://www.ncbi.nlm.nih.gov/)下載擬南芥、水稻和高粱等ERF氨基酸序列,利用DNAMAN軟件進行氨基酸序列比對。
1.3 ZmEREB93的擴增
采用RNA提取試劑盒(大連寶生物工程有限公司)提取上述各樣品總RNA,用cDNA反轉錄試劑盒(北京全式金生物技術有限公司)將其反轉錄成cDNA。同時,利用DNA提取試劑盒提取上述混合組織樣品的基因組DNA(gDNA)。根據的開放閱讀框(open reading frame,ORF)序列,采用Primer5設計擴增ORF序列引物(上游引物F:5′-ATGCAGCAGATGGAGTGGGACA-3′,下游引物R:5′-CGGAGACAGCGAGCCGAACAGC-3′)。分別以cDNA和gDNA為模板,用PrimeSTAR高保真DNA聚合酶進行PCR擴增,擴增體系為2×PrimeSTAR Buffer(Mg2+)12.5 μL、dNTPmix 2.0 μL、上游引物0.5 μL、下游引物0.5 μL、模板0.5 μL、PrimeSTAR 0. 25 μL和ddH2O 8.72 μL。擴增程序為94℃ 3 min,98℃ 10 s,66℃ 10 s,72℃ 50 s,35個循環;72℃ 5 min,12℃保存。產物經2%瓊脂糖凝膠電泳檢測,回收,并與載體pMD19-T連接,轉化大腸桿菌,送至上海生工生物工程股份有限公司測序。
1.4 qRT-PCR分析
采用NCBI網站在線引物設計軟件Primer-BLAST(https://blast.ncbi.nlm.nih.gov)設計的qRT-PCR引物(上游引物為93-F:5′-CCCTGGCACGT CGTCTATCT-3′,下游引物為93-R:5′-TCGTTGAGGT CGAACGGGAG-3′),擴增片段長185 bp。以玉米為內參基因(上游引物為EF-1a-F:5′-TGGGC CTACTGGTCTTACTACTGA-3′,下游引物為EF-1a- R:5′-ACATACCCACGCTTCAGATCCT-3′),擴增片段長135 bp。采用SYBR green法在CFX96TMReal- Time System進行qRT-PCR。qRT-PCR反應體系及程序按照SYBR Greensuper Mix試劑盒(北京全式金生物技術有限公司)說明,分別設置3個生物學和技術重復。采用2-ΔΔCT法計算在各組織中的相對表達量。
1.5 轉錄激活活性分析
采用Primer5設計的ORF擴增引物,根據同源重組酶及選取的限制性內切酶位點要求,在上下游引物分別引入pGBKT7多克隆位點側翼序列(上游引物為93-BF:5′-aggccgaattcccggggatccATGCA GCAGATGGAGTGGGA-3′,下游引物為93-BR:5′- atgcggccgctgcaggtcgacCGGAGACAGCGAGCCGAA-3′。小寫字母表示pGBKT7載體上的序列)。PCR擴增體系、程序及純化回收參照1.3方法。采用HⅠ和Ⅰ限制性內切酶切割pGBKT7載體,使其線性化并純化回收。使用同源重組試劑盒ClonExpress II One Step Cloning Kit(南京諾唯贊生物科技股份有限公司)構建pGBKT7-重組質粒,轉化酵母AH109菌株。前期證實玉米ZmBES1/BZR1轉錄因子具有轉錄激活活性,以轉化pGBKT7-ZmBES1/BZR1作為陽性對照,以轉化pGBKT7空載體作為陰性對照。將轉化后的菌液涂布在單缺SD/-Trp和三缺SD/-Trp/-His/ -Ade+X-α-gal的培養基上,28℃倒置培養3 d,觀察菌落顏色及生長狀況。
1.6 亞細胞定位
亞細胞定位使用植物表達載體pCAMBIA2300,同1.5引物設計方法,分別設計特異PCR引物(上游引物為93-GFPf:5′-ggacagggtacccggggatcc ATGCAGCAGATGGAGTGGGA-3′,下游引物去掉終止密碼子TGA,引物為93-GFPr:5′-ggtactagtgtcga ctctagaCGGAGACAGCGAGCCGAA-3′)。參考1.3進行PCR擴增及擴增產物純化回收。同1.5載體構建方法,用HⅠ和Ⅰ限制性內切酶切割pCAMBIA2300-載體,使其線性化并純化回收,構建pCAMBIA2300-表達載體。參考Sun等[31]方法,將其轉入GV3101農桿菌感受態,通過煙草注射的方法將其轉化至本氏煙草中,正常培養36—48 h,于激光共聚焦顯微鏡下觀察并拍照。
1.7 轉基因擬南芥創制及表型分析
將上述pCAMBIA2300-表達載體轉化GV3101農桿菌感受態,采用花絮浸染法轉化擬南芥哥倫比亞生態型Col-0。收獲T0代種子,播種至含50 mg·L-1卡那霉素的1/2 MS培養基,并于光照培養室培養2周,篩選陽性轉化株系。每個株系獨立收種,用同樣方法篩選陽性株系至T3代純合株系,并進行PCR檢測,于激光共聚焦顯微鏡下觀察熒光以確定目的基因是否表達。選取2個有目的基因插入且成功表達的純合株系與野生型對照種植于相同條件下,直至成熟收種。每個株系隨機取約1 000粒種子經1/10000天平進行稱重,并用徠卡普通光學顯微鏡DM500于40倍物鏡下拍照,測量每個株系種子的長和寬。每個株系種植30棵幼苗,并重復3次。
1.8 Chip-seq分析
體外Chip試驗使用pCold-TF載體,同1.5引物設計方法,分別設計特異PCR引物(上游引物為93-Cf:5′-ctcggtaccctcgagggatccATGCAGCAGATGGA GTGGGA-3′;下游引物去掉終止密碼子TGA,引物為93-Cr:5′-ctatctagactgcaggtcgacCGGAGACAGCGAG CCGAA-3′)。參考方法1.3進行PCR擴增及擴增產物純化回收。同1.5載體構建方法,用HⅠ和Ⅰ限制性內切酶切割pCold-TF載體,使其線性化并純化回收,構建pCold表達載體,使與His標簽融合。隨后,將其轉化至大腸桿菌菌株Rosetta(DE3),在含有50 mg·mL-1氨芐青霉素的LB液體培養基培養OD600至0.6。參考Sun等[30]方法,用0.5 mol·L-1異丙基-β-D-硫代半乳糖苷(IPTG)誘導表達ZmEREB93蛋白,使用Ni-TED 1 mL SefinoseTM柱(上海生工生物工程股份有限公司)純化ZmEREB93蛋白。隨后,提取B73總DNA,超聲波破碎為100—500 bp片段,并用乙醇沉淀純化DNA。將純化后DNA片段與帶His標簽的ZmEREB93蛋白加入孵化液(50 mmol·L-1Tris、1 mmol·L-1EDTA、100 mmol·L-1KCl、0.1% Triton X-100和1 mmol·L-1DTT,pH=7.0),4℃過夜。用孵育緩沖液洗滌3次,加入100 μL 5 mol·L-1NaCl,在65℃孵育2 h,解交聯His-ZmEREB93和DNA片段。隨后,提取DNA片段,并送至中國諾沃金公司進行Illumina測序,測序結果用于候選基因分析。
1.9 共表達分析
參考馮文奇等[32]方法,從MaizeGDB數據庫(https://download.maizegdb.org/)下載B73轉錄組數據[33],利用R語言編寫程序,將與玉米其他基因各組織中表達數據(FPKM值)進行相關性分析,選擇相關系數大于0.7或者小于-0.7的基因作為候選目標。結合Chip-seq和共表達分析結果,篩選兩方面共有的基因,并對這些基因起始密碼子(ATG)上游2 000 bp序列進行分析,確認其啟動子區域是否包含ERF結合基序GCC-box或DRE/CRT core元件,以鑒定ZmEREB93轉錄因子的候選靶基因。
1.10 酵母單雜
同1.5方法設計擴增候選靶基因啟動子的特異PCR引物(分別為016pf:5′-tttgatattggatcggaattcTGTCA ACGTCGATCCGATGG-3′,016pr:5′-atacagagcacatgcc tcgagGGCTCCTCGTGCGCCTGG-3′;611pf:5′-tttgatatt ggatcggaattcCTATGACGACGGCGGCTCT-3′,611pr:5′-atacagagcacatgcctcgagCGGCGGCCGCGCCCGTCG-3′;488pf:5′-tttgatattggatcggaattcACAAACACAGGCA GAGAGGCA-3′,488pr:5′-atacagagcacatgcctcgagCGGT GAGTCGCGCGGCGC-3′;991pf:5′-tttgatattggatcggaa ttcACACACAGGTGGCTTTTCAAAGG-3′,991pr:5′-atacagagcacatgcctcgagGGAATAAGTTGGAGGAAAGGAAGC-3′;的ORF擴增引物為93f:5′-gattatgcctctcccgaattcATGCAGCAGATGGAGTGGGA-3′,93r:5′-agaagtccaaagcttctcgagCGGAGACAGCGA GCCGAA-3′)。參考Xiong等[34]方法,實施酵母單雜交試驗。將的ORF構建至pJG4-5載體,靶基因啟動子構建至pLacZi2u載體。利用酵母轉化試劑盒(北京酷來搏科技有限公司),將已構建pLacZi2u啟動子載體分別與pJG4-5空載體、pJG4-5與pLacZi2u啟動子載體共同轉化EYG48酵母感受態細胞(上海昂羽生物技術有限公司)。吸取2 μL轉化后的菌液點至SD/-Trp/-Ura缺陷型培養基28℃培養2 d篩選陽性轉化子,培養至菌斑2—3 mm。隨后,挑取菌落于5 mL EP管過夜培養,取2 μL點于SD/-Trp/-Ura/X-gal(含20% Galactose+20% Raffiose+ 10 x Buffer salt)的缺陷型培養基28℃培養2 d并觀察菌落生長情況與顯色狀態。
2 結果
2.1 ZmEREB93的序列特征
生物信息學分析結果表明,長618 bp,含一個外顯子,無內含子和非編碼區,編碼205個氨基酸。ZmEREB93蛋白分子量21.62 kD,等電點10.24,親水指數-0.33(<0),穩定系數43(>40),無跨膜區,預測定位于細胞核。其二級結構含19.02% α螺旋,8.29%β折疊,51.22%無規則卷曲。以上結果表明,ZmEREB93蛋白是一個不穩定的親水蛋白。
以B73的cDNA和gDNA作模板,擴增的ORF,電泳結果顯示,擴增產物大小一致且均位于500—700 bp(圖1)。測序結果證實,Z的ORF長618 bp,無內含子。通過多序列比對發現,ZmEREB93與其他物種來源的ERF12具有較高的同源性,均含有一個高度保守的AP2結構域,屬于ERF亞家族(圖2)。
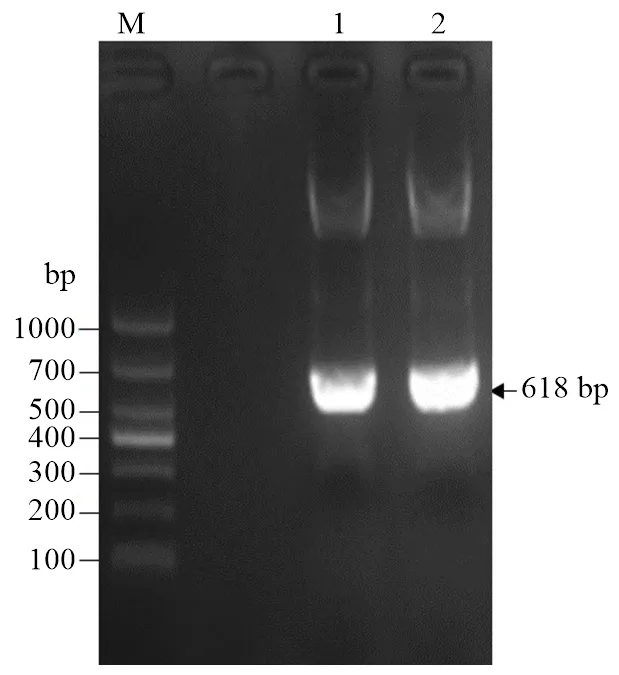
M:1000 bp DNA標準;1和2:分別以cDNA和gDNA為模板擴增
2.2 ZmEREB93在種子中的特異性表達
通過對玉米在不同組織中的qRT- PCR分析,結果表明,在授粉后15和25 d的種子中表達量較高,其中,在授粉后25 d的種子中表達量達到授粉后15 d種子中表達量的11倍左右。同時,成熟期的莖和根中也有表達,但是表達量較低,在雄穗、花絲和苞葉中無表達(圖3)。
2.3 ZmEREB93無轉錄激活活性
為鑒定ZmEREB93轉錄激活活性,將其ORF構建至pGBKT7載體,以pGBKT7為陰性對照,pGBKT7為陽性對照,分別轉化AH109酵母感受態細胞。結果(圖4)表明,各載體轉化菌株在SD/-Trp缺陷型培養基上均可正常生長,說明質粒轉化成功。隨后,將陽性轉化子點至SD/-Trp/-His/ -Ade/X-α-gal三缺培養基上,轉化pGBKT7的陽性對照菌株可正常生長且變藍,而轉化pGBKT7載體的陰性對照與pGBKT7菌株均不能生長。說明ZmEREB93在酵母細胞中以單體形式無轉錄激活活性。
2.4 ZmEREB93定位于細胞核
將和質粒轉化農桿菌后,并注射煙草葉片進行瞬時表達。激光共聚焦顯微鏡成像結果顯示,轉化空載體的煙草葉片中,綠色熒光分布在整個細胞中。但是,在轉化載體的煙草葉片中,熒光僅分布在細胞核(圖5),該結果證實ZmEREB93轉錄因子位于細胞核發揮功能。
2.5 ZmEREB93負調控轉基因擬南芥種子大小
對純合轉基因株系進行PCR和熒光檢測(圖6),在轉基因株系L#1和L#2中可擴增到目的基因且檢測到熒光,而野生型對照中不能檢測到目的基因,說明插入擬南芥基因組且能夠表達。對各株系的種子表型進行分析表明,與野生型對照相比,轉基因株系L#1的粒長和粒寬均顯著降低,轉基因株系L#2種子的粒長無顯著變化,但其粒寬顯著降低。同時,L#1和L#2株系種子的千粒重均顯著低于野生型株系(圖7)。以上結果表明,負調控轉基因擬南芥種子大小。
2.6 ZmEREB93靶基因
體外Chip-seq結果顯示,通過Chip試驗得到1 395個DNA片段,-value(-log10)值均大于2。進一步基因序列比對及GO注釋發現,共注釋1 226個基因。共表達分析結果顯示,玉米中有100個基因的表達與表達相關性較高,相關系數大于0.7。結合兩方面結果,篩選到共有基因4個,分別為、、和。隨后,啟動子元件分析發現,這4個基因啟動子區均存在DRE/CRT core元件,無GCC-box(表1)。初步推測這些候選基因是ZmEREB93轉錄因子調控的靶基因。

AtERF12:擬南芥,AT1G28360;SbERF12:高粱,SORBI_3009G184300;OsERF12:水稻,OS05G0497200;SiERF12:谷子,XP_004970252.1;AlERF12:馬絆草,QYY53011.1;ZjERF12:大棗,XP_015890508.1;DzERF12:榴蓮,XP_022777342.1;CiERF12:山核桃,XP_042944848.1;CqERF12:藜麥,XP_021740383.1;ItERF12:三裂葉薯,XP_031112454.1
Y1H試驗結果表明,在SD/-Trp/-Ura二缺培養基上,所有轉化后的酵母均可正常生長形成菌落。但是,在SD/-Trp/-Ura/X-gal培養基上,只有共轉化ZmEREB93與啟動子載體的菌落呈現深藍色,說明ZmEREB93可以直接結合的啟動子(圖8-a)。進一步構建啟動子截短片段并實施Y1H,結果發現,ZmEREB93結合啟動子的-1 621— -621 bp區段,該片段有3個DRE/CRT core元件(圖8-b)。說明是ZmEREB93的靶基因。

圖3 ZmEREB93的表達模式分析

圖4 ZmEREB93轉錄激活活性分析
3 討論
3.1 ZmEREB93的特征
在SUN等[30]前期研究中發現,玉米ZmBES1/ BZR1-5轉錄因子正向調控種子大小,作為其靶基因且受其負調控。因此,推測可能參與調控籽粒發育過程。本研究成功克隆到,發現其僅有一個外顯子,無內含子(圖1),在玉米種子中特異性表達,尤其在授粉后25 d的玉米種子中高表達(圖3),表明其可能在玉米籽粒發育中起重要作用。ZmEREB93氨基酸序列僅有1個高度保守的AP2典型的特征結構域(圖2),屬于玉米ERF 家族成員[37]。進一步研究證實,ZmEREB93蛋白位于細胞核但無轉錄激活活性(圖4—圖5),可能的原因是ZmEREB93在酵母細胞中以單體形式存在不能發揮轉錄激活活性。已有研究證實,植物中一些轉錄因子單獨存在時無法轉錄激活下游靶基因,只有形成二聚體或與其他蛋白互作后調控下游的表達。例如,玉米轉錄因子ZmBES1/BZR1-5單體無轉錄激活活性,但會自身形成二聚體[30]。水稻OsERF115轉錄因子與OsNF-YB1形成蛋白復合體進而發揮功能[38]。
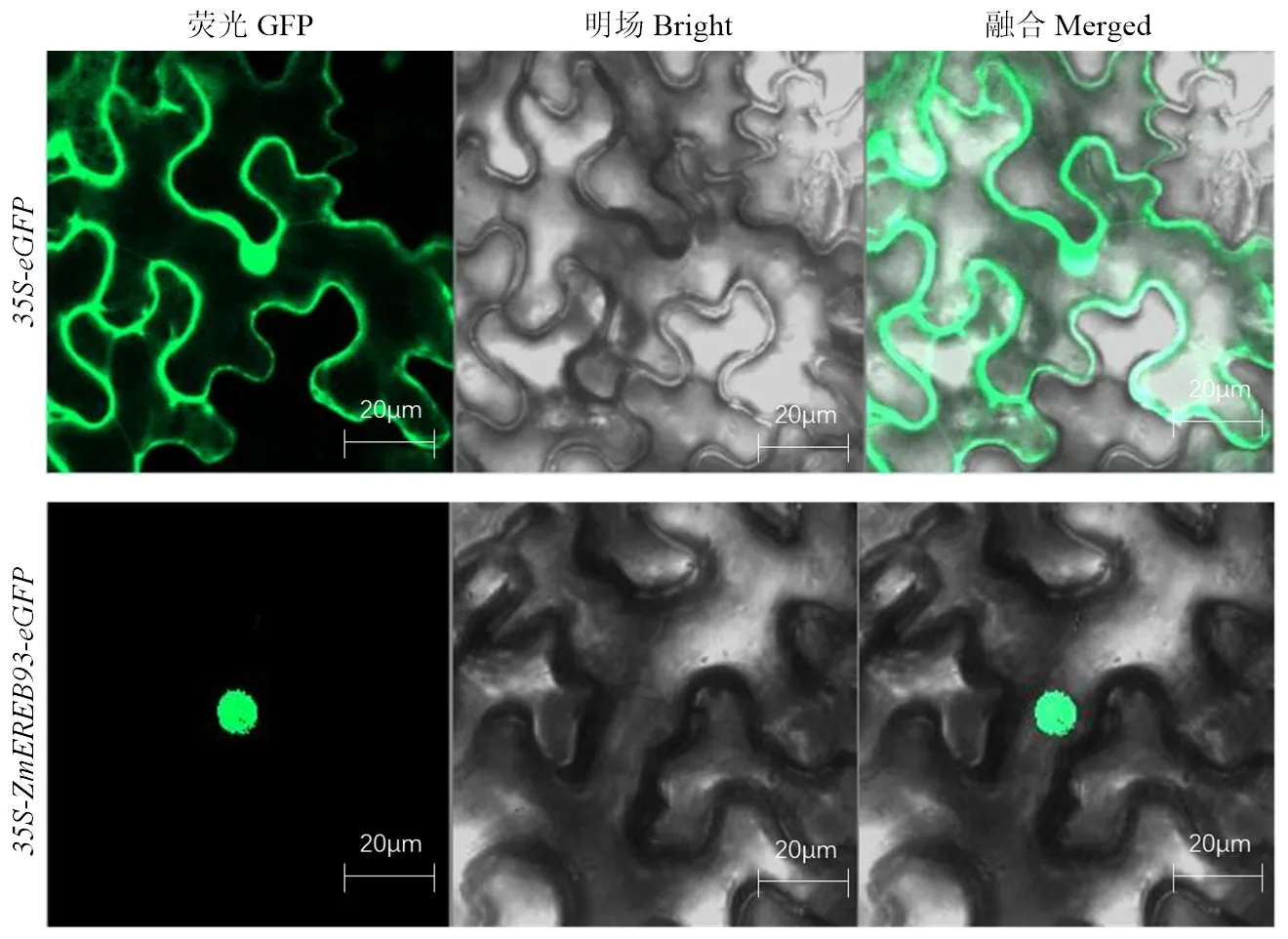
圖5 ZmEREB93的亞細胞定位
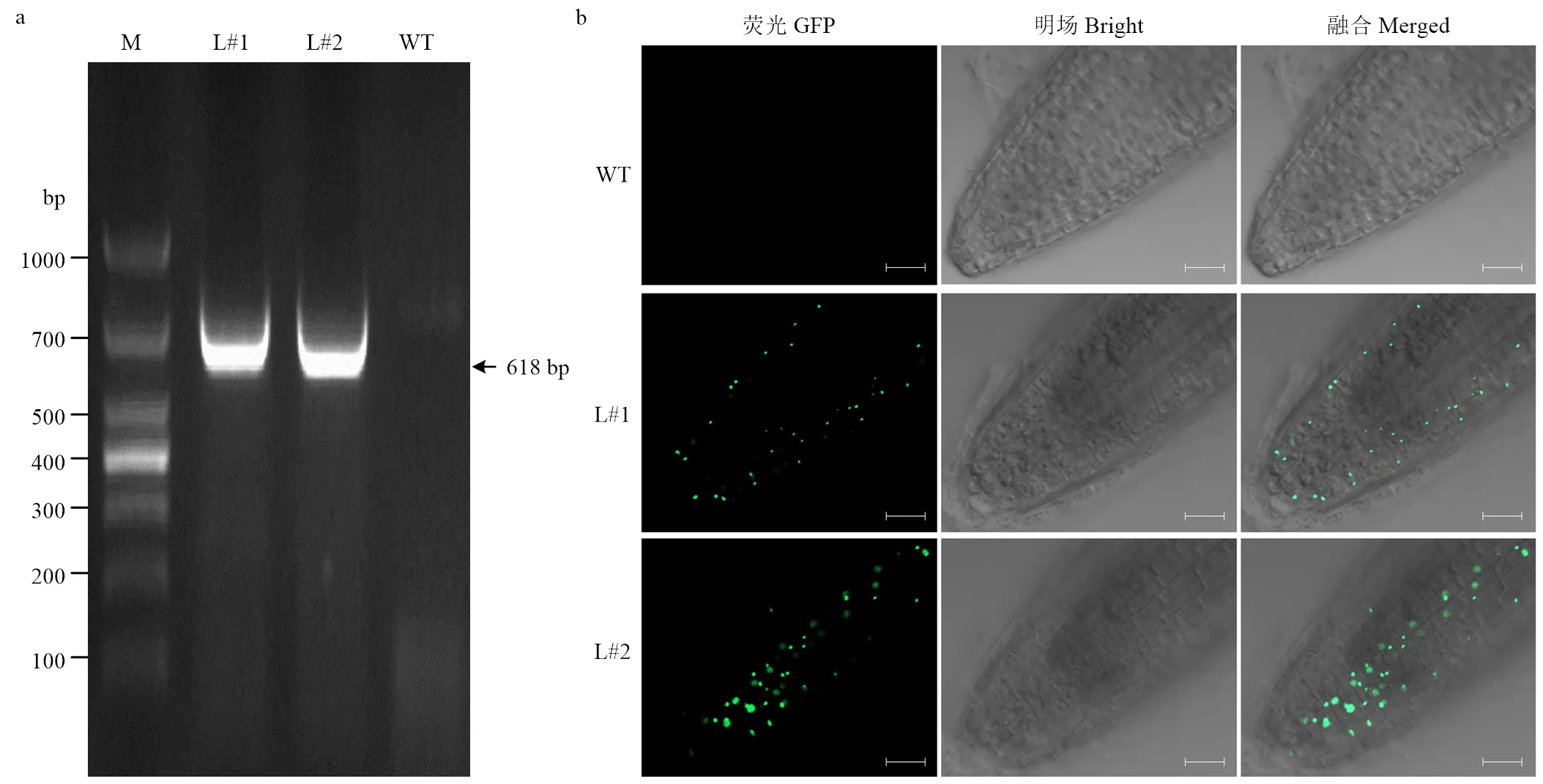
a:各株系PCR檢測;b:各株系熒光檢測;M:1000 bp DNA標準;L#1和L#2:分別為轉化ZmEREB93的擬南芥株系;WT:野生型。下同
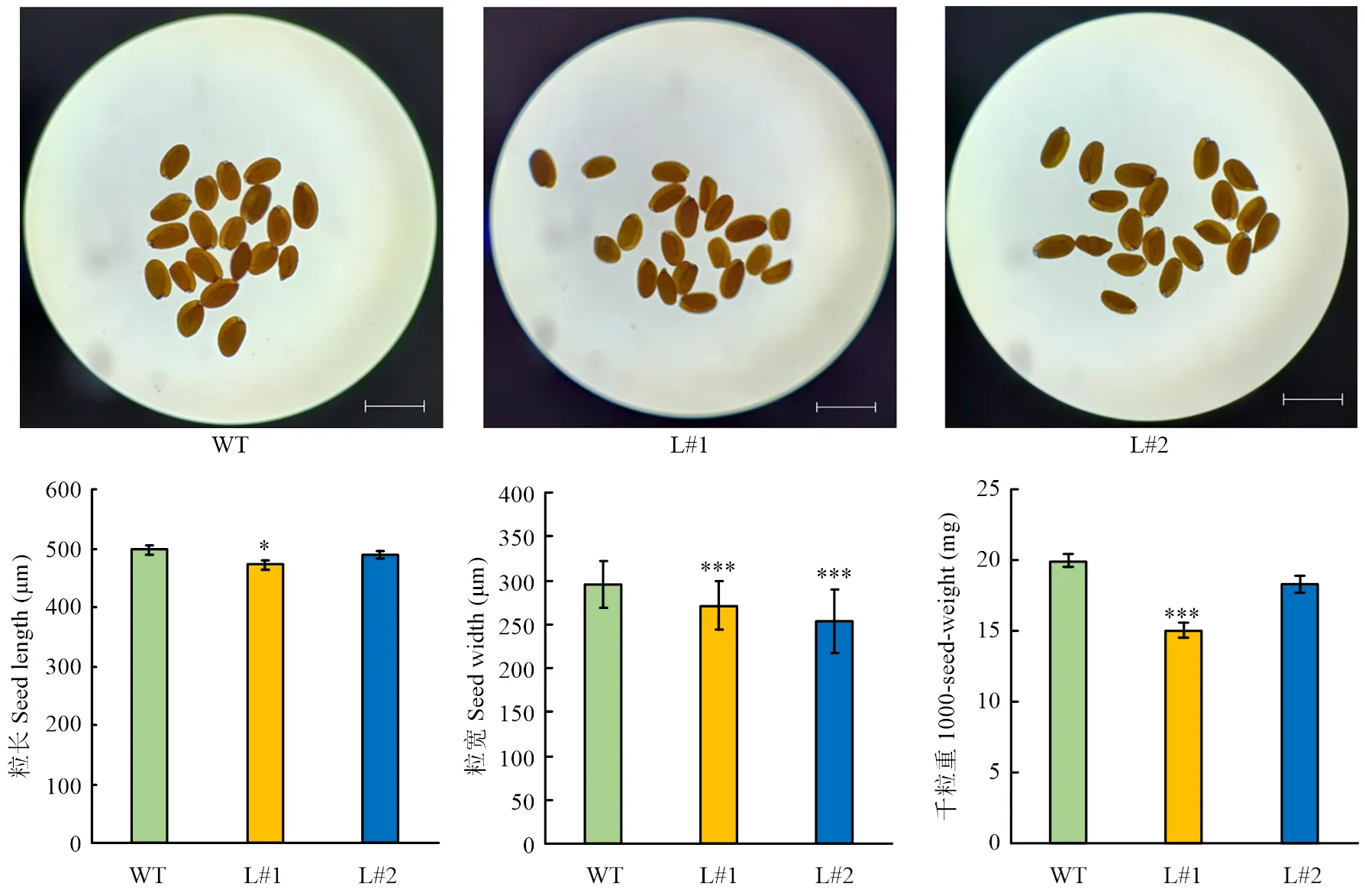
*:p<0.05;**:p<0.01;***:p<0.0001

a:Y1H驗證ZmEREB93與Zm00001d013611、Zm00001d006016、Zm00001d027448、Zm00001d039991啟動子結合。P016、P488、P991、P611表示Zm00001d006016、Zm00001d027448、Zm00001d039991、Zm00001d013611啟動子;b:Y1H驗證ZmEREB93與Zm00001d013611啟動子不同片段結合。P1、P2、P3表示不同片段啟動子;△表示DRE/CRT core元件
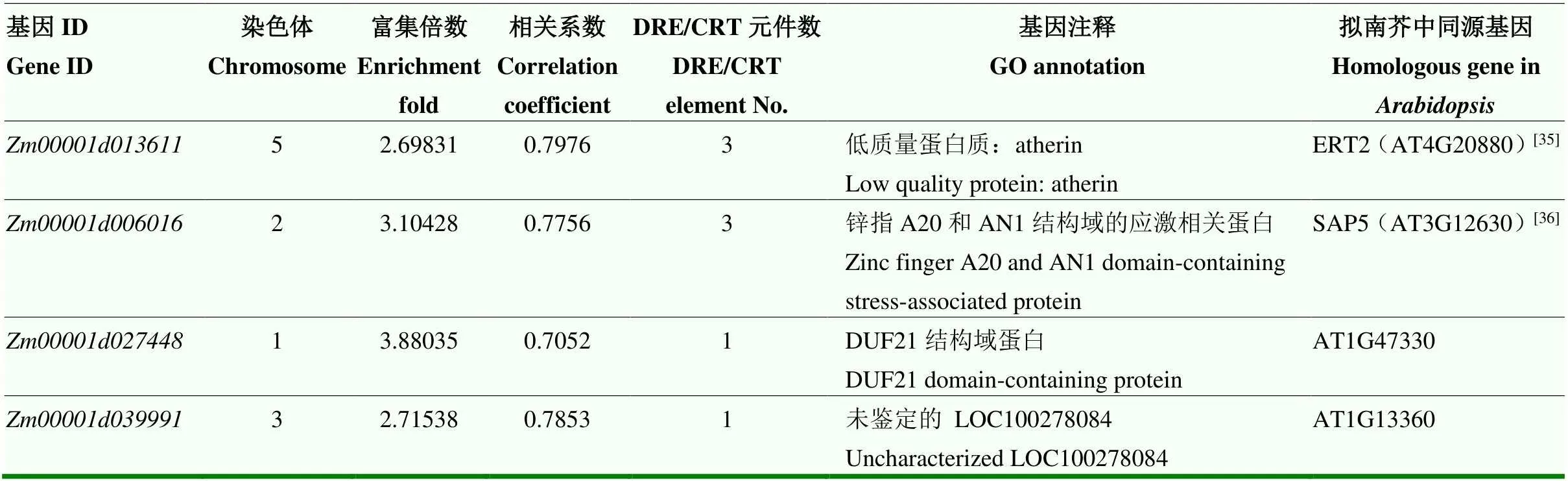
表1 ZmEREB93候選靶基因
3.2 ERF亞類轉錄因子調控植物生長發育
植物中,AP2轉錄因子家族的ERF亞類成員主要參與調控初級次生代謝、激素信號轉導、生長發育及逆境應答等過程[17-20]。玉米中,AP2轉錄因子的AP2亞族成員主要調控生長發育過程[22-26],而ERF亞類主要參與調控逆境應答過程[22, 27-28]。但是,研究人員也發現,植物中ERF亞類成員作為轉錄抑制因子也參與調控籽粒發育過程。例如,菊花CmERF12與花和胚發育相關蛋白CmSUF4相互作用,進而抑制其靶基因(EGG CELL1),最終負調控胚的發育[39]。水稻OsERF115與OsNF-YB1形成蛋白復合體參與調控水稻籽粒灌漿和胚乳發育[38]。近期研究還發現,OsERF115通過抑制籽粒大小負調控因子OsGW2和OsGS6的啟動子活性,進而調控籽粒大小和粒重[38, 40]。在本研究中,玉米在擬南芥中的同源基因參與調控花的發育[41],但是,將玉米轉化擬南芥過量表達,轉基因株系的種子較對照顯著變小(圖7),說明ZmEREB93轉錄因子抑制種子發育。
3.3 ZmEREB93調控的下游途徑
眾所周知,ERF亞類成員通過結合下游基因啟動子區的GCC-box或DRE/CRT core順式作用元件調控靶基因表達[7-9]。結合chip-seq與共表達分析篩選到4個候選基因可能作為ZmEREB93的靶基因,且它們的啟動子區均有DRE/CRT core元件(表1)。其中,和在擬南芥中的同源基因分別為和,表達無組織特異性且受乙烯誘導,則參與逆境脅迫應答[34-35]。通過Y1H進一步證實,ZmEREB93可直接結合啟動子的-1 621—-621 bp區段,該片段有3個DRE/CRT core元件(圖8)。雖然,其余候選靶基因在Y1H試驗中未得到驗證,但不能排除其真實性,由于酵母表達系統存在假陽性和假陰性,仍需體內試驗驗證。同時,及均為未知基因,也可能調控籽粒發育。而且,在Chip-seq中鑒定到8個啟動子上具有DRE/CRT順式作用元件的PPR家族基因,而PPR成員通過影響線粒體發育,進而調控玉米的內核發育以及籽粒發育[41]。PPR突變通常造成線粒體的形態與功能缺陷,最終導致玉米籽粒發育的嚴重缺陷[42-43]。因此,在后續研究中,將通過轉基因或敲除試驗重點評價在玉米籽粒發育中的功能,并在體內進行其靶基因的驗證及功能研究。
4 結論
從玉米B73中克隆到,長度為618 bp,不含內含子,編碼205個氨基酸,有一個高度保守AP2結構域,屬于AP2家族的ERF亞類。ZmEREB93在授粉后25 d的種子中高表達,定位于細胞核,且負調控轉基因擬南芥的種子大小。為ZmEREB93調控的候選靶基因。
[1] DOLL N M, DEPEGE-FARGEIX N, ROGOWSKY P M, WIDIEZ T. Signaling in early maize kernel development. Molecular Plant, 2017, 10(3): 375-388.
[2] 高春艷, 吳芮, 袁玉, 劉同玥, 任莉萍. 植物AP2/ERF轉錄因子及其在非生物脅迫應答中的作用. 江漢大學學報(自然科學版), 2017, 45(3): 236-240.
GAO C Y, WU R, YUAN Y, LIU T Y, REN L P. Function of AP2/ERF transcription factors in plant tolerance to abiotic stress. Journal of Jianghan University (Natural Science Education), 2017, 45(3): 236-240. (in Chinese)
[3] JOFUKU K D, DEN BOER B G, VAN MONTAGU M, OKAMURO J K. Control offlower and seed development by the homeotic gene APETALA2. The Plant Cell, 1994, 6(9): 1211-1225.
[4] CHAKRAVARTHY S, TUORI R P, D’ASCENZO M D, FOBERT P R, DESPRES C, MARTIN G B. The tomato transcription factor Pti4 regulates defense-related gene expression via GCC box and non-GCC box cis elements. The Plant Cell, 2003, 15(12): 3033-3050.
[5] LICAUSI F, OHME-TAKAGI M, PERATA P. APETALA2/ethylene responsive factor (AP2/ERF) transcription factors: mediators of stress responses and developmental programs. New Phytologist, 2013, 199(3): 639-649.
[6] Wen K. The important role of AP2 functional genes in plant floral development. Biotechnology Bulletin, 2010, 20(2): 1-7.
[7] 豐錦, 陳信波. 抗逆相關AP2/EREBP轉錄因子研究進展. 生物技術通報, 2011(7): 1-7.
Feng J, Chen X B. Research progress of AP2/EREBP transcription factors in stress tolerance. Biotechnology Bulletin, 2011(7): 1-7. (in Chinese)
[8] RIECHMANN J L, MEYEROWITZ E M. The AP2/EREBP family of plant transcription factors. Biological Chemistry, 1998, 379(6): 633-646.
[9] RAO G, SUI J, ZENG Y, HE C, ZHANG J. Genome-wide analysis of the AP2/ERF gene family in. FEBS Open Bio, 2015, 24(5): 132-137.
[10] SHIGYO M, HASEBE M, ITO M. Molecular evolution of the AP2 subfamily. Gene, 2006, 366(2): 256-265.
[11] SHOESMITH J R, SOLOMON C U, YANG X, WILKINSON L G, SHELDRICK S, EIJDEN E V, COUWENBERG S, PUGH M L, ESKAN M, STEPHENS J, BARAKATE A, DREA S, HOUSTON K, TUCKER M R, MCKIM S M. APETALA2 functions as a temporal factor together with BLADE-ON-PETIOLE2 and MADS29 to control flower and grain development in barley. Development, 2021, 148(5): 1-5.
[12] JIANG W, ZHANG X, SONG X, YANG J, PANG Y. Genome-wide identification and characterization of APETALA2/ethylene-responsive element binding factor superfamily genes in soybean seed development. Frontiers in Plant Science, 2020, 11: 566647.
[13] LEI M, LI Z Y, WANG J B, FU Y L, XU L. Ectopic expression of theAPETALA2 gene AfAP2-2 reduces seed size and delays flowering in. Plant Physiology and Biochemistry, 2019, 139: 642-650.
[14] JOFUKU K D, OMIDYAR P K, GEE Z, OKAMURO J K. Control of seed mass and seed yield by the floral homeotic gene APETALA2. Proceedings of the National Academy of Sciences of the USA, 2005, 102(8): 3117-3122.
[15] JIANG L, MA X, ZHAO S, TANG Y, LIU F, GU P, FU Y, ZHU Z, CAI H, SUN C, TAN L. The APETALA2-Like transcription factor SUPERNUMERARY BRACT controls rice seed shattering and seed size. The Plant Cell, 2019, 31(1): 17-36.
[16] LUO C, WANG S, NING K, CHEN Z, WANG Y, YANG J, QI M, WANG Q. The APETALA2 transcription factor LsAP2 regulates seed shape in lettuce.Journal of Experimental Botany, 2021, 72(7): 2463-2476.
[17] CHEN Y, FENG P, TANG B, HU Z, XIE Q, ZHOU S, CHEN G. The AP2/ERF transcription factor SlERF.F5 functions in leaf senescence in tomato. Plant Cell Reports, 2022, 41: 1181-1195.
[18] RITONGA F N, NGATIA J N, WANG Y, KHOSO M A, FAROOQ U, CHEN S. AP2/ERF, an important cold stress-related transcription factor family in plants: A review. Physiology and Molecular Biology of Plants, 2021, 27(9): 1953-1968.
[19] YU Z X, LI J X, YANG C Q, HU W L, WANG L J, CHEN X Y. The jasmonate-responsive AP2/ERF transcription factors AaERF1 and AaERF2 positively regulate artemisinin biosynthesis inL.Molecular Plant, 2012, 5(2): 353-365.
[20] XU S, HOU H, WU Z, ZHAO J, ZHANG F, TENG R, CHEN F, TENG N.embryo development is negatively affected by a novel ERF transcription factor, CmERF12. Journal of Experimental Botany, 2022, 73(1): 197-212.
[21] FENG C Z, CHEN Y, WANG C, KONG Y H, WU W H, CHEN Y F.RAV1 transcription factor, phosphorylated by SnRK2 kinases, regulates the expression of ABI3, ABI4, and ABI5 during seed germination and early seedling development. The Plant Journal, 2014, 80(4): 654-668.
[22] ZHANG J, LIAO J, LING Q, XI Y, QIAN Y. Genome-wide identification and expression profiling analysis of maize AP2/ERF superfamily genes reveal essential roles in abiotic stress tolerance. BMC Genomics, 2022, 23(1): 125.
[23] CHUCK G, MEELEY R, HAKE S. Floral meristem initiation and meristem cell fate are regulated by the maize AP2 genes ids1 and sid1.Development, 2008, 135(18): 3013-3019.
[24] LIU W Y, LIN H H, YU C P, CHANG C K, CHEN H J, LIN J J, LU M J, TU S L, SHIU S H, WU S H, KU M S B, LI W H. Maize ANT1 modulates vascular development, chloroplast development, photosynthesis, and plant growth. Proceedings of the National Academy of Sciences, USA2020, 117(35): 21747-21756.
[25] LID S E, AL R H, KREKLING T, MEELEY R B, RANCH J, OPSAHL-FERSTAD H G, OLSEN O A. The maize disorganized aleurone layer 1 and 2 (dil1, dil2) mutants lack control of the mitotic division plane in the aleurone layer of developing endosperm. Planta, 2004, 218(3): 370-378.
[26] LI J, CHEN F, LI Y, LI P, WANG Y, MI G, YUAN L. ZmRAP2.7, an AP2 transcription factor, is involved in maize brace roots development.Frontiers in Plant Science, 2019, 10: 820.
[27] FU J, ZHU C, WANG C, LIU L, SHEN Q, XU D, WANG Q. Maize transcription factor ZmEREB20 enhanced salt tolerance in transgenic. Plant Physiology and Biochemistry, 2021, 159:257-267.
[28] ZANG Z, WANG Z, ZHAO F, YANG W, CI J, REN X, JIANG L, YANG W. Maize ethylene response factoris required for resistance to. Frontiers in Plant Science, 2021, 12: 630413.
[29] LI S, WANG H, LI F, CHEN Z, LI X, ZHU L, WANG G, YU J, HUANG D, LANG Z. The maize transcription factor EREB58 mediates the jasmonate-induced production of sesquiterpene volatiles.ThePlant Journal, 2015, 84(2): 296-308.
[30] SUN F, DING L, FENG W, CAO Y, LU F, YANG Q, LI W, LU Y, SHABEK N, FU F, YU H. Maize transcription factor ZmBES1/ BZR1-5 positively regulates kernel size. Journal of Experimental Botany, 2021, 72(5): 1714-1726.
[31] SUN F A, YU H Q, QU J T, CAO Y, DING L, FENG W Q, MUHAMMAD H B K, LI W C, FU F L. Maize ZmBES1/BZR1-5 decreases ABA sensitivity and confers tolerance to osmotic stress in transgenic. International Journal of Molecular Sciences, 2020, 21(3): 996.
[32] 馮文奇, 孫福艾, 丁磊, 郭新, 李晚忱, 付鳳玲, 于好強. 玉米轉錄因子基因克隆及功能分析. 核農學報, 2020, 34(1): 17-25.
FENG W Q, SUN F A, DING L, GUO X, LI W C, FU F L, YU H Q. Cloning and function analysis ofgene in maize. Journal of Nuclear Agricultural Sciences, 2020, 34(1): 17-25. (in Chinese)
[33] WALLEY J W, SARTOR R C, SHEN Z, SCHMITZ R J, WU K J, URICH M A, NERY J R, SMITH L G, SCHNABLE J C, ECKER J R, BRIGGS S P. Integration of omic networks in a developmental atlas of maize. Science, 2016, 353(6301): 814-818.
[34] XIONG F, ZHANG B K, LIU H H, WEI G, WU J H, WU Y N, ZHANG Y, LI S. Transcriptional regulation of PLETHORA1 in the root meristem through an importin and its two. The Plant Cell, 2020, 32(12): 3812-3824.
[35] TRENTMANN S M. ERN1, a novel ethylene-regulated nuclear protein of. Plant Molecular Biology, 2000, 44(1): 11-25.
[36] KANG M, FOKAR M, ABDELMAGEED H, ALLEN R D.SAP5 functions as a positive regulator of stress responses and exhibits E3 ubiquitin ligase activity.Plant Molecular Biology, 2011, 75(4/5): 451-466.
[37] ZHANG J, LIAO J, LING Q, XI Y, QIAN Y. Genome-wide identification and expression profiling analysis of maize AP2/ERF superfamily genes reveal essential roles in abiotic stress tolerance. BMC Genomics, 2022, 23: 125.
[38] XU J J, ZHANG X F, XUE H W. Rice aleurone layer specific OsNF-YB1 regulates grain filling and endosperm development by interacting with an ERF transcription factor. Journal of Experimental Botany, 2016, 67(22): 6399-6411.
[39] XU S, HOU H, WU Z, ZHAO J, ZHANG F, TENG R, CHEN F, TENG N. Chrysanthemum embryo development is negatively affected by a novel ERF transcription factor, CmERF12. Journal of Experimental Botany, 2022, 73(1): 197-212.
[40] LIU C, MA T, YUAN D, ZHOU Y, LONG Y, LI Z, DONG Z, DUAN M, YU D, JING Y, BAI X, WANG Y, HOU Q, LIU S, ZHANG J S, CHEN S Y, LI D, LIU X, LI Z, WANG W, LI J, WEI X, MA B, WAN X. The OsEIL1-OsERF115-target gene regulatory module controls grain size and weight in rice. Plant Biotechnology Journal, 2022. Doi: 10.1111/pbi.13825.
[41] CHANDLER J W, WERR W. A phylogenetically conserved APETALA2/ETHYLENE RESPONSE FACTOR, ERF12, regulatesfloral development. Plant Molecular Biology, 2020, 102: 39-54.
[42] CHEN X, FENG F, QI W, XU L, YAO D, WANG Q, SONG R.encodes a PPR protein that affects cis-splicing of mitochondrial nad4 intron 1 and seed development in maize. Molecular Plant, 2017, 10(3): 427-441.
[43] CAI M, LI S, SUN F, SUN Q, ZHAO H, REN X, ZHAO Y, TAN B C, ZHANG Z, QIU F.encodes a mitochondrial PPR protein that affects the-splicing of nad2 intron 1 and seed development in maize. The Plant Journal, 2017, 91(1): 132-144.
Maize Transcription Factor ZmEREB93 negatively regulates kernel development
PANG HaoWan1, FU QianKun1, YANG QingQing1, ZHANG YuanYuan2, FU FengLing1, YU HaoQiang1
1Maize Research Institute, Sichuan Agricultural University, Chengdu 611130;2College of Life Science & Biotechnology, Mianyang Tearchers’ College, Mianyang 621000, Sichuan
【Objective】Maize, a kind of crucial crop, is widely used in food supply, livestock feed, and industry. AP2/EREBP (APETALA2/ethylene response element-binding protein) transcription factor (TF) plays an important role in plant growth, development, and stress response. Previous study showed thatmight regulate seed size as a target gene of ZmBES1/BZR1-5 TF.was cloned and used to analyze its expression pattern and function, which lays foundation to clarify the function and mechanism ofregulating seed size. 【Method】The full length ofwas cloned from maize inbred line B73 by PCR. The characters of nucleotide and amino acid sequences were analyzed by informatic methods. Subsequently, the tissue expression specificity ofwas analyzed via quantitative real time PCR (qRT-PCR). The expression vector in plant and yeast was constructed and used for subcellular localization and transcription activation assay, respectively.was transformed intomediated by agrobacterium transformation. The seed phenotype of transgenic lines was analyzed. Finally, the potential target genes of ZmEREB93 were screened by chromatin immunoprecipitation sequencing (Chip-seq) and co-expression analysis, and further confirmed by yeast one hybrid (Y1H). 【Result】Thegene was cloned by PCR. Sequence analysis showed thathad no intron and a 618 bp ORF, encoding 205 amino acids with a highly conserved AP2 domain and belongs to the ERF subclade of AP2 family. The results of qRT-PCR showed that thegene highly expressed in kernels of 15 and 25 days after pollination (DAP), and slightly expressed in stem and root, but did not express in tassel, silk and bract. The expression level ofwas the highest in 25 DAP kernels reached 11 times of that in 15 DAP kernels. The results of transcriptional activation and subcellular localization assay exhibited that ZmEREB93 protein had no transcriptional activation activity in yeast cells and was localized in the nucleus, respectively. Compared to wild type, the seeds of transgenic lines were significantly smaller and showed lower thousand-seed-weight. Chip-seq and co-expression analysis suggested that the,,andgenes were candidate target genes regulated by ZmEREB93 TF. The result of Y1H showed that ZmEREB93 directly bind topromoter. 【Conclusion】Maize ZmEREB93 TF specifically expressed in seeds and negatively regulated seed size.
maize; transcription factors; AP2/EREBP; grain

10.3864/j.issn.0578-1752.2022.19.001
2022-04-18;
2022-06-16
成都市科學技術局技術創新研發項目(2021-YF05-02024-SN)、四川省科學技術廳國際科技創新合作項目(2022YFH0067)
龐浩婉,E-mail:1421781500@qq.com。通信作者于好強,E-mail:yhq1801@sicau.edu.cn
(責任編輯 李莉)

