結(jié)直腸癌組織T 細胞受體組庫多樣性與患者臨床特征和具核梭形桿菌豐度的關(guān)聯(lián)分析
胡慕妮,季林華,張昕雨,沈超琴,洪 潔,陳豪燕
1.上海交通大學醫(yī)學院附屬仁濟醫(yī)院消化科,上海 200001;2.上海交通大學醫(yī)學院附屬仁濟醫(yī)院胃腸外科,上海 200127
結(jié)直腸癌(colorectal cancer,CRC)是全球第五大確診的惡性腫瘤,為癌癥相關(guān)死亡的第三大常見原因;據(jù)估計全球每年新增CRC病例超過10萬例[1]。CRC的發(fā)生和發(fā)展與遺傳、免疫、環(huán)境及腸道微生物等多種因素密切相關(guān)。研究[2]表明具核梭形桿菌(Fusobacterium nucleatum,F(xiàn)n)在CRC患者糞便及腫瘤組織中的豐度較正常人群明顯升高。同時Fn能夠?qū)е履c上皮細胞DNA損傷,誘發(fā)慢性炎癥反應(yīng),促進CRC的發(fā)生和發(fā)展[3-4]。CRC患者腫瘤組織中高豐度的Fn與患者不良的預后也密切相關(guān)[4]。研究CRC發(fā)生和發(fā)展過程中的分子變化,尋找與之相關(guān)的新型生物學標志物對CRC相關(guān)研究的開展具有重要意義。
隨著高通量技術(shù)的快速發(fā)展,多組學相關(guān)研究促進了腫瘤相關(guān)生物學標志物的發(fā)掘。既往研究[5]發(fā)現(xiàn)T 細胞受體(T cell receptor,TCR)組庫可以反映特定時間段內(nèi)機體內(nèi)T細胞的多樣性以及機體免疫系統(tǒng)應(yīng)對外界刺激應(yīng)答的能力。當患者受到某一抗原的刺激時,其相對應(yīng)的T細胞會呈現(xiàn)單克隆或寡克隆反應(yīng)性增生,導致TCR 組庫多樣性有所下降[6],這提示TCR 組庫多樣性可作為潛在的新型生物學標志物來評價機體的疾病狀態(tài)。
T 細胞受體測序(T cell receptor sequencing,TCR-seq)以T 細胞為研究對象,應(yīng)用多重聚合酶鏈式反應(yīng)或5'cDNA 末端快速擴增(rapid amplification of 5'cDNA end,5'RACE)技術(shù)擴增決定TCR 多樣性的互補決定區(qū)(complementary determining regions,CDR);該技術(shù)結(jié)合高通量測序技術(shù)常應(yīng)用于TCR 組庫多樣性的評估中[7]。2021 年,來自哈佛大學的X Shirley Liu 團隊開發(fā)的一種新的算法TRUST4[8],可以針對組織或者外周血的RNA 測序(RNA sequencing,RNA-seq)數(shù)據(jù)挖掘免疫組庫信息;其能 與SMART-seq (switching mechanism at 5'end of the RNA transcript sequencing)和10× Genomics 平臺兼容,可用于后續(xù)進一步的數(shù)據(jù)分析,有助于快速準確地評估TCR 序列信息,并深入研究免疫組庫與疾病間的聯(lián)系。TCR 組庫多樣性的分析主要在TCRα、β 鏈的基因水平(基因種類、頻率、克隆數(shù)量及基因組合的差異表達等)與氨基酸水平(TCR β 鏈CDR3氨基酸的種類、頻率、長度分布等)上開展[9],通常采用的指標包括香農(nóng)熵(Shannon entropy)、Chao1指數(shù)、Simpson 指數(shù)、Hill 值等[10-11]。香農(nóng)熵用于評估T細胞克隆的多樣性,通過估計克隆型的相對數(shù)目和每種克隆型的豐度或分布反映TCR 的變異程度;香農(nóng)熵值越高,提示樣本中T 細胞克隆的多樣性越高。較高的Chao1 指數(shù)和Hill 值以及更小的Simpson指數(shù)也可反映更高的TCR 多樣性。通過分析TCR 組庫多樣性的特點,能夠進一步了解機體免疫應(yīng)答狀態(tài),從而有助于評估疾病進展和治療效果[12]。
近年來,不少學者圍繞TCR 組庫多樣性、腫瘤狀態(tài)以及腫瘤免疫治療等相關(guān)問題展開研究,并揭示了其中的密切聯(lián)系,尤其在惡性黑色素細胞瘤、非小細胞肺癌等腫瘤中取得較多研究成果[13-14]。然而,TCR 組庫分析技術(shù)在CRC 中的相關(guān)研究和應(yīng)用較少。本研究利用TRUST4 算法分析53 例CRC 患者腫瘤組織及其匹配的癌周正常組織的RNA-seq 數(shù)據(jù),探究CRC 組織TCR 組庫多樣性與臨床特征以及腫瘤組織中Fn 豐度的相關(guān)性,以期為CRC 發(fā)生和發(fā)展機制研究提供參考。
1 對象與方法
1.1 研究對象
選擇2016 年7 月—2016 年12 月上海交通大學醫(yī)學院附屬仁濟醫(yī)院胃腸外科收治的63 例行CRC 根治術(shù)患者作為研究對象。所有病例均經(jīng)病理活檢確診。收集患者性別、年齡、腫瘤位置、腫瘤TNM 分期等臨床資料,獲取術(shù)中切除的腫瘤組織及其癌周正常黏膜組織樣本(距離腫瘤組織邊緣>5 cm)。
1.2 研究方法
1.2.1 組織處理 在組織離體0.5 h 內(nèi),使用生理鹽水對其進行沖洗,隨后放入裝有RNAlater 組織保存液的10 mL 離心管中。4 ℃冰箱過夜之后使用DEPC水洗去組織表面的RNAlater,然后轉(zhuǎn)入凍存管中,保存于-80 ℃冰箱內(nèi)。
1.2.2 RNA-seq以及16S rDNA測序 使用TRizol法提取63對新鮮CRC腫瘤組織及配對癌旁組織的總RNA,通過RNeasy Mini Column(Qiagen,美國)以及DNA酶進行純化,并通過Agilent 2100 Bioanalyzer進行質(zhì)量檢測。其中10對樣本的RNA完整性指標(RNA integrity number,RIN)低于6,未達到測序平臺的最低標準,予以排除。最終對質(zhì)量合格的53 對樣本進行深度RNA-seq,測序方案為雙端測序(pair-end 150 bp,PE150),每例樣品測序數(shù)據(jù)量為10 Gb。選取16S rDNA的特定序列進行高通量測序分析以估計細菌豐度。以純化的DNA作為模板,利用PCR擴增特定目的片段,對回收后的PCR 純化產(chǎn)物的樣品使用Qubit dsDNA HS Assay Kit試劑盒(Life Technologies公司,美國)進行定量檢測,最后在MiSeq高通量測序儀(Illumina公司,美國)PE250平臺進行測序。
1.2.3 測序數(shù)據(jù)處理 使用FastQC軟件對原始RNAseq數(shù)據(jù)進行質(zhì)控處理,質(zhì)控處理后的序列通過HISAT2.45軟件與人類基因組序列GRCh38進行比對,利用基因組注釋軟件GENCODE v22對轉(zhuǎn)錄組進行定量分析。本研究RNA-seq 結(jié)果已上傳至基因表達數(shù)據(jù)庫(Gene Expression Omnibus,GEO)(GSE158559)。原始RNAseq數(shù)據(jù)利用TRUST4算法(https://github.com/liulabdfci/TRUST4)進行處理,獲得每個樣本的免疫組庫信息。TRUST4首先將所測測序片段(reads)比對到參考基因組上,將比對上的reads組裝成重疊群(Contig),然后根據(jù)國際免疫遺傳學信息系統(tǒng)(International Immunogenetics Information System,IMGT)進行注釋。使用FastQC軟件對16S rDNA測序樣本數(shù)據(jù)進行質(zhì)量控制處理,再通過UCHIME算法去除嵌合體。利用USEARCH序列分析軟件將拼接好的標簽(Tag)在97%的相似度水平下進行聚類,獲得OTU信息,并基于rdp_16s_v16.fa分類學數(shù)據(jù)庫對OTU進行分類學注釋。
1.2.4 TCR 組庫多樣性評估 基于TRUST4 算法處理后的免疫組庫信息數(shù)據(jù),使用immunarch R 進一步對免疫組庫數(shù)據(jù)進行可視化分析,采用repClonality函數(shù)比較腫瘤組織及其正常組織中克隆型的數(shù)目、不同擴增程度克隆型的占比、不同克隆型的豐度;使用repExplore 函數(shù)比較不同長度CDR3 氨基酸序列分布上克隆型數(shù)目,并利用repDiversity 函數(shù)計算多樣性指數(shù)(Chao1 指數(shù)、逆Simpson 指數(shù)、Hill 數(shù)值)。Chao1 指數(shù)、逆Simpson 指數(shù)、Hill 數(shù)值越高,TCR多樣性越高。
1.2.5 臨床特征及Fn 豐度評估 臨床特征評估納入性別、年齡、腫瘤位置、腫瘤TNM 分期等因素。年齡以65 歲作為臨界值。根據(jù)腫瘤所在部位將腫瘤位置定義為直腸和非直腸。TNM 分期參照美國癌癥聯(lián)合 委 員 會(American Joint Committee on Cancer,AJCC)結(jié)直腸癌TNM 分期第八版[15],分為Ⅰ/Ⅱ期和Ⅳ/Ⅲ期。將性別、腫瘤位置、腫瘤TNM 分期轉(zhuǎn)化為二分類變量進行分組統(tǒng)計。Fn 豐度評估使用OTU的reads數(shù)目的中位數(shù)作為臨界值,>臨界值被定義為Fn高負荷,≤臨界值被定義為Fn低負荷。
1.3 統(tǒng)計學分析
使用R studio(4.1.1 版)軟件進行統(tǒng)計學分析。符合正態(tài)分布的定量資料采用表示,2 組間差異比較使用t檢驗。對于非正態(tài)分布的定量資料,采用M表示,2 組間差異比較使用Wilcoxon 檢驗。P<0.05表示差異有統(tǒng)計學意義。
2 結(jié)果
2.1 腫瘤組織及癌周正常黏膜組織TCR克隆型分析
2.1.1 克隆型數(shù)目 對53 對樣本的TCR 克隆型數(shù)目進行分析,總體上來看,腫瘤組織中克隆型數(shù)目較癌周正常黏膜組織下降(圖1A)。統(tǒng)計分析結(jié)果顯示,腫瘤組織TCR 克隆型數(shù)目明顯少于癌周正常黏膜組織,差異具有統(tǒng)計學意義(P=0.000,圖1B),且腫瘤組織中占據(jù)免疫組庫10%的克隆型數(shù)目也低于癌周正常黏膜組織(P=0.000,圖1C)。此外,對腫瘤組織和癌周正常黏膜組織TCR β 鏈CDR3氨基酸序列不同長度分布上的克隆型數(shù)目進行分析,結(jié)果顯示,在不同長度的CDR3序列上,腫瘤組織的克隆型數(shù)目均低于癌周正常黏膜組織(圖1D)。以上結(jié)果提示癌周正常組織TCR組庫多樣性更高。

圖1 腫瘤組織和癌周正常組織的TCR克隆型數(shù)目差異分析Fig 1 Comparison of the number of TCR clonotypes between tumor and adjacent normal tissues
2.1.2 不同擴增水平的克隆型 將TCR 克隆型按照擴增水平從高到低排序,分析排在前10、11~100、101~1 000、1 001~3 000、3 001~10 000、10 001~30 000、30 001~50 000位的克隆型所占的比例。腫瘤組織中擴增水平排在前10 位的TCR 克隆型所占比例明顯高于癌周黏膜正常組織,差異有統(tǒng)計學意義(P=0.000,圖2),提示腫瘤組織的TCR 的擴增程度高于癌周正常黏膜組織。

圖2 腫瘤和癌周正常組織不同擴增水平的TCR克隆型差異分析Fig 2 Comparison of proportion of TCR clonotypes with different amplification levels between tumor and adjacent normal tissues
2.1.3 不同頻率克隆型的相對豐度 進一步分析腫瘤組織和癌周正常黏膜組織不同頻率TCR克隆型在整個克隆空間內(nèi)的相對豐度。克隆型的頻率包括:稀有頻率(>0且≤10-5)、小頻率(>10-5且≤10-4)、中頻率(>10-4且≤0.001)、高頻率(>0.001且≤0.01)、超高頻率(>0.01且≤1)。腫瘤組織和癌周正常黏膜組織中,TCR克隆型均以小頻率(>10-5且≤10-4)和中頻率(>10-4且≤0.001)為主。腫瘤組織中高頻率(>0.001且≤0.01)和超高頻率(>0.01且≤1)克隆型的相對豐度高于癌周正常黏膜組織,稀有頻率(>0且≤10-5)和小頻率(>10-5且≤10-4)克隆型的相對豐度低于癌周正常黏膜組織,差異具有統(tǒng)計學意義(均P=0.000,圖3)。
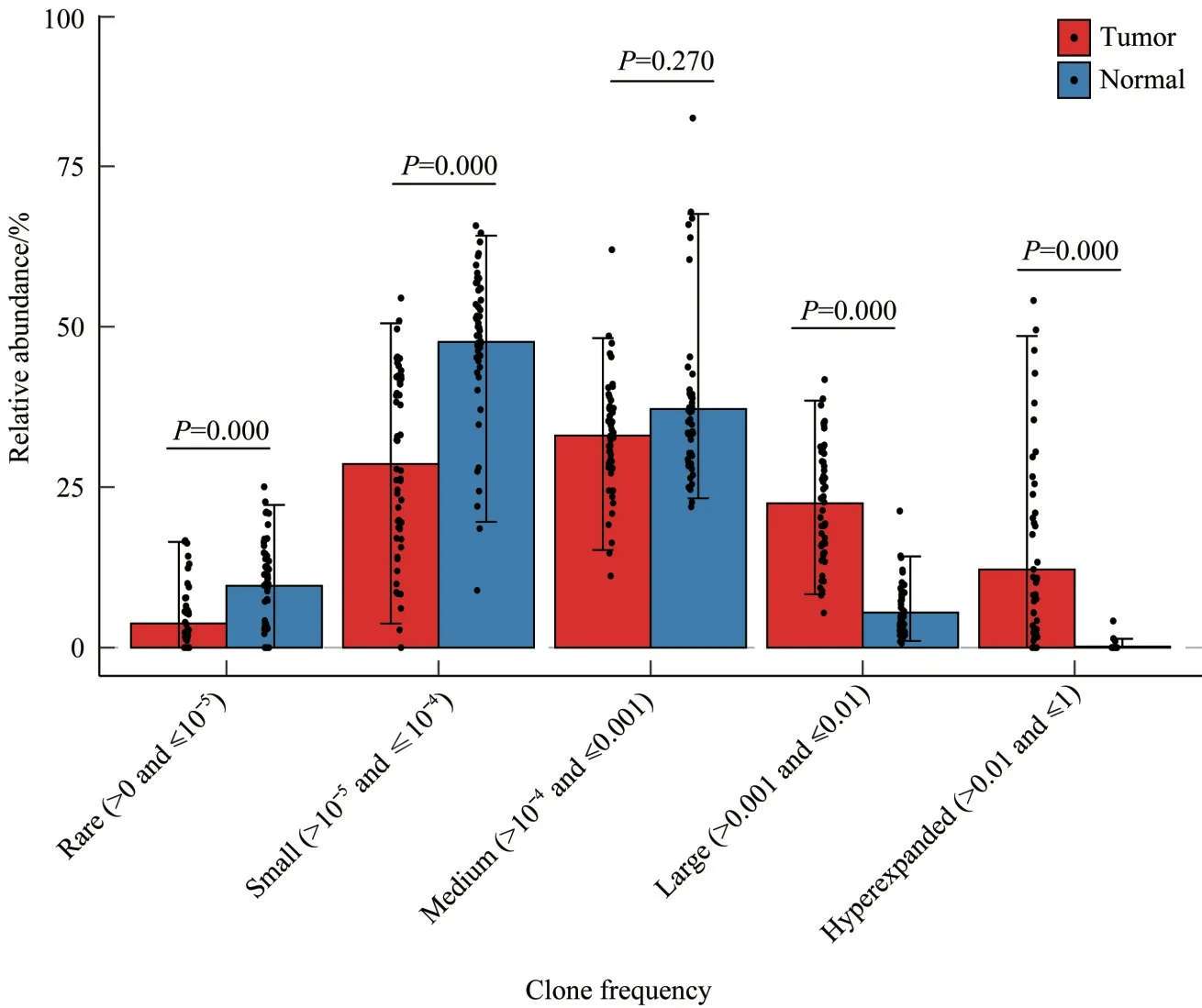
圖3 腫瘤和癌周正常組織不同頻率TCR克隆型的豐度差異Fig 3 Comparison of relative abundances in different TCR clonotypes with specific frequencies between tumor and adjacent normal tissues
2.2 腫瘤組織及癌周正常黏膜組織TCR 組庫多樣性指數(shù)評估
利用Chao1 指數(shù)、逆Simpson 指數(shù)以及Hill 數(shù)值曲線對腫瘤組織及癌周正常黏膜組織的TCR 組庫多樣性進行分析比較。腫瘤組織的Chao1 指數(shù)和逆Simpson 指數(shù)均小于癌周正常黏膜組織,差異具有統(tǒng)計學意義(均P=0.000,圖4A、B)。Hill 數(shù)值曲線也顯示,腫瘤組織TCR 組庫多樣性較癌周正常黏膜組織明顯下降(P=0.000,圖4C)。

圖4 腫瘤和癌周正常組織間TCR組庫多樣性指數(shù)比較Fig 4 Comparison of TCR repertoire diversity indexes between tumor and adjacent normal tissues
2.3 不同臨床特征腫瘤患者的TCR組庫多樣性分析
發(fā)生于直腸的CRC 患者TCR 克隆型的數(shù)目高于非直腸位部(P=0.040,圖5A),提示直腸部位腫瘤TCR 組庫多樣性更高。Fn 高負荷CRC 腫瘤組織的克隆型的數(shù)目(P=0.040,圖5B)和Chao1 指數(shù)(P=0.030,表1)低于Fn 低負荷CRC 腫瘤組織,差異具有統(tǒng)計學意義;提示不同F(xiàn)n 豐度下CRC 腫瘤組織的TCR 組庫多樣性具有差異,F(xiàn)n 豐度越高的腫瘤組織TCR 組庫多樣性越低。而不同年齡、性別(≥65 歲vs<65 歲)、TNM 分期(Ⅰ/Ⅱ期vsⅢ/Ⅳ期)的CRC患者,腫瘤組織Chao1指數(shù)的差異無統(tǒng)計學意義(均P>0.05,表1)。

表1 不同臨床特征和不同F(xiàn)n豐度下Chao1指數(shù)比較Tab 1 Comparison of Chao1 indexes in different clinical features and different Fn loads

圖5 不同腫瘤部位以及不同F(xiàn)n豐度下TCR克隆型比較Fig 5 Comparison of TCR clonotypes in different tumor locations and different Fn loads
3 討論
近年來,不少實體腫瘤的研究試圖深入挖掘免疫組庫信息,從而為疾病的診斷、免疫狀態(tài)監(jiān)測和患者預后評估提供新型生物學標志物[16-18]。然而關(guān)于TCR 組庫在CRC 中的相關(guān)研究開展得較少。SHERWOOD 等[19]通過對比分析15 例CRC 患者的TCR β 鏈測序的結(jié)果,發(fā)現(xiàn)腫瘤浸潤性T 細胞的多樣性變異遠低于癌周正常黏膜組織,同時腫瘤組織中TCR 組庫的寡克隆性較高,提示腫瘤組織和周圍正常黏膜組織內(nèi)的免疫應(yīng)答反應(yīng)不同。既往關(guān)于TCR組庫多樣性的研究大多依靠TCR-seq技術(shù)獲得樣本的TCR 序列。盡管測序技術(shù)能夠較為準確地呈現(xiàn)TCR序列特征,但測序費用高昂,且既往研究納入的樣本量都比較少。本研究使用TRUST 算法挖掘CRC 腫瘤組織和其匹配的癌周正常黏膜組織RNA-seq數(shù)據(jù)中的免疫組庫信息,相比于單獨針對樣本進行TCR-seq處理,經(jīng)濟成本有所降低,數(shù)據(jù)處理更加高效,并能獲取機體更多的生物學信息。此外,基于既往研究[3]發(fā)現(xiàn)的Fn 在CRC 的發(fā)生和發(fā)展過程的重要作用,我們試圖尋找CRC 組織中的TCR 組庫多樣性與Fn 豐度之間的關(guān)系。
本研究對比分析53 例CRC 患者的腫瘤組織及其匹配的癌周正常黏膜組織的TCR 組庫特點,發(fā)現(xiàn)在腫瘤組織中擴增水平前10 位克隆型的占比顯著高于癌周正常黏膜組織,這提示腫瘤狀態(tài)下T細胞受到外界抗原刺激,可能存在單克隆或寡克隆反應(yīng)性增生,導致TCR 某一序列克隆比例顯著上升,而多樣性卻呈現(xiàn)總體下降趨勢。腫瘤組織和癌周正常組織間TCR 組庫多樣性的差異,對于研究腫瘤發(fā)生和發(fā)展的機制具有重要的參考價值。CRC 中發(fā)生于直腸部位的腫瘤通常表現(xiàn)出對治療的高敏感性以及更好的預后[1]。本研究中,發(fā)生于直腸部位的腫瘤的TCR 組庫多樣性較非直腸部位腫瘤更高,這提示TCR 組庫可能有助于CRC 的臨床治療和預后評估。此外,本研究發(fā)現(xiàn)CRC 腫瘤組織內(nèi)的Fn 豐度與TCR 組庫多樣性有關(guān),在較低的Fn 負荷下,腫瘤的TCR 組庫多樣性更高。Fn 與CRC 的發(fā)生和發(fā)展密切相關(guān),腫瘤組織內(nèi)Fn 的高負荷與患者的不良預后密切相關(guān)[2]。TCR 組庫多樣性特點與CRC 患者的Fn 豐度相關(guān),進一步提示TCR 組庫多樣性能夠作為潛在的生物學標志物應(yīng)用于CRC發(fā)生和發(fā)展機制的研究中。
本研究尚存在一定的局限性。TCR β 鏈的CDR3序列信息是通過TRUST 算法計算得出的,盡管該算法十分高效,但難以避免存在一定的誤差。這需要未來開發(fā)更多更高效的檢測手段或者計算方法來盡可能地還原生物原始的CDR3序列信息。同時,TCR 組庫多樣性在CRC 的臨床診治及預后評估過程中的相關(guān)價值也有待于擴大樣本量進一步研究。在CRC 的發(fā)生和發(fā)展過程中,TCR 組庫多樣性發(fā)生改變機制如何,是否存在疾病特征性的TCR 單克隆序列,TCR組庫多樣性特點是否可以用于直接評估CRC 患者的預后和免疫治療效果,都有待于進一步探討。
綜上所述,本研究利用TRUST 算法從53 例CRC患者的RNA-seq數(shù)據(jù)中挖掘患者的免疫組庫信息,發(fā)現(xiàn)TCR 組庫多樣性與患者多項臨床特征以及腫瘤組織內(nèi)的Fn 豐度有關(guān),為CRC 發(fā)生和發(fā)展的機制研究提供了重要信息。
利益沖突聲明/Conflict of Interests
所有作者聲明不存在利益沖突。
All authors disclose no relevant conflict of interests.
倫理批準和知情同意/Ethics Approval and Patient Consent
本研究涉及的所有實驗均已通過上海交通大學醫(yī)學院附屬仁濟醫(yī)院科學倫理委員會的審核批準(2016-01-15)。受試對象或其親屬已經(jīng)簽署知情同意書。All experimental protocols in this study were reviewed and approved by Renji Hospital,Shanghai Jiao Tong University School of Medicine(2016-01-15). Consent letters have been signed by the research participants or their relatives.
作者貢獻/Authors'Contributions
陳豪燕、洪潔參與了實驗設(shè)計;胡慕妮完成主要統(tǒng)計學分析和繪圖;季林華、張昕雨和沈超琴主要負責標本收集和實驗部分;胡慕妮、陳豪燕參與了論文的寫作和修改。所有作者均閱讀并同意了最終稿件的提交。
CHEN Haoyan and HONG Jie were responsible for the experimental design. HU Muni completed the main statistical analysis and mapping.JI Linhua,ZHANG Xinyu and SHEN Chaoqin mainly prepared for specimen collection and experiments.CHEN Haoyan and HU Muni participated in the writing and revision of the paper. All authors have read and approved the submission of the final manuscript.
·Received:2022-01-24
·Accepted:2022-05-27
·Published online:2022-07-25
參·考·文·獻
[1] SIEGEL R L, MILLER K D, GODING SAUER A, et al. Colorectal cancer statistics,2020[J]. CA Cancer J Clin,2020,70(3):145-164.
[2] HARUKI K, KOSUMI K, HAMADA T, et al. Association of autophagy status with amount ofFusobacterium nucleatumin colorectal cancer[J]. J Pathol,2020,250(4):397-408.
[3] YU T, GUO F F,YU Y N, et al.Fusobacterium nucleatumpromotes chemoresistance to colorectal cancer by modulating autophagy[J].Cell,2017,170(3):548-563.e16.
[4] MIMA K, NISHIHARA R, QIAN Z R, et al.Fusobacterium nucleatumin colorectal carcinoma tissue and patient prognosis[J].Gut,2016,65(12):1973-1980.
[5] VUJOVIC M, DEGN K F, MARIN F I, et al. T cell receptor sequence clustering and antigen specificity[J]. Comput Struct Biotechnol J,2020,18:2166-2173.
[6] CHEN M, CHEN R Z, JIN Y, et al. Cold and heterogeneous T cell repertoire is associated with copy number aberrations and loss of immune genes in small-cell lung cancer[J]. Nat Commun,2021,12(1):6655.
[7] SIMNICA D,AKYüZ N,SCHLIFFKE S,et al. T cell receptor nextgeneration sequencing reveals cancer-associated repertoire metrics and reconstitution after chemotherapy in patients with hematological and solid tumors[J]. Oncoimmunology,2019,8(11):e1644110.
[8] SONG L, COHEN D, OUYANG Z Y, et al. TRUST4: immune repertoire reconstruction from bulk and single-cell RNA-seq data[J].Nat Methods,2021,18(6):627-630.
[9] SHAO H W, LIN Y M, WANG T, et al. Identification of peptidespecific TCR genes byin vitropeptide stimulation and CDR3 length polymorphism analysis[J]. Cancer Lett,2015,363(1):83-91.
[10] LOONEY T J, TOPACIO-HALL D, LOWMAN G, et al. TCR convergence in individuals treated with immune checkpoint inhibition for cancer[J]. Front Immunol,2020,10:2985.
[11] STEWART J J, LEE C Y, IBRAHIM S, et al. A Shannon entropy analysis of immunoglobulin and T cell receptor[J]. Mol Immunol,1997,34(15):1067-1082.
[12] HOPKINS A C, YARCHOAN M, DURHAM J N, et al. T cell receptor repertoire features associated with survival in immunotherapy-treated pancreatic ductal adenocarcinoma[J]. JCI Insight,2018,3(13):e122092.
[13] HAN J F, DUAN J C, BAI H, et al. TCR repertoire diversity of peripheral PD-1+CD8+T cells predicts clinical outcomes after immunotherapy in patients with non-small cell lung cancer[J].Cancer Immunol Res,2020,8(1):146-154.
[14] JOSHI K, MILIGHETTI M, CHAIN B M. Application of T cell receptor (TCR) repertoire analysis for the advancement of cancer immunotherapy[J]. Curr Opin Immunol,2022,74:1-8.
[15] AMIN M B, GREENE F L, EDGE S B, et al. The Eighth EditionAJCC Cancer Staging Manual: continuing to build a bridge from a population-based to a more“personalized”approach to cancer staging[J]. CA Cancer J Clin,2017,67(2):93-99.
[16] BUHLER S,BETTENS F,DANTIN C,et al. Genetic T-cell receptor diversity at 1 year following allogeneic hematopoietic stem cell transplantation[J]. Leukemia,2020,34(5):1422-1432.
[17] FARMANBAR A, KNELLER R, FIROUZI S. RNA sequencing identifies clonal structure of T-cell repertoires in patients with adult Tcell leukemia/lymphoma[J]. NPJ Genom Med,2019,4:10.
[18] CHARLES J, MOURET S, CHALLENDE I, et al. T-cell receptor diversity as a prognostic biomarker in melanoma patients[J].Pigment Cell Melanoma Res,2020,33(4):612-624.
[19] SHERWOOD A M, EMERSON R O, SCHERER D, et al. Tumorinfiltrating lymphocytes in colorectal tumors display a diversity of T cell receptor sequences that differ from the T cells in adjacent mucosal tissue[J]. Cancer Immunol Immunother,2013,62(9):1453-1461.

