染色質免疫共沉淀測序技術研究進展
陳桂芳 楊佳怡 高運華 任歌
(1.中國計量科學研究院,北京 100029;2.沈陽化工大學,沈陽 110142)
組蛋白修飾和轉錄因子對基因表達具有重要的調控作用。在真核細胞中,DNA纏繞著由H2A、H2B、H3、H4構成的核心組蛋白八聚體形成核小體,通過連接DNA串聯形成染色質。組蛋白修飾的發生將影響組蛋白與DNA的親和性,改變染色質的可及性,進而影響基因的表達。轉錄因子識別并結合基因上游區域特定DNA序列,調控相關基因的表達。染色質免疫共沉淀(chromatin immunoprecipitation,ChIP)利用目的蛋白特異性抗體,與可溶性染色質免疫共沉淀,特異性地富集目的蛋白結合的DNA。與芯片技術相結合的ChIP-chip(chromatin immunoprecipitation-chip)是利用表面覆蓋已知序列的核苷酸探針的芯片,對染色質免疫共沉淀捕獲的DNA進行核酸雜交,通過傳感器檢測堿基互補配對產生的熒光信號,進一步分析目的蛋白結合位點[1]。ChIP-chip檢測覆蓋率受限于芯片上預先設定的DNA序列,存在分辨率低、靈敏度有限,對探針設計要求高等局限性[2]。隨著高通量測序技術的發展以及測序成本不斷降低,染色質免疫共沉淀與測序相結合的ChIP-seq被廣泛使用。研究人員利用生物信息學工具對高通量測序生成的大量數據進行分析,通過將測序序列比對到全基因組,定位轉錄因子結合、組蛋白修飾的區域[3]。近年來,ChIP-seq被越來越廣泛地用于擬南芥、水稻及其他植物的基因表達調控研究中。
ChIP-seq通常需要數百萬個細胞,染色質免疫共沉淀和測序文庫制備包含多個實驗步驟,免疫共沉淀可能受到非特異性結合的影響,產生背景噪音,文庫中DNA中GC含量過高或過低,將導致PCR擴增偏倚,進而影響分析結果的準確性。研究人員利用流式細胞術、微流控芯片等技術分離少量細胞或單細胞,優化染色質片段化、免疫共沉淀以及測序文庫構建等實驗流程;通過特異性抗體引導將MNase或Tn5轉座酶間接結合到目的蛋白,并在蛋白結合位點附近使染色質斷裂,替代了ChIP-seq染色質片段化和免疫共沉淀操作,簡化實驗操作流程。在上述基礎上,實現少量細胞或單細胞水平的ChIP-seq檢測。本文簡述了ChIP-seq原理,詳細介紹其數據分析方法,討論近年來發展的ChIP-seq優化方法和衍生技術,分析并比較不同方法的特點,總結了植物轉錄因子和組蛋白修飾在生物鐘調控、激素信號轉導、光信號途徑、脅迫響應等方面研究與應用。
1 ChIP-seq原理及數據分析
1.1 ChIP-seq原理
組蛋白修飾主要發生在組蛋白的N端,核小體組蛋白被DNA環繞,兩者結合較穩定。轉錄因子一般具有DNA結合結構域,識別靶基因并以序列特異性方式結合DNA,轉錄因子與DNA相互作用通常是動態的[4-5]。根據目的蛋白與DNA結合特性,ChIP中制備染色質片段的方式不同,主要包括甲醛交聯染色質免疫共沉淀(formaldehyde cross-linking and sonication followed by chromatin immunoprecipitation,X-ChIP)和非交聯染色質免疫 共 沉 淀(native chromatin immunoprecipitation,N-ChIP)[6-7]。甲醛能交聯固定蛋白與DNA,X-ChIP通過甲醛交聯與超聲處理形成可溶性染色質片段,利用特異性抗體沉淀目的蛋白與DNA復合物,通過解交聯、蛋白酶消化等分離純化DNA,常用于檢測轉錄因子與DNA的相互作用[7]。微球菌核酸酶(micrococcal nuclease,MNase)兼具核酸內切酶和核酸外切酶活性,主要作用于核小體之間的連接DNA(linker DNA)。N-ChIP一般無需甲醛交聯,MNase使染色質在連接DNA區域斷裂,形成以核小體為單元的染色質片段,酶切后采用組蛋白修飾特異性抗體進行免疫共沉淀并分離純化DNA,常用于組蛋白修飾的檢測[8-9]。
通過超聲或酶切斷裂的DNA帶有凸出的粘性末端,為連接具有平末端的測序接頭,在構建文庫過程中,需要對DNA進行末端修復。延伸DNA 3'端直至與5'端平齊,隨后對5'和3'末端分別進行磷酸化和加dA修飾,獲得具有5'磷酸化和3' dA的DNA片段,進一步連接測序接頭[4,10]。圖1顯示了ChIP-seq基本流程:通過超聲或酶切進行染色質片段化,利用特異性抗體免疫共沉淀目的蛋白和DNA,分離純化的DNA經末端修復、連接測序接頭,通過PCR擴增構建文庫、測序并分析[11]。

圖1 ChIP-seq基本流程Fig.1 Major steps of ChIP-seq
1.2 ChIP-seq數據分析
ChIP-seq數據分析是通過將測序序列比對到參考基因組,識別富集區域內具有顯著信號的峰、定位目的蛋白結合位點[12-13]。ChIP-seq數據分析基本流程包括:預處理及質量控制、序列比對、峰識別、可視化及高級分析等[14]。研究人員開發了多種基于不同算法的軟件工具對高通量測序數據進行處理。其中,Linux系統的Conda和基于R語言的Bioconductor是開放式軟件平臺,具有ChIP-seq數據分析的大量工具和軟件包[15]。
1.2.1 ChIP-seq數據分析軟件及算法 測序數據質量控制包括質量評估和預處理。測序中,每個堿基具有一個質量值Q-score(Q-score= -log10e,e為測序錯誤率),用于衡量測序準確度,如Q30表明堿基識別發生錯誤的概率為0.1%,Q20指堿基識別發生錯誤的概率為1%。通過FastQC軟件對測序的原始讀長(raw reads)進行質量評估,一般使用質量值大于20或30的堿基占總體堿基的百分比(Q20或Q30)來評估測序數據質量。接頭序列、擴增不均勻等將影響測序數據質量,這些情況可利用FastQC查看[16]。根據質量評估結果,可選擇TrimGalore、Picard以及SAMTools等工具進行預處理,去除低質量堿基、接頭序列以及PCR擴增重復(PCR duplicates)等,獲得較高質量的數據(clean data)[17]。
序列比對通過將測序序列比對到參考基因組(或序列已知的基因組)進行定位[18]。高通量測序將產生大量的短序列數據,包含許多重復序列。絕大多數的序列比對算法構建索引數據庫,通過索引篩選短序列在基因組中候選位置,減少搜索空間,提高比對效率[19]。根據建立索引數據結構方法的不同,短序列比對軟件主要分為兩類:基于哈希表(Hash table)數據結構和基于BWT壓縮算法的索引數據結構(Burrows Wheeler transform,BWT)[20-22]。 哈 希表數據結構序列比對過程中,測序讀段(reads)將以種子序列(seed)為單元生成序列集合,排列種子序列并建立索引數據結構進行比對[14,23]。該方法也可以對參考基因組生成種子序列,建立索引數據結構。BWT算法通過掃描短序列識別堿基重復的序列,將重復序列排列在一起,進一步壓縮索引數據結構并重排列,以利于快速搜索和比對。目前,較為常用的短序列比對軟件有Bowtie2、BWA、SOAP2和MAQ,不同的比對軟件在比對數目、運行時間、內存消耗等方面各具優勢和不足[24]。較短讀長為單元時,可能的匹配區域很多,種子序列位點定位效率將降低,在進行序列比對過程中,基于Hash table數據結構的MAQ難以實現準確比對;基于BWT算法的比對工具有Bowtie2、BWA、SOAP2等,該算法的重排列利于短讀長在基因組中候選位點進行快速搜索和比對[25]。測序堿基的深度與基因組覆蓋率成正比例相關,隨測序深度增加,基因組覆蓋率增加,數據量更大。在HPV全基因組測序數據比對分析中,研究者采用上述4種工具將部分HPV測序數據與已知HPV基因組數據庫進行比對,結果表明BWT算法的比對效率和計算速度優于Hash table算法[26]。
ChIP-seq數據分析中的重要環節是峰識別(peak calling)。峰(peak)被定義為基因組上reads富集的區域,峰識別是通過掃描比對到基因組上的短序列數據,進行樣本數據和對照組數據的比較,識別富集區域[14]。MACS算法通過滑動窗口(sliding window)掃描,基于泊松分布模型統計顯著的峰[27]。MACS采用具有固定大小的滑動窗口移動,可能產生窗口邊緣識別模糊的問題。QuEST算法基于連續覆蓋掃描,通過高斯核密度函數對reads富集密度進行評估,其中reads所在位置為窗口中心,具有最高密度值[28-29]。MACS通過計算全基因組范圍內每個檢測峰(peak)顯著性P值,進一步分析差異peak,鑒定具有統計顯著性的差異蛋白質結合位點,為較常用的峰識別工具[29]。
基于不同的需求,后續的數據處理和分析方法有所不同,如DNA序列特征的Motif分析、目的蛋白結合位點在基因組不同區域的偏好性分析、預測結合位點關聯基因功能的GO注釋及預測基因調控通路的Pathway分析等[30-31]。圖2顯示ChIP-seq數據分析流程及軟件。

圖2 ChIP-seq數據分析流程及軟件Fig.2 Protocol for computational analysis of ChIP-seq data and software
1.2.2 ChIP-seq常見數據格式及可視化 數據格式對于合理組織數據存儲,有效降低存儲空間以及加快下游分析速度至關重要。圖3顯示了ChIP-seq數據常見格式、格式轉變軟件及可視化方法。fasta和fastq格式是存儲核酸序列的常用格式,為二進制文本。其中,fastq格式包含短讀序列和質量分數等信息[32]。為方便后續分析,可利用sratoolkit軟件將測序原始數據格式轉換為標準的fastq格式。SAM和BAM格式專用于存儲參考序列的比對序列,是由基因組序列比對得到的輸出格式[33]。BAM數據格式具有索引功能,為SAM格式的二進制,通過SAMTools軟件可將SAM文件轉換為BAM,有利于降低儲存空間。UCSC基因瀏覽器可以讀取BAM格式的數據,實現快速瀏覽[34]。儲存轉錄因子、組蛋白修飾等結合位點在全基因組上的信號分布情況的數據格式為BedGraph(Bed)格式。該格式包含有染色體名稱、染色體起始位點以及檢出信號值。將MACS軟件輸出Bed格式文件轉化為bigwig文件,上傳到UCSC或IGV瀏覽器以實現數據可視化[35-36]。其他可視化工具有基于R語言的ChIPseeker和基于Python的deepTools等。
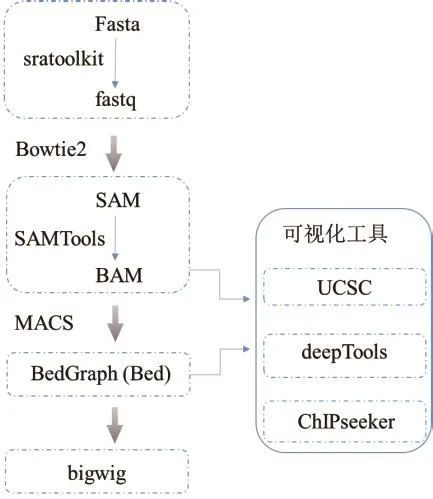
圖3 ChIP-seq數據格式及可視化Fig.3 Standard data formats and visualization tools for ChIP-seq
2 基于ChIP-seq的優化技術
ChIP-seq需要大量細胞,面對稀少樣本,收集足夠多的細胞存在困難。免疫共沉淀過程,可能受到甲醛交聯,非特異性結合的影響[37]。DNA片段中GC含量過高或過低,將導致PCR擴增偏倚,影響測序質量[38]。近年來研究人員針對上述問題進行優化并提出相應的技術。
2.1 基于細胞分離優化的ChIP-seq技術
流式細胞分選(fluorescence activated cell sorting,FACS)是利用鞘液包裹細胞形成樣品流,通過流式細胞儀檢測細胞攜帶的熒光信號,由分選器將特定的細胞從樣本中分離出來。Amour等[39]通過FACS將細胞分離到含有裂解緩沖液的反應池中,進行細胞核分離與MNase酶切,提出基于MNase酶切的非交聯免疫共沉淀ChIP-seq技術(nultra low input micrococcal nuclease-based native ChIP,ULI-NChIP),該技術適用于微量樣品建庫測序。該方法利用連續裝置簡化實驗操作流程、減少分離純化過程的洗滌次數,降低樣品損失,利于在少量細胞中進行ChIP-seq 實驗[40]。
微流控芯片通過產生非連續的液滴包裹單個細胞,實現單細胞分離[41]。Rotem等[42]基于液滴微流控芯片建立scChIP-seq(single-cell ChIP-seq):在具有多通道結構的芯片上,細胞裂解緩沖液包裹著標簽序列與單細胞懸液匯聚并通過油相,形成“油包水”液滴,液滴內發生細胞裂解反應,隨后與含MNase的凝膠微珠融合進行染色質片段化,進一步對數千個單細胞獨立建庫。scChIP-seq具有高度集成化、自動化優勢,但微流控芯片使用成本較高,且微流控液滴操作對實驗人員有較高技術要求。
2.2 基于酶切優化的ChIP-seq技術
lambda核酸外切酶、RecJf核酸外切酶具有5'-3'外切酶活性,分別作用于雙鏈DNA和單鏈DNA,水解核苷酸之間的磷酸二酯鍵。Ho和Pugh將兩種核酸外切酶引入到X-ChIP實驗,提出ChIP-exo(ChIP combined with lambda exonuclease digestion)[43-44]。在特異性抗體沉淀目的蛋白與DNA交聯復合物之后,通過lambda核酸外切酶消化,使雙鏈DNA斷裂末端最大程度的靠近蛋白,減少背景干擾,提高蛋白質結合區域分析的準確性。進一步解交聯、分離純化DNA,利用RecJf核酸外切酶消化單鏈DNA,減少背景噪音。ChIP-exo核酸外切酶處理,有利于提高檢測轉錄因子結合位點的分辨率[2,44]。
2.3 基于免疫共沉淀優化的ChIP-seq技術
染色質免疫共沉淀實驗過程中,通常使用磁珠富集抗體、蛋白和DNA復合物,但難以避免非特異性結合的影響[37]。Zhu等[45]基于微流控芯片優化ChIP實驗操作流程,提出MOW ChIP-seq(microfluidic oscillatory washing-based ChIP-seq)。將超聲處理的染色質載入芯片,采用偶聯有目的蛋白特異性抗體的磁珠富集可溶性片段。通過控制芯片上微量流道內樣品與磁珠的流動速度,促進兩者混合均勻,提高免疫磁珠的捕獲效率。另外利用微流控振蕩輔助,降低非特異性結合,提高結果準確度。MOW ChIP-seq通過結合免疫磁珠、微流控芯片對樣品進行分離與富集,具有試劑和樣品消耗量少、自動化等優勢[37]。
2.4 基于測序文庫優化的ChIP-seq技術
ChIP-exo增加的酶切步驟以及多次洗滌,使用于建庫的起始DNA量減少,需要增加PCR循環擴增數,可能導致過多的重復序列[46]。在ChIP-exo基礎上,He等[47]設計含特異性序列的測序接頭,通過DNA自環化(self-circularization)方法優化建庫,提 出 ChIP-nexus(ChIP experiments with nucleotide resolution through exonuclease,unique barcode and single ligation)。建庫過程中,將帶有限制性內切酶BamH I酶切位點序列的測序接頭連接到DNA,利用環化連接酶將DNA自身環化。環化DNA通過BamH I酶切處理,其斷裂產物兩端將帶有測序接頭,利于直接擴增建庫。ChIP-nexus通過分子內自身環化進行接頭連接構建文庫,其連接效率比ChIP-seq中DNA連接酶的連接效率更高,有利于降低建庫DNA需求量。
擴增構建測序文庫過程中,高GC含量DNA片段易產生擴增偏倚,擴增體系中的引物二聚體將造成背景信號[48]。Adli等[49]對文庫構建方案優化,并提出Nano-ChIP-seq。在PCR擴增建庫過程中,使用發夾結構引物,減少引物二聚體。針對高GC含量序列,使用Phusion高保真DNA聚合酶,并優化緩沖液和擴增循環次數。此外,在引物序列中引入限制性內切酶BciV I的酶切位點序列,擴增產物在內切酶作用下產生3'A突出末端,利于直接連接測序接頭。研究表明,利用Nano-ChIP-seq建庫方案可從少量細胞中進行ChIP-seq檢測[49-50]。表1比較了上述ChIP-seq優化技術。
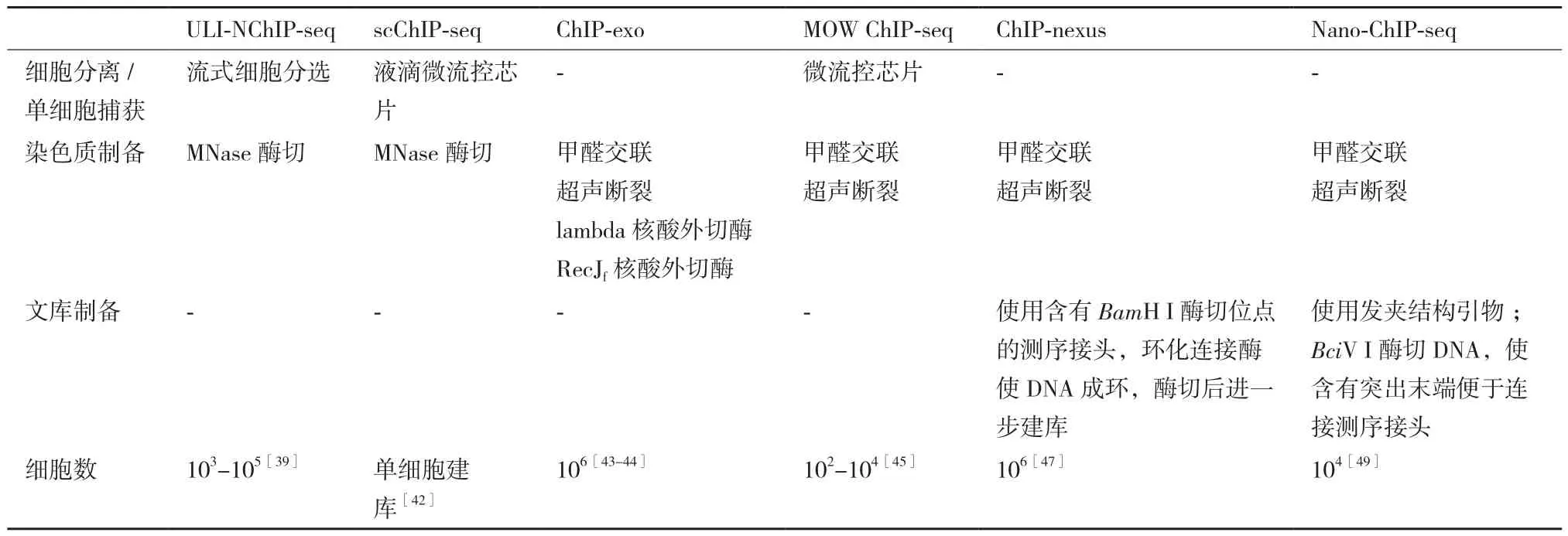
表1 ChIP-seq優化技術比較Table 1 Comparison of optimization techniques of ChIP-seq
3 CUT&RUN、CUT&Tag及CoBATCH技術
ChIP包含細胞裂解、細胞核提取、染色質制備以及免疫共沉淀等多個連續步驟,需要的細胞量較大,常規ChIP-seq很難進行少量細胞或單細胞水平的檢測[51]。最近,研究人員利用目的蛋白特異性抗體使MNase或Tn5轉座酶“靶向”作用于目的蛋白結合位點附近的染色質,提出CUT&RUN、CUT&Tag等方法。與ChIP-seq相比較,這些方法無需獲取可溶性染色質進行免疫共沉淀,具有實驗流程簡單、耗時短等優勢,可以在少量細胞或單細胞水平進行檢測。
3.1 CUT&RUN技術
Schmid等[52]建立了以酶“靶向”切割染色質的 方 法:ChIC(chromatin immunocleavage)。 為 使MNase選擇性地作用于目的蛋白結合區域,研究者利用對抗體具有親和力的Protein A融合MNase(pAMN),通過抗體結合pA-MN融合蛋白將MNase間接結合到目的蛋白。在ChIC實驗中,首先利用螯合劑EDTA、EGTA等抑制MNase酶活性,將細胞與含有目的蛋白特異性抗體、pA-MN的緩沖液反應后,用Ca2+激活MNase,使其在特定位點斷裂DNA。2016年,Skene等[51]將ChIC與高通量測序技術結合,提出CUT&RUN。
CUT&RUN中,特異性抗體先與目的蛋白結合,進一步與pA-MN反應,募集pA-MN到結合位點。在0℃下保持較低的酶活性,抗體固定酶后,采用Ca2+激活MNase使其在目的蛋白結合位點附近作用于染色質開放區域,酶切后染色質片段釋放,進一步建庫測序[51,53]。研究者利用伴刀豆球蛋白A(concanavalin A,Con A)能與細胞的多糖、糖蛋白特異結合特性,將Con A包被在磁珠表面,固定細胞或細胞核,以利于洗滌。2018年,Skene等[54]對CUT&RUN實驗優化,通過洋地黃皂苷(digitonin)增加細胞膜通透性,釋放酶切染色質片段到細胞外,由于未被切割的染色質仍留在細胞內,有利于降低背景噪音。2019年,Hainer等[55]利用流式細胞分選將單個細胞分離到多孔板中進行CUT&RUN實驗,提出可對極少量細胞進行檢測的ULI-CUT&RUN(ultra-low input CUT&RUN)。
3.2 CUT&Tag及CoBATCH技術
Tn5轉座子由核心序列和兩末端序列組成。Tn5轉座酶可以與Tn5轉座子的末端序列結合形成復合物,該復合物具有“剪切-粘貼”(cut and paste)催化活性,兩者協同完成Tn5轉座子末端序列的切割和轉移。在Tn5轉座復合體作用下,轉座子末端DNA的磷酸二酯鍵被水解并產生3'- OH羥基末端,隨后Tn5轉座酶進攻目標DNA形成9 bp的切口,同時轉座子羥基末端與目標DNA的磷酸基團之間形成共價鍵,從而將轉座子序列插入到目標DNA[56]。由于這一特性,Tn5轉座酶可將測序接頭序列隨機插入染色質開放區域,進行DNA片段化和測序接頭連接[57]。Schmidl等[58]首先將 Tn5轉座酶工具與ChIP相結合,代替ChIP-seq構建測序文庫中的末端補平、3'末端加A等處理,提出ChIPmentation。其中,測序接頭和Tn5轉座酶組裝形成轉座復合物,與X-ChIP獲得的目的蛋白結合DNA反應,進行接頭連接。
2019年,Kaya-Okur等[59]參考 CUT&RUN的實驗流程,將帶有測序接頭的Tn5轉座酶與Protein A融合(pA-Tn5),利用pA-Tn5替換pA-MN,提出CUT&Tag。特異性抗體結合目的蛋白后,進一步與pA-Tn5反應,利用Mg2+激活Tn5轉座酶活性進行染色質切割與接頭連接。Tn5轉座酶建庫有利于減少損失,與CUT&RUN比較,CUT&Tag的樣本需求量更小。2020年,Bartosovic等[60]將液滴微流控技術與CUT&Tag相結合,進行單細胞建庫測序,提出 scCUT&Tag(single-cell Cut&Tag)。 表2比 較 了CUT&RUN、CUT&Tag與 ChIP-seq。

表2 CUT&RUN、CUT&Tag與ChIP-seq比較Table 2 Comparison of CUT&RUN,CUT&Tag and ChIP-seq
標簽組合(combinatorial indexing)建庫是利用不同的barcode序列為不同樣品的DNA進行組合標簽標記,通過一次建庫可區別成千上萬單細胞,利于提高單細胞測序的通量,獲得更多單細胞信息[61]。2019年,Wang等[62]基于標簽組合和pA-Tn5提出 CoBATCH(combinatorial barcoding and targeted chromatin release)。通過流式細胞儀和微孔板進行單個細胞的分選和分離,采用帶有T5/T7組合標簽的pA-Tn5對染色質進行轉座酶切割和標簽標記,進一步對帶有不同標簽的DNA建庫與測序。CoBATCH利用組合標簽區分不同樣本來源的細胞,同時組合標簽增加了測序文庫的復雜度,利于進行高通量的單細胞檢測。
4 在植物基因表達調控研究中的應用
研究人員使用ChIP-seq檢測植物轉錄因子結合位點、組蛋白修飾分布,已廣泛應用于生物鐘調控、激素信號轉導、光信號途徑、脅迫響應等研究[63-65]。CUT&RUN和CUT&Tag方法具有流程簡單、良好的可重復性、需要的細胞數量少等優勢,近年來,被初步應用于植物轉錄因子、組蛋白修飾H3K27me3和 H3K4me3 等研究[66-68]。
4.1 植物轉錄因子結合位點相關研究
植物的生長發育除了受自身遺傳因素的調控外,還受到環境脅迫、內源激素變化等影響。轉錄因子在植物的生物鐘調控、激素信號轉導、生長和代謝等過程中發揮重要作用[69-70]。GRF7(growthregulating factor 7)是水稻(Oryza sativa)生長調節因子類轉錄因子,Chen等[71]選用不同發育時期的水稻幼穗進行ChIP-seq檢測,發現OsGRF7與細胞色素P450基因OsCYP714B1和生長素響應基因OsARF12啟動子中的ACRGDA motif結合,激活基因轉錄,參與赤霉素的合成和生長素的信號傳導途徑,調控幼穗發育。生物鐘是影響生物晝夜節律的重要因素,CAA1(circadian clock-associated 1)是擬南芥(Arabidopsis thaliana)生物鐘重要轉錄因子,PRRs家族基因參與生物鐘調控,Kamioka等[69]通過ChIP-seq發現CCA1直接結合在基因PRR5的啟動子區域,抑制PRR5表達,并發現CCA1與PRR9、PRR7、PRR5等基因啟動子上多個motif結合,包括G-box、EEs、CT重復、TCP等,調控生物鐘周期。光是調控植物生長和發育的重要環境因素,FHY3(far-red elongated hypocotyl 3)是擬南芥光信號轉錄因子,Ouyang等[72]利用ChIP-seq在遠紅光條件下鑒定到FHY3結合在基因FHY1和ELF4啟動子的FBS motif(CACGCGC),激活基因表達,促進光敏色素A在細胞核的積累,進而調控光信號途徑;并發現FHY3與葉綠體分裂相關基因ARC5啟動子區域的FBS motif結合,激活其轉錄,進而影響葉綠體發育。葉夾角是影響株型與作物產量的重要農藝性狀,油菜素內酯(brassinosteroid,BR)是植物重要的促生長類激素。BR促進細胞伸長與分裂,對水稻葉夾角發育有影響,Guo等[68]通過CUT&RUN分析發現,水稻bHLH(basic-helix-loop-helix)轉錄因子家族的OsbHLH98,結合水稻BR信號轉導途徑相關基因BUL1的啟動子上的G-box、E-box等motif,抑制基因表達,調控水稻葉夾角發育。
4.2 植物組蛋白修飾及分布相關研究
在植物發育過程中,除了轉錄因子激活或抑制基因表達的作用外,染色質組蛋白翻譯后修飾通過影響染色質結構來調控基因的表達。組蛋白修飾H3K27me3抑制基因轉錄,Wu等[73]通過ChIP-seq發現在高氮素條件下水稻分蘗抑制基因D14和OsSPL14啟動子區域的H3K27me3富集水平顯著升高。進一步研究表明,水稻分蘗期氮素應答關鍵蛋白NGR5通過與PRC2相互作用,將PRC2招募到D14和OsSPL14的啟動子上催化H3K27me3修飾并抑制基因表達,進而調控氮濃度對水稻氮素吸收和分蘗發生的影響。Nishio等[74]以鼠耳芥(Arabidopsis halleri)為材料,通過ChIP-seq分析組蛋白修飾H3K27me3對其季節性和晝夜節律基因表達的影響,并與組蛋白修飾H3K4me3進行比較。發現H3K27me3具有季節性的可塑性與晝夜節律穩定性,H3K27me3的信號變化晚于H3K4me3出現,在環境變化中進行長期的基因表達調控。
研究者將CUT&RUN、CUT&Tag用于植物組蛋白修飾分析,并與ChIP-seq進行比較。Zheng等[66]利用流式細胞儀分選擬南芥胚乳細胞核,通過CUT&RUN分析發現胚乳細胞周期中有絲分裂間期的相關基因被H3K27me3修飾,H3K27me3影響親本等位基因的差異表達與胚乳發育。與ChIP-seq相比較,CUT&RUN檢測所需細胞數量更少。Tao等[67]將CUT&Tag用于對棉纖維細胞基因組及外顯子、內含子、啟動子等區域組蛋白修飾H3K4me3的分布特征分析,發現H3K4me3顯著富集在基因啟動子上(轉錄起始位點上游1-2 kb)。使用相同數量細胞進行CUT&Tag和ChIP-seq檢測,發現CUT&Tag分析結果具有較好重復性,顯示出更高分辨率和更低的背景信號,所需實驗時間更短。
5 總結與展望
ChIP-seq已被廣泛用于轉錄因子和組蛋白修飾研究。近年來,研究人員將流式細胞分選、微流控芯片與ChIP-seq相結合,優化細胞分離、染色質片段化、免疫共沉淀以及測序文庫構建等關鍵步驟并提出優化方法。CUT&RUN、CUT&Tag利用“靶向”酶切和Tn5轉座酶建庫,簡化實驗流程。在上述基礎上,使得ChIP-seq在少量細胞或在單細胞水平的檢測成為可能。其他測序技術在轉錄因子和組蛋白修飾研究中具有重要作用:微球菌核酸酶測序MNase-seq(micrococcal nuclease sequencing) 利 用MNase切割染色質,獲取核小體DNA建庫測序,繪制核小體定位圖譜[75]。MNase-seq與ChIP-seq聯合將利于分析目的蛋白結合位點附近的核小體定位狀態。染色質轉座酶可及性測序ATAC-seq(assay for transposase-accessible chromatin with high throughput sequencing)利用Tn5轉座酶進行染色質切割與接頭連接,檢測染色質開放區域[76]。ATAC-seq結合ChIP-seq可進一步分析轉錄因子、組蛋白修飾對染色質開放性的影響。ChIP-seq與轉錄組測序技術(RNA-seq)的聯合分析,有利于進一步確認轉錄因子以及組蛋白的修飾對于基因表達的調控作用。
隨著技術的不斷發展,ATAC-seq、RNA-seq,CUT&RUN、CUT&Tag等均可以實現單細胞水平的檢測。進行單細胞的組學分析,克服細胞異質性對有效信號的“干擾”問題,有利于珍稀樣品的準確分析。將ChIP-seq納入多組學聯合分析,幫助更全面的理解細胞內蛋白與DNA相互作用,具有重要的研究意義。

