擬柱孢藻N8全基因組測序及磷吸收轉運通路的比較分析
陳志江 阮紫曦 程 楠 肖利娟, 彭 亮, 韓博平, 雷臘梅,
(1. 暨南大學生命科學技術學院生態學系, 廣州 510632; 2. 廣東省水庫藍藻水華防治中心, 廣州 510632)
拉氏擬柱孢藻(Cylindrospermopsis raciborskii,簡稱擬柱孢藻)為念珠藻科, 柱孢藻屬, 最近有研究者建議將尖頭藻和擬柱孢藻合并, 稱該藻為拉氏尖頭藻(Raphidiopsis raciborskii)[1]。擬柱孢藻是一種具有固氮能力的絲狀水華藍藻, 對氮、磷營養鹽的利用效率高, 對溫度和光照波動的適應性強, 目前已廣泛分布世界各地的淡水水體中, 可在熱帶地區的湖泊和水庫中常年形成水華, 在亞熱帶和溫帶水體形成季節性水華[2,3]。擬柱孢藻還可產生擬柱孢藻毒素(Cylindrospermopsin, CYN)和石房蛤毒素(Saxitoxin, STX)等藍藻毒素, 嚴重危害人類健康和淡水生態系統安全[4—6]。
全基因組測序是對未知基因組序列的物種進行個體基因組測序的一項技術, 1977年第一代測序技術誕生, 至21世紀以Roche公司的454技術、Illumina公司的Solexa技術和ABI公司的SOLiD技術為代表的第二代高通量測序的廣泛應用, 極大推動了不同物種基因組研究進展[7], 隨后的第三代技術向序列讀長長, 精度高方向發展[8]。全基因組測序可揭示基因組的復雜性和多樣性, 對了解一個物種的分子進化、基因組成和基因調控等方面具有重要的意義[9]。目前三代測序技術已在水華藍藻中得到廣泛應用, 如2016年發布的片狀微囊藻(Microcystis panniformis)FACHB-1757基因組數據[10]及2018年發布的銅綠微囊藻(Microcystis aeruginosa)NIES-2481基因組數據[11], 都使用了三代測序技術。研究者們利用測序技術對藍藻基因組的解析促進了他們對該物種的生態學、生理學和毒素合成機制等相關特性的了解, 同時基于比較基因組的分析進一步揭示了它們的全球地理分布與進化起源[12,13]。
相比較而言, 擬柱孢藻的基因組研究相對較晚。2009年, 美國國立生物技術信息中心(NCBI)公布了最早完成全基因組測序的擬柱孢藻藻株CS-505(C. raciborskiiCS-505), 該藻株的基因組相對較小, 為3.89 Mb, 在當時已完成全基因組測序的絲狀藍藻中, 擬柱孢藻是除尖頭藻外基因組最小的絲狀藍藻[14]。Stucken等[14]通過基因組分析發現擬柱孢藻CS-505基因組存在水平基因轉移, 并探討了其絲體形成、異形胞發育及固氮能力的分子基礎。Sinha等[15]于2014年通過比較基因組發現澳大利亞產毒與非產毒擬柱孢藻的最大區別是擬柱孢藻毒素合成酶基因簇是否存在。隨著擬柱孢藻在全球的快速擴張, 人們對更多來源不同的擬柱孢藻開展了全基因組測序, 如2018年Willis等[16]和Abreu等[17]分別進行了同一地區的9株擬柱孢藻和不同地區的8株擬柱孢藻的比較基因組分析, 以期為該藻靈活的生理生態策略、個體發育及逆境應答等研究提供參考[16,17]。
磷是藍藻生長必需的營養元素, 分無機磷、有機磷兩大類, 藍藻基因組中存在多種用于吸收不同磷源的機制[18], 其中低親和力無機磷轉運系統存在于部分藍藻中, 以適應在高磷環境下吸收無機磷,高親和力無機磷轉運系統在藍藻中普遍存在, 可在低磷環境下吸收無機磷。此外, 藍藻中還存在雙組分調節系統用于調節磷代謝、有機磷酸鹽轉運復合體介導磷酸酯的轉運、C-P裂解酶裂解磷酸酯和堿性磷酸酶特異性水解磷酸單酯[18—22], 藍藻的磷調控機制高度多樣化, 以適應不同的生活環境[18]。擬柱孢藻擁有快速吸收和儲存無機磷的能力, 不但能在高磷環境中生長良好, 還能適應低磷環境, 在無機磷濃度極低的環境中占優勢[6,23,24], 已有研究發現在擬柱孢藻CS-505和CS-506中存在以上完整的磷調控機制[25]。目前關于擬柱孢藻磷代謝基因排列特征的描述較為匱乏, 從基因組水平上分析擬柱孢藻磷代謝途徑基因將加深對擬柱孢藻生存優勢形成的認識, 進一步了解磷代謝以及磷在基因型選擇和進化中發揮的作用[18]。
迄今為止, 美國國立生物技術信息中心(NCBI)數據庫已有27株擬柱孢藻全基因組測序信息, 其中一半以上的藻株來自澳大利亞, 目前尚未有中國地區擬柱孢藻全基因組數據的發布。近年來, 擬柱孢藻在廣東省大部分水庫常年存在并取代微囊藻成為危害該地區水庫供水安全的首要藍藻, 在廣東省江門市鎮海水庫中常年優勢度可達93.5%, 目前已有的研究多關注于環境因子對擬柱孢藻優勢形成的影響[21,22]。本文以從鎮海水庫中分離的擬柱孢藻N8為材料, 進行測序及基因組分析, 并針對其親緣關系、毒素合成基因和磷吸收轉運基因進行了初步探索。本研究的開展有助于了解華南地區擬柱孢藻的基因組特性, 豐富擬柱孢藻基因組數據庫,從分子水平上加深人們對擬柱孢藻生態優勢形成的認識, 為有效防控擬柱孢藻水華提供理論依據。
1 材料與方法
1.1 擬柱孢藻N8的來源與培養
擬柱孢藻N8分離自廣東省江門市鎮海水庫(22°34′7′′N、112°32′58′′E), 其藻絲體筆直不彎曲,可形成端生的長圓形或卵圓形異形胞[21]。藻株置于光照強度為35 μmol/(m2·s)、溫度為(25.5±1)℃、光暗周期比為12h﹕12h的培養箱中進行培養, 使用的培養基為BG11[25]。
1.2 擬柱孢藻N8基因組DNA的提取
使用植物基因組DNA提取試劑盒(天根生化科技有限公司)按照制造商的說明從樣本中提取高質量的基因組DNA。使用Nanodrop One核酸分析儀測定DNA樣品260和280 nm處的光吸收值, 根據A260/A280的比值確定DNA純度(1.8 使用PacBio RS測序系統的SMRT(Single Molecule Real-Time)[26]技術進行測序, 利用PacBio?SMRTbellTMTemplate Prep Kit(PacBio, 美國)構建文庫, 在PacBio RS II平臺上進行測序反應。用Pac-Bio SMRT Analysis 5.1[23]軟件進行質控并組裝成一條環狀的基因組序列。以上工作委托北京諾禾致源生物科技有限公司完成。 使用RepeatMasker 4.1.2[27]、RNAmmer 1.2[28]、tRNA-scan-SE 2.0[29]和GeneMarkS 4.28[30]分別進行重復序列、非編碼RNA、tRNA和編碼基因的預測;后將編碼基因與COG數據庫[31]、SwissProt蛋白庫[32]、GO數據庫[33]和KEGG數據庫[34]進行比對, 使用Circos 0.69軟件[35]繪制全基因組圖譜。 我們挑選了24株來自不同產地及不同產毒特性擬柱孢藻全基因組單拷貝基因進行親緣關系分析, 手動將同一株藻的全基因組單拷貝基因序列進行排序和串聯, 采用MEGA 6.0[36]對25株藻的全基因組單拷貝基因進行序列比對, 以串聯序列為材料利用iqtree軟件包構建系統發育樹, 程序中的ModelFinder模塊自動計算出最大似然法(ML)的最佳擬合進化模型為GTR+F+G4模型, 系統樹各分支的置信度由重復抽樣法(Bootstrap)1000次重復檢測,同時使用MEGA 6.0構建最大似然(ML)進化樹, 以確定進化樹的可靠性, 進化樹的編輯器使用iTOL的在線版本(https://itol.embl.de)。 通過antiSMASH 6.0軟件(微生物次生代謝物合成基因簇數據庫)[37], 結合注釋信息搜索對應于次生代謝物非核糖體肽合成酶(NRPS)、聚酮化合物合酶(PKS)產生的CYN合成酶基因簇和STX合成酶基因簇。 根據KEGG代謝通路注釋結果結合基因的序列相似性和位置相關性, 找到對應的核苷酸/蛋白質序列, 放到NCBI上的Nucleotice/Protein Blast上比對, 從而確定基因組中磷吸收轉運基因, 包括雙組分調節系統, 低親和力、高親和力無機磷轉運系統,有機磷酸鹽轉運復合體, C-P裂解酶和堿性磷酸酶基因, 繪制基因簇排列圖。 經過我們的分析, 挑選出8株擬柱孢藻進行磷吸收轉運基因分析, 8株藻株分別代表了三大地區(歐亞地區、南美地區和澳大利亞地區)和不同產毒特性(產CYN、STX和非產毒)的擬柱孢藻。 擬柱孢藻N8的基因組覆蓋率為121, 平均測序質量為0.85, Contig N50值為3865242 bp, 基因組大小為3857174 bp, 呈現一條環狀的染色體, GC含量為40.13%, 注釋得到的基因總數為3598, 結構RNA基因為51個, 其中tRNA基因42個, 5S-16S-23S rRNA基因各3個(圖 1)。該藻株的全基因組序列已上傳至NCBI數據庫, 索引編號為GCA_018139025.1。 圖1 擬柱孢藻N8的全基因組圖譜Fig. 1 The whole genome map of C. raciborskii N8 COG注釋(圖 2a): 在擬柱孢藻N8功能分布的基因數目中, 細胞壁/膜/包膜合成相關基因(M)有210個, 所占比例最高, 為5.92%; 而染色質結構與動力學相關基因(B)只有1個, 所占比例最低, 為0.03%。 KEGG注釋(圖 2b): KEGG中PATHWAY數據庫注釋到擬柱孢藻N8涉及的基因通路有142條, 其中完整的或者有關鍵酶可以完成代謝的通路有102條, 新陳代謝系統部分存在的完整通路最多, 有76條。 GO注釋(圖 2c): 擬柱孢藻N8在分子功能類注釋到了2814個相關基因, 其中所占比例較大的有黏合物相關基因1135個、催化活性相關基因1267個及運輸活性相關基因137個, 占該組分基因的90.23%; 在細胞學組件分類部分注釋到了2190個相關基因, 其中所占比例較大的有細胞器相關基因177個、高分子復合物相關基因236個、復雜大分子及細胞和細胞元件相關基因各783個, 占該組分注釋基因的90.37%; 在生物學途徑類注釋到了4869個相關基因, 其中占比較大的有代謝過程相關基因1417個; 細胞過程相關基因1332個、定位相關基因365個、形成定位相關基因358個、生物調節相關基因329個、生物過程監管相關基因、應激反應相關基因220個和細胞成分組織或生物起源相關基因122個, 占該組分注釋基因的85.01%。 圖2 擬柱孢藻N8的COG (a)、KEGG (b)和GO (c)數據庫注釋圖Fig. 2 COG (a), KEGG (b) and GO (c) database notes of C. raciborskii N8 在NCBI數據庫中搜尋了27株擬柱孢藻的全基因組序列(其中CS-505和CS505為同一藻株, 采用了不同測序方法, 部分藻株數據未完善故未采納), 表 1顯示了27株擬柱孢藻的基因組基本信息。由表 1可知, 當前數據庫中完成測序的藻株主要來自澳大利亞和巴西, 分別有13株和6株, 烏拉圭和美國各有2株, 新加坡、韓國、荷蘭各有1株。不同來源的擬柱孢藻基因組的GC含量穩定, 維持在40.1 %左右,大小在2.74—4.16 Mb, 其中美國藻株LB2897的基因組最小, 約為2.749 Mb, 澳大利亞藻株CS505基因組最大, 約為4.159 Mb。在這些藻株中(除KL1藻株產毒未知外), 僅8株藻株不產毒, 18株藻株為產毒株, 其中來自南美的巴西和烏拉圭的擬柱孢藻產毒素STX, 來自澳大利亞(11株)和新加坡(1株)的擬柱孢藻產毒素CYN及其異構體。采用的測序技術以第二代測序技術為主, 分為Roche 454技術、Illumina Solexa技術和ABI SOLID技術, 擬柱孢藻N8、CS505和GIHE 2018采用第三代測序技術—SMRT技術。 表1 NCBI數據庫中已公布的擬柱孢藻藻株基因組基本信息Tab. 1 Basic information on the genome of C. raciborskii strains downloaded from NCBI 基于擬柱孢藻的全基因組單拷貝基因構建了擬柱孢藻N8的親緣關系樹, 采用最大似然法(ML)。由圖 3可知24株擬柱孢藻有3大分支, 來自南美地區的擬柱孢藻屬第Ⅰ分支, 來自亞歐地區的擬柱孢藻屬第Ⅱ分支, 來自澳大利亞地區的擬柱孢藻屬第Ⅲ分支。 圖3 基于全基因組單拷貝基因構建的系統進化樹(ML)Fig. 3 Phylogenetic tree based on whole genome single copy gene construction (ML) 擬柱孢藻N8屬第Ⅱ分支, 與來自韓國的擬柱孢藻GIHE 2018的親緣關系最近。 在擬柱孢藻N8的基因組中未發現CYN合成酶基因簇和STX合成酶基因簇, 推斷擬柱孢藻N8不能產生毒素CYN和STX。 對N8和其他7株來自不同地區的擬柱孢藻進行比較分析[Cr2010(荷蘭), GIHE 2018(韓國), CS505、CO3和SO1(澳大利亞), CENA302和MVCC19(南美洲)], 結果表明不同地區的擬柱孢藻, 其磷代謝基因拷貝數目不同, 磷調控多樣化。研究的8株擬柱孢藻基因組中都存在雙組分調控系統phoB和phoR, 低親和力無機磷轉運系統與高親和力轉運系統。多數藻株有2個有機磷酸鹽轉運系統phnD-C-E, 來自巴西的擬柱孢藻CENA302只有1個phnD, 擬柱孢藻N8比其他藻株多1個轉錄調節蛋白-phnF和1個有機磷酸鹽代謝蛋白(金屬依賴性)-phnM。8株藻中大多有2個或2個以上phoA堿性磷酸酶基因, 來自荷蘭的擬柱孢藻Cr2010僅有1個phoA基因拷貝(表 2)。 表2 八株擬柱孢藻磷吸收、調節、轉運、代謝基因及拷貝數Tab. 2 Phosphorus absorption, regulation, transport, metabolism genes and copies of eight C. raciborskii strains 如圖 4和圖 5所示, 8株擬柱孢藻的高親和力無機磷轉運系統按照pstS-C-A-B順序排列的,sphX在擬柱孢藻的位置不定, 可位于pstS-C-A-B的上游或下游, 擬柱孢藻的雙組分調節系統phoB-phoR形成簇, 擬柱孢藻的2個有機磷酸鹽轉運操縱子phnC-DE, 兩個轉運操縱子的方向可同可不同, C-P 裂解酶基因按phnFHGIJKLM排列, 除擬柱孢藻N8和擬柱孢藻GIHE 2018外, 其余藻株的phnFH與phn-GIJKLM簇距離較遠。 圖4 8株擬柱孢藻的雙組分調節系統和高親和力無機磷轉運系統Fig. 4 Two-component regulatory system and high affinity inorganic phosphorus transport system of eight strains of C. raciborskii 圖5 8株擬柱孢藻的有機磷酸鹽轉運復合體和C-P裂解酶基因Fig. 5 Organic phosphonate transport complex and C-P lyase gene of eight strains of C. raciborskii 自Stucken等[14]發布第一個擬柱孢藻全基因組信息后, 近10年來陸續有不同來源的擬柱孢藻完成測序。已公布全基因組序列的擬柱孢藻主要來自澳洲和南美洲, 2019年, 2020年發表了荷蘭藻株Cr2010[43]、韓國藻株GIHE2018[42]、美國藻株LB2897與KL1[45,47], 目前NCBI數據庫上還沒有中國地區擬柱孢藻的全基因組信息, 本研究測序的擬柱孢藻N8將成為中國地區首株具有全基因組信息的藻株。 擬柱孢藻N8與NCBI上已公布的其他國家的藻株在基因組大小、GC含量和基因數量等基本特征較為一致, NBCI上已發布的擬柱孢藻大都采用第二代測序法進行測序, 僅CS505、GIHE 2018藻株采用了三代測序技術。二代測序技術具有高效率、高通量和高精度的特點, 應用廣泛, 但拼接的基因組受序列讀長的限制, 其結果的準確性依賴于較高的測序覆蓋度和準確的序列拼接技術[48]。擬柱孢藻N8采用第三代測序技術, 該技術改進了序列組裝, 序列讀長長, 精度高, 在測序的同時能夠獲取全基因組甲基化修飾信息, 其測序結果也相對完善[8]。N8的contig N50值為3865242 bp, 這在通過Pac-Bio平臺測序的已公布的擬柱孢藻基因組中, 擬柱孢藻N8的N50值僅次于CS505的4011384 bp, 測序得到N8的基因組大小為3.857 Mb, 與澳大利亞藻株S01、S14等基因組大小相近, 其GC含量穩定, 維持在40.1 %左右。擬柱孢藻N8的編碼基因有3598個,這可能與擬柱孢藻N8的重疊基因較多, 除核心基因組外, 本身也需更多特異的編碼蛋白維持機體功能相關[14—17]。但也可能是本研究采用了精度更高的測序技術, 且隨著數據庫的不斷完善, 從而有更多編碼蛋白被檢測到。 擬柱孢藻N8數據庫注釋結果與其他擬柱孢藻的注釋結果有極大相似性[15,17], 具有注釋功能的基因主要涉及: 蛋白質代謝、協同因子和維生素代謝、氨基酸及其衍生物、碳水化合物代謝。但不同產地、不同形狀、不同產毒特征的藻株注釋到的基因比例不同[15—17], 如來自南美藻株在生物學途徑分類部分中涉及刺激反應、化學反應的基因占比最多, 非南美藻株在細胞學組件分類部分的膜基因占比最多, 產STX藻株在細胞學組件分類部分的包膜基因占比最多, 產CYN藻株在細胞學組件分類部分的膜基因占比最多[17], 卷曲藻株基因組中存在直藻株所沒有的與卷曲形態相關的涉及碳水化合物代謝的蛋白質[16], 擬柱孢藻N8在COG注釋中, 細胞壁/膜/包膜合成相關基因所占比例最多。在GO注釋中, 細胞學組件分類部分所占比例較大的是高分子復合物相關基因、復雜大分子及細胞和細胞元件相關基因; 生物學途徑類占比最多的是代謝過程相關基因。注釋基因比例的不同反映了擬柱孢藻間不同的生態適應與生理可塑性能力[14]。 目前擬柱孢藻的全基因組數據相對匱乏, 非洲藻株尚未有全基因組數據發表。在探討世界范圍內擬柱孢藻的系統進化和起源時, 有研究認為多個遺傳標記的共建樹比單個遺傳標記的區分能力更強大, 例如Haande等[53,54]發現pc-IGS、nifH、ITSL和rpoC1等單個基因建樹不能區分澳大利亞、非洲和歐洲的藻株, 但4個遺傳標記共建樹能夠將非洲、澳大利亞、歐洲和美洲的藻株完全區分, 基于多個基因構建的系統發育樹會比單個基因構建的系統發育樹的結果更加可靠, 我們通過全基因組單拷貝基因構建擬柱孢藻N8的系統發育樹(ML), 發現來自廣東省的擬柱孢藻N8與亞歐地區的擬柱孢藻GIHE 2018、CR12和Cr2010的親緣關系相近, 可見同種不同株系的擬柱孢藻, 雖然地理距離隔得遠,但從枝長信息看, 親緣關系也很接近。 產毒與非產毒藻株的最大的區別是非產毒藻株中沒有毒素合成酶基因簇的存在[15], 擬柱孢藻N8基因組里沒有發現CYN合成酶基因簇和STX合成酶基因簇, 推斷N8不產毒素CYN和STX。在NCBI公布的全基因組數據里, CS-508、CS-509、CYLP、GIHE 2018和LB2897也均為非產毒藻株[15,38,41,42,45]。 擬柱孢藻N8分離自廣東省鎮海水庫, 該水庫中TN/TP比為37—243, 顯著高于浮游植物和大多數其他生物對氮、磷兩種元素的相對需求-Redfield比(N/P=16), 表明該水庫全年均處于磷限制性狀態[24],而擬柱孢藻在鎮海水庫中常年的優勢度可達93.5%,表明該藻能適應低磷環境。我們的前期研究也發現, N8藻株對磷的適應范圍很寬, 在0.02—5.12 mg/L磷濃度下均能生長[23]。本研究進一步證實擬柱孢藻N8的基因組擁有完整的磷吸收代謝通路, 可能是該藻能適應低磷和磷波動環境的分子基礎。 Bai等[55]比較了非產毒的擬柱孢藻Cylindrospermopsis raciborskiiFACHB 1096和銅綠微囊藻Microcystis aeruginosa205在不同磷源的生長情況,在K2HPO4, β-甘油磷酸酯, (2-氨基乙基)-次膦酸、草甘膦和無磷的條件下, 擬柱孢藻FACHB 1096的比生長速率和內源性堿性磷酸酶活性比銅綠微囊藻 205的更高, 這側面反映了擬柱孢藻比其他藻類擁有更有效的磷利用機制。擬柱孢藻擁有高親和力無機磷轉運系統, 該系統在藍藻中保守存在, 排列方式為pstS-C-A-B[18], 受低磷環境的影響, 有些藍藻可擁有多個高親和力無機磷轉運系統[50], 在擬柱孢藻N8基因組中僅有一個完整的pstS-C-A-B基因簇, 額外存在pstS拷貝, 盡管存在多個pstS拷貝, 但一般只有一個pstS的基因表達能對磷變化做出有效性的響應[51]; 在某些藻類基因組中, 高親和力無機磷轉運復合體還包括了1個磷酸鹽結合亞基sphX[18,52],sphX與pstS-C-A-B形成操縱子, 擬柱孢藻N8的sphX位于pstS-C-A-B上游, 擬柱孢藻N8具有較高的磷親和性, 在低磷環境下能維持生長[23],sphX的存在與否及位置差異在不同藍藻中情況不同[18], 是否對磷吸收親和力產生影響還需進一步實驗驗證。低親和力無機磷轉運系統在藍藻中不是普遍存在的[20], 而擬柱孢藻N8與其他擬柱孢藻一樣擁有低親和力無機磷轉運系統[25], 推斷該系統的存在有利于其在高磷環境下吸收無機磷, 更能適應環境中磷濃度的變化[6,23,25]。擬柱孢藻N8的C-P裂解酶基因按phnFHGIJKLM排列, 有2個操縱子phnCDE, 用于有機膦的吸收和同化, 擬柱孢藻N8比其他7株藻株多一個phnF與phnM, 這可能導致藻株間有機磷轉化能力的差異[48,51]。擬柱孢藻N8中編碼堿性磷酸酶基因為phoA, 有2個拷貝, 與其他擬柱孢藻的堿性磷酸酶拷貝數不一致[50], 藍藻中的堿性磷酸酶活性可以隨磷濃度大小而變化的, 拷貝數目不同反映了藻株間不同的有機磷利用效率[22]。由以上討論可見,擬柱孢藻N8的磷吸收轉運通路在基因拷貝數目與排列順序等與其他藻株存在差異, 今后可從比較基因組學結合生理實驗入手, 進一步探討不同擬柱孢藻間磷吸收調節機制, 從而解析擬柱孢藻在磷限制水體中占優勢的原因。1.3 全基因組測序與組裝
1.4 基因組注釋
1.5 親緣關系分析
1.6 產毒潛力分析
1.7 磷吸收轉運基因分析
2 結果
2.1 擬柱孢藻N8基因組的基本信息

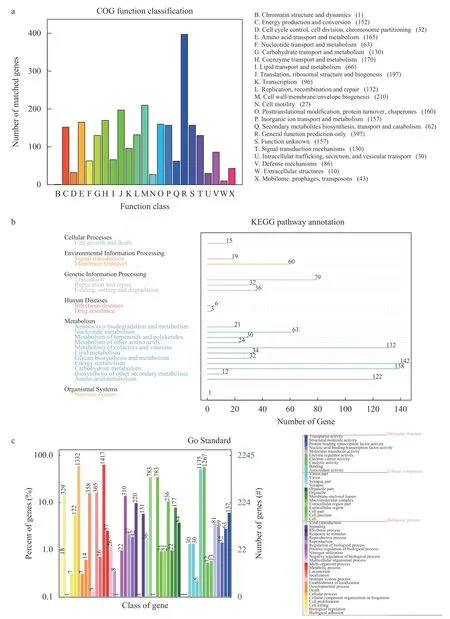
2.2 擬柱孢藻N8的數據庫注釋
2.3 擬柱孢藻基因組基本信息的比較

2.4 親緣關系分析

2.5 擬柱孢藻N8的毒素合成酶基因簇分析
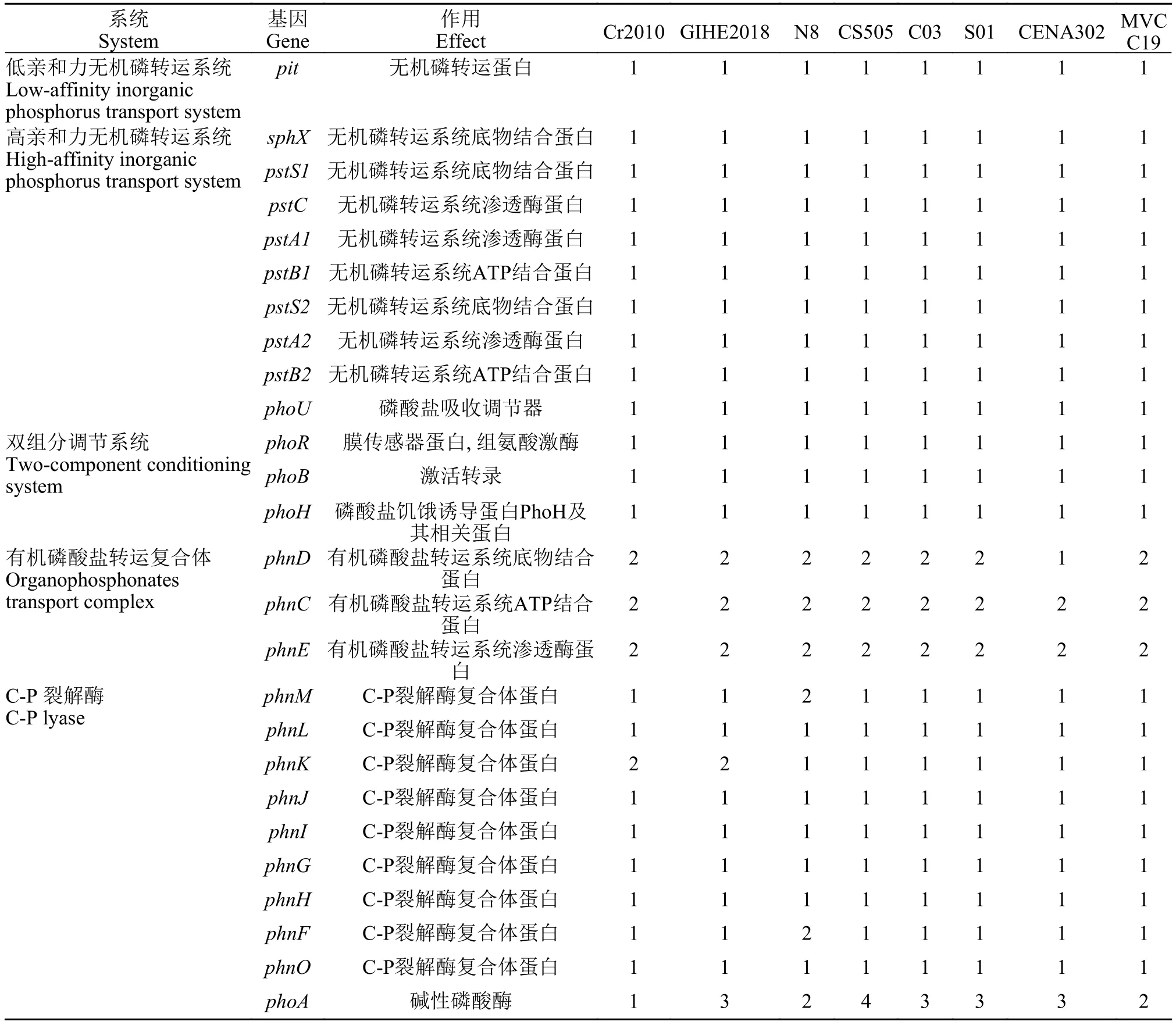
2.6 八株擬柱孢藻的磷代謝基因組成分析

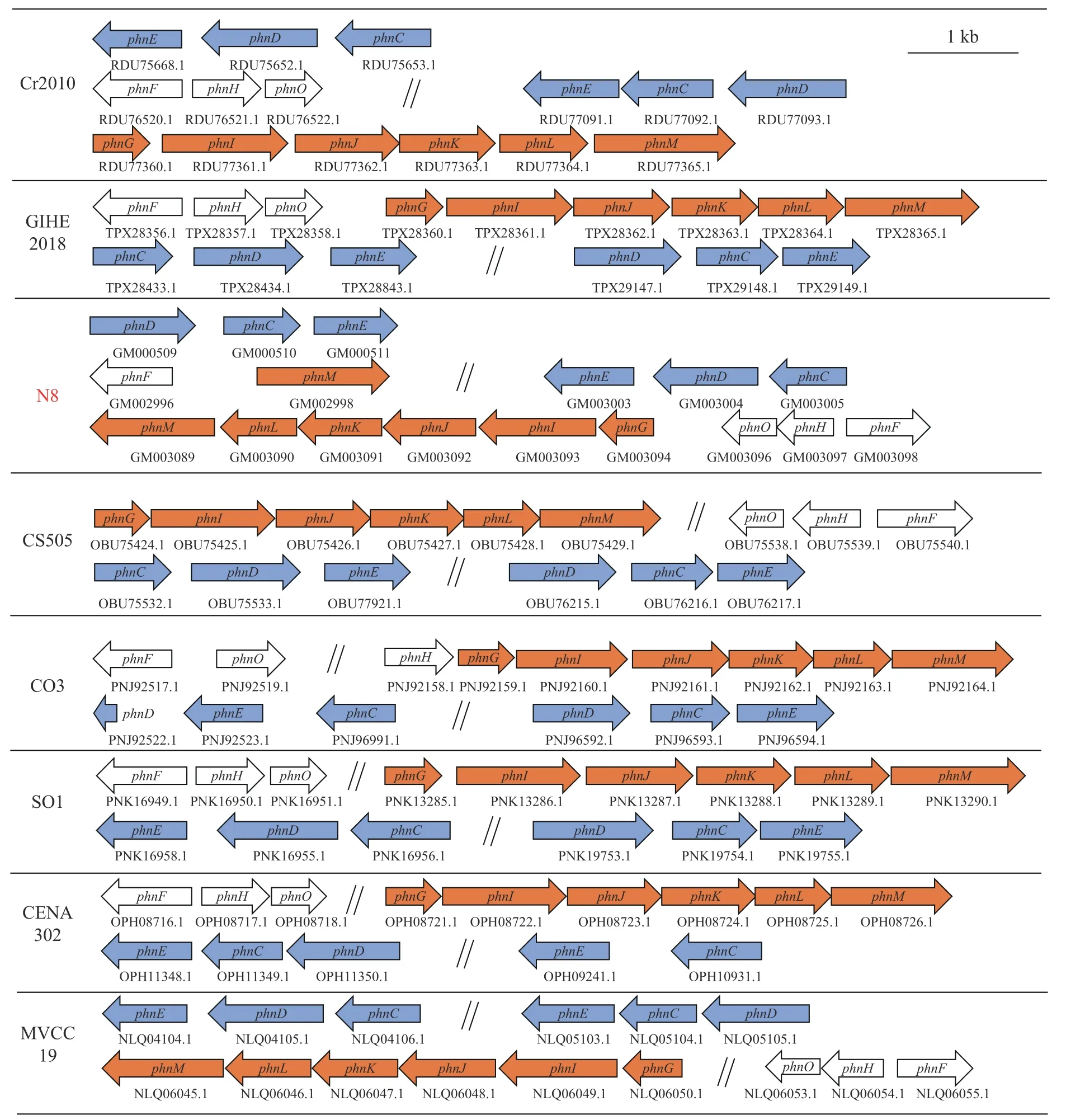
3 討論
3.1 擬柱孢藻的基因組特征
3.2 擬柱孢藻的親緣關系與產毒特征
3.3 擬柱孢藻的磷吸收代謝通路

