基于4個線粒體基因的不同海拔高原林蛙群體遺傳多樣性研究
鄭海錢,徐志旺,徐康寧,李卉,李樹然,張永普
(溫州大學生命與環境科學學院,浙江 溫州 325035)
受人類活動的影響,地球環境正面臨前所未有的巨變,工業革命后劇烈的氣候變化對全球生態系統產生了巨大的負面影響(Hoegh-Guldberg.,2019)。由于獨特的生活史特征,兩棲動物不僅是水生生態系統中重要的生物類群,還是聯系水生和陸生環境的代表(徐士霞等,2004)。受環境變化的影響,全球超過2 000種兩棲動物的生存正面臨威脅,其中一半可能極度瀕危或瀕臨滅絕(González-Del-Pliego.,2019)。群體滅絕率的升高與遺傳多樣性的喪失密切相關(Saccheri.,1998),低遺傳多樣性的群體通常表現出低適合度和高滅絕率(Markert.,2010)。研究兩棲動物的遺傳多樣性有助于評估現有物種或群體的生存狀態,從而為保護策略的制定提供科學的理論指導(崔朝霞等,2011)。
mtDNA以結構簡單、進化速率較快、嚴格遵循母系遺傳,以及幾乎不發生重組的特點,被廣泛應用于系統進化研究和生物遺傳多樣性評價(Avise.,1987;宋潔等,2008;Chen.,2009;楊文嘉,2015;杜民等,2021)。其中,部分mtDNA單個基因的分析已經得到普遍應用,但是不同基因進化速率往往存在差異,c是唯一由mtDNA編碼的細胞色素,參與線粒體呼吸鏈電子傳遞,進化速率適中;基因是mtDNA中比較保守的部位,進化速率略低于基因;而線粒體基因和基因最保守(Hwang.,1999)。這導致同一物種采用不同基因分析可能會得出不同的結論,因此采用多基因聯合分析物種系統發育與遺傳多樣性,以期得到更準確的結果(董依萌等,2020)。
青藏高原以其特殊的地理位置和氣候條件(如溫度、氧濃度、紫外線輻射等),孕育了一大批獨特的生物資源,使青藏高原成為中國重要的生態安全屏障,以及研究生物對高原環境適應性進化的熱點地區(魯春霞等,2004;高偉等,2019)。高原林蛙是青藏高原常見的一種兩棲動物,分布海拔為2 000~4 400 m,背面皮膚較粗糙,呈灰褐色、棕褐色、棕紅色或灰棕色。對高原林蛙的研究集中于活動特征(齊銀等,2007a,2007b)、種間競爭(張晉東等,2007;陳偉等,2018)、生活史(Zhao.,2014;Yu.,2018)和種間遺傳關系(Yang.,2017;Wang.,2020)等方面,遺傳多樣性研究相對較少(Zhou.,2013),海拔梯度差異研究更少。本研究基于線粒體、、和基因對不同海拔(2 000 m、2 600 m、3 200 m和3 800 m)的高原林蛙群體進行遺傳多樣性研究,并通過對和2個蛋白編碼基因的正選擇分析,探討高原林蛙的高原適應機制,以期為其保護提供科學依據。
1 材料與方法
1.1 樣品采集
樣品于2020年7—8月和2021年7月采自青海省民和縣 巴 州 鎮(100°45′E,36°12′N,海 拔2 000 m;=22)、海南藏族自治州共和縣龍羊峽水庫(100°41′E,36°4′N,海拔 2 600 m;=30)、共和縣湖東種羊場(100°46′E,36°42′N,海拔 3 200 m;=24)和果 洛 藏 族 自 治 州 瑪 沁 縣(100°14′E,34°27′N,海拔3 800 m;=24),分別定義為2 000 m群體、2 600 m群體、3 200 m群體和3 800 m群體。剪取高原林蛙左后肢的第三趾(Perry.,2011),95%乙醇固定后-80℃保存。
1.2 DNA提取,PCR擴增與測序
采用EZUP柱式動物基因組DNA抽提試劑盒(上海生工生物工程股份有限公司),并參考說明書,提取樣本總DNA,將DNA溶于CE Buffer中,用超微量核酸蛋白測定儀(Nano-600,上海嘉鵬科技有限公司)檢測濃度,合格樣品-80℃保存。
PCR 反應體系為 25 μL:2×Taq PCR StarMix(北京康潤誠業生物科技有限公司)12.5 μL,去離子水9.5 μL,DNA模板1 μL,上、下游引物(表 1)各1 μL。PCR反應條件為:95℃預變性3 min;95℃變性30 s、退火30 s(退火溫度分別為:57.1℃、:50℃、:50℃、:58℃)、72℃延伸30 s,35個循環;72℃延伸5 min,通過1%瓊脂糖凝膠電泳(150 V,25 min)檢測PCR產物,送北京擎科生物技術有限公司雙向測序。
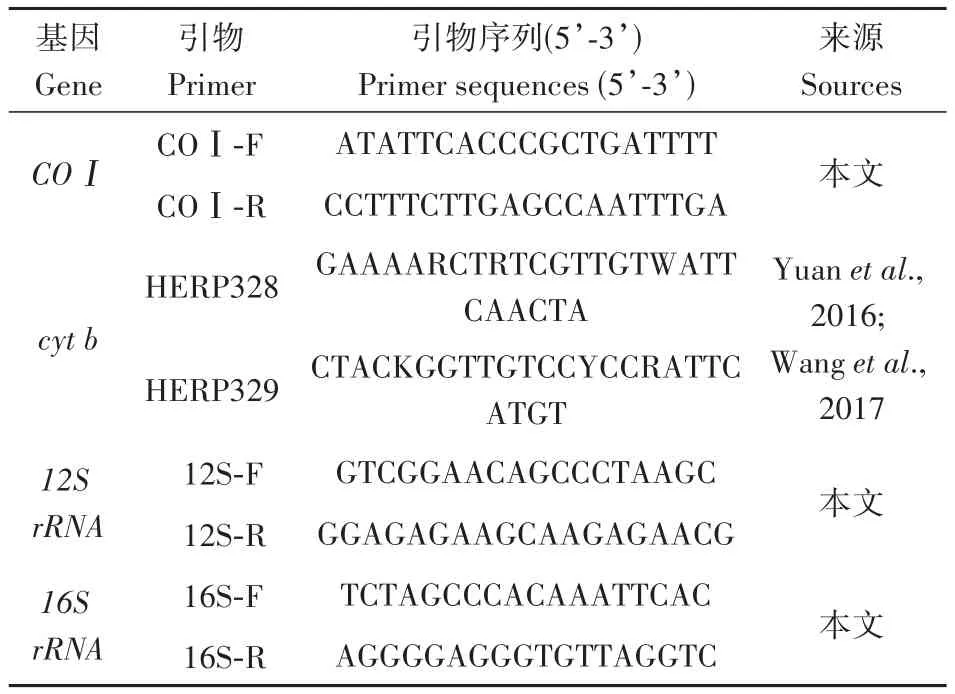
表1 本研究中使用的引物Table 1 The primers used in this study
1.3 數據處理
將所得雙向序列經ContigExpress拼接后與NCBI數據庫進行BLAST比對,排除假基因的可能,用DNAMAN(Ji.,2020)進行序列對比,剪去多余的片段,保存成fasta格式,使用Sequence Matrix將4個基因合并成聯合數據集。
利用DNASP 5.0(Librado.,2009)分析多態位點數、單倍型多樣性()、核苷酸多樣性()、平均核苷酸差異數、錯配分布;利用MEGA7.0(Kumar.,2016)分析堿基組成,計算變異位點數、簡約信息位點數、單突變位點;以倭蛙和桓仁林蛙為外 群(NCBI),用 IQtree構 建 最 大 似 然 樹(Nguyen.,2015)。
基于Easycodeml(Gao.,2019)分別對線粒體的2個蛋白編碼基因(和)進行選擇壓力分析,在分支模型上選擇位點模型,位點模型主要選用M0(單一比率)、M1a(近中性)、M2a(正選擇)、M3(離散)、M7(beta)、M8(beta& ω)、M8a(beta& ω=1),利用似然比檢驗(LRT)統計評估成對模型:M0 vs.M3、M1a vs.M2a、M7 vs.M8、M8 vs.M8a,檢測正選擇壓力。使用Arlequin3.5(Excoffier&Lischer,2010)計算群體遺傳分化系數(),并利用分子變異分析(analysis of molecular variance,AMOVA)檢測遺傳變異的來源,通過1 000次重復抽樣檢驗不同遺傳結構水平上協方差的顯著性,進行 Tajima’s和 Fu’s中性檢驗(Tajima,1989)評估高原林蛙群體動態,基因流()=(1-)/4(Slatkin.,1987)。通過 POPART(Leigh.,2015)繪制單倍型TCS網絡圖,推算各個單倍型間的進化關系。
2 結果與分析
2.1 線粒體基因序列特征遺傳多樣性分析
共得到100只高原林蛙的、、和基因序列片段,將每只個體的4條線粒體基因片段合并為1條長度為4 055 bp的線粒體序列,即4個線粒體基因聯合數據集。檢測到63個變異位點,其中單一信息位點和簡約信息位點分別有31個和32個,A+T含量為55.4%。
4個群體線粒體基因聯合數據集定義了34個單倍型,其中,2 000 m群體、2 600 m群體、3 200 m群體、3 800 m群體分別為10個、9個、5個、11個,共55個多態位點,、和平均核苷酸差異數分別為0.001 62±0.000 08、0.935±0.012和 6.443。3 200 m群體的遺傳多樣性最低,基于4個線粒體基因聯合數據集的和分別為0.587±0.102和0.000 19±0.000 05;2 600 m群體的遺傳多樣性最高,基于4個線粒體基因聯合數據集的和分別為0.860±0.036和0.001 21±0.000 21;2 000 m群體的遺傳多樣性高于3 800 m群體(表2)。
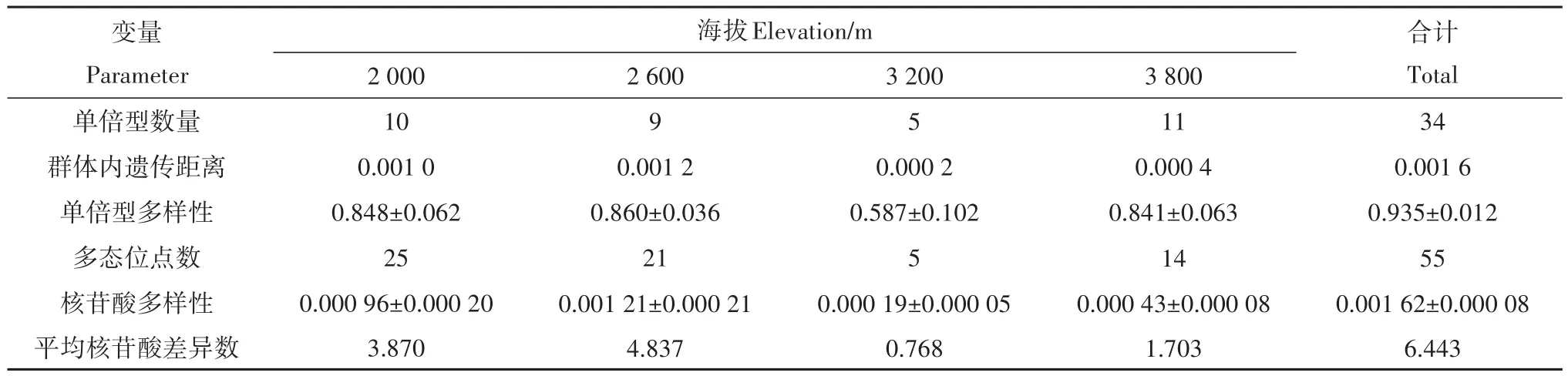
表2 基于線粒體基因分析青海省4個高原林蛙群體的遺傳多樣性Table 2 Genetic diversity of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
2.2 群體遺傳結構與遺傳分化
TCS單倍型網絡圖顯示(圖1),高原林蛙2 000 m群體和2 600 m群體組成一個進化單元。4個基因聯合建樹結果表明(圖2),系統發育樹分為2個支系,2 000 m群體多數樣本(15/22)、2 600 m群體多數樣本(24/30)和3 200 m群體全部樣本為1個支系,2 000 m群體少數樣本(7/22)、2 600 m群體少數樣本(6/30)和3 800 m群體全部樣本為另一支系。
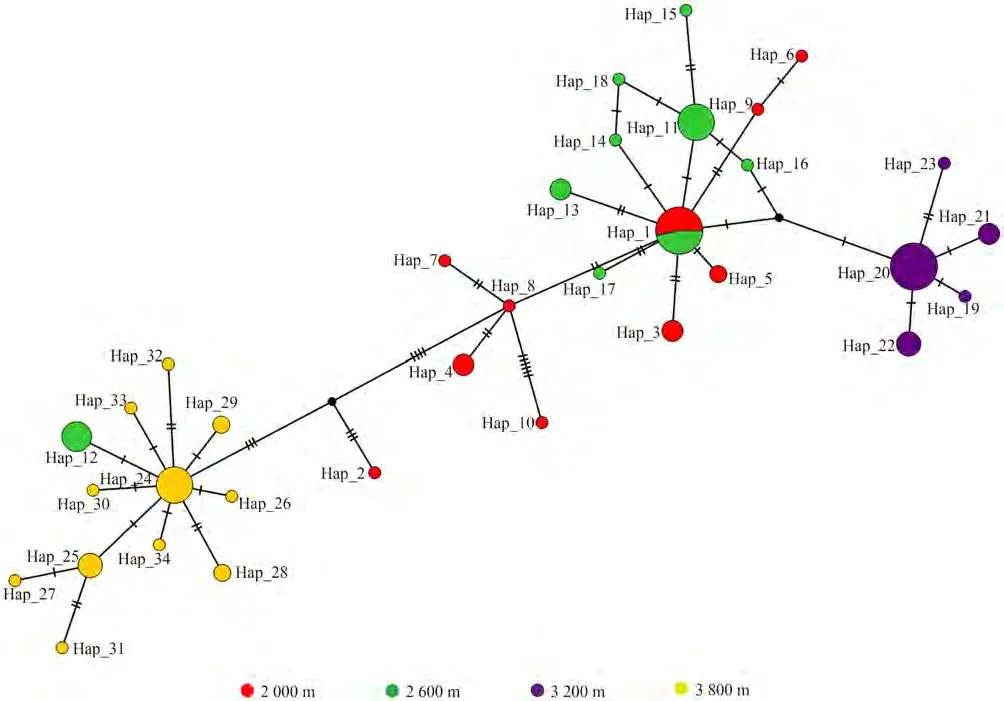
圖1 青海省4個高原林蛙群體線粒體基因的TCS網絡Fig.1 TCS network of mitochondrial gene of 4 Rana kukunoris populations in Qinghai Province
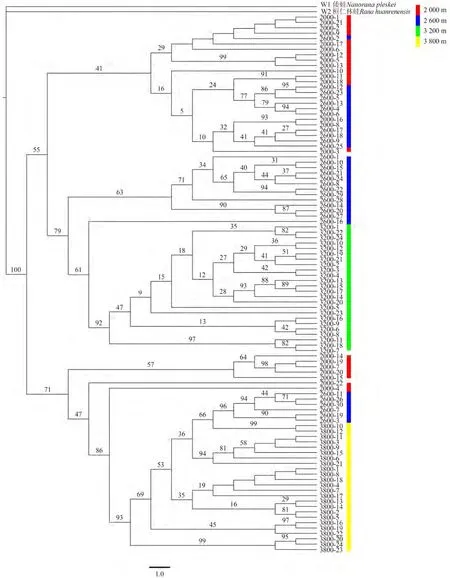
圖2 基于線粒體基因構建的青海省4個高原林蛙群體最大似然法系統發育樹Fig.2 Maximum-likelihood phylogenetic tree of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
群體間的為0.090~0.859,除了2 000 m群體和2 600 m群體外,其他兩兩群體間均發生了極顯著的遺傳分化(表3)。兩兩群體間的為0.09~2.53。AMOVA結果顯示(表4),線粒體基因群體間分子變異和群體內分子變異分別占63.01%和36.99%,分子變異主要發生在群體間。總為0.630,且<0.001。群體間的是 2.52。基于和基因的選擇壓力分析結果表明(表5),M8模型更靈敏,M8模型中,基因檢測到2個正選擇位點,包括2 600 m群體檢測到1個正選擇位點(天冬氨酸D→天冬酰胺N),3 200 m和3 800 m群體檢測到1個正選擇位點(天冬酰胺N→賴氨酸K),基因沒有檢測到正選擇位點。
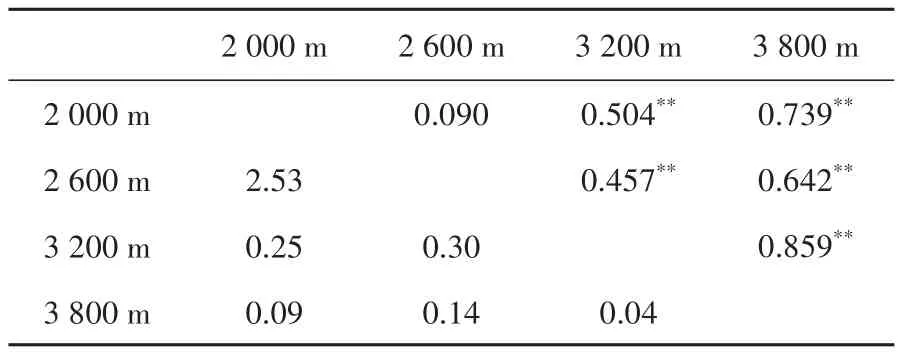
表3 基于線粒體基因的青海省4個高原林蛙群體間遺傳分化系數(對角線上)和基因流(對角線下)Table 3 Genetic differentiation coefficient(above the diagonal)and gene flow(below the diagonal)among 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
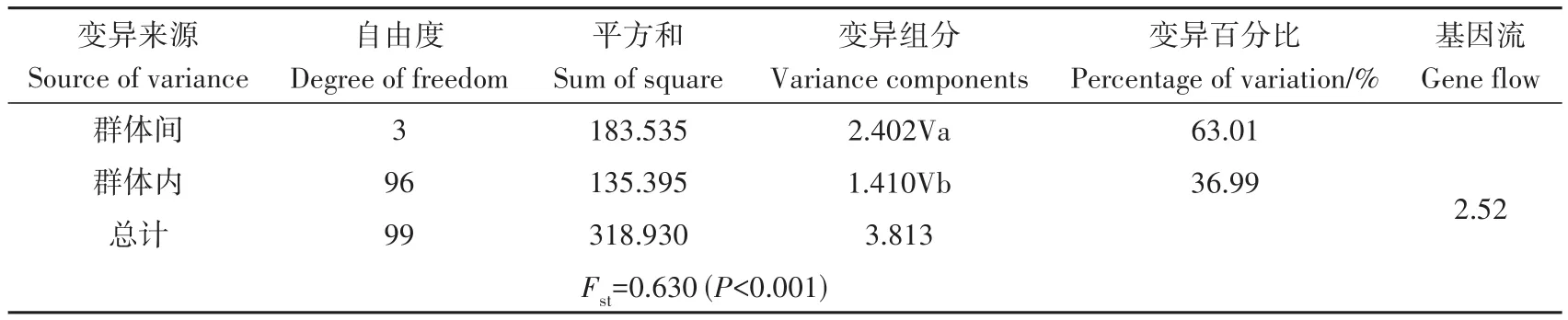
表4 基于線粒體基因的青海省高原林蛙4個群體分子方差分析結果Table 4 Results of molecular variance analysis of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
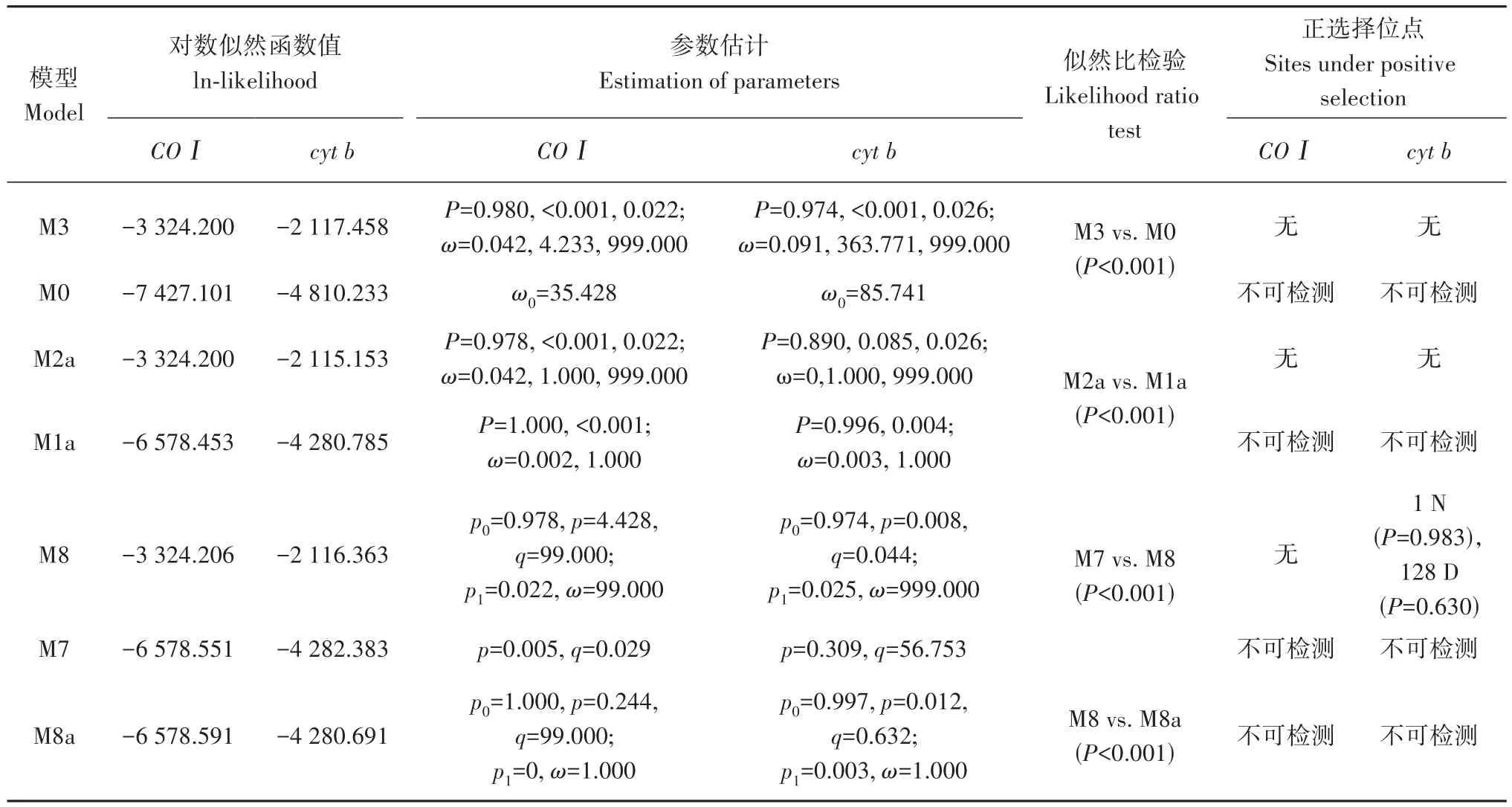
表5 青海省高原林蛙4個群體線粒體基因選擇壓力分析Table 5 Analysis of selection pressure of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
2.3 群體歷史動態
4個群體中,除2 600 m群體的Fu’s檢驗值為正值(0.877),其余3個群體均為負值,3 800 m群體的Tajima’s和Fu’s絕對值最高、2 600 m群體的最低,Tajima’s統計顯示2 000 m群體和3 800 m群體差異顯著,Fu’s統計顯示3 800 m群體差異顯著(表6)。基于線粒體基因聯合數據集的高原林蛙整體的Fu’s和Tajima’s均為負值,但差異都不顯著,就單個群體而言,大多數群體的Tajima’s與Fu’s均為負值,且只有少數群體出現顯著差異,另外,線粒體基因聯合數據集結果顯示(圖3),4個群體的錯配分布曲線呈雙峰。
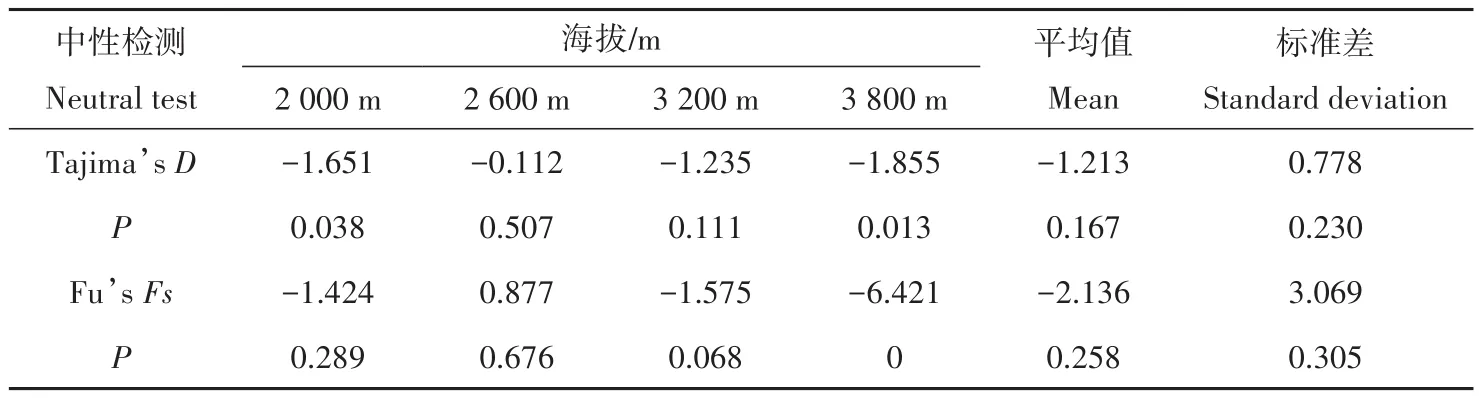
表6 青海省4個高原林蛙群體線粒體基因序列中性檢測Table 6 Neutral test of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
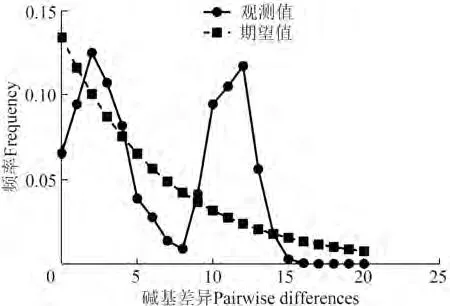
圖3 基于線粒體基因的青海省4個高原林蛙群體的錯配分布曲線Fig.3 Mismatch distribution curve of 4 Rana kukunoris populations in Qinghai Province based on mitochondrial genes
3 討論
3.1 高原林蛙群體的遺傳多樣性
和是衡量遺傳多樣性的2個重要參數(劉紹平等,2010)。Grant和Bowen(1998)提出,和的標準臨界值分別為0.5和0.005,將2個指數的搭配分為 4 種類型:<0.5,<0.005;>0.5,<0.005;<0.5,>0.005;>0.5,>0.005。基于線粒體4個基因聯合數據集分析的遺傳多樣性結果屬于第2種類型,這可能是高原林蛙群體曾經歷過瓶頸效應后,伴隨了迅速的群體擴張與變異的積累。這與北非綠蛙的遺傳多樣性相似(Farjallah.,2012)。此外,基于線粒體基因對不同地理群體的高原林蛙的遺傳多樣性研究表明,青藏高原北部地區適宜生境多于其他地區,其遺傳多樣性也高于其他地區(Zhou.,2013)。由此推測適宜生境的數量或面積影響遺傳多樣性,本研究中線粒體4個基因聯合數據集對不同海拔的高原林蛙遺傳多樣性結果表明,2 000 m群體和2 600 m群體的和高于其他群體,推測低海拔生境適宜度整體上高于高海拔,但整體遺傳多樣性仍處于較低水平。而物種的遺傳多樣性降低將導致群體生存能力降低,甚至滅絕(Pauls.,2013)。目前高原林蛙被世界自然保護聯盟(IUCN)列為無危(LC),但其較低的群體遺傳多樣性提示該物種同樣值得進一步關注。
3.2 群體遺傳結構和遺傳分化
系統發育樹的結果顯示,2 000 m和2 600 m群體的多數樣本以及3 200 m群體聚為一支,表明這3個群體的遺傳關系較近。基于4個線粒體基因聯合數據集的所有單倍型中,無4個群體共享單倍型,且大部分單倍型是群體獨有,說明在進化過程中,每個群體為了適應外界環境形成了獨特單倍型。3 800 m群體的單倍型數量高于其他群體,可能是高海拔地區人煙稀少、人為干擾較少,自然環境中高原林蛙的基因交流較頻繁。AMOVA結果表明,群體間的差異大于群體內,遺傳分化主要發生在群體間。Wright等(1978)建議:為0~0.05表示群體間遺傳分化很小、0.05~0.15表示存在中等程度的遺傳分化、0.15~0.25表示遺傳分化較大、>0.25表示有很大的遺傳分化。本研究中基于4個線粒體基因聯合數據集的為0.630,表明高原林蛙群體遺傳分化程度很大。
群體間的基因流與遺傳分化負相關,當<1時,基因流弱,遺傳分化處于較強水平;當1<<4時,基因流中等,遺傳分化處于中等水平;當>4時,基因流強,遺傳分化水平較弱(Slatkin.,1987;姚佩君,2020)。4個線粒體基因聯合數據集的N為2.52,說明4個群體間的基因交流中等,但除了2 000 m群體和2 600 m群體間的N為2.53外,其他兩兩群體間的N均<1,和上述遺傳分化結果相對應,青藏高原高原林蛙不同海拔群體間存在一定的地理隔離,這可能與兩棲動物的遷移能力較弱,對所處生境較敏感有關(李郊,2014)。
檢測正選擇壓力對于揭示生物的適應性進化機制具有重要意義(許瑜婷,高芳鑾,2021)。本研究中,基因上沒有檢測到受正選擇的氨基酸位點,說明基因未受到選擇壓力作用;而除了最低的2 000 m群體,其余3個群體中基因都檢測到了正選擇氨基酸位點。考慮到基因在有氧呼吸中的重要作用,其可能在高原林蛙適應高海拔低溫、低氧環境的過程中起到了積極的作用。在高原林蛙的核基因中也發現了與能量代謝相關的基因受到了正選擇(Yang.,2012)。
3.3 群體歷史動態
應用Tajima’s與Fu’s中性檢驗推測群體歷史時,如果均為負值,且達到顯著差異(<0.05),則說明序列中含有比中性進化模型更多的核苷酸位點變化,預示著被研究群體可能經歷過擴張(Tajima.,1989)。4個線粒體基因聯合數據集的Tajima’s和Fu’s均為負值,但差異都不顯著。單個群體中,大多數群體的Tajima’s與Fu’s也均為負值,且只有少數群體出現顯著差異;從錯配分布曲線來看,4個群體總體曲線為雙峰。高原林蛙群體可能未經歷過群體擴張事件。
2 000 m和2 600 m的高原林蛙群體遺傳多樣性高于3 200 m和3 800 m群體,4個海拔的群體遺傳分化程度高,基因可能對其適應高海拔環境起到了積極作用,群體可能未經歷擴張事件。本研究可為理解高原林蛙適應高原環境及其保護提供參考。

