基于宏基因測序及分箱技術的復合乳酸菌發酵乳菌種鑒定及定量
程坤,趙婷,劉蕊,周立光,馮會粉,于學健,李宏,姚粟*
1(中國食品發酵工業研究院有限公司,中國工業微生物菌種保藏管理中心,北京,100015) 2(中國合格評定國家認可中心,北京,100062)
發酵乳是以生牛(羊)乳或乳粉為原料,經殺菌、發酵后制成的pH值降低的產品(GB 19302—2010《食品安全國家標準發酵乳》)。發酵乳行業的規模和質量飛速發展,符合“健康中國”、“國民營養計劃”等國家戰略,是關系國計民生的重要產業。嗜熱鏈球菌(Streptococcusthermophilus)和德氏乳桿菌保加利亞亞種(Lactobacillusdelbrueckiisubsp.bulgaricus)是發酵乳常用發酵劑菌種,二者之間存在共生關系,德氏乳桿菌保加利亞亞種代謝生成的氨基酸可刺激嗜熱鏈球菌的生長,而嗜熱鏈球菌在代謝過程中產生的甲酸可以促進德氏乳桿菌保加利亞亞種的生長[1]。發酵乳通常添加對人體有益的益生菌,如:動物雙歧桿菌乳亞種(Bifidobacteriumanimalissubsp.lactis)和干酪乳酪桿菌(Lacticaseibacilluscasei),有助于促進腸道微生物的定殖,治療和預防腹瀉、緩解便秘、增強人體免疫力等[2],某一株益生菌在產品中需要達到一定數量才能起到保健作用,因此發酵乳中菌種的鑒定和定量尤其重要。
目前我國對于發酵乳中乳酸菌的使用和監管,標準主要為GB 4789.34—2016《食品安全國家標準 食品微生物學檢驗 雙歧桿菌檢驗》和GB 4789.35—2016《食品安全國家標準 食品微生物學檢驗 乳酸菌檢驗》,采用傳統培養方法和生理生化鑒定方法進行發酵乳中乳酸菌的鑒定和定量,具有成本低,易于操作等優點,但需要獲得菌種純培養物,且存在檢測結果不準確等問題。建立快速、準確的復合乳酸菌發酵乳菌種鑒定和定量檢測方法,成為發酵乳行業亟待解決的熱點、難點問題之一[3]。近幾年來,高通量測序技術迅速發展,采用宏基因組測序分箱分析可將包含不同生物體的序列進行組裝,重現復合樣品中的微生物種類,解析產品組成。該方法以復合樣品總基因組DNA為分析對象,有效避免了菌種分離培養步驟,目前已被應用于復合益生菌產品的物種組成分析[4]。平均核苷酸一致性(average nucleotide identity,ANI)是通過全基因組序列鑒定細菌物種的常用方法之一,其域值95%~96%與DNA-DNA雜交值70%相對應,可作為細菌種水平界定的標準[5]。ANI分析具有方便、快捷、分辨率高的優點[5],其與宏基因組分箱分析相結合,為復合乳酸菌發酵乳菌種的種水平精準鑒定提供了一種可靠方法。
復合乳酸菌發酵乳產品的菌種組成與定量分析是產品質量的關鍵。本文以3款市售復合乳酸菌發酵乳產品為研究對象,利用宏基因組測序和分箱技術、ANI分析等技術,研究復合乳酸菌發酵乳產品中菌種種水平鑒定及相對定量。對復合菌發酵乳產品的質量控制和行業監管具有重要參考意義。
1 材料與方法
1.1 實驗材料
1.1.1 復合乳酸菌發酵乳產品
本研究選取3款不同品牌的復合乳酸菌發酵乳產品A、B、C,均購自北京大型超市。產品標簽標識菌株類別及活菌數如下:
樣品1:產品A(菌種名稱:動物雙歧桿菌BB-12、嗜熱鏈球菌、德氏乳桿菌保加利亞亞種;保加利亞乳桿菌和嗜熱鏈球菌總數1×106CFU/g,雙歧桿菌1×107CFU/g);
樣品2:產品B(菌種名稱:保加利亞乳桿菌、嗜熱鏈球菌、干酪乳桿菌,乳酸菌活菌數≥1×106CFU/g);
樣品3:產品C(菌種名稱:乳雙歧桿菌BL-99、嗜熱鏈球菌、保加利亞乳桿菌;乳雙歧桿菌總數≥1×106CFU/g)。
1.1.2 試劑與儀器
DNA提取試劑盒:E.Z.N.A.?Bacterial DNA Kit,美國OMEGA公司;文庫構建試劑盒NEXTflex Rapid DNA-Seq Kit,美國Bioo Scientific公司。
Eppendorf微量可調節移液器、5424R小型臺式冷凍離心機,德國Eppendorf公司;HG-50高壓滅菌鍋,日本HIRAYAMA公司;AC2-4S1生物安全柜,新加坡Esco公司;BioDrop核酸蛋白分析儀,豪沃生物科技(上海)有限公司;Qubit 3.0 核酸熒光定量儀,美國ThermoFisher Scientific公司;Illumina HiSeq 3000基因測序平臺,美國Illumina公司。
1.2 實驗方法
1.2.1 基因組DNA的提取
取20 mL發酵乳樣品,4 000 r/min離心5 min,吸取上清液,12 000 r/min離心10 min,取沉淀按照試劑盒E.Z.N.A.?Bacterial DNA Kit的說明書提取基因組DNA。使用Qubit 3.0核酸熒光定量儀、BioDrop核酸蛋白分析儀分別檢測基因組DNA的濃度和純度,使用1%瓊脂糖凝膠電泳檢測基因組DNA的完整度。
1.2.2 宏基因組文庫構建與測序
通過物理方法將質檢合格的基因組DNA隨機打斷成300~400 bp的片段,使用NEXTflex Rapid DNA-SeqKit按照說明書進行文庫制備。使用Illumina HiSeq 3000測序平臺(上海美吉生物醫藥科技有限公司)進行宏基因組測序,宏基因組序列NCBI登錄號分別為:SAMN22013808,SAMN22013809,SAMN22013810。
1.2.3 下機數據質控與組裝
采用MetaWRAP流程[6]對宏基因組下機數據進行質控和組裝,采用Trim Galore(http://www.bioinformat- ics.babraham.ac.uk/projects/trim_galore/)去除reads 的adapter、過濾質量值低于20的reads,參數采用默認設置。使用Megahit[7]軟件進行序列組裝,采用succinct de Bruijn graph方法進行拼接,k-mer參數從小至大迭代,選擇長度≥1 000 bp的contigs用于后續分析。
1.2.4 分箱分析
采用MetaWRAP流程對拼接后的contig開展分箱分析,獲得各樣品的分箱結果(bin)。利用checkM[8]評估各bin的完整度和污染度,選取完整度>70%和污染度<10%的bin[9],利用blobology模塊繪制各樣品中bin的豐度散點圖。通過Circos[10](版本0.69-8)軟件對各樣品bins 進行可視化分析,每個bin的contigs重新基于模式菌株有參組裝,展示最終的基因組圈圖。
1.2.5 ANI分析
從NCBI genome 數據庫下載相關種模式菌株的基因組序列,使用fastANI[11](版本:1.32)計算各bin與相關物種模式株基因組序列之間的ANI值。以ANI值為95%~96%作為細菌物種界定的標準[5],對各樣品的分箱結果進行物種鑒定。
1.2.6 物種定量分析
采用Quant_bins模塊分析各bin的相對豐度。
2 結果與分析
2.1 測序數據質控和組裝
針對樣品的宏基因組測序下機數據,采用Fastp[12]軟件,堿基質量值≥20,對raw data進行質控。質控后A、B、C 3個產品的clean data數分別為13 667 335 968、12 529 436 956和12 225 941 409 bp,堿基質量值Q30均大于90%,測序數據質量能夠滿足后續分析(表1)。

表1 樣品宏基因組測序數據統計Table 1 Data statistics for metagenomic sequencing data
對clean data進行組裝,各產品獲得的contigs數目及相關參數見表2。
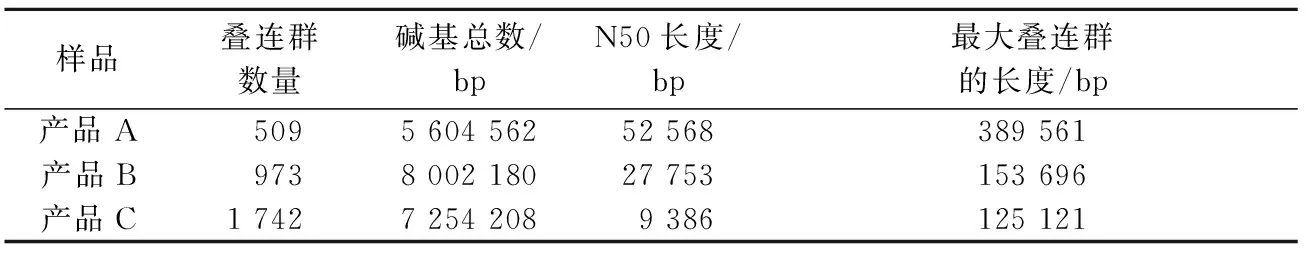
表2 樣品宏基因組數據組裝結果Table 2 Assembly result of the metagenomic sequencing data
2.2 復合乳酸菌產品中物種基因組重建
使用MetaWRAP宏基因組分析流程對組裝后的contigs進行分箱分析,獲得分箱結果(bin),即重建的基因組。產品A、B、C的contigs 均分別分箱成3個bin(表3),與各產品標識所聲稱添加的菌種數量一致。通過checkM來評估分箱后bin的完整度和污染度,結果表明,各樣品分箱產生的bin的完整度均高于85%,污染度均低于5%(表3)。各Bin豐度散點圖見圖1。
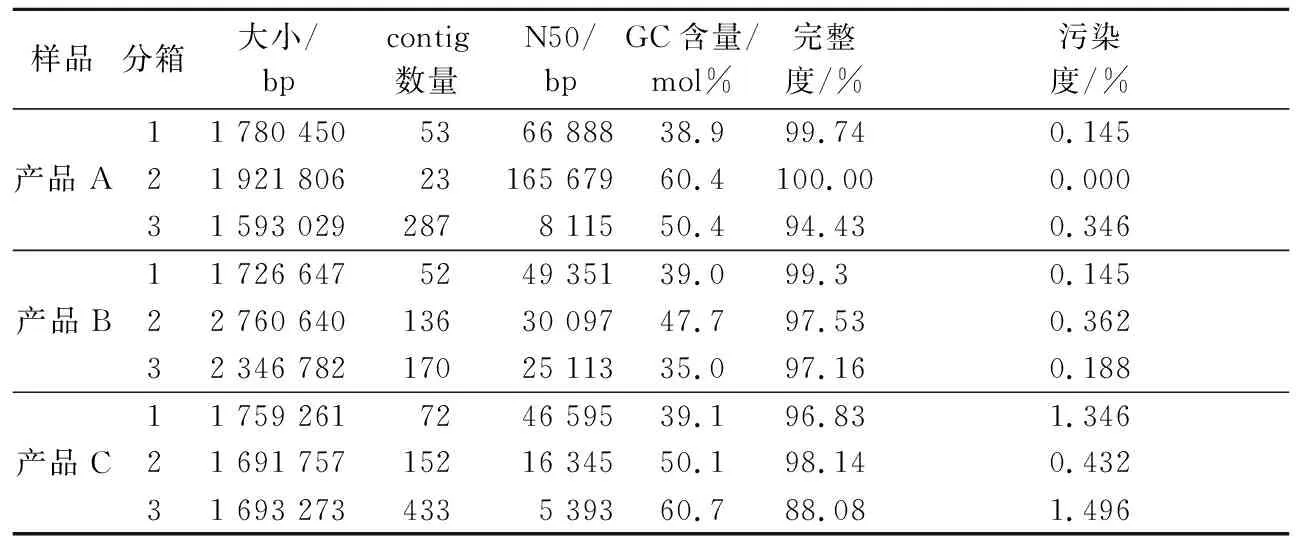
表3 宏基因組分箱后各bin的參數Table 3 The results of metagenomics binning

a-產品A;b-產品B;c-產品C圖1 Bin豐度散點圖Fig.1 Scatter plots of bins abundance注:一個點代表一個contig,相同顏色的contig來自同一個bin
2.3 復合乳酸菌產品中物種的鑒定
將分箱獲得的各bin通過NCBI-Blast進行初步比對,確定其近緣物種,利用fastANI軟件計算各bin與近緣物種模式株之間的ANI值(表4)。每個樣品中的bin與最近緣物種模式菌株基因組ANI分析的值均>95%,表明每個bin 代表的菌種與模式菌株為同一個種(表4)。

表4 分箱結果與參考模式菌基因組ANI 分析Table 4 The analysis results of ANI between bins and genomes of type strains
產品A和產品C中3個bin通過ANI分析鑒定為嗜熱鏈球菌、動物雙歧桿菌和德氏乳桿菌,與該產品聲稱添加的菌種一致。產品B中3個bin通過ANI分析鑒定為嗜熱鏈球菌、干酪乳酪桿菌和乳酸乳球菌(Lactococcuslactis),其中,嗜熱鏈球菌和干酪乳酪桿菌與該產品聲稱添加的菌種一致,未檢測到該產品聲稱添加的德氏乳桿菌,檢測到的乳酸乳球菌未出現在該產品聲稱中。
Contig利用模式基因組進行有參組裝后,通過Circos v 0.69-8軟件對各樣品bin進行可視化,基因組圈圖見圖2。

圖2 產品宏基因組菌種基因組重建Fig.2 Re-constructed genomes from samples by means of the metagenomics binning注:每一個產品的bins 都由橫向排列的遺傳圖譜來表示,相同物種用同一種顏色顯示,綠色“√”表示分箱得到的物種與產品聲稱菌種一致
動物雙歧桿菌和干酪乳桿菌在我國《可用于食品的菌種名單(衛辦監督發〔2010〕65號)》之列,是
發酵乳常用的益生菌,在A、B和C 3個產品中也均有添加。其中,動物雙歧桿菌乳亞種曾用名為乳雙歧桿菌(Bifidobacteriumlactis)[13];德氏乳桿菌保加利亞亞種的曾用名為保加利亞乳桿菌(Lactobacillusbulgaricus)[14]。干酪乳桿菌(Lactobacillucasei)在2020年其分類學地位變更為干酪乳酪桿菌[15]。
德氏乳桿菌在產品B中未被檢測到,進一步分析發現,在利用完整度>70%、污染度<10%參數對產品B宏基因組分箱初步結果的過濾前,檢測到1個完整度僅為6.61%的bin,通過ANI分析顯示為德氏乳桿菌,定量結果顯示相對豐度為0.09%,由于bin完整度較低,可能會引起該定量檢測結果的偏差,該菌種按照宏基因組數據過濾參數未形成完整bin,說明產品中菌種的相對豐度較低時,可能引起分箱bin的完整度不夠而沒有被檢測到。產品B中檢測到的乳酸乳球菌,推測可能為原料、環境、生產過程等環節引入,或未經菌種鑒定誤按嗜熱鏈球菌添加進產品,尚需進一步的驗證確認。
2.4 復合乳酸菌產品中各物種的定量分析
利用Quant_bins模塊計算各樣品中bin的相對豐度,結果如表5所示。
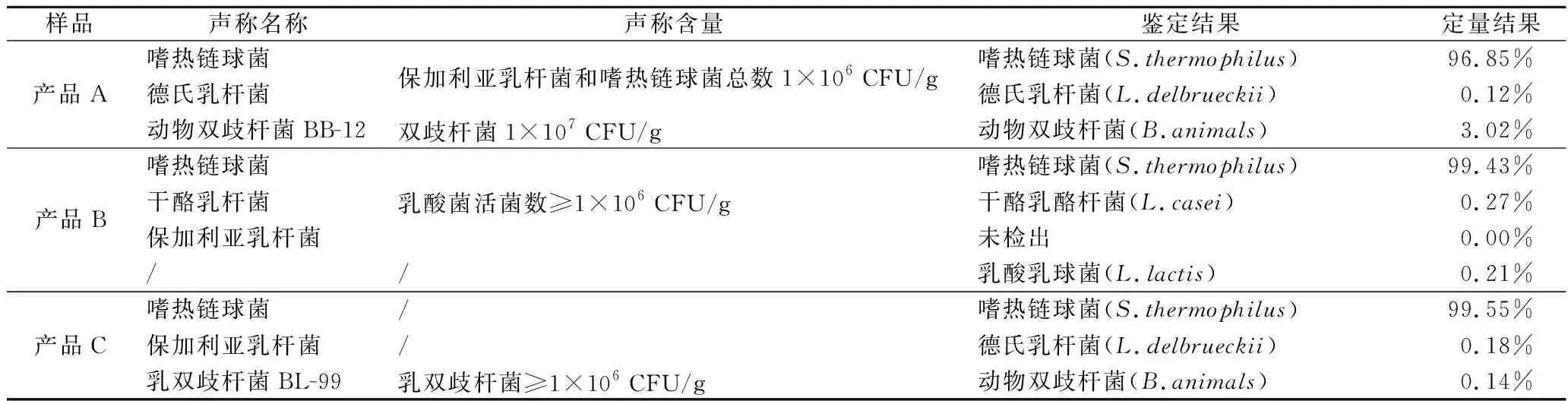
表5 各分箱結果的定量分析Table 5 Quantitative analysis of bins
對于產品A,其分箱分析所檢測到的嗜熱鏈球菌、德氏乳桿菌和動物雙歧桿菌3個物種的相對豐度分別為96.85%、0.12%和3.02%,嗜熱鏈球菌為絕對優勢物種。該產品標識聲稱嗜熱鏈球菌和德氏乳桿菌總量為1×106CFU/g,定量結果表明,產品中二者的相對比例約為807∶1。產品B標識聲稱活菌總數為1×106CFU/g,通過定量分析顯示,分箱分析所檢測到的嗜熱鏈球菌、干酪乳酪桿菌和乳酸乳球菌3個物種的相對豐度分別為99.43%、0.27%和0.21%,嗜熱鏈球菌為絕對優勢物種。產品C聲稱添加的物種類別與產品A相同,且其中動物雙歧桿菌的添加量為1×106CFU/g。定量結果表明,嗜熱鏈球菌的相對豐度為99.55%,德氏乳桿菌和動物雙歧桿菌的相對豐度分別為0.18%和0.14%。復合乳酸菌發酵乳產品中對所添加各菌種定量主要是以乳酸菌活菌數或某一種/株益生菌活菌數為指標,本研究中基于宏基因組的定量分析為相對定量,以活菌數和死菌數總和的百分比表示,隨著后生元(postbiotics)[16]概念逐漸被大眾所熟悉及相應產品的開發,相較于傳統定量檢測方法,本文提供了新的思路。
3 結論
本研究以3款市售復合乳酸菌發酵乳產品為研究對象,利用宏基因組測序和分箱分析、ANI分析等技術,研究復合乳酸菌發酵乳產品中菌種種水平鑒定及相對定量。產品A和產品C分箱獲得的3個bin通過ANI分析鑒定為嗜熱鏈球菌、德氏乳桿菌和動物雙歧桿菌,與產品聲稱添加菌種一致,產品A 3個物種的相對豐度分別為96.85%、0.12%和3.02%,產品C 3個物種的相對豐度分別為99.55%、0.18%和0.14%。產品B分箱獲得的3個bin通過ANI分析鑒定為嗜熱鏈球菌、干酪乳酪桿菌和乳酸乳球菌,與產品聲稱添加菌種部分一致,3個物種的相對豐度分別為99.43%、0.27%和0.21%。本研究為復合乳酸菌發酵乳產品的菌種鑒定與定量提供了一種可行的分析方法,與傳統基于可培養的分析方法相比,具有準確、可靠等優點,且不受菌種死活狀態的影響。隨著測序技術的不斷革新,高通量測序在成本與周期方面必將進一步下降和縮短,宏基因測序及分箱作為一項新興技術在食品檢測領域將進一步推廣應用,有望發展成為一種常規分析方法。本研究對復合乳酸菌發酵乳產品的質量控制和行業監管具有重要參考意義。

