基于高通量測序研究甘松根際細菌及藥用部位內生細菌群落結構及多樣性
李 瑩,鐘海蓉,李奕松,威則日沙,李文兵,劉 圓*
基于高通量測序研究甘松根際細菌及藥用部位內生細菌群落結構及多樣性
李 瑩1, 2, 3,鐘海蓉2, 4,李奕松1,威則日沙3, 5,李文兵2, 3, 6,劉 圓2, 3, 6*
1. 西南民族大學藥學院,四川 成都 610041 2. 四川省羌彝藥用資源保護與利用工程實驗室,四川 成都 610225 3. 青藏高原民族藥用資源保護與利用國家民委重點實驗室,四川 成都 610225 4. 四川中醫藥高等專科學校,四川 綿陽 621000 5. 涼山彝族自治州第二人民醫院,四川 西昌 615000 6. 西南民族大學民族醫藥研究院,四川 成都 610225
探析并比較野生甘松與仿野生栽培甘松的根際細菌與內生細菌的種群組成、豐富度、多樣性及差異性。以野生甘松和仿野生栽培甘松的根際土壤、根及根莖為研究樣本,基于Illumina MiSeq第二代高通量測序技術對根際土壤細菌、根及根莖內生細菌的16S rDNA V4-V5區進行序列測定,并進行相關生物信息學分析。各組樣本基因組經16S測序,細菌群落的所有序列分別進行97%的相似水平下的物種分類單元(operational taxonomic units,OUT)分類,根際細菌共涉及22門、38綱、69目、119科、254屬、268種,內生細菌共涉及16門、29綱、60目、107科、199屬、145種。根際、內生細菌群落中共有的序列分別為498個、177個,主成分分析的最主要2類主成分(PC1和PC2)類群的比例綜合分別接近70%、60%,其中豐度排在前4位的依次為變形菌門、放線菌門、酸桿菌門和擬桿菌門,其他類群的類別及相對豐度存在一定差異。優勢細菌差異分析中,各組細菌群落組成及相對豐度差異很大。野生甘松和仿野生栽培甘松的優勢促生菌群可能主要集中于變形菌門、放線菌門、酸桿菌門和擬桿菌門,可從中篩選出核心功能微生物混合菌群,對改良仿野生培育甘松土壤的微生物環境具有理論指導意義。
甘松;根際細菌;內生細菌;群落結構;多樣性
甘松為敗醬科植物甘松DC.的干燥根及根莖,為中藏藥材,亦為古印度阿育吠陀(Ayurveda)和尤納尼醫(Unani)學體系的常用藥物[1-2]。甘松廣泛應用于藥品、日化及香料等領域,具有較高的藥用價值和經濟價值。近年,甘松的野生資源逐年枯竭,匙葉甘松DC.已被列為《瀕危野生動植物物種國際貿易公約》(2007、2013年版)的附錄II中。由于甘松對生態環境(海拔3400~5000 m)要求較為苛刻,人工種植處于尚未起步的狀態。
本課題組2015~2020年,在西南民族大學青藏基地對甘松進行了仿野生移栽馴化工作,研究發現與野生甘松相比較,仿野生栽培甘松的總根表面積、須根根尖數量均明顯增加,主根變細,氣味明顯變弱;嚴重影響仿野生栽培甘松藥材的品質。近年來研究發現土壤微生態,特別是細菌微生物結構和數量可能是影響根類、根及根莖類藥材品質的重要因素[3-10]。其中,根際細菌中的促生菌在抑制根際病原菌、促進植物生長發育及代謝等方面起到重要作用[6-10];內生細菌不僅參與植物從土壤中吸收養分、植物次生代謝產物的合成與轉化,還可獨立產生次生代謝產物,在藥材品質、道地性、協助植物抗逆與應對海拔、季節、氣候特殊環境等方面均有重要的影響[4, 7-12]。
目前,關于甘松的根際微生物與內生菌方面的研究尚未見報道。高通量測序技術因其較傳統方法具有更快速、準確、科學、全面地探析樣本的微生物群落結構[13-14],本實驗利用基于Illumina MiSeq平臺的第二代高通量測序技術,擬對甘松根際及藥用部位痕量細菌的宏基因組進行擴增測序,分析甘松根際細菌和根及根莖內生細菌群落分布和多樣性;并比較仿野生栽培和野生甘松在根際細菌和內生細菌群落的異同,為進一步探究與篩選出核心功能微生物混合菌群,對改良仿野生培育甘松的土壤、調節微生物環境、幫助仿野生繁育品抵抗青藏高原的特殊惡劣環境提供科學數據。
1 儀器與試藥
精密電子天平(上海卓精電子科技有限公司),振蕩器vortex-5(海門市其林貝爾儀器制造有限公司),HHS-2S型電子恒溫不銹鋼水浴鍋(上海予卓儀器有限公司),Eppendorf離心機(Eppendorf,德國),電泳儀和凝膠成像儀[Bio Rad公司,伯樂生命醫學產品(上海)有限公司],ABI9700 PCR儀(ABI,Applied Biosystems 美國應用生物系統公司),Axygen凝膠回收試劑盒(Axygen公司,美國),FTC-3000TM real-time PCR(上海楓嶺生物技術有限公司),Miseq測序儀(Illumina公司,美國)。
Phusion超保真PCR Master Mix(貨號M0531L,NEB公司,英國),DNA Marker(貨號3427A,DL9000欣百諾公司,DL2000 Takara),TB Green Premix Ex Taq(貨號RR420,ATakara公司,日本),MiSeq Reagent Kits v3(貨號MS-102-3003,Illumina公司,美國),MIO-BIO PowerSoil DNA Isolation Kit(貨號12888-100,Mo Bio Laboratories公司,美國)。
野生甘松和仿野生栽培甘松樣品及其根際土壤均于2019年7月采自四川省阿壩藏族羌族自治州紅原縣,其中仿野生栽培品采自西南民族大學青藏基地(2015年從野外移栽至基地后,仿野生人工栽培4年)。經西南民族大學劉圓教授鑒定為敗醬科植物甘松DC.,采集信息詳見表1。
2 方法
2.1 樣品前處理
采集新鮮、健康的帶土甘松,抖去多余泥土后,將一部分帶浮土的根、根莖用剪刀(經70%乙醇消毒)剪段,裝至無菌PE管保存于液氮罐中,作為檢測根際細菌樣品;另一部分立即用蒸餾水將根和根莖淋洗干凈,用70%乙醇擦拭表面至無菌后,切段裝至無菌PE管并保存于液氮罐中,作為檢測內生細菌樣品。
表1 甘松樣品及根際土壤采集信息
Table 1 Collection information of N. jatamansi and rhizosphere soil
樣本編號樣品來源與部位產地海拔/m A1-1栽培甘松-土根西南民族大學青藏基地3670 A1-2栽培甘松-凈根西南民族大學青藏基地3670 A1-3栽培甘松-土根莖西南民族大學青藏基地3670 A1-4栽培甘松-凈根莖西南民族大學青藏基地3670 A2-1栽培甘松-土根西南民族大學青藏基地3670 A2-2栽培甘松-土根莖西南民族大學青藏基地3670 A2-3栽培甘松-凈根西南民族大學青藏基地3670 B1-1野生甘松-土根安曲鎮下哈拉瑪村3561 B1-2野生甘松-凈根安曲鎮下哈拉瑪村3561 B1-3野生甘松-土根莖安曲鎮下哈拉瑪村3561 B1-4野生甘松-凈根莖安曲鎮下哈拉瑪村3561 B2-1野生甘松-土根阿木鄉峨扎村3560 B2-2野生甘松-凈根阿木鄉峨扎村3560 B2-3野生甘松-土根莖阿木鄉峨扎村3560 B2-4野生甘松-凈根莖阿木鄉峨扎村3560
2.2 DNA提取
2.2.1 根際細菌 于超凈臺內,將甘松根及根莖附著的根際土壤洗脫到95%乙醇中,于?80 ℃超低溫冰箱冷凍保存,按照MIO-BIO PowerSoil DNA Isolation Kit試劑盒的說明書對根際土壤細菌總DNA進行提取。
2.2.2 內生細菌 于超凈臺內取出樣品后,在75%乙醇中浸泡2.5 min,用無菌水清洗3次,切取中間段的植物組織,保證其無菌。再用PBS溶液反復沖洗,將沖洗液離心5 min,轉速8000 r/min,合并沉淀置于?80 ℃凍存。臨用前放于干冰浴中,于潔凈操作臺上取出,立即用液氮進行研磨、破壁。按蛋白酶K裂解法和酚氯仿抽提常規方法提取細菌總DNA。
2.3 16 S rDNA V4~V5區的PCR擴增
根據illumina Miseq高通量測序要求,進行雙向測序,設計目標區域和帶有“5’ Miseq接頭- barcode-測序引物-特異引物-3’”的融合引物。并采用2步PCR擴增的方法,確保擴增效率和準確性。
PCR所用的引物已經融合了Miseq測序平臺的V4-V5通用引物,外生菌采用引物515F:5’-GTGCCAGCMGCCGCGGTAA-3’和926R:5’-CCGTCAATTCMTTTGAGTTT-3’。內生菌引物為799F:5’-AACMGGATTAGATACCCKG-3’和1193R:5’-ACGTCATCCCCACCTTCC-3’。
采用兩步PCR擴增的方法,首先采用特異引物擴增目的片段,將目的片段進行膠回收,而后將回收產物作為模板進行2次PCR擴增,目的是將Illumina平臺測序所需的接頭測序引物Barcode添加到目的片段的兩端。
采用AXYGEN公司的AxyPrepDNA凝膠回收試劑盒回收。回收產物進行qRT-PCR定量。
2.4 RT-PCR定量
全部PCR產物已采用AxyPrepDNA凝膠回收試劑盒進行回收,FTC-3000TM RT-PCR儀對回收的DNA以精確定量,按照等比例進行混合后測序。
2.5 上機測序
樣品檢測合格后,DNA委托微基生物科技(上海)有限公司完成測序,測序平臺為美國Illumina公司Illumina Miseq PE250高通量測序儀。
2.6 數據處理與分析
Miseq測序得到的PEreads,根據barcode對樣本進行區分,對序列質量進行質控及過濾;再根據overlap關系進行拼接,拼接后的序列再次進行質控和濾過,最后得到的優化序列。利用軟件USEARCH在97%的相似水平上進行操作分類單元(operational taxonomic units,OUT)聚類分析和物種分類學分析,基于OTU聚類分析結果,用于對OTU進行樣本內的alpha多樣性分析,并對測序深度進行檢測。同時,基于分類學信息在各個分類水平上進行群落結構的統計分析,并對樣本之間進行beta多樣性分析。基于上述分析,再進行群落結構和系統發育等深入的統計學和可視化分析。
3 結果與分析
3.1 甘松根際細菌的分析結果
所有樣品擴增區域均為16S rDNA V4~V5,測序條數均超過4.5萬條,所有樣品的測序深度超過99%,均擴增成功。
3.1.1 序列長度分布 甘松根際土壤樣品所測得的8個樣本,有效序列數在30 301~39 040,優化序列平均長度為412 bp,見圖1。
圖1 甘松根際細菌的序列長度分布
3.1.2 各樣本OTU分布及物種信息(測序樣品的置信分析) 基于OUT-稀釋曲線(圖2)結果顯示,隨著測序數量的增加,稀釋曲線斜率逐漸降低,但未進入平臺期,說明再增加測序量也只會產生少量新的物種(OUTs),說明本次測序數據量合理。
結合Alpna多樣性分析結果,Simpson指數呈現出的差異稍明顯,表明根莖(B1-3、B2-3、A2-2)根際細菌的種群豐富度和多樣性最高,而總體來看仿野生人工栽培品根際細菌豐富度更高;Shannon-Wiener指數、Chao指數、Ace指數結果均表明測序數據量合理,可以反映樣品中絕大多數的微生物信息。同時,說明野生甘松與栽培甘松的根際菌群豐富度與均勻度較為一致。
3.1.3 甘松根際細菌群落分布特征 對測得的所有序列進行97%的相似水平下的OUT分類,并進行生物信息統計分析(圖3)。
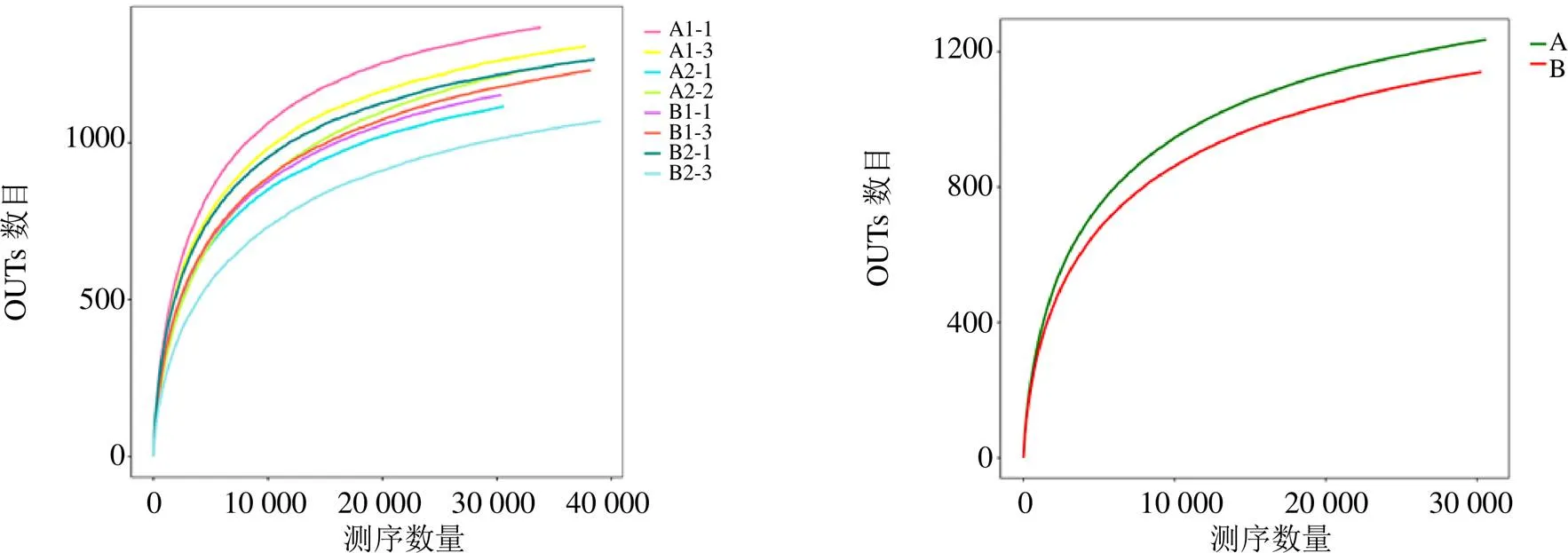
圖2 甘松根際細菌稀釋性曲線圖
在門的分類水平上,野生甘松和仿野生栽培甘松的根際細菌中共檢測出22個門,相對豐度較高且共有的8個細菌門類依次為變形菌門(Proteobacteria,46.55%~68.67%)、放線菌門(Actinobacteria,10.57%~27.44%)、酸桿菌門(Acidobacteria,2.62%~16.74%)、擬桿菌門(Bacteroidetes,2.99%~8.07%)、浮霉菌門(Planctomycetes,2.48%~5.08%)、綠彎菌門(Chloroflexi,1.43%~5.41%)、芽單胞菌門(Gemmatimonadetes,0.80%~2.57%)、厚壁菌門(Firmicutes,0.80%~3.28%)等。
在綱的分類水平上,共檢測出38個綱,其中相對占優勢的有α變形桿菌綱(Alphaproteobacteria,24.14%~44.98%)、-變形菌綱(Gammaproteobacteria,5.32%~21.93%)、放線菌綱(Actinobacteria,5.87%~20.13%)、嗜熱油菌綱(Thermoleophilia,3.46%~10.49%)、β-變形菌綱(Betaproteobacteria,2.80%~7.84%)、鞘脂桿菌綱(Sphingobacteriia,2.28%~5.87%)、酸桿菌綱(Acidobacteriia,0.11%~7.65%)、δ-變形菌綱(Deltaproteobacteria,1.29%~6.17%)、浮霉菌綱(Planctomycetacia,1.33%~3.00%)。
在目的分類水平上,共檢測出69個目,甘松根際細菌主要分布在根瘤菌目(Rhizobiales,2.38%~25.22%)、鞘脂桿菌目(Sphingobacteriales,2.28%~5.87%)、黃色單胞菌目(Xanthomonadales,2.86%~12.59%)、伯克霍爾德氏菌目(Burkholderiales,1.66%~8.99%)、鞘脂單胞菌目(Sphingomonadales,2.38%~25.22%)、酸桿菌目(Acidobacteriales,2.86%~12.59%)、紅螺菌目(Rhodospirillales,2.52%~5.16%)、Gaiellales(1.83%~5.65%)和弗蘭克氏菌目(Frankiales,2.42%~4.44%)等。
在科的分類水平上,共檢測出119個科,其中在鞘脂單胞菌科(Sphingomonadaceae,2.33%~8.86%)、慢生根瘤菌科(Bradyrhizobiaceae,3.21%~8.24%)、黃色單胞菌科(Xanthomonadaceae,1.33%~11.15%)、酸桿菌科(Acidobacteriaceae,0.11%~7.65%)、叢毛單胞菌科(Comamonadaceae,0.98%~4.34%)、根瘤菌科(Rhizobiaceae,0.97%~5.65%)、腸桿菌科(Enterobacteriacese,0.01%~17.73%)、Chitinophagaceae科(0.97%~3.51%)、黃色桿菌科(Xanthobacteraceae,0.88%~2.51%)等多有分布。
在屬的分類水平上,共檢測出254個屬,其中在慢生根瘤菌屬(3.06%~8.06%)、鞘氨醇單胞菌屬(0.65%~8.29%)、根瘤菌屬(0.85%~5.65%)、單胞菌屬(0.08%~6.69%)、鞘脂菌屬(0.15%~7.77%)、假黃色單胞菌屬(0.003%~1.58%)和屬(0.63%~1.71%)等多有分布在種的分類水平上,共檢測出268個種,其中有149種細菌尚未被人工條件下獲得培養的微生物,有1種未分類的細菌,這2部分細菌占有相對優勢。從甘松根際細菌的種水平樣品聚類數與柱狀圖組合分析圖(圖4)來分析,來自2個產地的野生甘松根的根際細菌聚成一類,根莖的根際細菌也聚成一類,但栽培甘松的根與根莖的根際細菌沒有這樣的規律。
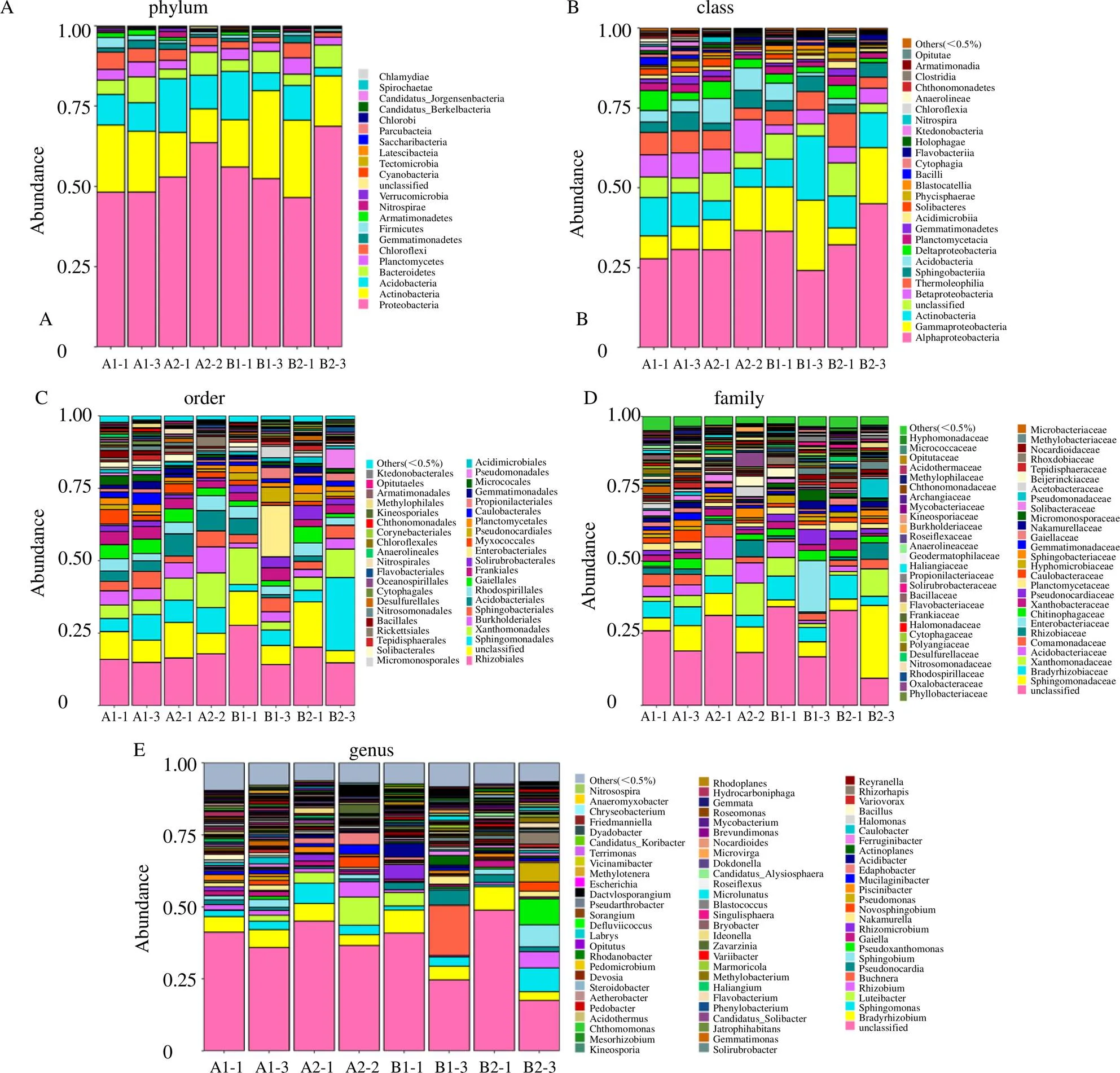
圖3 甘松根際細菌的門(A)、綱(B)、目(C)、科(D)、屬(E) 水平物種分布柱狀圖
3.1.4 甘松根際細菌群落主成分分析 基于相似水平為97%的OTU樣品表進行分析,得Venn圖(圖5),可比較直觀地觀察到野生甘松與仿野生栽培甘松的根際細菌群落均有1448序列被劃分入相同的OTU中,各樣品中共有的序列有498個。
再根據每個樣品OTU的豐度數據計算出每個OTU在每個樣品所占的百分比,利用這個百分比信息進行OTU的主成分分析(principal component analysis,PCA)。由圖6可知,PC1和PC2的比例綜合接近70%,基本可表明各樣本整體組成情況。其中,野生甘松根的根際細菌與仿野生栽培甘松根際細菌組成的相似度較高,而野生甘松的根際細菌組成離群較明顯。結合門、綱、目、科、屬和種水平上的分析結果,仿野生栽培甘松的根際細菌群落與野生甘松根際細菌群落更為相似。

圖4 甘松根際細菌的種水平樣品聚類數與柱狀圖組合分析圖

圖5 甘松根際細菌Venn圖-分組(左) 與花瓣圖(右)
3.1.5 野生甘松和仿野生栽培甘松根際優勢細菌差異分析 以所有野生甘松和仿野生栽培甘松的根際細菌類群為對象,使用LDA Effect Size組間群落差異分析兩者的根際細菌群落中主要差異物種(圖7)。分析結果表明,仿野生栽培甘松中起到重要作用的根際微生物物種包括假諾卡式菌目(Pseudonocardiales)、Solirubrobacterales目、微單孢菌目(Micromonosporales)等3個目,假諾卡式菌科(Pseudonocardiaceae)、微單孢菌科(Micromonosporaceae)、丙酸桿菌科(Propionibacteriaceae)、土壤紅桿菌科(Solirubrobacteraceae)、分支桿菌科(Mycobacteriaceae)和產堿桿菌科(Alcaligenaceae)等6個科,假諾卡式菌屬、游動放線菌屬、土壤紅桿菌屬、、小月菌屬、微枝形桿菌屬紅游動菌屬、土微菌屬、中慢生根瘤菌屬、、分枝桿菌屬和溶桿菌屬等12個屬。野生甘松中起到重要作用的根際微生物物種包括β-變形桿菌綱(Betaproteobacteria)、Solibacteres綱、芽孢桿菌綱(Bacilli)等3個綱,伯克霍爾德氏菌目(Burkholderiales)、黏球菌目(Myxococcales)、立克次氏體目(Rickettsiales)、Solibacterales、Nitrosomonadales和Bacillales等6個目,Solibacteraceae、多囊粘菌科(Polyangiaceae)、亞硝化單胞菌科(Nitrosomonadaceae)、伯克氏菌科(Burkholderianceae)等5個科,艾德昂菌屬、、未知嗜酸菌屬、食烴菌屬等5個屬。
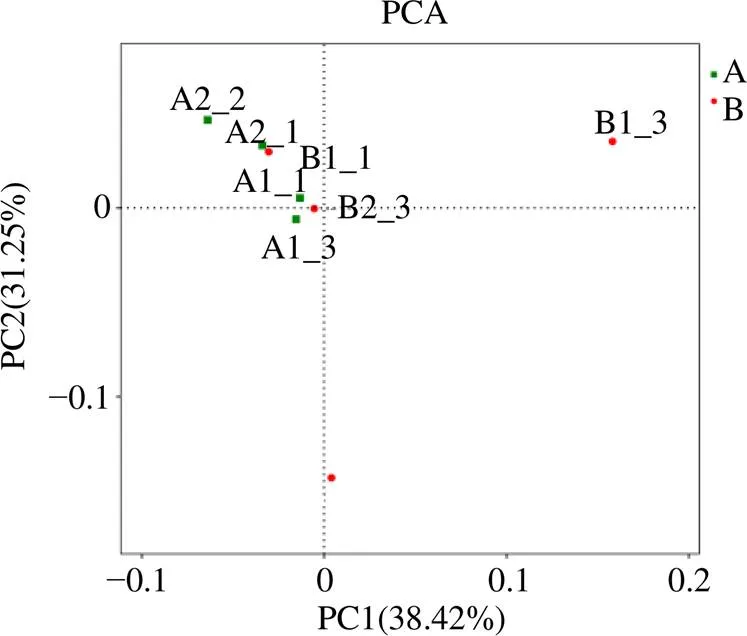
圖6 OUT水平上的甘松根際細菌主成分分析

圖7 野生甘松和仿野生栽培甘松根際優勢細菌LEfSe分析
3.2 甘松根和根莖內生菌的分析結果
所有樣品擴增區域均為16S rDNA V4-V5,除A1-2樣品的測序條數為逾2.2萬條外,測序條數均超過4萬條,所有樣品的測序深度超過97%,均擴增成功。
3.2.1 序列長度分布 甘松樣品所測得的7個樣本,有效序列數在30 301~39 040,優化序列平均長度為412 bp,見圖8。
3.2.2 各樣本OTU分布及物種信息(測序樣品的置信分析) 基于OUT-稀釋曲線(圖9)結果顯示,隨著測序數量的增加,稀釋曲線斜率逐漸降低,趨于進入平臺期,說明再增加測序量也只會產生少量新的OUTs。結合Alpna多樣性分析指數結果(圖10),Simpson指數呈現出的差異稍明顯,表明,其中以A2-3和甘松根與根莖的內生細菌的種群豐富度較高,差異也較大,B2-2的豐富度最高、多樣性最高;Shannon-Wiener指數、Chao指數、Ace指數野生甘松與仿野生栽培甘松的內生細菌菌群的豐富度與均勻度較為一致。結果均表明測序數據量合理,可以反映樣品中絕大多數的微生物信息。
圖8 甘松根及根莖的內生細菌的優化序列長度分布
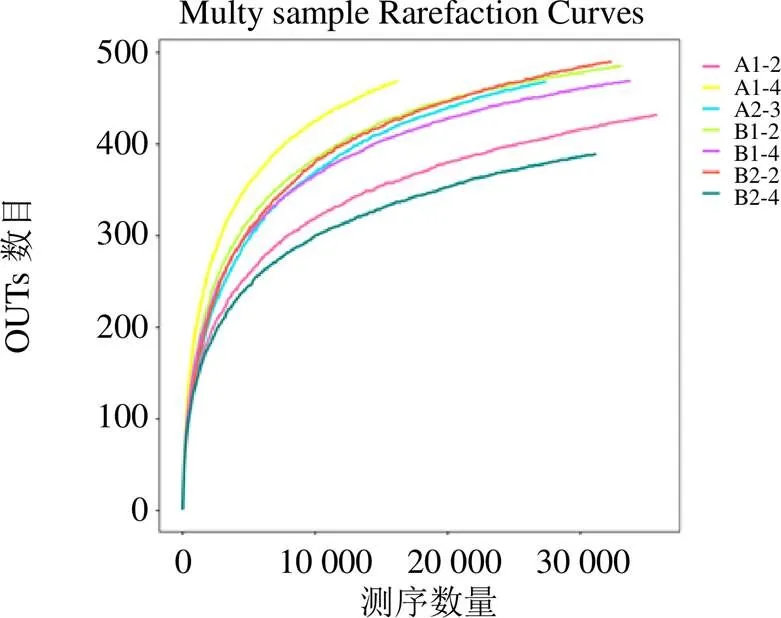
圖9 甘松內生細菌稀釋性曲線圖
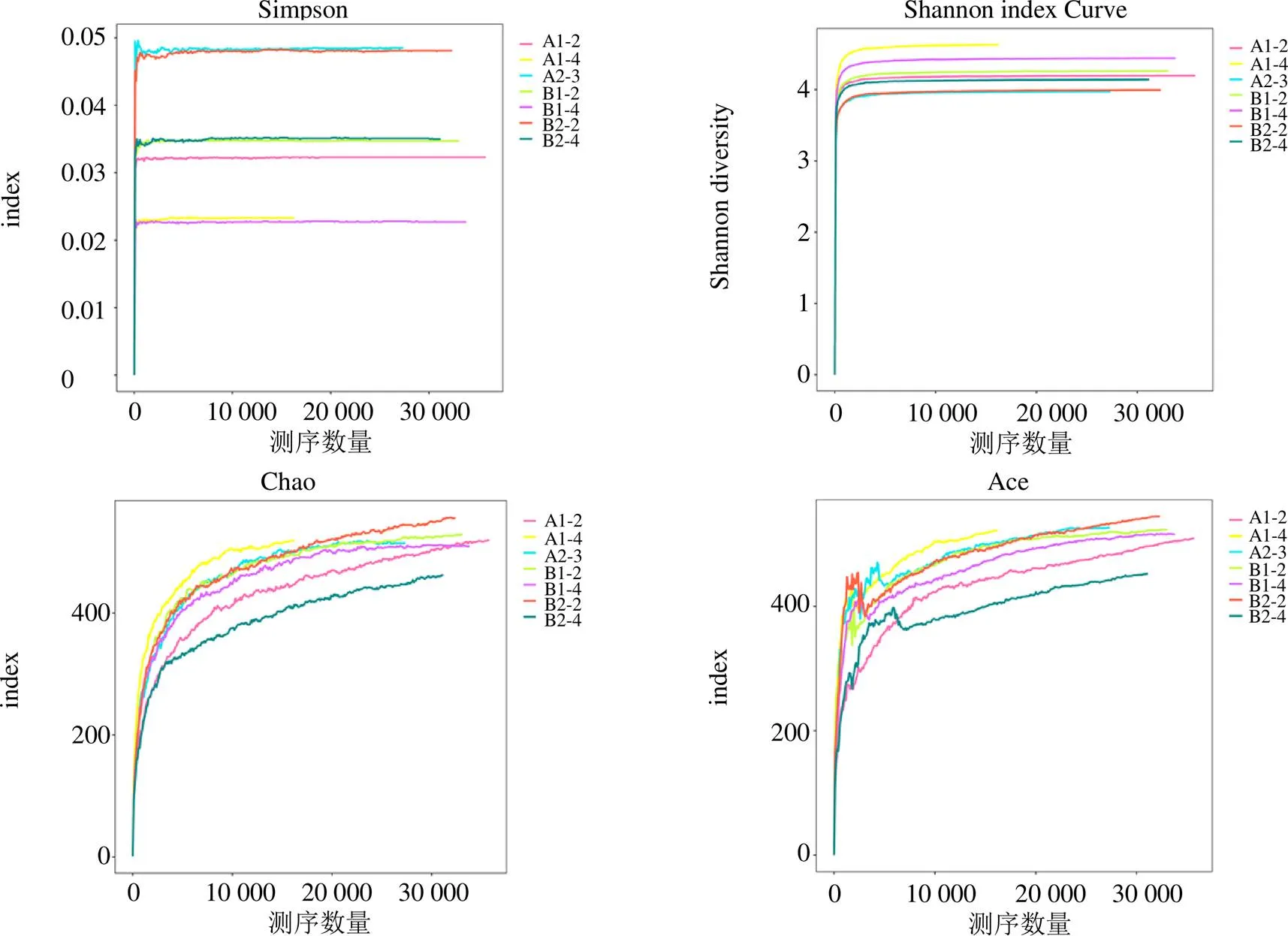
圖10 甘松內生細菌Simpson、Shannon、Chao、Ace指數圖
3.2.3 甘松根和根莖內生菌群落分布特征 對甘松根及根莖內生細菌測得的所有序列,進行97%的相似水平下的OUT分類,并進行生物信息統計分析(圖11)。在門的分類水平上,野生甘松和仿野生栽培甘松的根際內生細菌群落中共檢測出16個門,主要分布有變形菌門(Proteobacteria,65.54%~88.09%)、放線菌門(Actinobacteria,5.937%~26.61%)、酸桿菌門(Acidobacteria,0.38%~5.69%)、擬桿菌門(Bacteroidetes,0.22%~3.62%)、厚壁菌門(Firmicutes,0.45%~2.84%)、綠彎菌門(Chloroflexi,0.16%~1.37%)等。
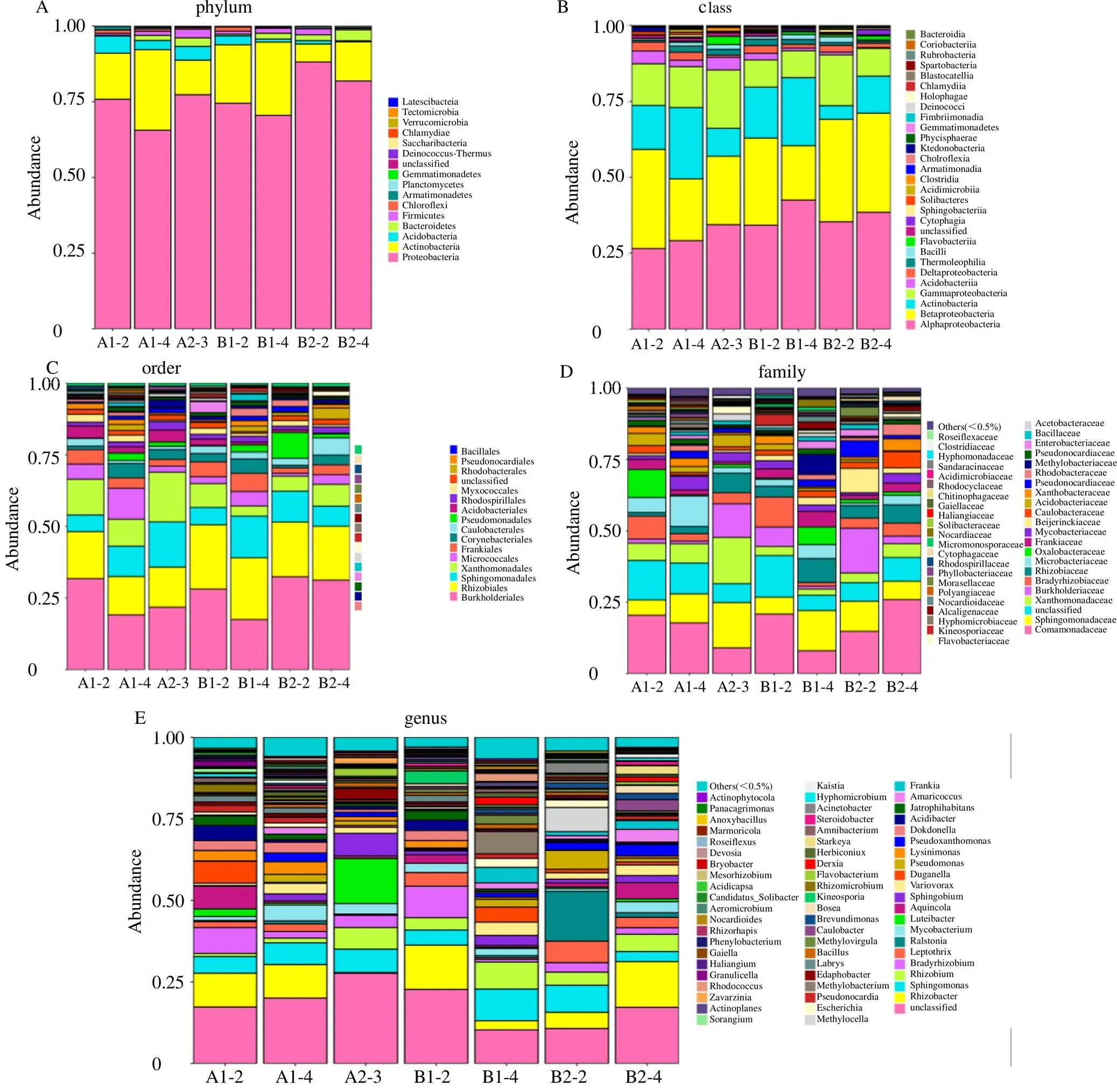
圖11 甘松內生細菌的門(A)、綱(B)、目(C)、科(D)、屬(E) 水平物種分布柱狀圖
在綱的分類水平上,共鑒別29個綱,甘松內生細菌在α-變形桿菌綱(Alphaproteobacteria,26.48%~42.47%)、β-變形菌綱(Betaproteobacteria,17.93%~33.78%)、放線菌綱(Actinobacteria,4.49%~23.54%)、γ-變形菌綱(Gammaproteobacteria,8.82%~19.13%)、酸桿菌綱(Acidobacteriia,0.24%~4.22%)、δ-變形菌綱(Deltaproteobacteria,0.70%~2.94%)、嗜熱油菌綱(Thermoleophilia,0.62%~2.12%)、芽孢桿菌綱(Bacilli,0.44%~1.91%)、黃桿菌綱(Flavobacteriia,0.06%~2.73%)等中均有分布。
在目的分類水平上,共鑒別60個目,甘松內生細菌在伯克霍爾德氏菌目(Burkholderiales,17.52%~32.36%)、根瘤菌目(Rhizobiales,13.28%~22.45%)、鞘脂單胞菌目(Sphingomonadales,6.07%~15.82%)、黃色單胞菌目(Xanthomonadales,3.51%~17.35%)、微球菌目(Micrococcales,5.83%~15.82%)、弗蘭克氏菌目(Frankiales,1.85%~6.34%)、棒狀桿菌目(Corynebacteriales,0.89%~5.03%)、柄桿菌目(Caulobacterales,1.02%~5.90%)、單胞菌目(Pseudomonadales,0.12%~8.93%)、酸桿菌目(Acidobacteriales,0.24%~4.22%)等均有分布。
在科的分類水平上,甘松內生細菌鑒別的有107個科,多分布在叢毛單胞菌科(Comamonadaceae,8.05%~25.91%)、鞘脂單胞菌科(Sphingomonadaceae,5.36%~15.80%)、黃色單胞菌科(Xanthomonadaceae,2.14%~6.76%)、伯克氏菌科(Burkholderiaceae,0.97%~15.63%)、根瘤菌科(Rhizobiaceae,1.30%~8.40%)、微桿菌科(Microbacteriaceae,0.91%~10.72%)、弗蘭克氏菌科(Frankiaceae,0.94%~5.48%)、分支桿菌科(Mycobacteriaceae,0.84%~4.86%)等科類。
在屬的分類水平上,甘松內生細菌共鑒別處出199個屬,其中在根瘤桿菌屬(0.23%~14.04%)、鞘氨醇單胞菌屬(3.13%~9.76%)、根瘤菌屬(0.96%~8.27%)、慢生根瘤菌屬(0.86%~9.68%)、纖毛菌屬(0.21%~6.63%)、分支桿菌屬(0.84%~4.86%)、屬(0.85%~5.65%)、屬(0.59%~7.01%)中多有分布。在種的水平上有145個種,其中68種細菌尚未被人工條件下獲得培養的微生物,1種未分類的細菌,這兩部分細菌占有相對優勢。從甘松內生細菌的種水平差異性矩陣熱圖(圖12)可得知,野生甘松和仿野生栽培甘松的內生細菌最先聚成一類,根際細菌也聚成一類,再與內生細菌聚為一類。
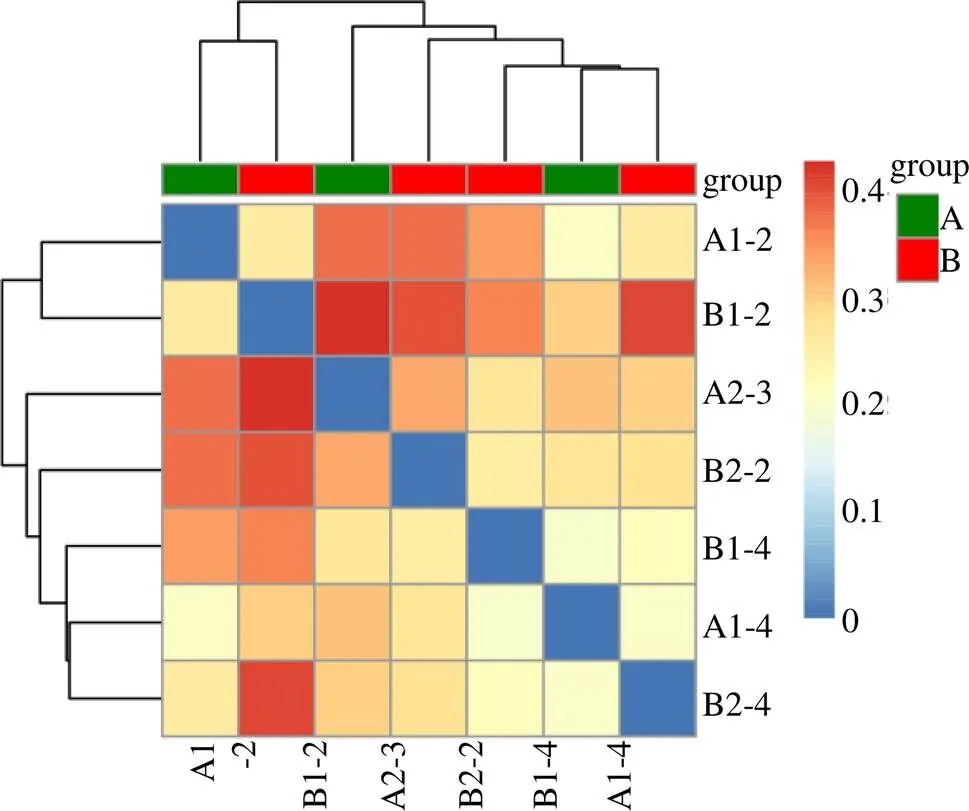
圖12 甘松內生細菌的種水平差異性矩陣熱圖
3.2.4 甘松根和根莖內生菌群落主成分分析 基于相似水平為97%的OTU樣品表進行分析,得Venn圖(圖13),可比較直觀地觀察到野生甘松與仿野生栽培甘松的內生細菌群落均有562序列被劃分入相同的OTU中,各樣品中共有的序列有177個。
再根據每個樣品OTU的豐度數據計算出每個OTU在每個樣品所占的百分比,利用這個百分比信息進行OTU的PCA分析。由圖14可知,PC1和PC2的比例綜合接近60%,基本可表明各樣本整體組成情況。其中,仿野生栽培甘松的樣品A1-2、A1-4的內生細菌群落與野生甘松樣品B1-2、B1-4和B2-4的內生細菌群落組成的相似度較高,而野生甘松樣品B2-2的內生細菌和仿野生栽培甘松樣品A2-3的內生細菌組成離群較遠。結合門、綱、目、科、屬水平和種水平的分析結果,仿野生栽培甘松的內生細菌群落與野生甘松根莖的內生細菌群落更為相似。

圖13 甘松內生細菌群落Venn圖-分組(左) 與花瓣圖(右)

圖14 OUT水平上的甘松內生細菌主成分分析
3.2.5 野生甘松和仿野生栽培甘松根及根莖的內生優勢細菌差異分析 以所有野生甘松和仿野生栽培甘松的內生細菌類群為對象,使用LDAEffect Size組間群落差異分析野生甘松與仿野生栽培甘松的內生細菌群落中主要差異物種(圖15)。分析結果表明,仿野生栽培甘松組別中起到重要作用的內生微生物物種包括酸桿菌門(Acidobacteria)和裝甲菌門(Armatimonadetes)2個門,梭狀芽胞桿菌綱(Clostridia)和Armatimonadia 2個綱,黃色單胞菌目(Xanthomonadales)、梭狀芽胞桿菌目(Clostridiales)、細鏈孢菌目(Catenulisporales)和Armatimonadales 4個目,黃色單胞菌科(Xanthomonadaceae)、細鏈孢菌科(Catenulisporaceae)和梭狀芽胞桿菌科(Clostridiaceae)等3個科,、細鏈孢菌屬、、羅河桿菌屬和5個屬。野生甘松組別中起到重要作用的內生微生物物種包括浮霉菌門(Planctomycetes),Phycisphaerae綱,根瘤菌目(Rhizobiales)和Phycisphaerales目,拜葉林克氏菌科(Beijerinckiaceae)、產堿桿菌科(Alcaligenaceae)、Phycisphaeraceae和生絲單胞菌科(Hyphomonadaceae)4個科,弗蘭克氏菌屬、、r 3個屬。
4 討論
高通量測序技術已被廣泛應用于環境[4]、食品[15]、作物培植[9,12,16]以及藥用植物[3,5-7,10-11]等微生物群落的多樣性研究中。本實驗首次運用高通量測序技術對甘松的仿野生栽培品和野生品的根際及內生細菌菌群進行結構分析。
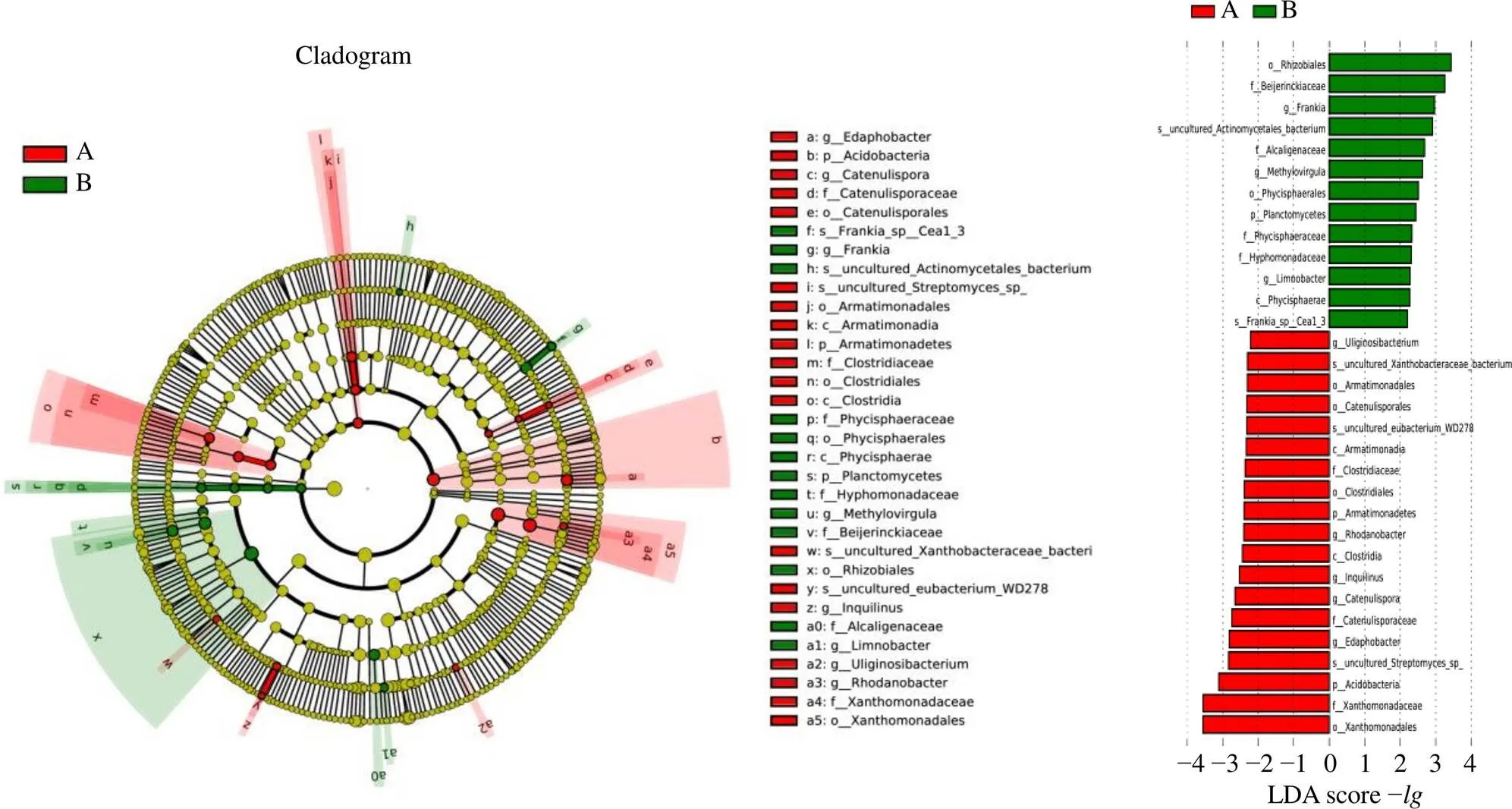
圖15 野生甘松和仿野生栽培甘松內生優勢細菌LEfSe分析
甘松是青藏高原高寒草甸重要的特有種之一,在保護青藏高原高寒生態系統方面具有重要的作用。在甘松的根際細菌與內生細菌群落中,優勢促生菌的功能主要是促進根際土壤養分的轉化,增強植物的抗環境脅迫能力和對病原微生物的抵抗力。
仿野生栽培甘松和野生甘松的優勢促生菌主要分布在根瘤菌目、鞘脂桿菌目、鞘脂單胞菌目、慢生根瘤菌科、酸桿菌科等。其中,根瘤菌目中的部分細菌具有固氮功能[8],鞘脂桿菌目中的部分細菌可參與鐵代謝以及少量的鉀和硫的代謝,影響植物的生理代謝,增強植物抗病和抗蟲能力[17-22]。鞘脂桿菌目中的部分細菌因能產生熱休克蛋白,可提高植物的耐寒性、耐熱性和耐旱性[23-25]。酸桿菌科功能的研究比較少,但其在生態系統中具有重要作用,有助于將腐殖質中的有機物轉化為土壤中的養分,主動與植物相互作用,起到促進植物生長的細菌作用,以及產生胞外多糖,支持細菌粘附到根表面等[26]。
仿野生栽培甘松根際細菌的優勢菌群有假諾卡式菌目、Solirubrobacterales和微單孢菌目等3個目,假諾卡式菌科、微單孢菌科和丙酸桿菌科等6個科;內生細菌的優勢菌群有黃色單胞菌目、梭狀芽胞桿菌目、細鏈孢菌目和Armatimonadales等4個目,黃色單胞菌科、細鏈孢菌科和梭狀芽胞桿菌科等3個科,相對豐度顯著高于野生甘松。這些差異菌群可能是影響仿野生甘松植物生長的主要細菌群落,例如細鏈孢菌科菌群是影響重組植物根際微生物共生網絡的關鍵,為根部提供了更多營養物質[27];屬的放線菌可抑制植物病原體在根際的生長、分解并利用土壤中的有機質,促進植物生長調節劑的產生[28]。
野生甘松根際細菌的優勢菌群有β-變形桿菌綱、Solibacteres綱、芽孢桿菌綱和豐祐菌綱等4個綱,伯克霍爾德氏菌目、黏球菌目、立克次氏體目、Solibacterales目等8個目,Solibacteraceae、多囊粘菌科、亞硝化單胞菌科等8個科;內生細菌的優勢菌群有根瘤菌目和Phycisphaerales2個目,拜葉林克氏菌科、產堿桿菌科、Phycisphaeraceae和生絲單胞菌科等4個科,弗蘭克氏菌屬、、等3個屬,相對豐度顯著高于仿野生人工栽培甘松。這些差異菌群是甘松植物適應青藏高原高寒的惡劣環境的重要菌群。例如弗蘭克氏菌屬可顯著提高在退化的土壤中移栽后植株的生長、生物量、莖根氮含量和成活率[29];屬為很強的甲基營養性菌,可促進植物的增殖、影響種子萌發和根系生長,幫助植物抵抗水分脅迫及幫助保護植物免受干燥和過度輻射等惡劣環境的影響[30]。
這些差異菌群可能是影響仿野生人工栽培甘松與野生甘松藥材品質差異的主要細菌群落,而相同的優勢促生菌是保持其品質一致的主要細菌群落,后續研究應重點關注。同時,課題組也在對移栽后的仿野生甘松種子與野生甘松種子育苗、移栽、仿野生繁育、藥材品質進行系列對比研究。課題后續將圍繞藥材品質一致性評價,篩選優化出甘松仿野生栽培所需的核心功能微生物混合菌群,并通過分離出的細菌菌群篩選對甘松植物生長的影響,有針對性地改良仿野生培育甘松土壤、調節仿野生繁育品的微生物環境,為開發出提高甘松藥材品質的生物菌肥提供重要的研究基礎。
利益沖突 所有作者均聲明不存在利益沖突
[1] 賈敏如, 張藝. 中國民族藥辭典 [M]. 北京: 中國醫藥科技出版社, 2016: 552.
[2] 中國藥典 [S]. 一部. 2020: 87-88.
[3] 劉珍珍, 谷萌萌, 張臣, 等. 基于高通量測序的三七微生物群落結構特征分析 [J]. 中草藥, 2020, 51(1): 204-209.
[4] 盧慧, 趙珩, 盛玉鈺, 等. 基于高通量測序的兩種高寒草甸土壤原核生物群落特征研究 [J]. 生態學報, 2018, 38(22): 8080-8087.
[5] 趙智靈, 劉學周, 魏曉雨, 等. 人參可利用內生菌株的篩選和鑒定 [J]. 中草藥, 2015, 46(14): 2143-2148.
[6] 蔣靖怡, 王鐵霖, 池秀蓮, 等. 基于高通量測序的紫花丹參與白花丹參根際細菌群落結構研究 [J]. 中國中藥雜志, 2019, 44(8): 1545-1551.
[7] 姜云, 冉超, 陳長卿, 等. 人參抗病、促生菌的篩選及田間效果評價 [J]. 中國中藥雜志, 2018, 43(11): 2230-2235.
[8] 陳文新, 汪恩濤. 中國根瘤菌 [M]. 北京: 科學出版社, 2011: 3-18.
[9] 李聰聰, 朱秉堅, 徐琳, 等. 高寒草甸優勢植物葉內、根內與土壤原核微生物群落的分異 [J]. 生態學報, 2020, 40(14): 4942-4953.
[10] 譚勇, 崔尹贍, 季秀玲, 等. 三七連作的根際、根內微生物變化與生態學研究進展 [J]. 中草藥, 2017, 48(2): 391-399.
[11] 劉蓬蓬, 陳江寧, 孟莉, 等. 基于Illumina MiSeq高通量測序分析黃芪內生細菌多樣性 [J]. 中草藥, 2018, 49(11): 2640-2645.
[12] 崔晉龍, 郭順星, 肖培根. 內生菌與植物的互作關系及對藥用植物的影響 [J]. 藥學學報, 2017, 52(2): 214-221.
[13] Delseny M, Han B, Hsing Y I. High throughput DNA sequencing: The new sequencing revolution [J]., 2010, 179(5): 407-422.
[14] Luo C W, Tsementzi D, Kyrpides N,. Direct comparisons of Illumina vs.454 sequencing technologies on the same microbial community DNA sample [J]., 2012, 7(2): e30087.
[15] 譚強來, 曾臻, 吳蘭蘭, 等. 基于高通量測序的江西特色發酵豆豉中微生物群落多樣性及其特征分析 [J]. 食品工業科技, 2019, 40(3): 85-89.
[16] 李金花, 高克祥, 萬利, 等. 微生物菌劑對楸樹幼苗生長及根際土細菌群落結構的影響 [J]. 生態學報, 2020, 40(21): 7588-7601.
[17] 崔曼, 尹彥舒, 張夢琦, 等. 一株大蒜根際細菌特性研究及其對田間大蒜產量和土壤酶活性的影響 [J]. 中國土壤與肥料, 2019(1): 173-179.
[18] Patel M, Kothari I L, Mohan J S S. Plant defense induced inpropagated banana () plantlets byderived elicitors [J]., 2004, 42(7): 728-731.
[19] 周冀衡, 李衛芳, 王丹丹, 等. 鉀對病毒侵染后煙草葉片內源保護酶活性的影響 [J]. 中國農業科學, 2000, 33(6): 98-100.
[20] 汪其同, 朱婉芮, 劉夢玲, 等. 基于高通量測序的楊樹人工林根際和非根際細菌群落結構比較[J]. 應用與環境生物學報, 2015, 21(5): 967-973.
[21] 程志強. 番茄根際和根內生細菌群落結構組成、多樣性、演替及其功能分析 [D]. 福州: 福建師范大學, 2018.
[22] 孫峰, 田偉, 張菲, 等. 丹江口庫區庫濱帶植被土壤細菌群落多樣性及PICRUSt功能預測分析 [J]. 環境科學, 2019, 40(1): 421-429.
[23] 汪其同. 楊樹人工林根際土壤微生物群落組成及結構研究 [D]. 泰安: 山東農業大學, 2017.
[24] Pegoraro C, Mertz L M, Maia L C,. Importance of heat shock proteins in maize [J]., 2011, 14(2): 85-95.
[25] 曹鵬熙, 劉怡萱, 許國琪, 等. 西藏那曲地區墊狀點地梅根際土壤細菌多樣性研究 [J]. 環境生態學, 2019, 1(5): 1-9.
[26] Kielak A M, Cipriano M A P, Kuramae E E. Acidobacteria strains from subdivision 1 act as plant growth-promoting bacteria [J]., 2016, 198(10): 987-993.
[27] Chen Y, Bonkowski M, Shen Y,. Root ethylene mediates rhizosphere microbial community reconstruction when chemically detecting cyanide produced by neighbouring plants [J]., 2020, 8(1): 4.
[28] Bhatti A A, Haq S, Bhat R A. Actinomycetes benefaction role in soil and plant health [J]., 2017, 111: 458-467.
[29] Diagne N, Arumugam K, Ngom M,. Use ofand actinorhizal plants for degraded lands reclamation [J]., 2013, 2013: 948258.
[30] Manish Kumar, Divjot Kour, Ajar Nath Yadav,Biodiversity of methylotrophic microbial communities and their potential role in mitigation of abiotic stresses in plants [J]., 2019, 74: 287-308.
Community structure and diversity analysis of rhizosphere bacterial ofand endophytic bacteria in medicinal parts based on high-throughput sequencing technology
LI Ying1, 2, 3, ZHONG Hai-rong2, 4, LI Yi-song1, WEIZE Ri-sha3,5, LI Wen-bing2, 3, 6, LIU Yuan2, 3, 6
1. College of Pharmacy, Southwest Minzu University, Chengdu 610041, China 2. Sichuan Provincial Qiang-Yi Medicinal Resources Protection and Utilization Technology and Engineering Laboratory, Chengdu 610225, China 3. Key Laboratory of Tibetan Plateau Ethnic Medicinal Resources Protection and Utilization, National Ethnic Affairs Commission of China, Chengdu 610225, China 4. Sichuan College of Traditional Chinese Medicine, Mianyang 621000, China 5. The Second People’s Hospital of Liangshan Yi Autonormous Prefecture, Xichang 615000, China 6. Ethnic Medicine Institute, Southwest Minzu University, Chengdu 610225, China
To analyze and compare the population composition, richness, diversity and difference in rhizosphere and endophytic bacteria of the wild and wild imitating cultivated.The 16S rDNA V4-V5 region of rhizosphere bacteria in soil and endophytic bacteria in roots and rhizomes ofthe wild and wild imitating cultivatedwas sequenced by using the Illumina Miseq second-generation high-throughput sequencing technology, and the related biological information was also analyzed.The genome of each sample was sequenced by 16S, and all sequences of bacterial community were OUT classified at 97% similar level, respectively. A total of 22 phyla, 38 classes, 69 orders, 119 families, 254 genera, and 268 species were detected in the rhizosphere bacteria of. And there were 16 phyla, 29 classes, 60 orders, 107 families, 199 genera, and 145 species in rhizosphere bacteria of. The common sequences of rhizosphere and endophytic bacterial communities were 498 and 177, respectively. The comprehensive proportions of PC1 and PC2 groups in principal component analysis were close to 70% and 60%, respectively. Among them, Proteobacteria, Actinobacteria, Acidobacteria and Bacteroidetes ranked the top four in the order of abundance, while there were some differences in the categories and relative abundance of other groups.The main growthpromoting phylums of wild and wild imitating cultivatedmay be mainly concentrated in Proteobacteria, Actinobacteria, Acidobacteria and Bacteroidetes, from which the core functional microorganism mixed flora can be screened out. These results can play a theoretical guiding significance for improving the soil microbial community of wild imitating cultivated
DC.; rhizosphere bacteria; endophytic bacteria; community structure; diversity
R282.2
A
0253 - 2670(2022)03 - 0835 - 13
;10.7501/j.issn.0253-2670.2022.03.024
2021-09-06
國家重點研發計劃(2018YFC1706101);四川省科技計劃應用基礎項目(2020YJ0099);中央高校基本科研業務費專項資金研究類項目(2018NQN13)
李 瑩(1982—),女,吉林伊通,漢族,副教授,碩士生導師,研究方向為民族藥品種與品質評價研究。E-mail: liying@swun.cn
劉 圓(1968—),女,教授,博士生導師,主要從事民族藥品種、品質評價及新藥資源保護與利用研究。Tel: (028)85528812 E-mail: 499769896@qq.com
[責任編輯 時圣明]

