寧夏枸杞花藥轉錄組分析及花藥發育相關基因的篩選
范文強,劉雪霞,馬小蘭,王路瑤,張 興,岳思君,唐建寧,朱金忠,姚新靈,鄭 蕊*
寧夏枸杞花藥轉錄組分析及花藥發育相關基因的篩選
范文強1,劉雪霞1,馬小蘭1,王路瑤1,張 興1,岳思君1,唐建寧2,朱金忠3,姚新靈1,鄭 蕊1*
1. 寧夏大學生命科學學院,西部特色生物資源保護與利用教育部重點實驗室,寧夏優勢特色作物現代分子育種重點實驗室,寧夏 銀川 750021 2. 寧夏枸杞產業發展中心,寧夏 銀川 750021 3. 中寧縣杞鑫枸杞苗木專業合作社,寧夏 中寧 755100
為了探知寧夏枸杞花藥發育過程及挖掘花藥發育相關基因。以單雙核花粉時期和成熟花粉時期的花藥為研究材料,通過高通量Illumina HiSeq2500測序平臺進行轉錄組分析。組裝共獲得128 151條Unigene,將Unigene與NCBI的非冗余蛋白質數據庫(non-redundant protein databasse,NR)、去冗余的蛋白序列數據庫(Swiss prot protein database,Swiss-Prot)、基因本體數據庫(gene ontology,GO)、直源同源群集數據庫(clusters of orthologous groups,COG)、真核同源群數據庫(euKaryotic orthologous groups,KOG)、基因功能和代謝途徑數據庫(Kyoto encyclopedia of genes and genomes,KEGG)數據庫比對,共有40 140條得到注釋。在錯誤發現率(false discovery rate,FDR)<0.05且差異倍數(fold change,FC)≥2的條件下,單雙核花粉時期與成熟花粉時期的差異表達基因共有46個。KEGG注釋發現有4個差異表達基因參與甘油酯代謝、亞油酸代謝、苯丙氨酸代謝和苯丙烷類生物合成4個途徑中。富含亮氨酸重復蛋白、細胞周期蛋白、類受體蛋白激酶、E3泛素蛋白連接酶等11個差異表達基因參與了花藥發育過程。隨機選取其中5個差異表達基因進行qRT-PCR驗證,表達結果與轉錄組數據基本一致,表明其數據真實可靠。為進一步解釋寧夏枸杞花藥發育的相關分子機制奠定了基礎,也為后續研究寧夏枸杞花藥發育的生物學特征提供了有益的基因資源。
寧夏枸杞;花藥發育;轉錄組;差異表達基因;生物學特征
寧夏枸杞L.屬于茄科(Solanaceae)枸杞屬Linn.落葉灌木,是重要的藥食兼用植物,具有很高的食用、藥用和生態保護價值[1]。枸杞是我國傳統的名貴中藥材和重要經濟作物,具有調節免疫、潤肺明目等功效,枸杞葉、果和根中都含有人體所需的蛋白質、氨基酸和微量元素[2]。枸杞子是一味常用的補肝益腎中藥,現代醫學研究證實其含有胡蘿卜素、維生素A、維生素C、維生素B1及鈣、磷、鐵、鋅、錳等營養成分,對造血功能有促進作用,還具有抗衰老、抗腫瘤、抗脂肪肝及降血糖等作用[3]。
花主要由花萼、花瓣、雄蕊和雌蕊4部分組成,其中花藥作為雄蕊最重要的組成部分,其發育過程復雜且精細,首先由雄蕊原基經過減數分裂形成小孢子四分體結構,然后花藥繼續膨大生長,小孢子由四分體結構發育成花粉粒,藥室破裂釋放花粉,最終花藥組織衰老退化[4]。研究植物的花藥發育,對于提高植物的抗逆能力、培育新品種以及提高產量具有重要的理論意義與實踐價值[5]。花藥發育相關基因的研究在許多植物已有報道,在番茄中,通過RNAi,使基因編碼的9-順式-環氧類胡蘿卜素雙加氧酶(9--epoxycarotenoid dioxygenase,NCED)活性降低,導致番茄花藥發育相關基因、、和的轉錄下調,導致花藥發育異常,從而產生了大量異常花粉[6];百合花藥發育相關的基因可以在突變體中適當表達,產生正常的花粉粒[7];水稻中基因的嚴格表達對水稻花藥發育至關重要[8]。關于枸杞生長發育[9],生理生態[10]以及基因克隆等方面的研究已有相關報導,其中枸杞基因已經被克隆并進行功能驗證,結果表明該基因參與枸杞花藥發育過程中淀粉供能的調控[2]。枸杞花藥的研究尚處于起步階段[11]。由于枸杞基因組和轉錄組相關信息相對缺乏,成為制約研究枸杞花藥發育及其調控機制的一個因素。因此,通過轉錄組學技術挖掘并研究一些與枸杞花藥發育相關基因的表達調控,對于解析枸杞花藥發育的分子機制具有重要意義。
目前,基于高通量測序技術的轉錄組分析,已經在植物、動物、微生物轉錄組研究中廣泛應用[12]。因此,本實驗利用RNA-Seq技術,選擇以體外更加容易培養的單雙核花粉時期和減數分裂完成以后的成熟花粉時期枸杞花藥進行轉錄組測序,得到的Unigene進行NCBI的非冗余蛋白質數據庫(non-redundant protein databasse,NR)、基因本體數據庫(gene ontology,GO)、直源同源群集數據庫(clusters of orthologous groups,COG)、真核同源群數據庫(euKaryotic orthologous groups,KOG)、基因功能和代謝途徑數據庫(Kyoto encyclopedia of genes and genomes,KEGG)、去冗余的蛋白序列數據庫(Swiss prot protein database,Swiss-Prot)、蛋白質家族數據庫(protein family,Pfam)注釋,篩選出與花藥發育相關的差異表達基因,結果為進一步研究枸杞花藥發育提供基因資源和研究基礎。
1 材料
“寧杞1號”是寧夏枸杞研究所鐘鉎元[13]選育的高產、優質、適應性強的枸杞新品種。于2016年6月取自寧夏育新枸杞種業有限公司(106°6'11.516"E,38°31'1.808"N)標準化管理的枸杞實驗田經寧夏大學生命科學學院鄭蕊博士鑒定為寧夏枸杞L.。參照徐青等[14]的方法,采集枸杞單雙核花粉時期(S1)和成熟花粉時期(S2)的花蕾,設置3個生物學重復,S1時期和S2時期的3個樣品分別編號為A1~A3和B1~B3。花蕾的采集結合形態觀察、縱橫徑測量和花藥壓片法,于干冰上剝離不同時期花蕾中的花藥(圖1),并立即置于液氮中速凍,長期保存于?80 ℃冰箱備用。
2 方法
2.1 RNA提取及測序文庫構建
RNA提取參照TaKaRa公司的RNAisoplus產品說明書進行,提取RNA樣品濃度由微量紫外檢測儀NanoDrop測定,并使用瓊脂糖凝膠電泳檢測RNA完整性,以確保枸杞轉錄組測序文庫滿足后續分析的需要。
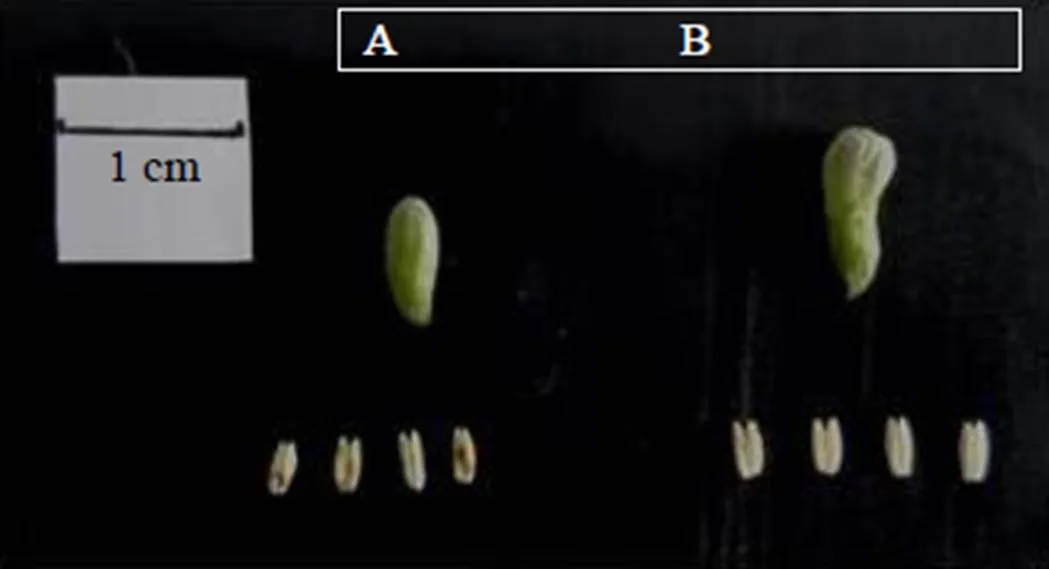
A-S1時期的花蕾和花藥 B-S2時期的花蕾和花藥
2.2 測序及原始數據的處理
通過高通量illuminaHiSeqTM2500測序平臺對已構建好的cDNA文庫進行測序,并獲得下機數據即原始讀段(raw reads)。利用Trinity軟件對測序獲得的Clean data進行組裝,獲得枸杞轉錄組Unigene數據庫,通過測序錯誤率和GC含量對測序數據的質量進行評估,得到Clean data進行分析。
2.3 差異基因的功能注釋
使用BLAST軟件將Unigene與NR、GO、COG、KOG、KEGG、Swiss-Prot、Pfam等數據庫比對(BLAST參數-value≤1.0×10?5和HMMER 參數-value≤1.0×10?10),獲得Unigene注釋信息。
2.4 花藥發育相關差異表達基因的篩選
使用DESeq軟件對“寧杞1號”花藥S1和S2 2個發育時期的差異表達基因進行分析。在差異表達分析過程中采用公認有效的Benjamini-Hochberg方法對原有假設檢驗得到的顯著性值進行校正,并最終采用校正后的值,即錯誤發現率(false discovery rate,FDR)<0.05為差異表達基因篩選的關鍵指標,以降低對大量基因的表達值進行獨立的統計假設檢驗帶來的假陽性。在篩選過程中,將FDR<0.05且差異倍數(fold change,FC)≥2作為篩選標準。其中,FC表示2樣品(組)間表達量的比值。
2.5 qRT-PCR分析
本研究為了驗證轉錄組數據的可靠性,以枸杞組成型表達基因為內參(GenBank登陸號:HQ415754.1),隨機選取5個相對表達量較高的差異表達基因進行qRT-PCR驗證,設計qRT-PCR特異引物(表1),利用反轉錄試劑盒將RNA反轉錄得到cDNA,采用TB Green Premix Ex Taq II試劑盒,在熒光定量PCR儀上進行定量檢測,樣品設置3個生物學重復,采用2?ΔΔCt法計算差異基因的相對表達量。
表1 qRT-PCR引物
Table 1 Primers used for real-time quantitative PCR
基因ID基因名稱正向引物反向引物 c68267.graph_c0PRX5’-CACCTGTGGATAGGAAGATCA-3’5’-GGAGCACTGTGGATTCATACT-3’ c68844.graph_c0AGPs5’-AGTGAGTGCGAACAATTAAC-3’5’-GTGGTAGTAAGAGATGCGTT-3’ c77007.graph_c0MBR15’-ACGGAAGCTCTGCACATAG-3’5’-GATGACATGGATATTCGTGAT-3’ c75034.graph_c0RPK5’-GAGTATATCTGATGGATATA-3’5’-ACAGGATTAGGTGGCCTA-3’ c72608.graph_c0LRR5’-GATGAACATTGAGTTAACTA-3’5’-CTTCACAACCATTTCAAT-3’ HQ415754.1LbActin5’-GACCTTCAATGTTCCCGCTATG-3’5’-GCCATCACCAGAGTCCAACAC-3’
2.6 統計學方法
本研究使用統計學軟件SPSS22和Excel2016對數據進行分析。
3 結果與分析
3.1 枸杞花藥轉錄組測序和clean data組裝
樣品經過轉錄組測序以后,共獲得44.72 Gb clean data,Q30堿基百分比在92.07%以上(表2),表明轉錄組數據豐富,質量較高。利用Trinity軟件對clean data數據進行組裝,共得到128151條Unigene,序列總長度為93 244 314 bp,平均長度為727.61 bp,N50為1170 bp,其中長度區間位于200~300 bp的Unigene數量最多,為42 785條,占33.39%(表3),表明組裝完整性較高,可用于后續分析。
3.2 Unigene功能注釋
使用BLAST軟件將Unigene序列與NR、GO、COG、KOG、KEGG、Swiss-Prot、Pfam等7個數據庫比對,最終獲得40 140個有注釋信息的Unigene,占Unigene總數的31.32%(表4)。
3.3 差異表達基因分析
采用DESeq方法篩選,設置篩選差異條件FDR<0.05,且FC≥2,作為篩選標準,共獲得差異表達基因46個,且相對于S1時期,這些基因均在S2時期下調表達(圖1)。
表2 轉錄組測序產量
Table 2 Statistics of transcriptome sequencing output
樣品讀取數堿基數GC/%Q30/% A129 290 3758 634 418 32643.1792.44 A223 477 5066 921 070 68243.0292.87 A321 597 6166 404 336 10043.5192.14 B122 641 0256 720 401 74042.9992.07 B228 977 3308 608 669 92443.1092.46 B325 044 7647 431 459 82442.8692.36
表3 組裝結果
Table 3 Statistics of assembly results
序列長度/nt基因(占比/%) 200~30042 785(33.39) 300~50034 095(26.61) 500~100027 280(21.29) 1000~200014 326(11.18) ≥20009665(7.54) 總數/條128 151 總長度/bp93 244 314 N50長度/bp1170 平均長度/bp727.61
表4 Unigene注釋信息
Table 4 Unigene annotation information
數據庫Unigene數量百分率/% NR39 35230.71 GO17 50113.66 COG9 184 7.17 KOG19 04014.86 KEGG11 092 8.66 Pfam22 73917.74 Swiss-prot21 53016.80 總計40 14031.32
3.3.1 差異表達基因的GO功能分類 對差異表達基因進行GO分析,結果表明有8個差異表達基因分別被注釋到生物學過程(biological process,BP)細胞組分(cellular component,CC)和分子功能(molecular function,MF)中(圖2)。
其中,分子功能中催化活性(catalytic activity)和蛋白結合(binding)所占比例最高,分別為7個(87.5%)和6個(75%);細胞組分中最多的是細胞膜部分(membrane),共有1個,占12.5%;生物學過程中則是代謝過程(metabolic process)、細胞過程(cellular process)和單組織過程(single-organism process)比重較大,分別為4個(50%)、3個(37.5%)和2個(25%),如表5所示。
3.3.2 差異表達基因COG/KOG分類 對差異表達基因進行COG分類(圖3),結果顯示,有17個差異表達基因分布于8類基因家族,注釋最多的是僅一般功能預測(general function prediction only,4);其次是翻譯(translation,3),復制、重組和修復(reproduction、reorganization and repair,3),信號轉導機制(signal transduction mechanisms,3);注釋最少的是氨基酸的運輸和代謝(transport and metabolism of amino acids,1),脂質的運輸和代謝(transport and metabolism of lipids,1),碳水化合物運輸與代謝(carbohydrate transport and metabolism,1)以及次生產物合成運輸及代謝(secondary metabolites biosynthesis transport and catabolism,1)。

綠色和紅色的點代表有顯著性表達差異的基因,綠色代表基因表達量下調,紅色代表基因表達量上調,黑色的點代表無顯著性表達差異的基因
KOG分類發現共有10個基因被注釋到5個功能中(圖4),其中,含轉錄本數目最多的是僅一般功能的預測(general function prediction only,5);其次是翻譯后修飾(post-translational modifications,3)、蛋白質轉換(protein turnover,3)和伴侶蛋白(chaperones,3);最少的是細胞周期調控(cell cycle control,1)、細胞分裂(celldivision,1)、染色體分裂(chromosome partitioning,1)、氨基酸的轉運和代謝(amino acid transport and metabolism,1)以及信號轉導機制(signal transduction mechanisms,1)。
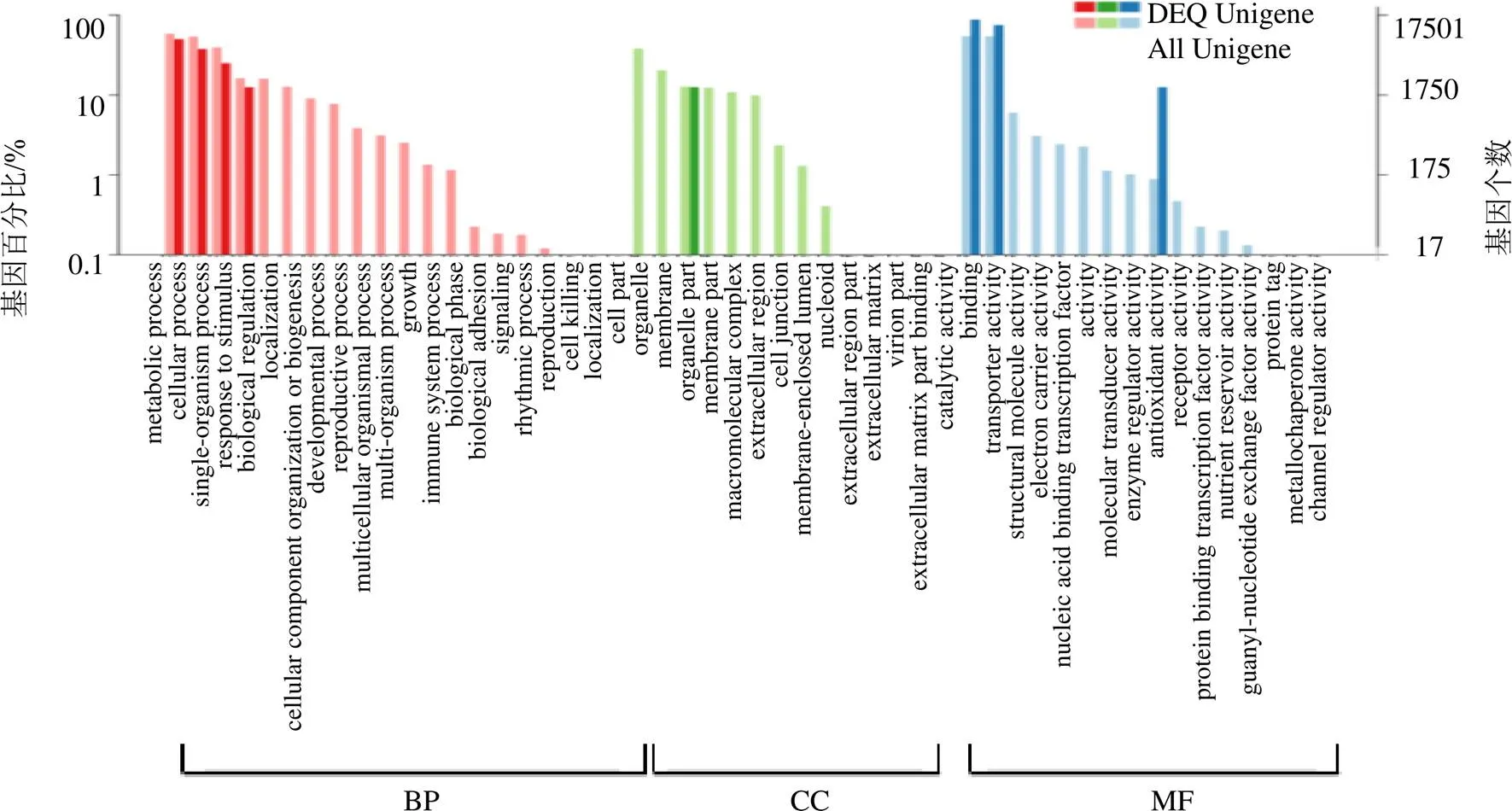
圖2 差異表達基因GO二級節點注釋
表5 差異表達基因GO功能分類
Table 5 GO functional classification of differentially expressed genes
GO功能分類功能注釋DEGs數量百分率/% CC細胞膜112.5 MF催化活性蛋白結合7687.575.0 BP代謝過程細胞過程單組織過程43250.037.525.0
3.3.3 差異表達基因KEGG注釋 對差異表達基因進行KEGG注釋,發現有3個差異表達基因分別參與甘油酯代謝、亞油酸代謝、苯丙氨酸代謝和苯丙烷類生物合成4條代謝通路(表6)。
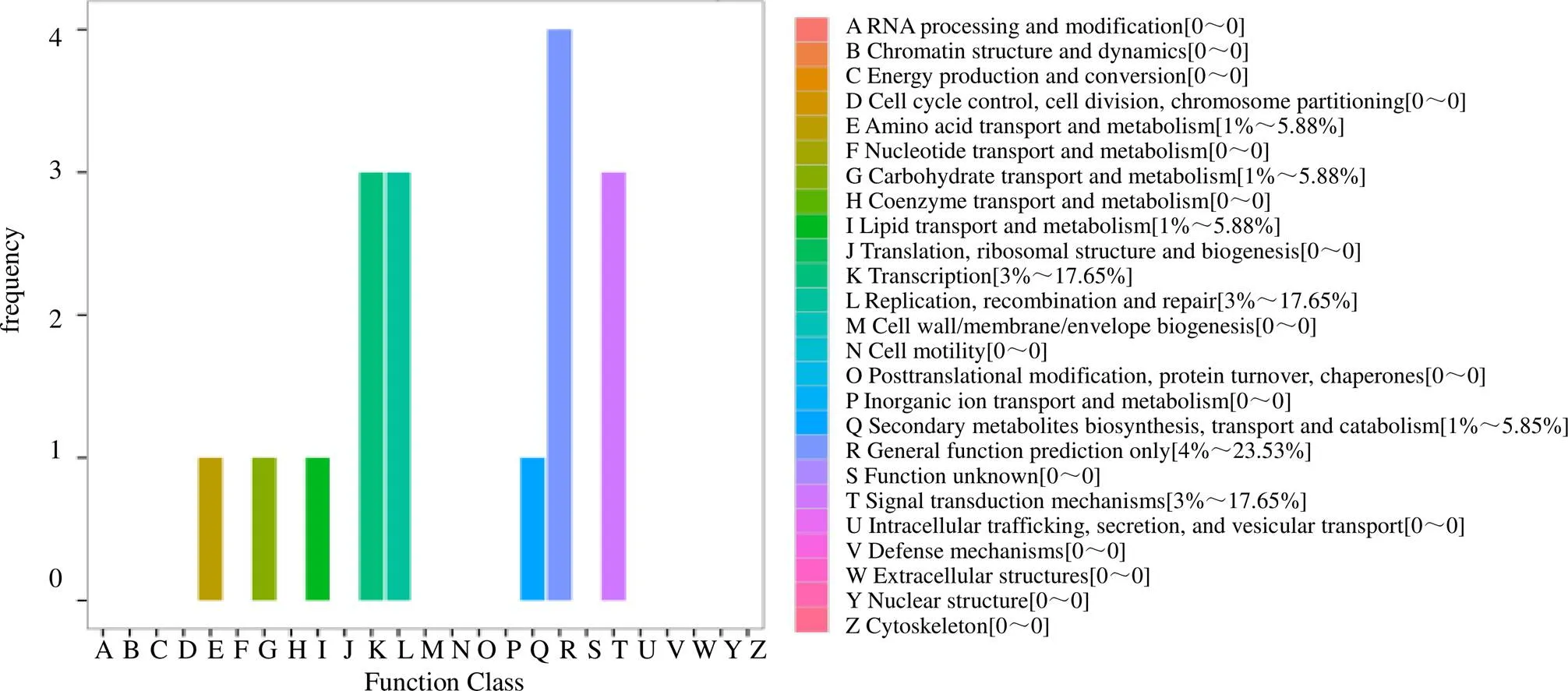
橫坐標為COG各分類內容,縱坐標為基因數目

橫坐標為KOG各分類內容,縱坐標為基因數目
表6 差異表達基因KEGG功能注釋
Table 6 KEGG functional annotation ofdifferentially expressed genes
編號KEGG功能注釋基因數量基因ID ko00561甘油酯代謝1c77096.graph_c1 ko00591亞油酸代謝1c31079.graph_c0 ko00940苯丙烷類生物合成1c68267.graph_c0 ko00360苯丙氨酸代謝1c68267.graph_c0
3.4 花藥發育相關基因篩選
通過對枸杞轉錄組測序結果進行功能注釋、功能分類及代謝途徑分析發現,共篩選到11個與花藥發育相關的差異表達基因(表7)。
3.5 差異表達基因的qRT-PCR驗證
隨機選取5個差異表達基因進行qRT-PCR分析。5個差異表達基因即過氧化物酶(peroxidase,PRX)、阿拉伯半乳糖蛋白(arabinogalactan protein,AGP)、E3泛素蛋白連接酶(E3 ubiquitin-protein ligase MBR1)、類受體蛋白激酶(receptor protein kinase-like,RPK)和富含亮氨酸重復蛋白(leucine-rich repeat protein,LRR),在S2時期的表達量相對于S1時期的表達量均下調,且5個差異表達基因表達水平在S2時期極顯著低于S1時期,與轉錄組測序結果基本一致(圖5),表明轉錄組數據結果可靠。
表7 花藥發育相關差異表達基因
Table 7 Differentially expressed genes related to anther development
基因ID基因描述變化倍數基因表達 c68267.graph_c0過氧化物酶?4.590 996 613下調 c77096.graph_c1二酰基甘油酰基轉移酶?2.919 934 769下調 c75034.graph_c0過氧化物酶?3.479 978 950下調 c72608.graph_c0富含亮氨酸重復蛋白?3.232 330 297下調 c73778.graph_c0硫氧還蛋白?2.932 284 773下調 c77007.graph_c0E3泛素蛋白連接酶?3.571 161 469下調 c68844.graph_c0阿拉伯半乳糖蛋白?3.730 754 896下調 c77579.graph_c0限制性內切酶?3.097 051 914下調 c77438.graph_c0內切酶?2.956 411 029下調 c69446.graph_c0細胞周期蛋白?3.158 118 976下調 c31079.graph_c0脂氧合酶?3.124 745 110下調

***代表差異極顯著,P<0.001
3.6 相關性分析
利用統計學軟件對S1和S2 2個時期的轉錄組數據和qRT-PCR結果進行相關性分析,結果表明S1、S2 2個時期差異表達基因的qRT-PCR結果與轉錄組數據相關系數分別為0.25、0.41,呈正相關,說明轉錄組數據可靠。
4 討論
本研究對“寧杞1號”花藥發育過程中S1時期和S2時期的花藥進行轉錄組測序,測序結果與NR、GO、COG、KOG、KEGG、Swiss-Prot、Pfam 7個數據庫比對,有注釋信息的Unigene占Unigene總數的31.32%,未被注釋到的Unigene占68.68%,可能原因是這些Unigene為非編碼序列,無法進行同源序列比對;另外,由于枸杞基因組信息缺乏,這些沒有被注釋到的基因可能是一些未知的基因。對于篩選的差異基因數目較少,推測原因可能是大多數基因在S1時期和S2時期差異表達不明顯。
花藥發育轉錄組分析顯示,在S1和S2 2個時期,有46個基因差異表達,且在S2時期全部下調表達。46個差異表達基因中,有33個基因在NR數據庫中得到注釋。將33個基因在PubMud數據庫檢索發現,PRX、二酰基甘油酰基轉移酶、RPK、富含亮氨酸重復基因、硫氧還蛋白、E3泛素蛋白連接酶、AGP、限制性內切酶等11個基因(表7)與擬南芥[15-16]等植物花藥發育過程有關。GO和KEGG功能分析表明,有4個差異表達基因被注釋到甘油酯代謝、亞油酸代謝、苯丙氨酸代謝和苯丙烷類生物合成4個代謝途徑中,其中編碼PRX的基因既在苯丙氨酸代謝中表達,也在苯丙烷生物合成中表達,說明該基因的功能與這2個代謝途徑比較密切。
PRX是苯丙烷類生物合成中的一類關鍵酶,苯丙烷類生物合成作為一個重要的代謝通路,是苯丙素代謝網絡的核心。苯丙素生物合成會影響植物直立生長,植物體內長途水分運輸[16]以及花器官的發育[17-18]。徐青等[14]的研究結果顯示,S1時期,絨氈層細胞退化,降解產物流入藥室,匯聚形成中央大液泡;S2時期,絨氈層細胞完全消失,表明絨氈層降解產物是花藥發育必須的營養物質;Jacobowitz等[15]的研究發現,基因是擬南芥花藥發育過程中維持絨氈層和小孢子細胞壁完整性必不可少的,PRX基因突變會導致絨氈層的肥大而導致雄性不育;同時,Hecht等[16]的研究發現,受體樣蛋白激酶2基因屬于LRR-RLK家族,是花藥中層和絨氈層分化所必需的,受體樣蛋白激酶2基因突變的花藥中不存在中間層,且絨氈層顯示肥大,表明受體樣蛋白激酶2會影響絨氈層的發育來控制花粉的成熟。本研究結果顯示,基因和受體樣蛋白激酶2基因在花藥發育S2時期的表達量下調,由此推測,在S1時期花藥發育需要絨氈層的降解產物提供營養,此時基因和受體樣蛋白激酶2基因的表達量較高,而S2時期花粉已完全成熟,不再需要絨氈層的降解來提供營養,所以這2個基因的表達量較S1時期下調。這與前人的研究結果一致,因此推測和受體樣蛋白激酶2基因是S1時期降解絨氈層為花藥發育提供營養物質必需的基因。此外,甘油酯代謝過程受到二酰基甘油酰基轉移酶基因(diacylglycerol acyltransferase,DGAT)的表達調控,Banilas等[19]的研究結果顯示,基因在成熟或衰老的橄欖組織中高度表達,同時基因轉錄物也在花藥和子房發育的后期積累,表明基因表達主要影響著花藥成熟時期的發育;這與本課題組的研究結果不同,推測可能原因是S2時期花藥的發育受到基因和其他基因共同調控。
隨機選取5個差異表達基因進行qRT-PCR驗證和相關性分析,qRT-PCR結果與轉錄組數據一致。相關性分析結果表明,S1、S2 2個時期5個差異表達基因的qRT-PCR結果與轉錄組數據呈正相關,說明轉錄組數據真實可靠。本研究篩選到的差異表達基因,在花藥發育過程中發揮重要的作用,這些差異表達基因的發現為后續研究枸杞花藥發育提供基因資源和奠定研究基礎。
利益沖突 所有作者均聲明不存在利益沖突
[1] Zhao J H, Li H, Yin Y,. Fruit ripening inandis associated with distinct gene expression patterns [J].2020, 10(8): 1550-1567.
[2] 張興, 周麗, 喬亞蕊, 等. 枸杞Lb14-3-3c基因克隆及轉化馬鈴薯的研究 [J]. 植物遺傳資源學報, 2019, 20(6): 1523-1534.
[3] 陳廷卓. 枸杞子的化學成分與藥理作用研究進展 [J]. 中醫臨床研究, 2010, 2(22): 24.
[4] 胡若琳, 袁超, 牛義, 等. 植物MYB轉錄因子在花藥發育中的調控作用 [J]. 生物工程學報, 2020, 36(11): 2277-2286.
[5] 張在寶, 趙海, 胡夢輝, 等. 組學在花藥發育研究中的應用進展I: 轉錄組學 [J]. 生物技術進展, 2019, 9(5): 433-439.
[6] Wang X H, Xu N, Dong K,. SlNCED1 affects pollen maturation in tomato by regulating the expression of anther-specific genes [J]., 2021, 95(2): 191-205.
[7] Yuan G, Wu Z, Liu X,. Characterization and functional analysis of LoUDT1, a bHLH transcription factor related to anther development in the lily oriental hybrid Siberia (spp.) [J]., 2021, 166: 1087-1095.
[8] Ortolan F, Fonini L S, Pastori T,. Tightly controlled expression of OsbHLH35 is critical for anther development in rice [J]., 2021, 302: 110716.
[9] 鄭國琦. 寧夏枸杞果實結構、發育與糖分積累關系研究 [D]. 西安: 西北大學, 2011.
[10] 李捷, 崔永濤, 柏延文, 等. 兩種枸杞對干旱脅迫的生理響應及抗旱性評價 [J]. 甘肅農業大學學報, 2019, 54(5): 79-87.
[11] 徐蕊. 枸杞LbMYB103基因在植物花器官發育中的功能初探 [D]. 銀川: 寧夏大學, 2018.
[12] Grabherr M G, Haas B J, Yassour M,. Full-length transcriptome assembly from RNA-Seq data without a reference genome [J]., 2011, 29(7): 644-652.
[13] 葉國盛. 枸杞特優品種寧1號 [J]. 農技服務, 1996, (9): 24.
[14] 徐青, 秦墾, 馮愛玲, 等. 枸杞不育系與可育系花藥發育的細胞學研究 [J]. 寧夏大學學報: 自然科學版, 2009, 30(3): 263-267.
[15] Jacobowitz J R, Doyle W C, Weng J K. PRX9 and PRX40 are extensin peroxidases essential for maintaining tapetum and microspore cell wall integrity duringanther development [J]., 2019, 31(4): 848-861.
[16] Hecht V, Vielle-Calzada J P, Hartog M V,. TheSOMATIC EMBRYOGENESIS RECEPTOR KINASE 1 gene is expressed in developing ovules and embryos and enhances embryogenic competence in culture [J]., 2001, 127(3): 803-816.
[17] Li Y, Kim J I, Pysh L,. Four isoforms of4-coumarate: CoA ligase have overlapping yet distinct roles in phenylpropanoid metabolism [J]., 2015, 169(4): 2409-2421.
[18] Kumar A, Ellis B E. The phenylalanine ammonia-lyase gene family in raspberry. Structure, expression, and evolution [J]., 2001, 127(1): 230-239.
[19] Banilas G, Karampelias M, Makariti I,. The olive DGAT2 gene is developmentally regulated and shares overlapping but distinct expression patterns with DGAT1 [J]., 2011, 62(2): 521-532.
Anther transcriptome and screening of genes related to anther development in
FAN Wen-qiang1, LIU Xue-xia1, MA Xiao-lan1, WANG Lu-yao1, ZHANG Xing1, YUE Si-jun1, TANG Jian-ning2, ZHU Jin-zhong3, YAO Xin-ling1, ZHENG Rui1
1. Key Laboratory of Protection and Utilization of Special Biological Resources in Western China, Ministry of Education, Key Laboratory of Modern Molecular Breeding for Dominant and Special Crops in Ningxia, College of Life Science, Ningxia University, Yinchuan 750021, China 2.Wolfberry Industry Development Center, Yinchuan 750021, China 3. Qixin Wolfberry Seedling Professional Cooperatives of Zhongning County, Zhongning 755100, China
To investigate the anther development process of, and explore the genes related.The anther transcriptome analysis was performed by the high-throughput Illumina HiSeq2500 sequencing platform using anthers of single microspore and bi-cellular pollen stage and mature pollen stage as research materials.A total of 128,151 unigene were obtained after assembly, and 40,140 unigenes were annotated by searching databases of NR, Swiss-Prot, GO, COG, KOG and KEGG. Under the condition of FDR (false discovery rate) <0.05 and FC (fold change) ≥2, a total of 46 differentially expressed genes were found between single microspore and bi-cellular pollen stage and mature pollen stage. KEGG annotations identified four differentially expressed genes involved in four pathways: glycerol ester metabolism, linoleic acid metabolism, phenylalanine metabolism and phenylpropanoid biosynthesis.There were eleven differentially expressed genes were involved in the anther development process, such as leucine-rich repeat protein (LRR), cyclin (Cyclin), receptor-like protein kinase (RPK), E3 ubiquitin protein ligase (MBR1), and so on. Five of the different expression genes among the eleven genes were randomly selected for qRT-PCR verification, and the expression results were basically consistent with the transcriptome data, indicating that the data were true and reliable.This study lays the foundation for further explaining the molecular mechanisms related to anther development in., and also provides useful genetic resources for subsequent studies on the biological characteristics of anther development in.
L.; anther development; transcriptome; differentially expressed genes; biological characteristics
R282.12
A
0253 - 2670(2022)03 - 0827 - 08
10.7501/j.issn.0253-2670.2022.03.023
2021-10-06
寧夏自然科學基金資助項目(2020AAC03097);寧夏枸杞產業發展中心項目“寧夏枸杞核心種質資源差異性分析及功能基因挖掘”;寧夏回族自治區重點研發計劃(重大)重點項目(2019BBF02022)
范文強,男,碩士,研究方向為植物生物學。E-mail: 1534204028@qq.com
鄭 蕊,女,博士,研究方向為植物生物技術。E-mail: xlzheng@126.com
[責任編輯 時圣明]

