基于轉錄組測序的椰心葉甲嚙小蜂SSR、SNP和InDel位點分析
劉華偉,李朝緒,李芬,呂朝軍,吳少英,覃偉權
摘? 要:椰心葉甲嚙小蜂(Tetrastichus brontispae)是外來有害生物椰心葉甲(Brontispa longissima)的蛹期寄生蜂,分析其轉錄組序列中的SSR、SNP和InDel位點信息,可以為開發新的分子標記,深入研究其遺傳多樣性、種群遺傳結構和歷史動態等提供數據支撐。本研究基于轉錄組數據,利用MISA軟件和Varscan軟件對Unigene進行SSR、SNP和InDel位點進行搜索。在11 802條Unigene中共獲得29 754個SSR位點,平均每1.72 kb含有1個SSR位點,發生率為39.96%。SSR片段為10~382 bp,長度具有顯著差異,平均長度為23.91 bp。SSR片段中,單堿基重復最多(60.82%),其次是二堿基重復(27.69%),再次為三堿基重復(10.79%)。其中優勢重復基元類型為A/T(59.32%),其次為AT/AT(15.28%)。在6895個Unigene中發掘出51 334個SNP位點,轉換位點37 445個(72.94%),顛換位點13 975個(27.22%),平均每條Unigene上含有7.45個SNP位點。還在6040個Unigene中篩選出15 644個InDel位點,平均每條Unigene上有2.59個InDel位點。椰心葉甲嚙小蜂轉錄組中SSR、SNP和InDel位點數量多,出現頻率高,類型豐富,具有較高的多態性潛能。
關鍵詞:椰心葉甲嚙小蜂;轉錄組;SSR;SNP;InDel
中圖分類號:S476? ? ? 文獻標識碼:A
SSR, SNP and InDel Analysis Based on Tetrastichus brontispae Transcriptome
LIU Huawei1,2,3,4,, LI Chaoxu1,3,4, LI Fen2, LYU Chaojun1,3,4, WU Shaoying2, QIN Weiquan1,3,4*
1. Coconut Research Institute, Chinese Academy of Tropical Agricultural Sciences, Wenchang, Hainan 571399, China; 2. Hainan University, Haikou, Hainan 570228, China; 3. Hainan Innovation Center of Academician Team (Integrated Management of Arecanut Yellow Leaf Disease), Wenchang, Hainan 571339, China; 4. Hainan Key Laboratory of Tropical Oil Crops Biology, Wenchang, Hainan 571399, China.
Abstract: Tetrastichus brontispae is the pupal parasitic wasp of Brontispa longissima, an exotically invasive pest. The analysis of SSR, SNP and InDel sites in the transcriptome sequences of T. brontispae can provide data support for the development of new molecular markers and the in-depth study of its genetic diversity, population genetic structure and historical dynamics. Based on transcriptional data, MISA software and Varscan software were used to search the SSR, SNP and InDel sites of Unigenes. A total of 29 754 SSR sites were obtained in 11 802 Unigenes, with an average of 1 SSR per 1.72 kb, with an incidence of 39.96%. The length of the SSR fragments was 10-382 bp, with an average length of 23.91 bp. In the SSR segment, mononucleotide was dominant (60.82%), followed by dinucleotide (27.69%) and trinucleotide (10.79%). In all repeating motifs, the dominant repeating motif was A/T (59.32%), followed by AT/AT (15.28%). Among 6 895 Unigenes, 51 334 SNP sites were discovered, and each Unigene contained 7.45 SNP on average. There were 37 445 transition sites (72.94%) and 13 975 transversion points (27.22%). 15 644 InDel sites were also identified out of 6 040 Unigene, with an average of 2.59 InDel per Unigene.SSR, SNP and InDel sites are abundant in the transcriptome of T. brontispae, with a large number, high occurrence frequency, rich type and polymorphism potential.
Keywords: Tetrastichus brontispae; transcriptome; SSR; SNP; InDel
DOI: 10.3969/j.issn.1000-2561.2021.10.011
椰心葉甲嚙小峰(Tetrastichus brontispae Ferrière)屬膜翅目(Hymenoptera)姬小蜂科(Eulophidae)嚙小蜂屬(Tetrastichus),是重大危險性外來有害生物椰心葉甲[Brontispa longissima (Gestro)]的寄生性天敵[1],主要寄生蛹期的椰心葉甲,通過野外釋放該蜂可以有效地防控椰心葉甲危害。目前國內外已對椰心葉甲嚙小蜂的成蟲習性、寄主適應性、生物生態學特性、室內大量繁殖和野外釋放等方面進行了相關研究[1-5]。
分子標記技術隨著分子生物學的快速發展在動植物中被廣泛應用。在各種分子標記中,簡單重復序列(simple sequence repeat,SSR)又稱作微衛星,因具有數量多易檢測、多態性信息豐富、呈共顯性遺傳等優點而成為研究群體遺傳學、遺傳育種、保護遺傳學以及系統進化等方面的有力工具,在動植物、微生物以及人類醫學等各領域都得到了廣泛的應用。隨著高通量測序技術的逐步發展,基于轉錄組數據篩選SSR位點在動植物研究中也被廣泛應用[6-7]。單核苷酸多態性(single nucleotide polymorphisms,SNP)由于其具有分布廣、位點多、易檢測、準確率高、遺傳穩定性高、并且在不同物種中具有顯著差異等特點而也被廣泛應用[8-9]。插入/缺失多態性(insenion-deletion,InDel)標記是根據核苷酸片段的插入或缺失而開發的,具有分布廣(僅次于SNP位點)、可重復性高、密度高、成本較低、變異率低、多態性強且易于檢測等優點,可以利用InDel進行優異基因挖掘、基因精細定位、遺傳多樣性分析等研究[10-12]。
由于椰心葉甲嚙小蜂在基因信息方面比較缺乏,所以目前國內外對其分子標記方面的研究仍是空白。本研究利用MISA和Varscan軟件,基于椰心葉甲嚙小蜂轉錄組數據對SSR、SNP和InDel位點進行搜索并分析,以期為椰心葉甲嚙小蜂及近緣種的SSR、SNP和InDel分子標記的開發深入研究其遺傳多樣性、種群遺傳結構和歷史動態提供參考依據。
1? 材料與方法
1.1? 材料
材料已上傳到NCBI SRA數據庫中,GenBank登錄號:PRJNA678031。轉錄組數據質量參照劉華偉等[13]的研究。
1.2? 方法
1.2.1? 轉錄組SSR的篩選及分析? 利用MISA軟件(https://webblast.ipk-gatersleben.de/misa/)從椰心葉甲嚙小蜂轉錄組的Unigene中進行SSR搜索,篩選標準:單核苷酸重復數≥10,二核苷酸重復數≥6,三核苷酸、四核苷酸、五核苷酸、六核苷酸≥5;運用Excel軟件對轉錄組的SSR各類型比例、序列分布和特征進行分析和統計。
SSR發生頻率=含SSR的Unigene數與Unigene總數的比值;SSR分布的平均距離=總Unigene長度與搜索到的SSR數量的比值。
1.2.2? 轉錄組中SNP和InDel的篩選及分析? 利用Varscan軟件(http://varscan.sourceforge.net/),搜索候選SNP和InDel位點。篩選標準:SNP/InDel位點堿基Q>20;覆蓋該位點的Reads數目>8;支持突變位點的Reads數目>2;SNP/InDel位點P<0.01。
2? 結果與分析
2.1? SSR重復基元的分布
通過MISA軟件,在11 802條Unigene檢測到29 754個SSR位點,SSR的發生頻率為39.96%,平均1.72 kb出現1個SSR位點。包含1個以上SSR位點的Unigene有6177條,復合型的SSR位點有4346個。
2.2? 椰心葉甲嚙小蜂轉錄組中SSR的數量和分布特點
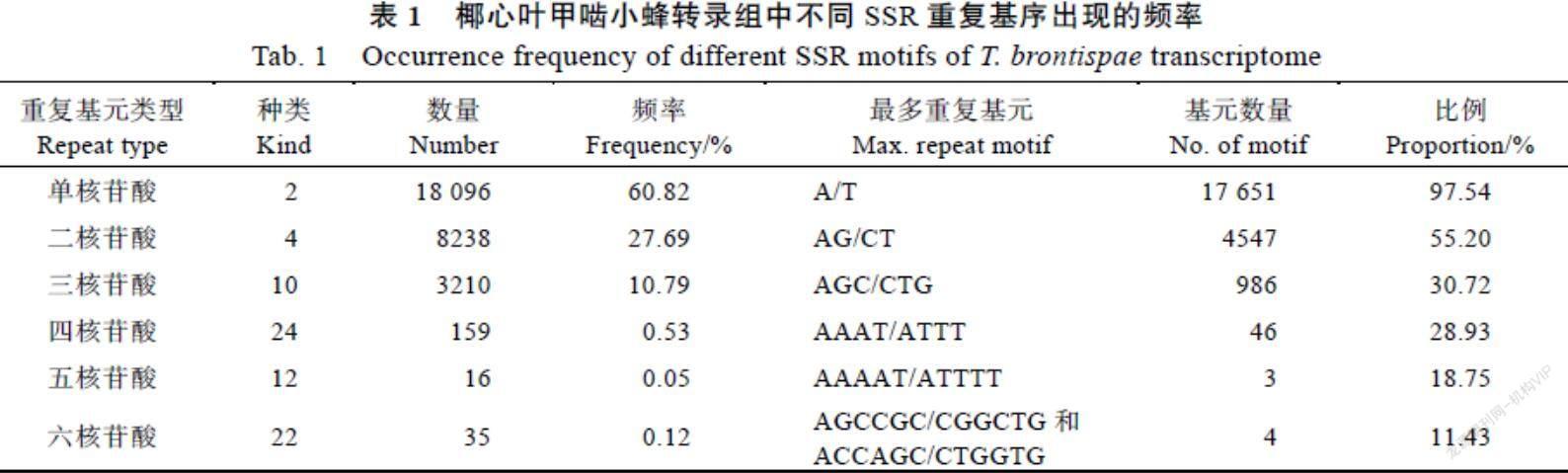
各堿基類型中的SSR序列占比差異較大(表1),共有74種重復基序。單核苷酸~六核苷酸堿基重復基元分別有2、4、10、24、12、22種。單核苷酸為優勢重復類型(60.82%),其中A/T出現的最多(97.54%);二堿基重復SSR含量約占總數的27.69%,AG/CT出現的頻率為55.20%;在三堿基重復SSR(10.79%)中,AGC/CTG出現的頻率為30.72%;而四堿基、五堿基、六堿基重復SSR占比較少(0.05%~0.53%)。出現頻率最高的重復基元分別是AAAT/ATTT(28.93%)、AAAAT/ATTTTT(18.75%)、AGCCGC/CGGCTG和ACCAGC/CTGGTG(11.43%)。在所有的堿基重復模式中,各種重復基元中在總SSR中的比例> 0.5%有13類,不同類型重復基序SSR比例分布見圖1。
2.3? SSR長度分析
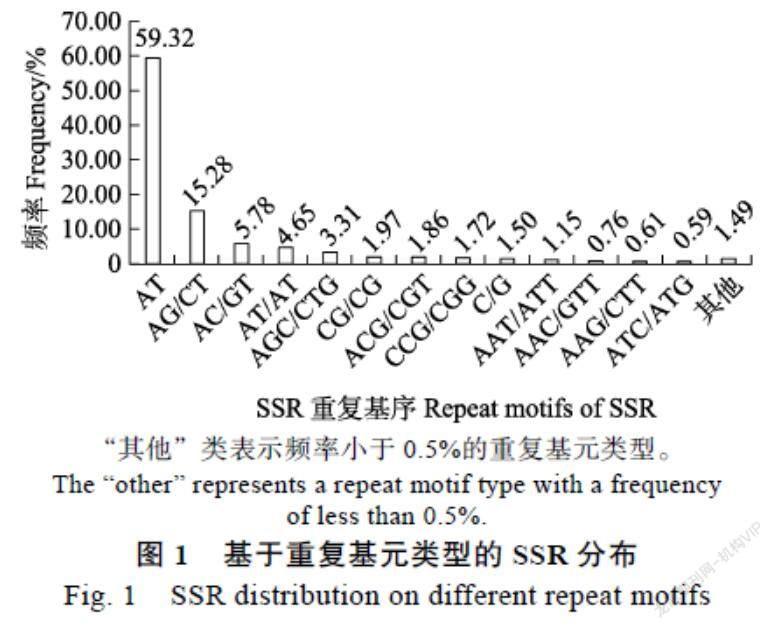
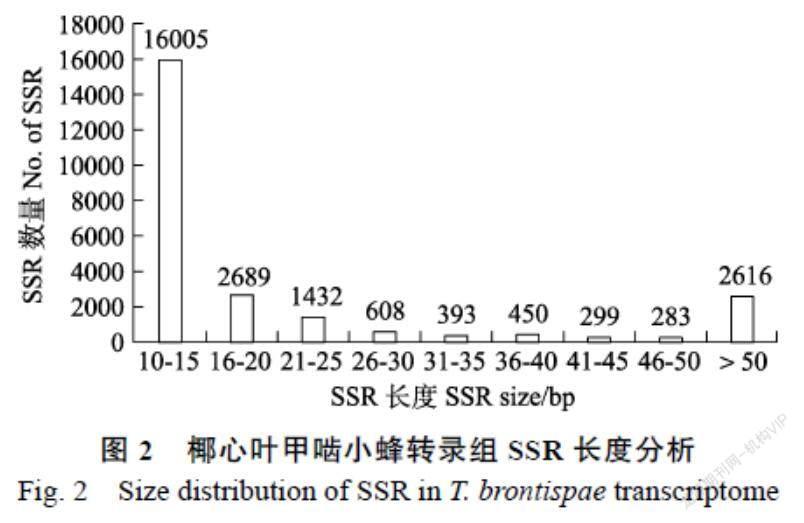
SSR長度也存在極顯著變異。椰心葉甲嚙小蜂轉錄組SSR片段長度由10~382 bp,平均長度23.91 bp。主要是重復長度≤20 bp的序列,有18 695條,占總數的73.58%,長度大于50 bp的長序列占SSR總數的10.30%(圖2)。
SSR數量與重復次數的關系如圖3所示,各種SSR的數量均與重復次數呈負相關。其中單核苷酸曲線的下降速率最大,當重復次數達到13次時,下降速率變慢。其他核苷酸曲線的下降趨勢趨于平緩。
2.4? 椰心葉甲嚙小蜂轉錄組SNP和InDel的特征分析
在獲得的轉錄組數據中,利用Varscan軟件進行SNP搜索,在6895個Unigene中檢測51 334個SNP位點,平均每1000 bp出現一個SNP位點,在所有的SNP位點中,轉換(Transition)位點有37 445個,顛換(Transversion)位點有13 975個,SNP位點統計見表2。
從圖4可見,含有1個SNP位點的Unigene最多(25.74%),其次是含有2個SNP位點的Unigene(14.58%),含有3~10個SNP位點的Unigene數量呈下降趨勢(136~756個Unigene),在1186個Unigene上發現了超過10個SNP位點,平均每條Unigene上有7.45個SNP位點。
利用Varscan軟件對InDel位點進行搜索,最終在6040個Unigene中共找到15 644個InDel位點,每3281 bp出現1個InDel位點。InDel位點分布趨勢和SNP位點的分布趨勢相同(圖5),含有1個SNP位點的Unigene最多(43.77%),其次是2個InDel位點(21.79%),含有3~10個InDel位點的Unigene呈下降趨勢(31~726個Unigene),在83個Unigene上發現了超過10個InDel位點,平均每條Unigene上有2.59個InDel位點。
3? 討論
近年來,隨著測序成本的降低和組學研究的深入,基于轉錄組數據篩選、分析SSR、SNP和InDel等分子標記位點在曼氏無針烏賊[7]、大黃魚[9]、甜菜[10]、大麥[11]等動植物中被大量研究,特別是在品種鑒定和種群關系分析方面。由于椰心葉甲嚙小蜂遺傳信息的缺乏,對其分子標記方面的研究仍是空白。
本研究在椰心葉甲嚙小蜂轉錄組數據中篩選出29 754個SSR位點,發生頻率為39.96%,比已報道的大部分昆蟲,如黑腹胃蠅(31.09%)[14]、溫帶臭蟲(18.86)[6]、印度谷螟(8.52%)[15]、沙蔥螢葉甲(4.53%)[16]、桔小實蠅(4.23%)[17]和黃粉甲(1.67%)[18]等數量都要高。并且其SSR平均分布距離(每1.72 kb出現一個SSR位點)也比印度谷螟(13.38 kb)[14]和桔小實蠅(10.21 kb)[17]要小得多。出現這種現象的原因可能與物種的特異性、測序數據量大小以及數據的篩選參數有關[6],在一定程度上說明椰心葉甲嚙小蜂中含有豐富的SSR標記位點,具有較高的多態性潛能。
在唐培安等[15]的研究中提出,普遍認為基于轉錄組數據挖掘的昆蟲SSR位點以三堿基重復為主。然而在椰心葉甲嚙小蜂中最優重復單元為單堿基重復(60.82%),主要重復基序是A/T(97.54%),這與溫帶臭蟲[6]、沙蔥螢葉甲[16]的研究結果相一致。另外有一些昆蟲以二堿基重復為主,如印度谷螟[15],還有一些昆蟲單堿基和三堿基占比相近,都是優勢重復單元,如黑腹胃蠅[14]和黃粉甲[18]。推測出現此現象的原因可能與物種本身的基因型和SSR的篩選參數有關[19]。
值得注意的是,在Meglécz等[20]和Yoon等[21]的研究中表示,由于胞嘧啶(C)容易甲基化突變為胸腺嘧啶(T),推測在動植物的轉錄組或基因組中,GC/CG的數量都幾乎接近0。但在李敏等[6]和唐培安等[15]的研究中表示,雖然大部分昆蟲中GC/GC的含量非常低甚至沒有,不過也在鱗翅目玉米粘蟲、二點委夜蛾和細梢小卷蛾中發現GC/GC基序不僅常見而且有較高的占比。本研究中,椰心葉甲嚙小蜂的GC/CG同樣是二堿基重復SSR中占比最少的重復基元(0.02%),但數量有586個,這與大部分昆蟲有差異。
Meglécz等[20]研究表示,SSR的長度越長,其核心基元的重復次數越多,多態性越高。Temnykh[22]的研究認為,當SSR長度≥20 bp時,多態性較高,當12 bp 本研究利用Varscan軟件,在6895個Unigene中檢測51 334個SNP位點,平均每1000 bp出現一個SNP位點,其中轉換位點有37 445個,顛換位點有13 975個。從理論上來說轉換∶顛換應該等于1∶2,在本研究中,轉換比顛換的比率達到2.68,遠大于理論值,這種現象稱為轉換偏差,這與堿基組成和進化過程中的選擇機制有關,說明堿基的轉換突變可能不是隨機產生的[23]。在轉換類型中,C-T的發生頻率最高,并且多是C轉換為T,這可能與CG中的胞嘧啶(C)常為甲基化的,脫氨后就變成胸腺嘧啶(T)有關[24]。另外還發現,轉換位點和顛換位點的總和與SNP位點總數不相等,這是因為在同一個位點同時發生轉換和顛換2種突變而導致。在椰心葉甲嚙小蜂轉錄組數據中,平均每條Unigene上含有7.45個SNP位點,其中含有1個SNP位點的Unigene最多(25.74%),其次是含有2個SNP位點的(14.58%)。除了SNP位點,本研究還利用Varscan軟件,在6040個Unigene中15 644個InDel位點,平均每3281 bp出現一個InDel位點。InDel位點的數量分布與SNP大致相同,也是一個InDel位點最多(43.77%),其次是2個InDel位點的(21.79%),平均每條Unigene上有2.59個InDel位點。 本研究利用轉錄組測序數據挖掘椰心葉甲嚙小蜂SSR、SNP和InDel位點,并對其特征進行了分析,為后續開發和應用分子標記的提供了數據支撐。通過進一步的設計、篩選引物,還可應用到椰心葉甲嚙小峰的遺傳多樣性分析、種群鑒定和遷飛能力評估等方面。 參考文獻 [1] 呂寶乾, 彭正強, 許春靄, 等. 椰心葉甲蛹寄生蜂: 椰心葉甲嚙小蜂的生物學特性[J]. 昆蟲學報, 2006, 49(4): 643-649. [2] 黃山春, 覃偉權, 周煥起, 等. 椰心葉甲嚙小蜂的繁殖生物學研究[J]. 華東昆蟲學報, 2007, 16(3): 168-171, 238. [3] 周? 祥, 黃光斗, 馬子龍, 等. 椰心葉甲嚙小蜂對寄主的選擇性、適宜性和功能反應[J]. 熱帶作物學報, 2006, 27(2): 74-77. [4] 周煥起, 馬子龍, 覃偉權, 等. 椰心葉甲的寄生性天敵: 椰心葉甲嚙小蜂和椰甲截脈姬小蜂的室內培育[J]. 中國生物防治, 2006, 22(S1): 6-10. [5] 金? 濤, 金啟安, 溫海波, 等. 利用寄生蜂防治椰心葉甲的概況及研究展望[J]. 熱帶農業科學, 2012, 32(7): 67-74. [6] 李? 敏, 王? 青, 陳? 晨, 等. 基于轉錄組測序的溫帶臭蟲SSR和SNP位點分析[J]. 山西農業大學學報(自然科學版), 2019, 39(4): 52-57. [7] 孫? 揚, 郭寶英, 祁鵬志, 等. 基于轉錄組的曼氏無針烏賊SSR與SNP位點信息分析[J]. 浙江海洋大學學報(自然科學版), 2019, 38(2): 100-106. [8] 李小白, 向? 林, 羅? 潔, 等. 轉錄組測序(RNA-seq)策略及其數據在分子標記開發上的應用[J]. 中國細胞生物學學報, 2013, 35(5): 720-726, 740. [9] 王盼盼. 大黃魚基因組和轉錄組SNP的挖掘與應用[D]. 廈門: 集美大學, 2016 [10] 黃平仙, 高永明, 劉乃新, 等. 基于全基因組重測序技術分析甜菜InDel標記[J]. 中國糖料, 2020, 42(3): 1-6. [11] 徐婷婷, 汪巧玲, 鄒淑瓊, 等. 基于高通量測序的大麥InDel標記開發及應用[J]. 作物學報, 2020, 46(9): 1340-1355. [12] 劉? 宇, 閆彩霞, 李春娟, 等. 花生栽培種InDel有效標記篩選與評估[J]. 核農學報, 2020, 34(2): 256-264. [13] 劉華偉, 李朝緒, 李? 芬, 等. 椰心葉甲嚙小蜂轉錄組分析及基因功能注釋[J]. 中國生物防治學報, 2021, 37(3): 412-419. [14] 陳亙濃, 黃河清, 張博茹, 等. 基于轉錄組數據的黑腹胃蠅微衛星位點信息分析[J]. 環境昆蟲學報, 2018, 40(6): 1219-1224. [15] 唐培安, 陶冶心, 薛? 昊, 等. 基于轉錄組數據的印度谷螟微衛星位點分析[J]. 植物保護, 2017, 43(3): 43-48, 75. [16] 張鵬飛, 周曉榕, 龐保平, 等. 基于轉錄組數據高通量發掘沙蔥螢葉甲微衛星引物[J]. 應用昆蟲學報, 2016, 53(5): 1058-1064. [17] 魏丹丹, 石俊霞, 張夏瑄, 等. 基于轉錄組數據的桔小實蠅微衛星位點信息分析[J]. 應用生態學報, 2014, 25(6): 1799-1805. [18] Zhu J, Wu G, Yang B. High-throughput discovery of SSR genetic markers in the yellow mealworm beetle, Tenebrio molitor (Coleoptera: Tenebrionidae), from its transcriptome database[J]. Acta Entomologica Sinica, 2013, 56(7): 724- 728. [19] 郭? 睿, 陳華枝, 莊天藝, 等. 利用轉錄組數據開發意大利蜜蜂的SSR分子標記[J]. 安徽農業大學學報, 2018, 45(3): 404-408. [20] Meglécz E, Nève G, Biffin E, et al. Breakdown of phylogenetic signal: a survey of microsatellite densities in 454 shotgun sequences from 154 non model eukaryote species[J]. PLoS One, 2012, 7(7): e40861. [21] Yoon J M. Genetic variations between hairtail (Trichiurus lepturus) populations from Korea and China[J]. Development and Reproduction, 2013, 17(4): 363-367. [22] Temnykh S. Computational and experimental analysis of microsatellites in rice (Oryza sativa L.): frequency, length variation, transposon associations, and genetic marker potential[J]. Genome Research, 2001, 11(8): 1441-1452. [23] Zhao H, Li Q Z, LI J, et al. The study of neighboring nucleotide composition and transition/transversion bias[J]. Science in China Series C: Life Sciences, 2006, 49(4): 395-402. [24] Garg K. Identification of candidate coding region single nucleotide polymorphisms in 165 human genes using assembled expressed sequence tags[J]. Genome Research, 1999, 9(11): 1087-1092. 責任編輯:黃東杰

