基于高維隨機矩陣的癌癥基因網絡識別方法
李 蓉,任喜梅,鐘春曉,王錦麗
(華東交通大學理工學院,江西 南昌 330100)
1 引言
癌癥由多階段的多基因共同參與而生成、發展,并與基因變化有著緊密聯系,癌癥的發生、促進、發展以及轉移,大部分都跟原癌基因[1]活化、抑癌基因[2]失活等基因突變密切相關。具有挖掘隱含生物學信息功能的微陣列數據,可以依據基因間的性能模塊中已知基因預測未知基因,但因其維數較高,存在較少數量與癌癥相關的基因。高維隨機矩陣理論(Random Matrix Theory,RMT)通過對比隨機的高維、多維序列屬性特征,發現實際數據與隨機因素之間的偏離程度,提取數據內的總體相關行為特征。
為此,本文將高維隨機矩陣與癌癥基因融合,提出一種癌癥基因網絡識別方法,將系數矩陣右邊增加一列,擴增隨機矩陣,提升隨機矩陣特征擬合度;通過規范化、中心化以及標準化隨機矩陣,提升矩陣適用性能;以互信息作為度量標準,依據各隨機矩陣奇異值矢量與初始特征奇異值矢量的差值,優化特征選擇;利用癌癥基因表達矩陣特征根的最近鄰間隔分布與高斯正態分布、泊松分布的標準誤差比值計算,增加有效信息的保留數量,增強噪聲濾除性能。
2 高維隨機矩陣下癌癥微陣列數據預處理
2.1 高維隨機矩陣構建
將維度較高的隨機變量作為矩陣組成元素,即可構成高維隨機矩陣,依據隨機矩陣理論含義,設定某一高維隨機矩陣為M,表達式如下所示

(1)
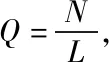
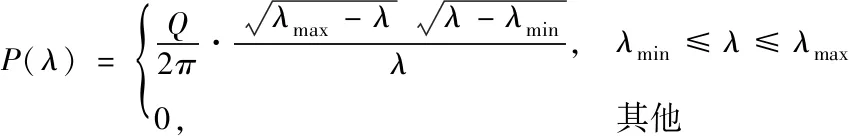
(2)
式中,隨機矩陣M的極大、極小特征值分別用λmax和λmin來表示,相應表達式如下所示
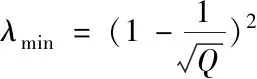
(3)

(4)
綜上所述,利用隨機矩陣M與相關矩陣C具有的屬性特征,劃分矩陣C為符合隨機矩陣M部分與差異部分,即隨機噪聲U與真實信息V,通過優化相關矩陣C,即可去除其中所含噪聲。
2.2 癌癥微陣列數據特征選擇優化
基因表達網絡[3]受實驗條件影響,一般會存在一些隨機因素:當實驗時間與樣本條件發生變化時,基因表達水平也將隨之改變;若實驗樣本有限,則有可能產生測量噪音。而此類隨機因素生成的虛假信息,會對真實信息造成干擾,影響識別結果的可靠性與準確性,因此,應在初始階段去除可能產生的隨機因素。
已知矩陣D是一個初始的數據矩陣,由特征集合F={f1,f2,…,ft}與類集合S={s1,s2,…,sk}架構而成,其中,t、k分別表示特征個數與類別數量,高維隨機矩陣M的構建公式如下所示
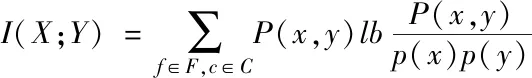
(5)
下列矩陣即為所得矩陣M的表達式

(6)
若上式中的k值較小,則無法理想地滿足隨機矩陣特征,所以,應在系數矩陣右邊增加一列,擴增[4]隨機矩陣M,復制m次后,得到下列表達式
M=[M,m(M)]
(7)
式中的m可通過下列計算公式完成求解,令初始的行列比值不發生改變

(8)
為確保該隨機矩陣M并非一種特例,可以代表大多數的普遍情況,采用下列式(9)與(10),規范化、中心化以及標準化隨機矩陣M,得到不失一般性的隨機矩陣Md
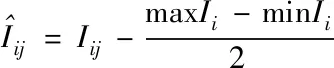
(9)
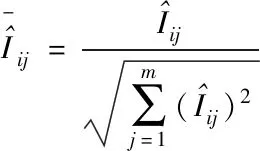
(10)
利用得到的隨機矩陣Md,推導出t×t的特征相關矩陣C表達式,如下所示

(11)
再通過下列奇異值分解[5]式,完成相關矩陣C的奇異值分解
C=UΛV
(12)
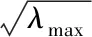
Cnew=UΛnewV
(13)
任意兩特征與初始類別的關聯程度,用Unew中含有的各項元素Kij來描述,各特征與新類別的關聯程度,用Vnew中含有的各項元素Eij來描述。通過對去噪的相關矩陣進行特征選擇,可去除冗余以及與類別不存在關聯性的特征。因為去噪后留存了j-1個奇異值,且選取特征數量與奇異值剩余個數相同,所以,特征選擇個數是j-1,各特征的重要程度計算公式如下所示

(14)
式中,第i個特征的重要程度為F(i),集合是F={f1,f2,…,fi},按照從大到小的順序降序排列F(i)后,得到重要程度最高的前j-1個特征,完成特征選取。
采用隨機矩陣進一步優化選擇的n個特征,各特征均是一個隨機變量,利用隨機特征矩陣與初始特征矩陣奇異值矢量的相關系數,描述特征與隨機變量的關聯度,相關系數越大,關聯度越高,所以,應留存較小的相關系數特征。
已知特征集合F={f1,f2,…,fn},類別數量為k個,計算初始特征矩陣M的奇異值過程中,以互信息[6]作為度量標準,采用式(5)和(6)架構互信息矩陣D,再依據式(9)~(12),逐步實施規范化、標準化、相關矩陣運算以及奇異值分解等操作,最終得到矩陣M的奇異值矢量e,該矢量的組成部分為n個奇異值,其中所含元素表示為εk;在求取隨機特征矩陣奇異值矢量階段,將隨機變量用各個特征表示,構建數量為n的隨機矩陣(M1,M2,…,Mn)。按照初始特征矩陣奇異值矢量計算流程,解得各隨機矩陣Mi(1≤i≤n)奇異值矢量ei,其中所含各元素用εik表示。關于各隨機矩陣奇異值矢量ei與初始特征奇異值矢量e的差di,可利用下列計算公式解得,通過取整差值di,并保留di≠0情況下的fi,實現特征選擇優化
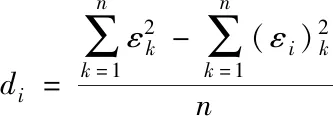
(15)
3 癌癥基因網絡識別
3.1 降噪點確定
利用隨機矩陣理論與下列皮爾森相關系數[7]公式,轉換癌癥基因微陣列數據為相關基因矩陣,使矩陣中含有全部基因之間的關聯程度:
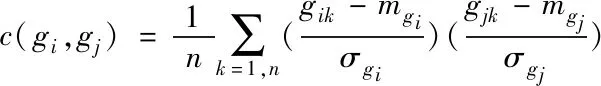
(16)
假設高維隨機矩陣理論的降噪參數是q,取值范圍為(0,1),將參數q值慢慢增大,去除較小的相關系數,求解對應于各降噪參數q的特征根NNSD(Nearest-neighbour Spacing Distribution,最近鄰間隔分布)。在不斷增大參數q值、去除相關矩陣的較小相關系數過程中,相關矩陣特征根最近鄰間隔分布形式由高斯正態分布過渡至泊松分布[8]。
通過標準誤差方法,可以準確、科學地確定癌癥基因特征根分布形式過渡至泊松體系的轉變點對應q值,所以,設定癌癥基因表達矩陣特征根的最近鄰間隔分布與高斯正態分布的標準誤差為SDGOE(q),與泊松分布的標準誤差為SDpoisson(q),利用下列兩項標準誤差界定公式,計算分布形式過渡的臨界點與降噪參數

(17)

(18)
式中,第i點對應特征根的最近鄰間隔分布是p(i),該點對應高斯正態分布與泊松分布特征根的最近鄰間隔分布分別用PGOE(i)、Ppoisson(i)表示。
如果兩個標準誤差值相同,特征根的最近鄰間隔分布體系元素相互效用較強,相關程度較大,相似性較高,多數為真實相關信息,極少數為隨機信息;如果標準誤差比值較大,分布體系則更趨近于泊松分布,偏離高斯正態分布,在留存有效信息的同時,充分濾除噪聲。因此,降噪點即為最大標準誤差比值的對應點。將降噪參數q從0逐漸增大至1,去除癌癥相關矩陣含有的隨機噪聲,才能得到真實的癌癥基因網絡。
3.2 癌癥基因網絡架構
依據癌癥基因初始數據與不同實驗條件的所有基因表達水平,采用cluster tree view軟件構建描述基因相互效用的層次樹形圖,該圖在聚合有相關性基因的同時,展現出層次結構的連接形式。基于明確的降噪點,選取出與其它基因相關系數不小于降噪參數q的基因,構建新的癌癥基因表達矩陣,并聚類分析經過噪聲濾除的癌癥基因。
假設新建癌癥基因表達矩陣的留存基因數量為361,且新矩陣的基因內涵蓋其中的所有信息,則該癌癥基因網絡樹形圖如圖1所示。
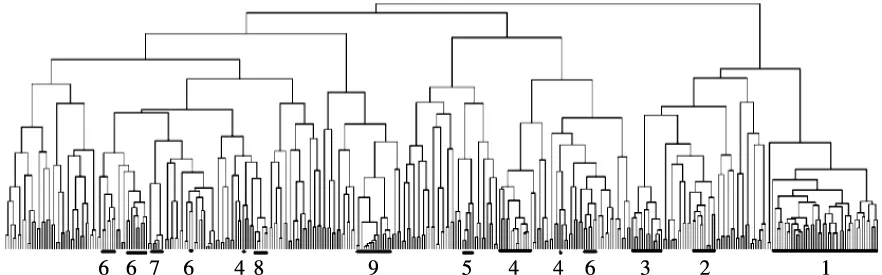
圖1 癌癥基因網絡樹形圖
樹形圖1中,基因之間的相關性用連線表示,相關程度的強弱用樹枝長度表示;連線與相關程度成反比。
4 實例分析
4.1 實驗數據采集
實驗環節以肝癌[9]為例,從http:∥ genome-www.stanford.edu hcc supplement.shtml.斯坦福微陣列數據庫中,挑選肝癌基因微陣列初始數據,得到的研究數據為基于82個HCC(Hepatocellular Carcinoma,肝細胞性肝癌)樣本1648個肝癌基因的微陣列數據,樣本基因信息如表1所示。

表1 肝癌基因數據統計表
4.2 降噪點獲取
從0到1逐漸增大高維隨機矩陣理論的降噪參數q,圖2所示為參數q取不同數值時,肝癌基因特征根的最近鄰間隔分布情況,圖中泊松分布用虛線表示,高斯正態分布用實線表示,最近鄰間隔分布用點線表示。
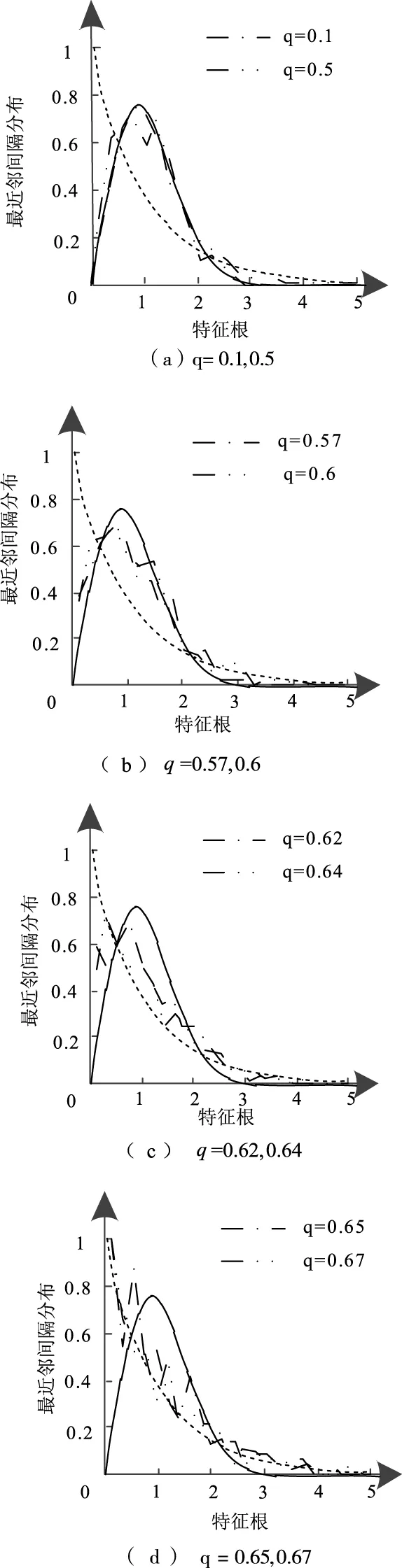
圖2 不同參數值的最近鄰間隔分布變化
通過圖2的曲線走勢可以看出,參數q在數值到達0.64后(見圖2(c)),基因特征根最近鄰間隔分布變化趨勢開始趨于泊松分布,經過標準誤差值運算,發現參數q在取值是0.67時(見圖2(d)),標準誤差比值為極大值,因此,1648×82肝癌微陣列數據的降噪點參數取值為0.67。
4.3 癌癥基因網絡識別結果
去噪后保留820個肝癌基因,并得到820×82的微陣列數據,利用Cluster 3.0軟件層次聚類肝癌基因,采用斯皮爾曼相關系數度量基因之間的相似性,通過cluster tree view軟件呈現的肝癌基因樹形圖,如圖3所示。

圖3 基因樹形圖
圖3的基因表達譜中,基因表達量上調用紅色表示,下調則用綠色表示。當基因被劃分為一類時,相同肝癌樣本的表達量上、下調一致。經分析得到的團簇分別是增殖簇、B淋巴細胞簇、細胞周期調控簇、基質細胞簇以及脂類酒精代謝簇,如圖4所示。
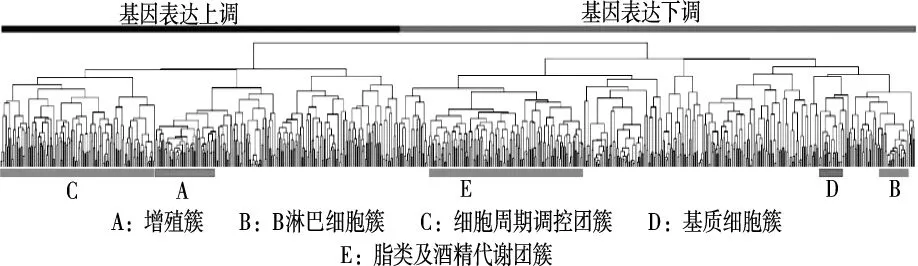
圖4 肝癌基因團簇分類示意圖
以關聯程度較強的B淋巴細胞簇為例,分析肝癌基因團簇,圖5所示為B淋巴細胞簇的基因樹形圖。從B淋巴細胞團簇中分別識別出B淋巴細胞的增長因子WNT4與編碼mRNA前剪接調控因子SLU7兩個基因。在團簇內層,兩基因緊密連通,該基因與B淋巴細胞免疫過程相關,說明淋巴細胞正浸潤肝組織。
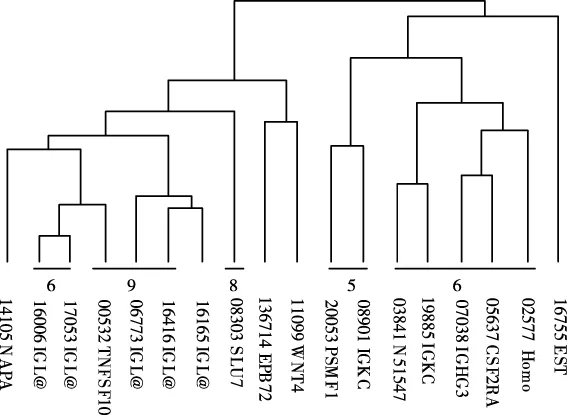
圖5 B淋巴細胞簇基因及其層次樹圖
為了驗證本文方法識別的有效性,采用基因網絡模塊劃分方法[10],得到圖6所示的肝癌基因處理結果。
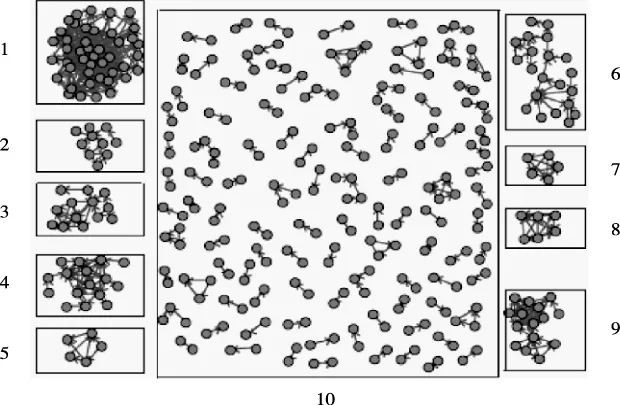
圖6 基因網絡模塊劃分方法下肝癌基因模塊圖
經過對比圖5和圖6可以看出,基因網絡模塊劃分方法識別出的肝癌基因中,只有1個基因在本文方法構建的樹形圖分支上與對應的主分支有所偏離,其它相同模塊的基因均與本文樹形圖所屬分支一致,說明本文方法能夠識別出基因的真實模塊,且相似度較高。
5 結論
癌癥作為一種復雜性疾病,對人類健康存在嚴重威脅,只有及時查出癌癥發生的相關基因,發現互相關聯,才能防止癌癥惡化,因此,本文針對癌癥基因的微陣列數據,以高維隨機矩陣為數據預處理策略,提出一種癌癥基因網絡識別方法,并制定出今后的研究探索方向:在過渡至泊松分布的過程中,最近鄰間隔分布體系仍有可能存在噪聲干擾,需設計出一個更加優化的降噪點判定方法,使噪聲能夠去除完全;為便于基因的后續調控,需量化模塊之間的連接大小與調控關系,并進一步研究基因之間相互調控的形式與力度,以及基因間相互影響程度。該方法對癌癥的生成檢測、惡化控制與治療,有著重要的現實意義。

