小麥TaARF20基因克隆及表達分析
郭曉鳳,王超杰,張莉莉,景豆豆,李政,都晶晶,馬守才
摘要:【目的】克隆TaARF20基因,并分析其表達特性,為解析小麥生長素響應因子(ARF)基因家族成員的調控機制提供理論依據。【方法】以普通小麥為材料,利用同源克隆技術獲得TaARF20基因的cDNA全長序列,利用生物信息學軟件分析其序列特征,并通過亞細胞定位試驗明確TaARF20蛋白的作用部位。將TaARF20基因與表達載體Pcold-TF連接,轉化大腸桿菌中進行原核表達。采用實時熒光定量PCR檢測TaARF20基因在普通小麥不同組織及普通小麥和多子房小麥不同發育時期幼穗中的表達模式。【結果】TaARF20基因包含1個內含子和2個外顯子,編碼區(CDS)長度為1116 bp,編碼371個氨基酸殘基,蛋白分子量約40.38 kD,理論等電點(pI)為4.96,脂溶性系數為19.80,疏水性系數為0.985,不穩定系數為50.51,屬于不穩定蛋白,定位在細胞核中,含有ARF家族蛋白的保守結構域和B3 DNA結合域,主要由α-螺旋(18.06%)、無規則卷曲(59.57%)和延伸連(22.10%)組成。啟動子區含有激素響應、光響應和低溫響應等多個順式作用元件。TaARF20蛋白與二粒小麥ARF20的氨基酸序列相似性最高,親緣關系也最近。TaARF20融合蛋白在原核系統中成功表達。TaARF20基因在小麥的根、葉、幼穗和籽粒中均有表達,但在幼穗中的相對表達量最高。對于長度為3和4 cm的幼穗來說,普通小麥TaARF20基因的相對表達量低于多子房小麥,但對于長度為5、6、8和9 cm的幼穗,普通小麥TaARF20基因的相對表達量高于多子房小麥。【結論】TaARF20基因在小麥穗生長發育中發揮重要調控作用,推測其是調控穗型、穗大小或穗粒數的關鍵基因。
關鍵詞: 小麥;TaARF20;基因克隆;亞細胞定位;原核表達;實時熒光定量PCR
中圖分類號: S512.103.53? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2021)09-2350-08
Cloning and expression analysis of TaARF20 gene in
wheat(Triticum aestivum L.)
GUO Xiao-feng1, WANG Chao-jie2, ZHANG Li-li1, JING Dou-dou1,
LI Zheng3, DU Jing-jing1, MA Shou-cai1*
(1College of Agronomy,Northwest A & F University/National Yangling Agricultural Biotechnology & Breeding Center/ Yangling Branch of State Wheat Improvement Center/Wheat Breeding Engineering Research Center,Ministry of Education/Key laboratory of Crop Hetersis of Shaanxi Province,Yangling, Shaanxi? 712100, China; 2Institute of Crop Science, Chinese Academy of Agricultural Sciences/Centre for Crop Genetics and Breeding/Wheat Mutation Breeding Centre, Beijing? 100081, China; 3College of Life Science,Henan University/State Key Laboratory of Crop
Adversity Adaptation and Improvement,Kaifeng, Henan? 475001, China)
Abstract:【Objective】To provide theoretical basis for analyzing the regulation mechanism of Auxin response factor(ARF) genes family in wheat by cloing and analyzing expression characteristic of TaARF20 gene. 【Method】The full-length cDNA sequence of TaARF20 gene was obtained from common wheat by homologous cloning technique, and its sequence characteristics were analyzed by bioinformatics. The active site of TaARF20 protein was determined by subcellular localization experiment. The TaARF20 gene was ligated with the expression vector Pcold-TF and transformed into Escherichia coli for prokaryotic expression. The expression patterns of TaARF20 gene in different tissues of common wheat and immature spike of common wheat and multi-ovary wheat at different development stages were analyzed by real-time fluorescence quantitative PCR. 【Result】Sequence analysis indicated that TaARF20 gene contained one intron and two exons, the coding region(CDS) was 1116 bp and encoded 371 amino acid residues. The molecular weight of TaARF20 protein was about 40.38 kD, theoretical isoelectric point (pI) was 4.96, fat solubility coefficient was 19.80, hydrophobicity coefficient was 0.985, and instability coefficient was 50.51 which indicated TaARF20 was an unstable protein. TaARF20 was located in the nucleus and contained ARF family domain and B3 DNA binding domain. Secondary structure analysis showed that the protein was mainly composed of alpha helix(18.06%), extended strand(59.57%), random coil(22.10%). The analysis of the promoter region revealed multiple cis-regulatory element responses, including hormonal responses, light and temperature responses. Alignment analysis of amino acid sequences and phylogenetic tree analysis showed that TaARF20 protein of common wheat had the highest amino acid sequence similarity and closest homology with Triticum dicoccoides ARF20. The result of SDS-PAGE showed that TaARF20 fusion protein was successfully expressed in prokar-yotic cells in vitro. Real-time fluorescence quantitative PCR analysis showed that TaARF20 gene was expressed differently in root, leaf, immature spike and seed of wheat, and the expression in immature spike was significantly higher than that in other tissues. The relative expression level of TaARF20 gene in common wheat was lower than that in multi-ovary wheat for immature spike of 3 and 4 cm, but higher than that in multi-ovary wheat for immature spike of 5, 6, 8 and 9 cm in length. 【Conclusion】TaARF20 gene plays an important regulatory role in immature spike of wheat, which is speculated that it may be a key gene regulating spike shape, spike size and grain number per spike.
Key words: wheat; TaARF20;gene cloning;subcellular localization;prokaryotic expression;real-time fluorescence quantitative PCR
Foundation item:Project of Seven Crops Breeding of National Key Research and Development Project(2018 YFD0100904); Shaanxi Key Research and Development Plan Project(2020NY-048);Post Subsidy Project of Shaanxi Provincial Key Laboratory(2018SZS-22)
0 引言
【研究意義】小麥(Triticum aestivum L.)是重要的糧食作物。由于近年來頻繁出現的氣候異常、土壤干旱和病蟲害等不利于小麥的正常生長發育,嚴重威脅我國糧食安全(陳文燁等,2020)。生長素響應因子(Auxin response factor,ARF)基因家族成員在植物根系形成、種子發育及適應逆境脅迫中起關鍵的調控作用(Kepinski and Leyser,2005;Sima and Zheng,2015;蘇麗艷,2019)。因此,克隆小麥TaARF20基因,并分析其表達特性,對深入解析ARF對小麥生長發育的調控作用機制具有重要意義。【前人研究進展】目前,對擬南芥ARF基因家族成員的研究較詳細,研究表明AtARFs基因參與植株生長發育活動的多個階段,且每個基因具有多種調控作用。如AtARF2、AtARF4和AtARF5基因在調節雌、雄配子體發育方面均發揮著重要作用,其下調表達或沉默會導致協同核缺陷和協同細胞活力的喪失(Liu et al.,2018),其中,AtARF2基因還調控種子發育,其突變后擬南芥種子粒大且重(Schruff et al.,2006),與油菜BnaARF18基因功能類似(Liu et al.,2015);AtARF10、AtARF16和AtARF17基因調控種子的休眠和萌發,其過表達能顯著延長種子休眠時間(Liu et al.,2013;唐桂英等,2020),其中AtARF17基因在初生外壁形成和花粉壁形成中也具有關鍵作用(Yang et al.,2013)。水稻ARF基因家族成員的相關研究也證明其參與調控植株生長發育和逆境抵御,如OsARF19基因過表達株系出現矮桿、窄葉和葉傾角增大等多個表型特點(張勝忠等,2017);OsARF12和OsARF16基因參與鐵和磷酸鹽的缺乏反應,維持磷酸鹽的體內穩態(Qi et al.,2011;Shen et al.,2013;Wang et al.,2014;Yu et al.,2015)。此外,番茄SlARF4基因過表達株系表現出對鹽脅迫和滲透脅迫較高的耐受性(Bouzroud et al.,2020)。【本研究切入點】目前有關小麥TaARF20基因克隆及其組織表達特性的研究鮮見報道。【擬解決的關鍵問題】利用基因同源克隆技術克隆小麥TaARF20基因序列,利用生物信息學軟件分析其序列和結構特征,并通過亞細胞定位試驗明確其在細胞中的作用部位,同時采用實時熒光定量PCR檢測TaARF20基因在小麥不同組織中的表達模式,為解析該基因在小麥生長發育中的調控機制提供理論參考。
1 材料與方法
1. 1 試驗材料
供試材料為普通小麥和多子房小麥,種植于西北農林科技大學楊凌試驗站。主要試劑:HiPure HP Plant RNA Mini Kit購自美基(上海)生物科技有限公司;HiScript 1st Strand cDNA Synthesis Kit反轉錄試劑盒購自諾唯贊(南京)生物科技有限公司;KOD-Plus-Neo試劑購自東洋紡(日本)生物科技有限公司;SGExcel FastSYBR Mixture(with ROX)試劑盒購自生工生物(西安)股份有限公司。主要設備儀器:ABI Life熒光定量PCR儀Q3(Thermo Fisher,美國)、T100TMThermal Cycler基因擴增儀(Bio-Rad,美國)、5810R高速冷凍離心機(Eppendorf,德國)、激光掃描共聚焦顯微鏡IX83-FV1200(Olympus,日本)、凝膠電泳儀(Bio-Rad,美國)等。
1. 2 試驗方法
1. 2. 1 總RNA提取及反轉錄合成cDNA 利用HiPure HP Plant RNA Mini Kit提取普通小麥葉片、幼穗、籽粒和根的總RNA及多子房小麥幼穗的總RNA,然后參照HiScript 1st Strand cDNA Synthesis Kit反轉錄合成第一鏈cDNA,存于-20 ℃保存備用。
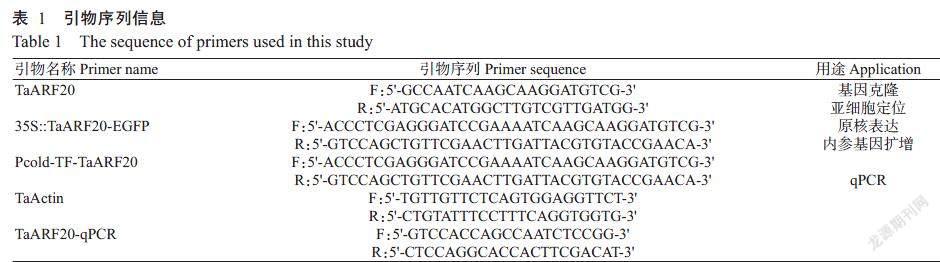
1. 2. 2 基因克隆及序列分析 參考Ensemble Plants數據庫(https://plants.ensembl.org/index.html)中的中國春小麥的TaARF20基因序列(TraesCS7A02G4757 00.1),利用Oligo 7.0設計克隆引物TaARF20-F/TaARF20-R(表1)。以普通小麥cDNA為模板,利用KOD-Plus-Neo試劑進行PCR擴增。反應體系50.0 ?L:10×KOD Neo Buffer 5.0 ?L,25 mmoL MgSO4 3.0 ?L,2 mmoL dNTPs 2.0 ?L,10 ?moL/L正、反向引物各1.5 ?L,100 ng/?L cDNA 2.0 ?L,1.0 U/?L KOD-Plus-Neo 1.0 ?L,ddH2O補足至50.0 ?L。擴增程序:94 ℃預變性5 min;98 ℃ 30 s,64 ℃ 30 s,68 ℃ 1 min,共進行35個循環;68 ℃延伸10 min。PCR產物經1%瓊脂糖凝膠電泳檢測并回收純化目的條帶,連接至T載體(Topsmart PBM16A),從而獲得重組質粒T-PBM16A-TaARF20,轉化大腸桿菌DH5α感受態細胞中并涂板培養。挑取單菌落,利用通用引物M13-F/M13-R進行菌液PCR鑒定后,將陽性克隆送生工生物(西安)股份有限公司測序。
利用DNAMAN 6.0將測序結果與參考基因序列進行比對分析,并用IBS(http://ibs.biocuckoo.org/)繪制基因結構。通過NCBI的CDD在線網站(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析該基因的保守結構域(Aron et al.,2017)。
1. 2. 3 生物信息學分析 利用ExPASy ProtParam(http://web.expasy.org/protparam/)分析TaARF20蛋白的理化性質(Gasteiger et al.,2005);采用NPS@ server(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_hnn.html)分析TaARF20蛋白的二級結構(Combet et al.,2000),以SubLocv 1.0(http://www.bioinfo.tsinghua.edu.cu/SobLoc/)預測TaARF20蛋白的亞細胞定位,利用PlantCare在線網站分析啟動子區的順式作用元件及生理功能。從Ensemble Plants數據庫中查找下載TaARF20蛋白的不同物種同源序列,并利用MEGA 7.0構建系統發育進化樹。
1. 2. 4 亞細胞定位 以測序正確的T-PBM16A-TaARF20質粒為模板,使用引物35S::TaARF20-EGFP-F/35S::TaARF20-EGFP-R(表1)擴增TaARF20基因。反應體系20.0 ?L:10×KOD Neo Buffer 5.0 ?L,2 mmoL/L dNTPs 2.0 ?L,25 mmoL/L MgSO4 3.0 ?L,10 ?moL/L上、下游引物各1.5 ?L,100 ng/?L DNA模板2.0 ?L,1.0 U/?L KOD-Plus-Neo 1.0 ?L,ddH2O補足至20.0 ?L。擴增程序:94 ℃預變性5 min;98 ℃ 30 s,64 ℃ 30 s,68 ℃ 1 min,進行35個循環;68 ℃延伸10 min。PCR產物經1%的瓊脂糖凝膠檢測并切膠回收目的片段,與1302EGFP載體連接,以構建35S::TaARF20-EGFP亞細胞定位載體,送至生工生物(西安)股份有限公司測序。將測序正確的載體轉化農桿菌GV3101,挑取單克隆于10 mL的液體LB培養基中搖菌,然后收集5 mL農桿菌菌液,用注射緩沖液(10 mmol/L MES? pH 5.6,10 mmol/L MgCl2,100 μmol/L AS)稀釋至OD600=0.6~0.8,以不攜帶目的基因的1302EGFP空載體為對照。注射后的煙草避光培養1 d,正常條件培養1 d,最后使用激光共聚焦掃描顯微鏡采集葉片細胞中的熒光信號。
1. 2. 5 原核表達載體構建及誘導表達 利用引物Pcold-TF-TaARF20-F/Pcold-TF-TaARF20-R PCR擴增TaARF20基因,回收純化目的片段,將其連接表達載體Pcold-TF上獲得重組質粒Pcold-TF-TaARF20。將空載體Pcold-TF和重組質粒Pcold-TF-TaARF20分別轉化大腸桿菌BL21(DE3)感受態細胞,接種于含有氨芐青霉素的液體LB培養基中,37 ℃下過夜培養。次日將菌液按1∶50擴大培養至OD600=0.6~0.8,取1 mL菌液作為對照,其余菌液中加入IPTG誘導表達16 h,通過SDS-PAGE凝膠電泳檢測蛋白表達情況。
1. 2. 6 實時熒光定量PCR檢測 采用SGExcel FastSYBR Mixture(with ROX)試劑盒,利用實時熒光定量PCR檢測TaARF20基因在小麥不同組織及幼穗不同發育時期的表達情況,所用引物見表1。反應體系20.0 μL:2×SGExcel FastSYBR Mixture(with ROX)10.0 μL,10 μmoL/L正、反向引物各0.4 μL,100 ng/μL cDNA 2.0 μL,ddH2O補足至20.0 μL。擴增程序:95 ℃預變性3 min,95 ℃ 5 s,60 ℃ 20 s,95 ℃ 15 s,60 ℃ 1 min,95 ℃ 1 s,共進行40個循環。溶解曲線為儀器默認設置。設3個生物學重復。按照2-ΔΔCt法計算目的基因在小麥不同組織及幼穗不同發育時期的相對表達量。
1. 3 統計分析
利用Excel 2017統計分析TaARF20基因的相對表達量,利用SPSS 22.0進行顯著性分析(One-way ANOVA)。
2 結果與分析
2. 1 TaARF20基因克隆及序列分析結果
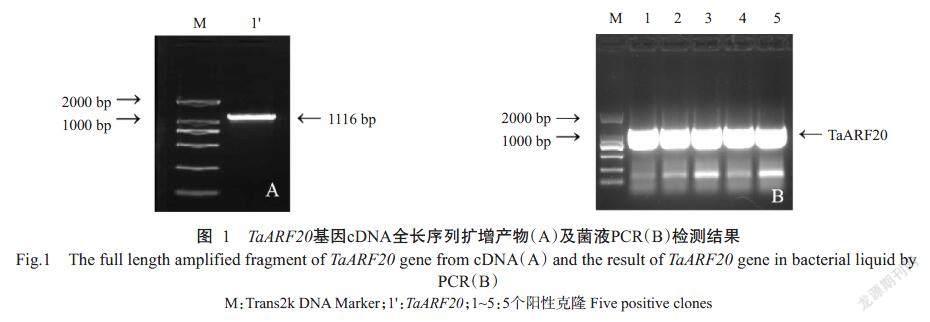
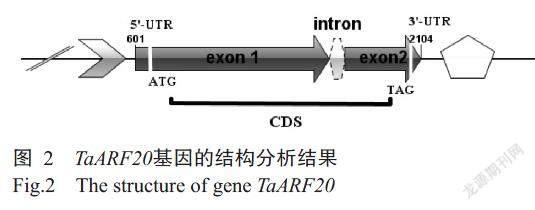
利用引物TaARF20-20F/TaARF20-20R對幼穗cDNA模板進行PCR擴增,結果(圖1-A)顯示,擴增條帶大小約1200 bp,與預期結果相符。菌液PCR檢測結果(圖1-B)顯示,重組質粒T-PBM16A-TaARF20已轉化大腸桿菌DH5α。由圖2可知,TaARF20基因包括1個內含子和2個外顯子,編碼區(CDS)長度為1116 bp,編碼371個氨基酸殘基。
2. 2 生物信息學分析結果
利用ExPASy ProtParam預測TaARF20蛋白的理化性質,結果顯示該蛋白的分子量為40.38 kD,理論等電點(pI)為4.96,脂溶性系數為19.80,疏水性系數為0.985,不穩定系數為50.51,屬于不穩定蛋白。NCBI的CDD分析結果顯示,TaARF20蛋白含有ARF家族蛋白的保守結構域和B3 DNA結合結構域。二級結構預測結果顯示,該蛋白主要由α-螺旋(18.06%)、無規則卷曲(59.57%)和延伸鏈(22.10%),其余為模糊狀態(0.27%)。亞細胞定位預測結果顯示,該蛋白定位在細胞核。
利用PlantCare分析TaARF20基因的啟動子序列,結果(表2)顯示,啟動子區含有許多不同類型的順式作用元件,主要涉及到激素響應、光響應和低溫響應等,其中激素響應元件包括脫落酸響應元件C-ABRE(ACGTG)、ABRE3a(TACGTG)和ABRE4(CACGTA)、茉莉酸甲酯響應元件CGTCA-motif(CGTCA)和TGACG-motif(TGACG)及赤霉素響應元件P-box(CCTTTTG)等;光響應元件有H-box(TACGTG)、GATA-motif(GATAGGG)和TCCC-motif(TCTCCCT);低溫響應元件有LTR(CCGAAA)。上述結果表明,TaARF20基因除了響應生長素外,還參與多種分子調控途徑從而影響植物的生長發育,主要包括激素代謝、脅迫應答和光溫反應等。
從Ensemble Plants數據庫中查找下載TaARF20蛋白的同源序列,與擬南芥、油菜等其他物種同源序列進行氨基酸序列比對(圖3),并構建系統發育進化樹(圖4),結果發現普通小麥TaARF20蛋白與二粒小麥ARF20蛋白的氨基酸序列相似性最高,與玉米、高粱、黍的相似性較低,表明普通小麥與二粒小麥親緣關系最近。
2. 3 TaARF20蛋白的亞細胞定位結果
利用農桿菌介導法將35S::TaARF20-EGFP載體注射入煙草葉片表皮細胞,在激光共聚焦顯微鏡下觀察綠色熒光信號在細胞中位置,結果顯示,TaARF20蛋白定位于細胞核(圖5),與上述預測結果一致,符合轉錄因子的基本特性。
2. 4 TaARF20基因的原核表達結果
將測序正確的重組質粒Pcold-TF-TaARF20轉化大腸桿菌BL21(DE3)感受態細胞,加入IPTG進行誘導表達,并用12% SDS-PAGE進行檢測,結果(圖6)發現,表達產物約60 kD,除去標簽蛋白后,目的蛋白TaARF20的分子量為40.38 kD,與預期結果相符。
2. 5 TaARF20基因的表達模式分析結果
采用實時熒光定量PCR檢測TaARF20基因在普通小麥幼穗、葉片、籽粒和根中的表達情況,結果(圖7)顯示,該基因在4種組織中均有表達,但在幼穗中的相對表達量最高,其次是葉片,籽粒中的相對表達量最低,表明TaARF20基因具有明顯的組織特異性。
為進一步探究TaARF20基因在普通小麥和多子房小麥幼穗中的表達差異,采集兩者不同發育時期的幼穗(幼穗長度為3、4、5、6、8和9 cm),采用實時熒光定量PCR檢測其TaARF20基因的表達情況,結果(圖8)顯示,對于3和4 cm的幼穗來說,普通小麥TaARF20基因的相對表達量低于多子房小麥,說明TaARF20基因可能在普通小麥幼穗發育早期發揮重要的調控作用;但對于5、6、8和9 cm的幼穗,普通小麥TaARF20基因的相對表達量高于多子房小麥,說明TaARF20基因可能在多子房小麥幼穗發育晚期發揮重要的調控作用。
3 討論
前人研究結果顯示,不同物種中的ARF20蛋白質氨基酸序列中均含有高度保守的ARF家族保守結構域和B3 DNA結合結構域(Ulmasov et al.,1999;Guilfoyle and Hagen,2007;Zhou et al.,2018)。本研究從普通小麥中克隆獲得TaARF20基因,其含有1個內含子和2個外顯子,CDS長度為1116 bp,編碼的蛋白分子量為40.38 kD,含有ARF家族保守結構域和B3 DNA結合結構域,與前人研究結果一致。轉錄因子主要在細胞核行使其功能,但目前缺乏ARF20分布于細胞核中的證據。本研究通過亞細胞定位試驗發現,TaARF20蛋白定位于細胞核。系統發育進化樹分析結果顯示,TaARF20蛋白與二粒小麥ARF蛋白的親緣關系最近,表明二者可能來源于同一個祖先進化而來,且TaARF20蛋白與大麥、白菜型油菜和擬南芥等物種ARF蛋白具有較高的氨基酸相似性,暗示ARF20在植物中具有相同的生理功能。目前已有研究證實,ARF蛋白通過在生長素和其他激素之間進行信號傳遞,從而調控植物的生長發育(Bishopp et al.,2011;Liu et al.,2011)。本研究對TaARF20基因啟動子區順式元件的分析結果表明,TaARF20基因不僅涉及到生長素響應,還可能參與脫落酸、赤霉素和茉莉酸甲酯信號途徑,因此,推測TaARF20蛋白通過脫落酸、赤霉素和茉莉酸甲酯間的信號傳遞從而調控小麥組織生長發育。但目前未見有關ARF20基因啟動子區含有響應光照、溫度的響應元件或其表達受光照、溫度誘導的相關報道,但本研究發現小麥TaARF20啟動子含有光照和溫度響應的順式作用元件,推測TaARF20基因對小麥生長發育的調控方式與其他物種存在差異。
多數ARFs基因表達模式具有一定的組織特異性(倪君,2011),如谷子中SiARF10基因在穗中的表達量顯著高于根、莖和葉(趙艷等,2016)。本研究發現,TaARF20基因具有明顯的組織特異性,其在小麥幼穗中的表達量明顯高于根、葉片和種子,表明TaARF20基因在小麥穗生長發育過程中發揮重要調控作用。但TaARF20基因的調控作用機制還有待進一步研究,特別是其是否調控穗型、穗大小或穗粒數。因此,今后應通過揭示不同品種中TaARF20基因單倍型與穗發育的關系,并深入研究小麥幼穗發育基因的轉錄水平,以期解析TaARF20基因在小麥穗型、穗大小或穗粒數形成和發育中的調控機制。
4 結論
TaARF20基因在小麥穗生長發育中發揮重要調控作用,推測其是調控穗型、穗大小或穗粒數的關鍵基因。
參考文獻:
陳文燁,楊帆,劉永偉,董福雙,趙和,柴建芳,呂孟雨,周碩. 2020. 小麥TaNAC-B072基因的克隆和表達分析[J]. 江西農業學報,32(8):1-7. [Chen W Y,Yang F,Liu Y W,Dong F S,Zhao H,Chai J F,Lü M Y,Zhou S. 2020. Cloning and expression analysis of TaNAC-B072 in wheat[J]. Acta Agriculturae Jiangxi,32(8):1-7.] doi:10.19386/j.cnki.jxnyxb.2020.08.01.
倪君. 2011. OsIAA23介導的生長素信號胚后維持水稻根靜止中心[D]. 杭州:浙江大學. [Ni J. 2011. OsIAA23-media-ted auxin signaling defines postembryonic maintenance of QC in primary roots in rice[D]. Hangzhou:Zhejiang University.]
蘇麗艷. 2019. 草莓FvARF5基因的克隆、生物信息學及表達分析[J]. 華北農學報,34(3):16-22. [Su L Y. 2019. Clo-ning,bioinformatics and expression analysis of FvARF5 gene in strawberry[J]. Acata Agriculturae Boreali-Sinica,34(3):16-22.] doi:10.7668/hbnxb.201751324.
唐桂英,彭振英,徐平麗,李鵬祥,朱潔瓊,單雷,萬書波. 2020. 花生AhARF基因家族鑒定與表達分析[J]. 中國油料作物學報,184(6):178-188. [Tang G Y,Peng Z Y,Xu P L,Li P Y,Zhu J Q,Shan L,Wan S B. 2020. Genome-wide identification and expression analysis of auxin response factor gene family in Arachis hypogaea L.[J]. Chinese Journal of Oil Crop Sciences,184(6):178-188.] doi:10.19802/j.issn.1007-9084.2020213.
張勝忠. 2017. 水稻花器官發育基因OsARF19和粒型基因DSS的分子鑒定和功能分析[D]. 南京:南京農業大學. [Zhang S Z. 2017. Identification and functional analysis of OsARF19 gene for floral organ development and DSS gene for grain sizein rice(Oryza sativa L.)[D]. Nanjing:Nanjing Agricultural University.]
趙艷,甕巧云,馬海蓮,宋晉輝,袁進成,王凌云,董志平,劉穎慧. 2016. 谷子ARF基因家族的鑒定與生物信息學分析[J]. 植物遺傳資源學報,17(3):547-554. [Zhao Y,Weng Q Y,Ma H L,Song J H,Yuan J C,Wang L Y,Dong Z P,Liu Y H. 2016. Genome-wide identification and bioinformatics analysis of ARF gene family in Setaria italica[J]. Journal of Plant Genetic Resources,17(3):547-554.] doi:10.13430/j.cnki.jpgr.2016.03.022.
Aron M B,Yu B,Han L Y,Jane H,Christopher J L,Lu S N,Farideh C,Myra K D,Renata C G,Noreen R G,Marc G,David I H,Lu F,Gabriele H M,James M S,Narmada T,Wang Z X,Roxanne A Y,Zhang D C,Zheng C J,Lewis Y G,Stephen H B. 2017. CDD/SPARCLE:Functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Research,45(1):200-203. doi:10.1093/nar/gkw1129.
Bishopp A,Help H,El-Showk S,Weijers D,Scheres B,Friml J,Benková E,M?h?nen A P,Helariutta Y. 2011. A mutua-lly inhibitory interaction between auxin and cytokinin specifies vascular pattern in roots[J]. Current Biology,21(11):917-926. doi:10.1016/j.cub.2011.04.017.
Bouzroud S,Gasparini K,Hu G,Barbosa M A M,Rosa B L,Fahr M,Bendaou N,Bouzayen M,Zs?g?n A,Smouni A,Zouine M. 2020. Down regulation and loss of Auxin Response Factor 4 function using CRISPR/Cas9 alters plant growth,stomatal function and improves tomato tolerance to salinity and osmotic stress[J]. Gene,11(3):272. doi:10.3390/genes11030272.
Combet C,Blanchet C,Geourjon C,Deléage G. 2000. NPS@:Network protein sequence analysis[J]. 25(3):147-150.
Gasteiger E,Hoogland C,Gattiker A,Duvaud S,Wilkins M,Appel R,Bairoch A. 2005. Protein identification and analysis tools on the ExPASy server[J]. The Proteomics Protocols Handbook,571-607.
Guilfoyle T J,Hagen G. 2007. Auxin response factors[J]. Current Opinion in Plant Biology,10(5):453-460. doi:10.1016/ j.pbi.2007.08.014.
Kepinski S,Leyser O. 2005. The Arabidopsis F-box protein TIR1 is an auxin receptor[J]. Nature,435(7041):446-451. doi:10.1038/nature03542.
Liu J,Hua W,Hu Z Y,Yang H L,Zhang L,Li R J,Deng L B,Sun X C,Wang X F,Wang H Z. 2015. Natural variation in ARF18 gene simultaneously affects seed weight and silique length in polyploid rapeseed[J]. Proceedings of the National Academy of Sciences of the United States of America,112(37):5123-5132. doi:10.1073/pnas. 1502160112.
Liu X D, Zhan H, Zhao Y, Feng Z,Li Q,Yang H Q,Luan S,Li J,He Z H. 2013. Auxin controls seed dormancy through stimulation of abscisic acid signaling by indu-cing ARF-mediated ABI3 activation in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America,110(38):15485-15490. doi:10. 1073/pnas.1304651110.
Liu Z H,Yu Y C,Xiang F N. 2011. Auxin response factors and plant growth and development[J]. Hereditas,33(12):1335-1346. doi:10.3724/SP.J.1005.2011.01335.
Liu Z,Miao L,Huo R,Johnson C,Kong L,Sundaresan V,Yu X. 2018. ARF2-ARF4 and ARF5 are essential for female and male gametophyte development in Arabidopsis[J].Plant & Cell Physiology,59(1):179-189.
Qi Y H,Wang S K,Shen C J,Zhang S N,Chen Y,Xu Y X,Liu Y,Wu Y R,Jiang D. 2011. OsARF12,a transcription activator on auxin response gene,regulates root elongation and affects iron accumulation in rice(Oryza sativa L.)[J]. New Phytologist,193(1):109-120. doi:10.1111/J.1469-8137.2011.03910.X.
Schruff M C,Spielman M,Tiwari S,Adams S,Fenby N,Scott R J. 2006. The AUXIN RESPONSE FACTOR 2 gene of Arabidopsis links auxin signalling,cell division,and the size of seeds and other organs[J]. Development,133(2):251-261.
Shen C,Wang S,Zhang S,Xu Y X,Qian Q,QI Y H,Jiang D. 2013. OsARF16,a transcription factor,is required for auxin and phosphate starvation response in rice(Oryza sativa L.)[J]. Plant Cell & Environment,36(3):607-620. doi:10.1111/pce.12001.
Sima X J,Zheng B. 2015. Advances in primary auxin-responsive Aux/IAA gene family:A review[J]. Journal of Zhejiang A & F University,32(2):313-318.
Ulmasov T,Hagen G,Guilfoyle T J. 1999. Dimerization and DNA binding of auxin response factors[J]. Plant Journal,19(3):309-319. doi:10.1046/j.1365-313X.1999.00538.x.
Wang S K,Zhang S,Sun C D,Xu Y X,Chen Y,Yu C L,Qian Q,Jiang D,Qi Y H. 2014. Auxin response factor (OsARF12),a novel regulator for phosphate homeostasis in rice(Oryza sativa L.)[J].The New Phytologist,201(1):91-103. doi:10.1111/NPH.12499.
Yang J,Lei T,Sun M X,Huang X Y,Zhu J,Guan Y F,Yang J Z N. 2013. AUXIN RESPONSE FACTOR 17 is essential for Pollen Wall Pattern Formation in Arabidopsis[J]. Plant Physiology,162:720-731.
Yu C L,Sun C D,Shen C,Wang S,Liu F,Liu Y,Chen Y L,Li C,Qian Q,Aryal B. 2015. The auxin transporter,OsAUX1,is involved in primary root and root hair elongation and in Cd stress responses in rice(Oryza sativa L.)[J]. The Plant Journal,83(5):818-830. doi:10.1111/tpj.12929.
Zhou X J,Wu X Z,Li T J,Jia M L,Liu X S,Zou Y L,Liu Z X,Feng W. 2018. Identification,characterization,and expression analysis of auxin response factor(ARF) gene family in Brachypodium distachyon[J]. Functional and Integrative Genomics,18(6):709-724. doi:10.1007/s10142-018-0622-z.
(責任編輯 陳 燕)

