仿刺參疣足數量SNP 遺傳力評估*
倪 萍 任 強 王 靜 丁 君 常亞青王揚帆① 胡景杰 包振民
(1. 中國海洋大學 海洋生物遺傳學與育種教育部重點實驗室 青島 266003;2. 大連海洋大學 農業農村部北方海水增養殖重點實驗室 大連 116023)
仿刺參(Apostichopus j aponicus)是中國傳統的海珍品之一,營養價值高,藥用價值廣泛(常亞青等,2004)。海參養殖業在我國經過十幾年的迅猛發展,已成為海水養殖的主要產業之一(王秀利等, 2006)。然而,由于刺參養殖的過速發展和不規范運作,基礎研究滯后于刺參養殖產業的發展,加之基礎設施、養殖工藝和方式的滯后,出現刺參病害頻發、種質資源匱乏、成品參品質下降、單位面積產量和產值下降等問題(李成林等, 2010; 張春云等, 2004; 常亞青等,2006; 趙帥等, 2016);野生資源過度捕撈和養殖刺參長期近親繁殖、累代養殖,也使刺參品種質量逐步下降(封巖, 2019),刺參的養殖業面臨嚴重威脅。隨著人們對仿刺參需求量的增加,迫切需要對其進行遺傳改良,選育具有優良性狀的新品種。遺傳參數的估計是水產動物選擇育種的一項基礎工作,其中,遺傳力是衡量育種進展和育種方法的關鍵性指標(Falconer et al, 2000)。
遺傳力反映親屬間的相似程度和對人工與自然選擇的反應速度,往往是育種工作的第一步。傳統方法利用親緣關系較近的個體表型間的相關系數及親緣系數計算遺傳力(Lstib?rek et al, 2018; 文超良等,2019),在仿刺參遺傳力估計中被廣泛應用,例如,對仿刺參前期浮游階段耳狀幼體體長(欒生等, 2006)、耳狀幼體和稚參體長(李云峰等, 2009)、幼參4 個生長性狀(孟思遠等, 2010)的遺傳力估計等。傳統方法利用系譜信息構建親緣相關矩陣(Numerator relationship matrix, A 陣),并利用線性混合模型估計遺傳力,對系譜信息的準確性和完整性要求較高(李晶等,2020);由于孟德爾抽樣誤差(Hill, 2014),基于系譜推斷的個體間親緣關系準確性有限,并且存在系譜記錄錯誤、部分群體系譜信息不完整或缺失的情況,使得傳統法估計遺傳力的準確性存在一定的局限。
隨著測序技術的發展,越來越多的學者利用單核苷酸多態性(SNP)分子標記推斷個體間的親緣關系(Thompson, 1975; Lynch, 1988; Queller, 1989),通過構建基因組親緣關系矩陣(Genomic relationship matrix,GRM)(Li et al, 2014; 文超良等, 2019),進行基于SNP分子標記的遺傳力估計(SNP 遺傳力)(Yang et al ,2017),并在動植物以及人類群體中得到廣泛應用(Ritland et al, 1996; Mousseau et al, 1998; Thomas et al, 2002; Visscher et al, 2006)。例如,Yang 等(2010)利用REML 分析方法估計了SNP 數據集解釋的人類身高表型的變異程度;Guo 等(2018)利用GCTA 的REML 估計了櫛孔扇貝(Chlamys fa rreri)的生長性狀(扇貝長、寬、高和濕重)的SNP 遺傳力;Fishback 等(2002)使用 REML 方法估計了虹鱒(Oncorhynchus mykiss)總長和重量的遺傳力。Benjamin 等(2012)將這種基于GRM 的REML 分析方法稱為GREML 法。GREML 法可以利用SNP 數據估計無親緣關系的個體之間的遺傳關系,然后,由全基因組SNP 解釋的表型變異比例推斷遺傳力。與傳統方法相比,REML 分析法適用于非均衡資料的方差組分估計,可大大提高方差組分的估計準確性。本研究利用已完成測序的仿刺參基因組(Li et al, 2018),對不同養殖地理位置仿刺參群體進行全基因組重測序,并針對仿刺參重要經濟性狀疣足數量,進行全基因組水平的SNP 遺傳力估計,為豐富仿刺參生長相關的分子育種理論、加快仿刺參優良品種的選育提供一定的理論依據。
1 材料與方法
1.1 仿刺參樣本與表型
本研究215 個仿刺參樣本分別來自于8 個不同的養殖地理位置:平島、西小磨、山東棲霞口、蚆蛸島、中俄雜交仿刺參、旅順、黃龍尾和俄羅斯海參崴。首先,對所有仿刺參樣本的疣足數量進行統計(表1)。

表1 仿刺參樣本表型統計Tab.1 Statistics of papillae number for A. japonicus
1.2 全基因組重測序與參考SNP 選擇
按照動物組織基因組DNA 提取試劑盒(天根,北京)說明書提取仿刺參基因組DNA。使用NanoDrop-2000 超微量紫外分光光度計進行定量分析。
檢驗合格的DNA 片段經末端修復、加polyA 尾、加測序接頭、純化和PCR 擴增完成整個文庫制備。庫檢合格的文庫通過Illumina Hiseq Xten 的PE150bp模式進行測序,每個樣品測序深度為10×。對測序獲得的測序數據進行質量過濾,使用 Cutadapt 軟件(Martin, 2011)去除接頭序列,使用SolexaQA 軟件(Cox et al, 2010)去除質量值低于20 的堿基,將得到的高質量測序數據利用BWA 軟件(Li et al, 2009)比對到仿刺參參考基因組。使用Samtools(Li et al, 2009)進行去重復,GATK(Ye et al, 2009)進行局部重比對,堿基質量值校正等處理以及 SNP 小片段插入缺失(Small INDEL)的檢測,按照條件“QD<2.0||MQ<40.0||FS>60.0||MQRankSum<-12.5||ReadPosRankSum<-8.0”對SNP 進行過濾,得到最終的SNP 位點集。
篩選SNP 是為了在重測序獲得的高密度SNP 中減少或刪除處于高度連鎖不平衡的SNP,特別是在有限群體中有相當數量的SNP 之間存在高度的連鎖不平衡關系,會降低對動物個體遺傳參數的估計準確度。本研究采用 SNP 基因頻率的平均歐式距離(Average Euclidean distance, AED) (Wu et al, 2016)來篩選SNPs,挑選出次要等位基因頻率(Minor allele frequency, MAF)大于0.05 和0.1 的50K SNP,在篩選出來的 50K SNP 中隨機均勻挑選不同密度的SNP(SNP 數量分別為5K、10K、15K、20K、25K、30K、35K、40K、45K 和50K)。
1.3 基于SNP 的遺傳力估計
本研究使用 GCTA(Yang et al , 2011)(gcta_1.93.2beta 版 本) 的 GREML(Genomic relatedness matrix restricted maximum likelihood)中的期望最大約束似然法(EM-REML)、平均信息約束似然法(AI-REML)對仿刺參疣足數量的SNP 遺傳力進行評估。EM 算法是由Dempster 等(1977)提出的一種迭代算法,可針對有缺失數據的數據資料,獲得參數的最大似然估計值。缺失數據在實際的統計分析中非常普遍,EM 算法的基本思想是用缺失數據在給定參數的某個估計值的條件下的期望代替缺失數據,從而將不完全數據變成完全數據,使得似然函數的最大化變得相對簡單。REML 的 AI 算法(平均信息算法)由Gilmour 等(1995)、Johnson 等(1995)和Jensen 等(1997)提出,它是將求似然函數最大值的 2 種常見的Newton-Raphson 算法和Fisher's scoring 方法結合起來的一種算法(張勤, 2007)。
GCTA 軟件的核心即如下所示線性混合模型(Yang et al, 2011):
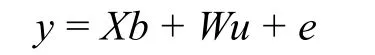
式中,y 表示表型,b 表示固定效應協變量的系數,u 表示隨機效應自變量的系數,這里的隨機效應指的是所有SNP 位點對表型的效應,e 表示隨機誤差。

其中,u 和e 服從如下正態分布:I為一個n × n 的單位矩陣,n 表示樣本量。
表型方差用如下公式表示:

W 為第ij 元素的標準化基因型矩陣,xij為第j 個個體的第i 個SNP 的參考等位基因的拷貝數,pi為參考等位基因頻率。
GCTA 對于樣本遺傳相似度的定義公式:
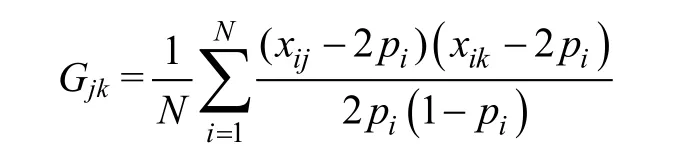
定義所有SNP 位點的方差:

式中,N 為SNP 位點數,G 為個體間的親緣關系矩陣(GRM)。
定義SNP 遺傳力的計算公式:
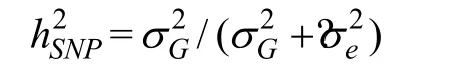
對來自不同養殖地理位置的215 個仿刺參樣本進行高通量重測序,獲得一組高質量SNP。構建GRM矩陣后,使用 GCTA 的 2 種遺傳參數估計方法(AI-REML 和EM-REML),添加協變量的校正,針對仿刺參疣足數量,分別計算不同密度SNP 下的SNP遺傳力估計值及染色體水平的SNP 遺傳力估計值。
2 結果與分析
2.1 仿刺參疣足數量的表型數據分布
從215 個仿刺參樣本疣足數量的頻率直方圖和概率密度曲線(圖1a)可以看出,本研究不同地域的仿刺參疣足數量大致符合正態分布,表明仿刺參疣足數量性狀不受地域環境的影響。考慮到圖1a 中出現大量疣足數量大于60 的個體,且主要集中在中俄雜交仿刺參和俄羅斯海參崴群體中,因此,利用R MASS包中nlminb 函數,對215 個樣本數據進行混合正態分布參數的最大似然估計(圖1b),結果顯示,樣本疣足數量符合2 個正態分布(均值39.8、方差6.79 和均值54.35、方差10.9)的最優擬合,說明樣本中存在多疣足仿刺參群體情況。進一步把8 個不同地域的仿刺參群體分為正常群體(平島、西小磨、山東棲霞口、蚆蛸島、旅順和黃龍尾)與多疣足群體(中俄雜交仿刺參和俄羅斯海參崴),并利用R t.test 函數對上述2 個群體疣足數量進行t 檢驗,結果差異顯著(P<0.01)。根據上述表型數據分布結果,本研究將不同地域的仿刺參群體分為正常群體與多疣足群體,并在SNP 遺傳力估計模型設計群體固定效應,以校群體分層對SNP 遺傳力估計的影響。

圖1 215 份仿刺參樣本疣足數量的頻率直方圖和概率密度曲線(a)和雙重高斯分布最優擬合(b)Fig.1 True frequency histogram and probability density curve (a) and optimal fitting of a double Gaussian distribution (b) of papillae number for the 215 A. japonicus samples
2.2 仿刺參疣足數量SNP 遺傳力估計
本研究全基因組重測序質量較高,GC 分布正常,對基因組的覆蓋度均超過90%,高質量SNPs 數量約為500 萬個。使用GCTA 的2 種REML 分析方法(EM-REML 和AI-REML)對仿刺參疣足數量的遺傳力進行評估,比較相同密度、相同次要等位基因頻率篩選條件下,不同分析方法得到的仿刺參疣足數量SNP遺傳力估計值。發現通過2 種方法得到的遺傳力估計值差異極小(表2,表3),證明本研究采用的分析方法有較好穩定性、可靠性。
除此之外,本研究對不同密度SNP 條件下的仿刺參疣足數量SNP 遺傳力估計值進行比較,結果顯示,當MAF>0.05 時(表2),不同密度SNP 條件下得到的仿刺參疣足數量SNP 遺傳力估計均值為(0.566±0.022)~(0.612±0.003),SNP 數量在5K、10K 時,得到的SNP 遺傳力估計均值分別為0.567±0.022、0.588±0.011(AI-REML)和0.566±0.022、0.587±0.011(EMREML);SNP 數量在15K~50K 時,得到的遺傳力估計均值相較于低密度SNP 遺傳力估計均值更大,且趨于穩定;SNP 數量為50K 時,得到的SNP 遺傳力估計均值最高,分別為0.612±0.003(AI-REML)和0.611±0.003(EM-REML)。當MAF>0.1 時(表3),不同密度SNP 條件下得到的仿刺參疣足數量SNP 遺傳力估計均值為(0.586±0.015)~(0.615±0.016),SNP 數量在5K、10K、15K 時,得到的遺傳力估計均值分別為0.593±0.026、0.596±0.017、0.587±0.014(AI-REML)和 0.592±0.026 、 0.595±0.017 、 0.586±0.015(EMREML);SNP 數量在20K~50K 時,得到的遺傳力估計均值相較于低密度SNP 遺傳力估計均值更大,且趨于穩定;SNP 數量為30K 時,得到的SNP 遺傳力估計均值最高,分別為0.615±0.016(AI-REML)和0.614±0.016 (EM-REML),并且當SNP 數量在35K、40K、50K 時得到的遺傳力估計均值均達到0.614。

表2 仿刺參疣足數量SNP 遺傳力(MAF>0.05)Tab.2 SNP heritability estimates for papillae number in A. japonicus (MAF>0.05)
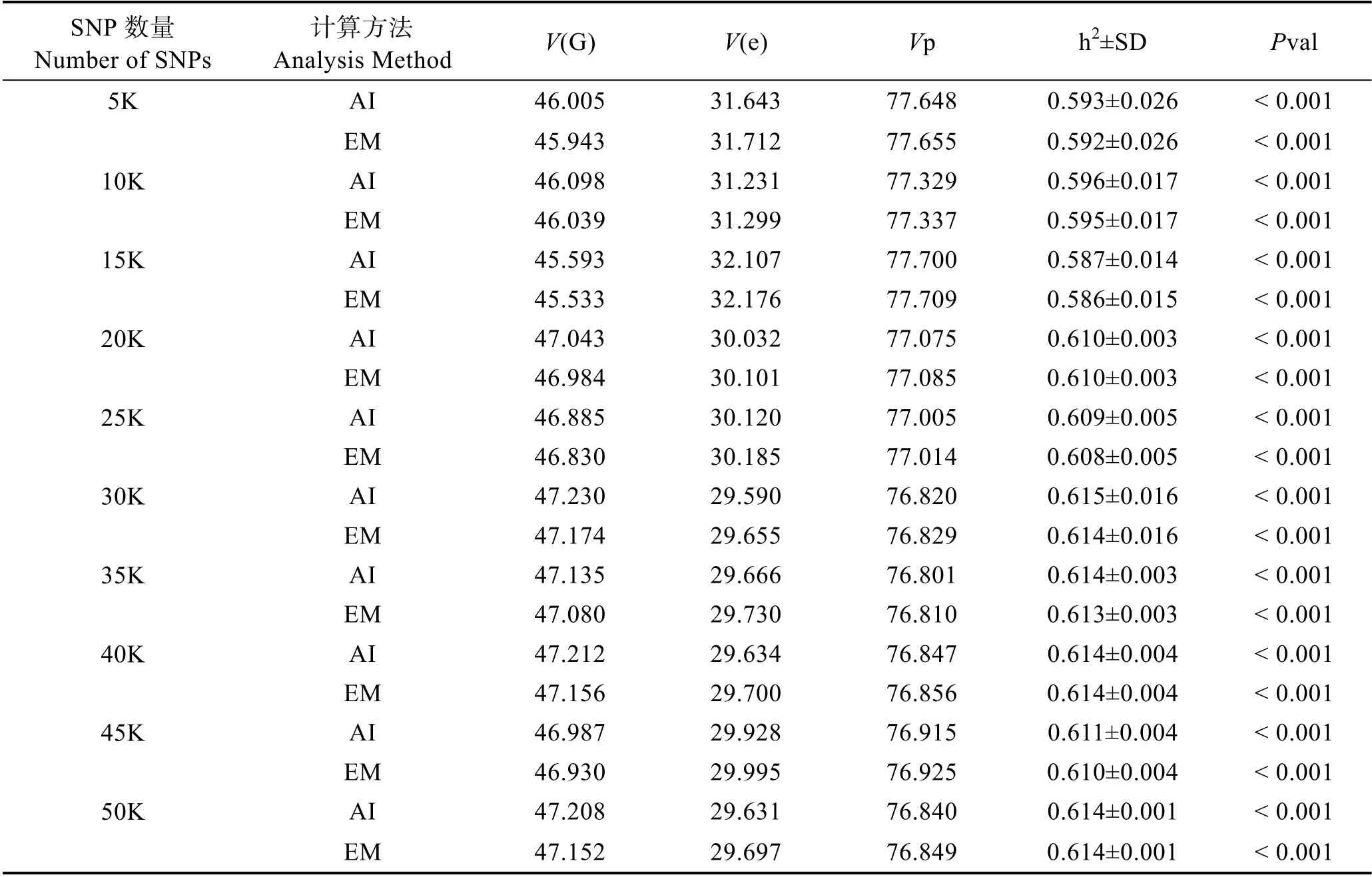
表3 仿刺參疣足數量SNP 遺傳力(MAF>0.1)Tab.3 SNP heritability estimates for papillae number in A. japonicus (MAF>0.1)
2.3 仿刺參疣足數量染色體水平SNP 遺傳力估計
根據已發表的仿刺參基因組和遺傳連鎖圖譜,共發現22 個連鎖群(Tian et al, 2015; Li et al, 2018),本研究將Scaffold 拼接到染色體水平,并對仿刺參疣足數量進行染色體水平的SNP 遺傳力估計,對染色體SNP 遺傳力估計值和染色體長度進行回歸分析(圖2),結果顯示,7 號染色體的SNP 遺傳力估計值最小,為0.085(MAF>0.05)、0.094(MAF>0.1);2 號染色體的SNP 遺傳力估計值最大,為0.598(MAF>0.05)、0.599(MAF>0.1);14 號染色體長度最小,SNP遺傳力估計值為 0.269(MAF>0.05)、0.282(MAF>0.1);1 號染色體長度最大,SNP 遺傳力估計值為0.522(MAF>0.05)、0.526(MAF>0.1)。在MAF>0.05和MAF>0.1 條件下,單個染色體的貢獻和其長度顯著相關,較長的染色體具有明顯的線性趨勢(P<0.05),且遺傳力估計值最小的7 號染色體和長度最小的14 號染色體均偏離線性回歸較遠。
3 討論
遺傳力是反映性狀遺傳能力大小的重要遺傳參數,準確合理的遺傳參數估計有助于更好地理解遺傳因素對特定群體某一性狀的表型影響程度。估計動物個體的SNP 遺傳力,首先需要篩選出一組適用的SNP。篩選這些SNP,一般需要滿足兩個條件:第一,篩選的 SNP 是該物種基因芯片中共同的SNP,這樣可以很方便地將這些基因SNP 芯片用于分子育種,而不需要重新設計新的芯片或增加芯片中的SNP 位點;第二,選出的SNP 有較高的信息含量,其統計準確度要高。Hulsegge 等(2013)采用LD 的r2>0.30 作為刪除SNP 的尺度,結果表明,在保持相同準確性的前提下,使用這個尺度來篩選SNP,可以明顯降低所需SNP 標記的數量。同時,需要篩選信息量高的SNP,篩選高信息量的SNP 可以依據不同的統計指標。例如,Hulsegge 等(2013)分別使用 Delta(群體間等位基因頻率的絕對差異)、Wright(1978)的FST 以及Weir 等(1984)的FST 衡量標記信息量的效果。信息熵(Entropy)(Mitt et al, 2017)也是衡量SNP 信息量的指標。本研究采用SNP 基因頻率的平均歐式距離(Average Euclidean distance,AED) (Wu et al, 2016)來篩選SNPs。后續工作將根據上述指標,挑選信息含量高并可進行準確的遺傳參數估計的低密度SNP 標記。

圖2 仿刺參疣足數量染色體水平SNP 遺傳力估計Fig.2 Chromosome-wise heritability estimates for papillae number in A. japonicus
群體的分層效應會對遺傳力估計值的準確性產生影響。本研究根據215 個仿刺參樣本疣足數量的頻率直方圖和概率密度曲線(圖1)初步將研究樣本分為兩大類,一類為來自中國的品種(正常群體),另一類為俄羅斯以及中國與俄羅斯雜交的品種(多疣足群體)。計算時使用群體分層這一因素作為協變量進行校正,并得到了穩定結果,證明本研究使用的分組依據可靠。本研究使用了基于GCTA 的期望最大約束似然法(EM-REML)和平均信息約束似然法(AI-REML)對仿刺參疣足數量的SNP 遺傳力進行估計,由估計值結果(表2、表3)可知,此方法得到的SNP 遺傳力估計值均具有較高的穩定性和可信度。此前,孟思遠等(2010)通過建立家系,使用傳統方法估算仿刺參幼參階段肉刺數目的遺傳力,得到仿刺參肉刺(疣足)數量遺傳力為0.191~0.404。本研究使用GCTA 的REML分析法得到了與傳統方法相比更高、更穩定的遺傳力估計值,說明疣足數量主要受加性效應的控制,對仿刺參的選擇育種具有較大潛力。通過比較不同SNP密度下仿刺參疣足數量SNP 遺傳力估計值,發現當SNP 數量在50K 時,得到的遺傳力估計值均已達到穩定,說明50K SNP 的密度足夠捕獲數量性狀基因座(QTL)大效應和小效應,證實了低密度SNP 用于準確估計遺傳參數的可行性。同時,在全基因組選擇方面,Wu 等(2016)通過設計牛的低密度SNP 芯片,對育種值估計的準確性進行評估,獲得了較準確的結果,也證實了低密度SNP 芯片應用于分子育種的可靠性。由于全基因組分析對樣本量要求較高,通常在500 以上(Yang et al, 2010),本研究的樣本量為215,統計量較少,容易導致統計結果出現一定程度的偏差,但所得結果與已報道數據(孟思遠等, 2010; 和飛等, 2017)一致。后續工作將擴大樣本統計量進行仿刺參數量性狀的遺傳力評估。
本研究在染色體水平對仿刺參疣足數量SNP 遺傳力進行估計(圖2),提供性狀遺傳結構的信息。結果顯示,仿刺參疣足數量是一個復雜的數量性狀,不同染色體上的變異導致了仿刺參疣足數量的變異,與該性狀相關的效應位點散布在各染色體上,說明了通過重測序的技術手段,在全基因組上尋找SNP 的重要性。單個染色體的貢獻和其長度顯著相關,表示在每條染色體上可能有多個小到中等效應的變異,而不是少數具有主要效應的變異。Goddard 等(2009)關于家畜復雜性狀的分子標記輔助選擇結果也支持這一假設,該報道稱,性狀變異有很大的遺傳因素,但由于統計力量不足,大多數具有小到中度影響的變異尚未在遺傳關聯分析中被確認。而GCTA 可以克服分子標記輔助選擇的缺陷,通過全基因組標記面板,所有QTL 都能和至少1 個marker 處于連鎖不平衡狀態。分析發現,7 號染色體雖不是最小染色體,但其遺傳力估計值最低,與此相反,2 號染色體雖不是最大染色體,卻具有最大遺傳力估計值,說明較大的基因效應位點可能不在7 號染色體上,而位于2 號染色體,意味著QTL 出現在2 號染色體的概率較大,出現在7 號染色體上的概率較小。此結果的發現為將來開展仿刺參疣足數量的全基因組關聯分析(GWAS)提供了支持。
4 總結
本研究對不同養殖地理位置仿刺參群體進行全基因組重測序,并針對仿刺參重要經濟性狀疣足數目,使用GCTA 軟件的GREML 進行全基因組水平的SNP 遺傳力估計,染色體水平SNP 遺傳力估計,同時比較不同密度SNP 標記條件下的仿刺參疣足數量SNP 遺傳力估計值;結果表明,仿刺參疣足數量SNP遺傳力為中等遺傳力(MAF>0.05 時,0.566~0.612;MAF>0.1 時,0.586~0.615),當SNP 數量在50K 時,得到的遺傳力估計值均已達到穩定,且染色體SNP遺傳力與其長度顯著相關;說明仿刺參疣足數量是復雜數量性狀,由全基因組水平多基因共同作用,且50K SNP 密度足夠捕獲QTL 大效應和小效應。本研究通過分子學研究方法得到可靠的遺傳力估計值,有效解決了傳統方法系譜記錄繁瑣,遺傳力估計準確性差等問題,為分子育種技術提供了一定理論依據;同時,證明低密度SNP 標記用于仿刺參分子育種的可能性,可為低密度SNP 芯片的設計開發提供支持。

