利用EvolvR系統對酶基因實現多窗口定向進化的研究
林峻, 陳玨, 黃佳敏
(福州大學生物科學與工程學院, 福州大學應用基因組學研究所, 福建 福州 350108)
0 引言
基因定向進化是一種廣泛應用的生物工程策略, 定向進化也稱體外進化或實驗室進化, 在無需完全了解蛋白質結構的情況下, 即可直接改進蛋白質功能. 定向進化的成功主要取決于兩個方面: 高效構建突變文庫和高通量篩選突變體[1-2]. 研究表明, 單個氨基酸突變就可以增強蛋白的催化活性或穩定性[3], 但目前尚不清楚隨機突變整個編碼序列還是僅突變其活性位點更有效[4]. 研究人員已經開發出多種策略來產生分子多樣性, 最廣泛的方法就是隨機突變. 隨機突變與高通量篩選相結合, 是改善蛋白質功能或產生人工酶的一種通用策略[5-6].
傳統的定向進化要求在一定的范圍內突變目標核苷酸. 目前, 還沒有一種高效的方法可以連續地改變目標區域的所有核苷酸[7-8]. 2018年, Halperin等開發了一種名為EvolvR的新型突變工具[9]. EvolvR將CRISPR/Cas9的特異性識別元件與易錯DNA聚合酶的突變能力結合起來, 通過自主設計的含20個堿基的向導RNA(sgRNA)來實現精準定位. EvolvR有兩個重要元件, 一是變體Cas9(nickaseCas9; nCas9)[10], 其RuvC核酸酶結構域具有缺陷, 僅能誘發DNA單鏈斷裂; 二是直接與nCas9融合的易錯聚合酶(PolI3M)[11]. 聚合酶的保真度決定了突變率的高低. 研究表明, PolI3M是PolI中最容易發生聚合出錯的變體. PolI3M包含3個點突變(D424A、 I709N和A759R), 這些突變會增加其聚合酶活性的錯誤率并消除其校對活性. 該研究證明了這兩個元件的融合, 可使PolI3M的突變活性指向nCas9靶向的基因組目標位點, 而不是在基因組中任意突變DNA. EvolvR可以在研究者選定的可調控窗口長度內, 不斷地使該區域的核苷酸發生隨機變異, 它是CRISPR工具箱中的一種最新的獨創性工具.
目前, 僅有研究報導了EvolvR用于抗性基因的突變, 以及串聯最多2個sgRNAs以擴大突變窗口長度, 還沒有EvolvR用于酶定向進化的報道, 也沒有大于2個sgRNAs串聯的應用報道. 因此, EvolvR的實用性仍然有待商榷.
本研究利用EvolvR突變質粒(enCas9-PolI3M-TBD)對T7 DNA 聚合酶誘導突變, 并嘗試將4個能高效表達的 sgRNA 串聯, 觀察其是否能同時靶向同一基因的不同靶標位點. T7 DNA 聚合酶是一種分子生物學基因操作的工具酶, 在末端標記、 第二鏈cDNA合成、 長片段引物延伸等方面均有用途. 對其進行定向進化, 可以提高活性、 甚至有可能進化出新的生物學功能. 本研究期望探究EvolvR在酶基因定向進化上的潛力, 尤其是多位點同時編輯, 為將來更好地利用EvolvR系統打下基礎.
1 材料與方法
1.1 材料
1.1.1 菌株與質粒
本研究使用的EvolvR質粒pEvolvR-enCas9-PolI3M-TBD(Plasmid#113077)和pEvolvR-enCas9-PolI5M(Plasmid # 113078)均購于美國addgene官網. 目標質粒pCDFDuet-T7poly(含噬菌體T7 DNA聚合酶基因)由本實驗室保存. 4個sgRNA串聯的基因序列由安徽通用生物有限公司合成.
1.1.2主要工具酶和試劑
研究用的2×TaqMasterMix購自北京康為世紀生物技術有限公司; Bsm BI、 NEBNext Ultra II Q5 Master Mix和T4 DNA ligase購自美國NEB公司; Tn5轉座酶購自天津強微特公司; 卡那霉素Kanamycin、 氨芐青霉素Ampicillin、 大觀霉素Spectinomycin以及超級感受態細胞細胞制備試劑盒均購自生物工程(上海)股份有限公司; 蛋白胨、 酵母粉購自英國OXCID公司; 250 bp DNA ladder marker和In-Fusion HD Cloning Kit購自寶生物工程(大連)有限公司; TG1感受態細胞購自上海唯地生物技術有限公司; 膠回收試劑盒購自美國Thermo Fisher公司.
1.1.3引物序列
實驗所用引物見表1.
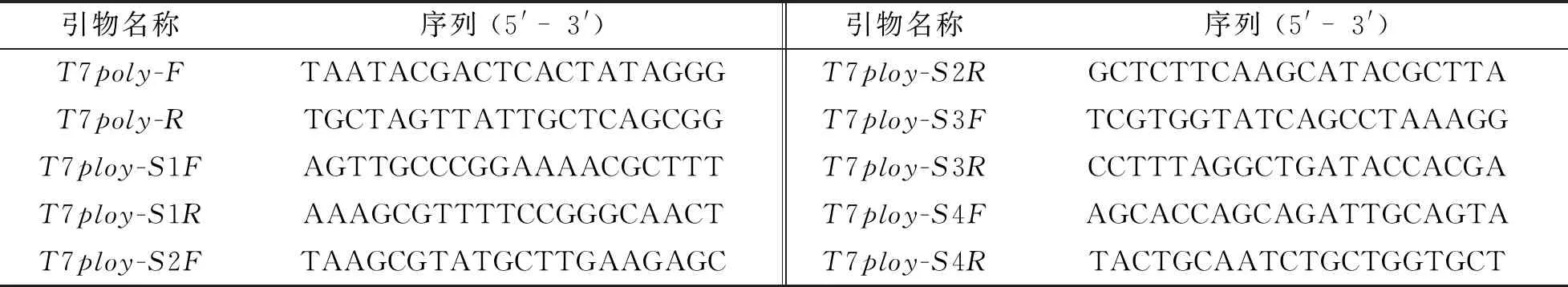
表1 實驗所用引物
1.2 方法
1.2.1 目標質粒pTarget的獲取
pCDFDuet-T7poly(大觀霉素抗性, 100 μg·mL-1)質粒由本課題組保存. 原始菌種劃線活化培養, 挑取單菌落過夜培養, 使用上海生工質粒小量提取試劑盒提取質粒, 并用Nanodrop 2000 (購自美國Thermo Fisher公司)測定濃度, -20 ℃保存備用.

表2 質粒酶切體系
1.2.2串聯sgRNA質粒的構建
將兩種EvolvR質粒pEvolvR-enCas9-PolI3M-TBD和pEvolvR-enCas9-PolI5M在平板上劃線活化, 挑取單菌落于LB液體培養基(卡那霉素抗性, 250 μg·mL-1)中過夜培養, 小提質粒-20 ℃保存備用. 使用限制性內切酶Bsm BI對載體進行酶切, 酶切體系如表2, 酶切反應條件為55 ℃反應45 min, 然后80 ℃加熱20 min使酶徹底變性. 最后, 膠回收酶切產物, 測定濃度后-20 ℃保存備用. 利用在線設計軟件http://crispr.mit.edu/進行sgRNA序列設計, 4種sgRNA串聯共表達結構如下:
J23115 promoter-[Insert 1st gRNA 20N]-Single gRNA body-L3S2P21 terminator-J23116 promoter-[Insert 2nd gRNA 20N]-Single gRNA body-L3S2P56 terminator-J23116 promoter-[Insert 3rd gRNA 20N]-Single gRNA body-L3S2P21 terminator-J23115 promoter-[Insert 4th gRNA 20N]-載體上的Single gRNA body-載體上的TrrnB terminator
在通用生物(安徽)有限公司訂購合成上述全基因序列, 在序列兩端添加Bsm BI酶切位點, 將該合成基因產物進行Bsm BI酶切, 并連入enCas9-poll3M-TBD和enCas9-poll5M膠回收的線性載體上, 連接反應體系如表3. 連接體系置于16 ℃過夜反應. 轉化NEB-10β感受態細胞, 轉化產物涂抗性平板(卡那霉素)過夜培養. 挑取單菌落送測序驗證, 測序引物使用PBAD-R反向通用測序引物, 驗證正確后, 提取質粒-20 ℃保存備用.

表3 EvolvR線性化載體與串聯sgRNA酶切產物連接反應體系
1.2.3 EvolvR突變條件的確定
本研究采用雙質粒共轉化方案. 兩種質粒的工作濃度控制在100 ng·μL-1, 各取0.5 μL(50 ng)充分混合用于共轉化大腸桿菌TG1.
1.2.4利用EvolvR突變質粒構建目標酶的突變文庫
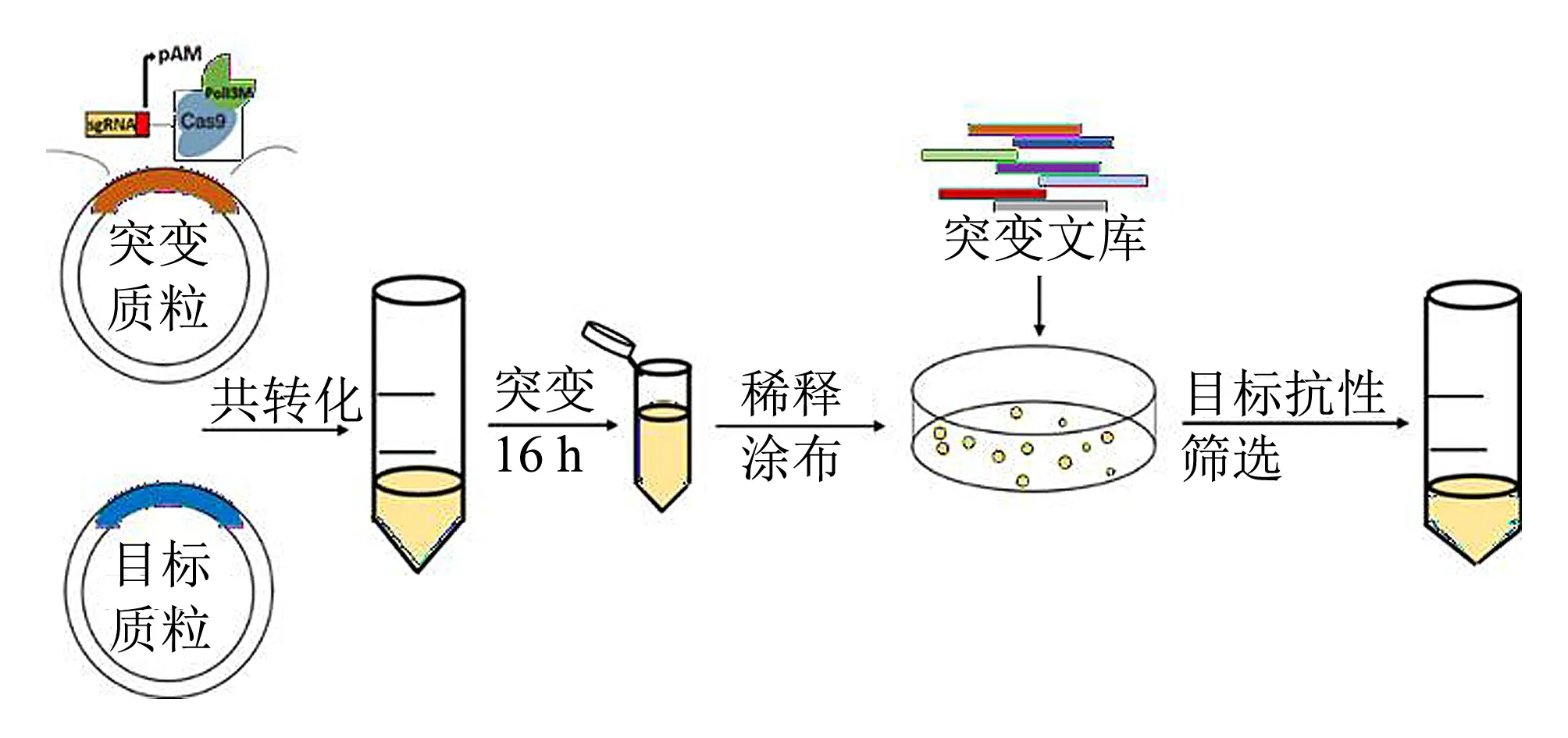
圖1 突變文庫的構建過程
目標質粒(大觀霉素抗性)和4個sgRNA串聯表達的突變質粒(卡那霉素抗性)共轉化構建突變文庫的過程如圖1所示. 在突變前將兩個質粒濃度調整為100 ng·μL-1. 各取50 ng質粒混合充分, 共轉化至TG1感受態細胞. 加入到具有目標質粒和突變質粒兩種抗性的LB液體培養基中震蕩培養. 之后再將混合菌液稀釋涂布在含目標質粒相應抗性的平板上, 篩選突變轉化子, 送測序驗證突變情況.
1.2.5建庫高通量測序分析突變文庫
從突變反應后的菌液(混合文庫)中提取混合質粒, 以該質粒為模板進行PCR反應, 擴增得到突變后的T7poly基因全長. 將其回收, 用1×TE稀釋至50 ng·μL-1并進行高通量測序建庫, 建庫流程如圖2所示.
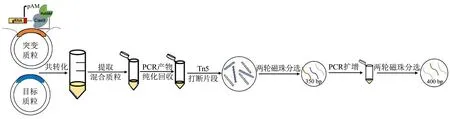
圖2 高通量測序建庫過程
配制10.5 μL反應體系, 利用Tn5復合體將DNA片段隨機打斷, 反應體系如表4所示. 之后, 進行磁珠分選, 分選目的分子量為350 bp的DNA碎片. 再利用NEBNext Ultra II Q5 Master Mix進行擴增, 配制20 μL的擴增反應體系, 如表5所示.

表4 DNA片段化
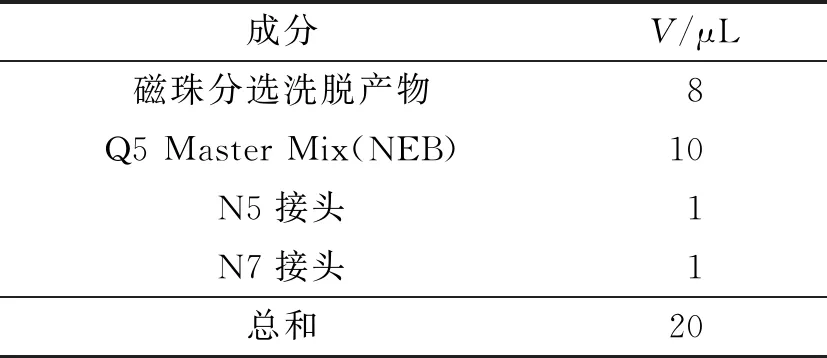
表5 NEB M0544s酶擴增
PCR的反應條件: 75 ℃, 3 min; 98 ℃, 30 s; (98 ℃, 10 s; 65 ℃, 1 min 15 s)×15 循環; 65 ℃, 5 min. 對文庫擴增產物再次進行磁珠分選, 分選目的相對分子質量為400 bp的DNA. 分選產物取2 μL樣品進行毛管電泳文庫質控. 通過文庫質控的產物, 使用Illumina Novaseq 6000測序儀, 在Pair end 150模式下, 對自建文庫進行高通量測序.
將poll3M-TBD突變質粒作用后的高通量測序數據和poll5M作用后的高通量測序數據進行數據質控分析. 首先, 將rawdata的原始文件上傳至Galaxy網頁版(https://usegalaxy.org/), 用FastQC(Galaxy Version 0.72)程序得到rawdata的質量報告; 然后用Trim Galore!(Galaxy Version 0.6.3)程序對rawdata文件進行修剪; 之后用BWA-MEM(Galaxy Version 0.7.17.1)程序將修剪后的文件與T7poly基因全長序列進行比對, 從而得到后續分析所需要的bam結果文件. 將該結果經IGV軟件[11]作可視化處理分析.
2 結果與分析
2.1 EvolvR串聯突變質粒的構建
EvolvR質粒酶切電泳圖如圖3所示. 將pUC57-串聯sgRNA質粒也進行酶切回收, 純化回收750 bp的目的條帶(見圖4). 將串聯sgRNA插入酶切回收后的EvolvR質粒載體上. EvolvR質粒含有綠色熒光蛋白基因, 轉化大腸桿菌后, 菌液呈現偏綠色. 酶切質粒會切除綠色熒光蛋白基因, 所轉化的菌液就會失去綠色. 這可作為是否成功構建突變質粒的一個判斷依據. 構建得到的重組質粒送Sanger測序驗證.

圖3 EvolvR質粒酶切電泳結果
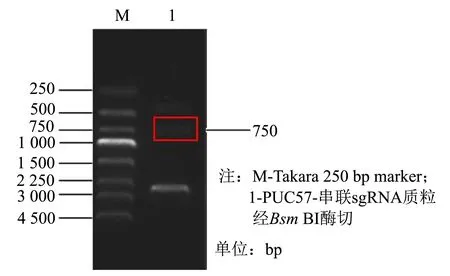
圖4 PUC57-串聯sgRNA質粒酶切
2.2 串聯sgRNA突變效果的高通量測序分析
針對目標質粒pCDFDuet-T7poly, 將4個能高效表達的sgRNA串聯, 對T7poly進行隨機突變. 從enCas9-poll3M-TBD串聯突變質粒作用后的突變混合菌液中提取質粒, 進行高通量測序建庫, 文庫質控的結果如圖5所示. 使用fastQC軟件對測序結果質控如圖6所示. 該文庫數據量為0.5 G, 與目標序列mapping后得到337 533條reads, 高通量測序結果經IGV軟件可視化處理分析, 如圖7所示, 發現4個串聯sgRNA有3個發揮了作用.
1號sgRNA靶向區域, 發現兩個位點發生突變, 343 bp和359 bp存在堿基置換突變. 343 bp(靶向18 bp)的總測序量為303 032條, 其中有1%的T置換為G. 359 bp(靶向2 bp)的總測序量為295 672條, 其中有1%的T置換為G. 2號sgRNA靶向區域中有一個位點檢測到突變. 382 bp(靶向8 bp)的總測序量為220 823條, 其中有1%的T置換為G. 3號sgRNA的813 bp(靶向-7 bp)的總測序量為144 177條, 其中有2%的T置換為G. 為了直觀反映靶向位置的堿基突變, 統計作圖如圖8所示.
圖5 enCas9-poll3M-TBD串聯突變質粒突變文庫毛細管電泳質控結果
圖6 enCas9-poll3M-TBD串聯突變質粒突變文庫fastQC質控圖

圖7 enCas9-poll3M-TBD串聯突變文庫高通量測序結果經IGV軟件可視化處理分析
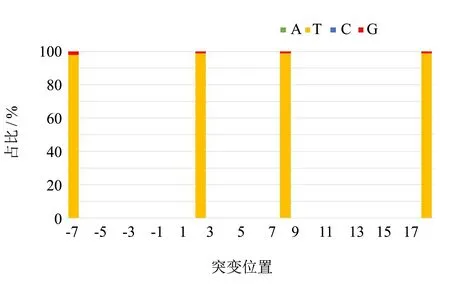
圖8 enCas9-poll3M-TBD串聯突變質粒突變高通量測序結果
從enCas9-poll5M串聯突變質粒作用后的突變混合菌液中提取質粒進行高通量測序建庫, 文庫質控的結果如圖9所示. 該文庫測序數據量為0.5 G, 與目標序列mapping后得到367 001條reads, 文庫的fastQC質控如圖10所示, 高通量測序結果經IGV軟件可視化處理分析, 如圖11所示. 研究發現, 4個串聯sgRNA中只有1個發揮了作用, 1號sgRNA靶向序列的358 bp和359 bp處存在堿基置換突變. 358 bp處覆蓋有的總測序量為317 084條reads, 其中有1%的T置換為G. 359 bp處覆蓋有的總測序量為316 898條reads, 其中有1%的T置換為G. 為了直觀反映靶向位置的堿基突變, 統計作圖如圖12所示. 由此也能判斷enCas9-poll5M突變效率確實低于enCas9-poll3M-TBD.
定向進化是一個突變和選擇的循環過程, 在此過程中目標DNA的序列發生突變. 不可否認, 定向進化技術正成為現在及未來幾年生命科學研究領域的熱點技術之一. 定向進化技術在不斷發展中, EvolvR利用CRISPR/Cas9的特異性與易錯DNA聚合酶的突變能力, 可以簡單快速地構建隨機突變文庫. 它通過自主設計的20 nt向導RNA(sgRNA)實現精準定位, 一個sgRNA能靶向一個靶向位點[12-14].
傳統的定向進化技術, 諸如Error-prone PCR和DNA shuffling, 較難對靶標基因的目的區域進行有選擇性的定向進化, 且實驗步驟繁瑣, 需要很多分子克隆操作, 門檻高. 而EvolvR技術利用了CRISPR的靶向能力, 可以針對靶標基因上的特定區域, 進行基因突變, 結合蛋白結構與功能研究, 無疑可以更加有目的性和理性地對目標基因進行定向進化, 大大提高了工作效率. 同時, EvolvR操作簡便, 只要把目的基因和sgRNA構建于質粒載體上, 剩余的工作即可交給大腸桿菌, 通過不停的傳代繁殖, 實現快速進化, 減少了工作量.
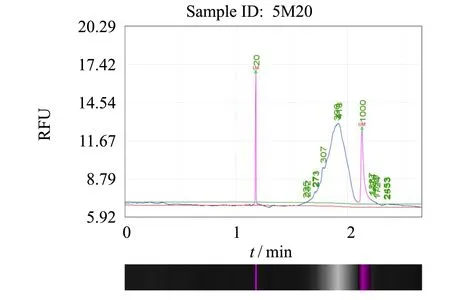
圖9 enCas9-poll5M串聯突變質粒突變文庫毛細管電泳350 bp文庫質控結果
圖10 enCas9-poll5M串聯突變質粒突變文庫fastQC質控圖
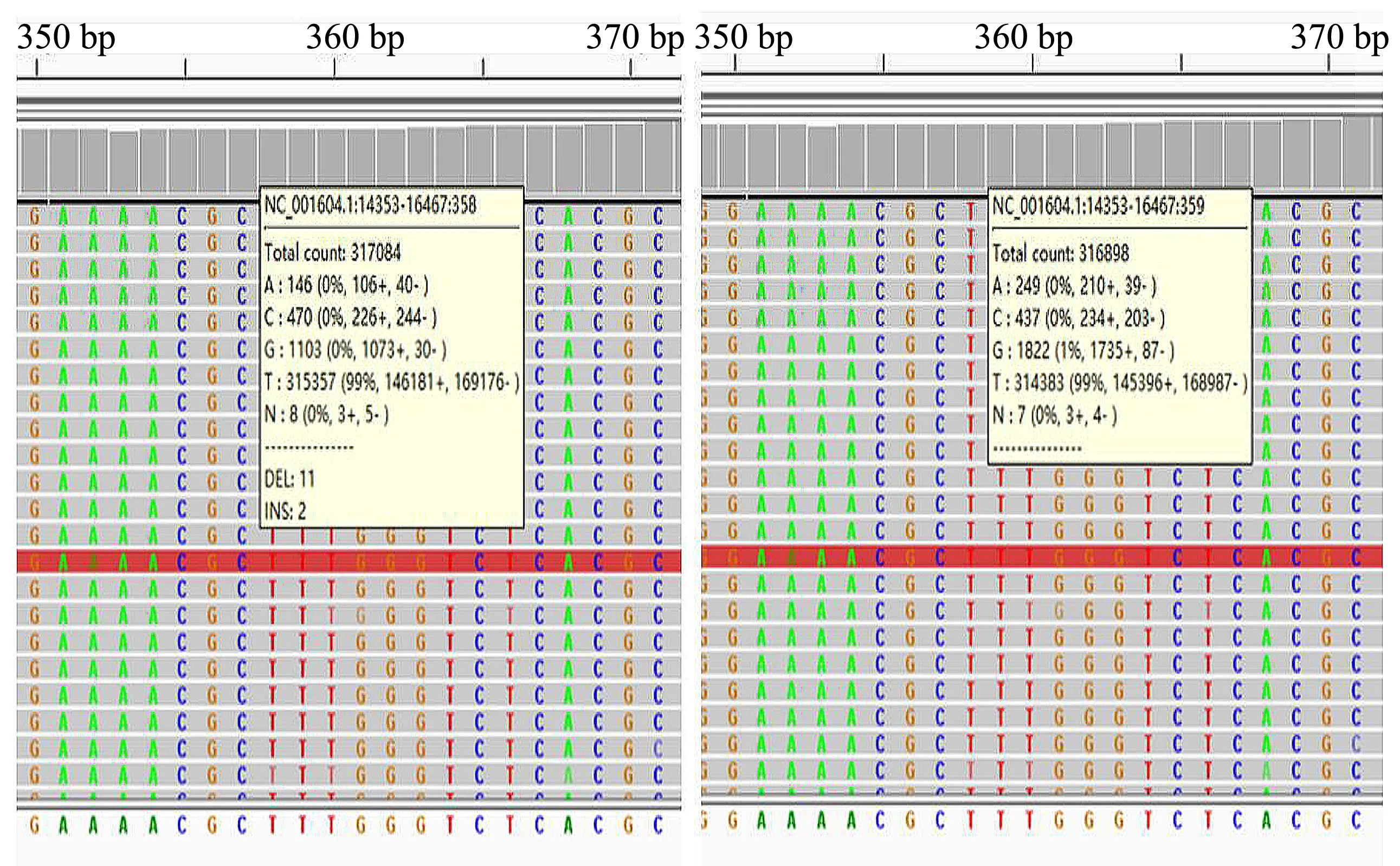
圖11 enCas9-poll5M串聯突變文庫高通量測序結果經IGV軟件可視化處理分析
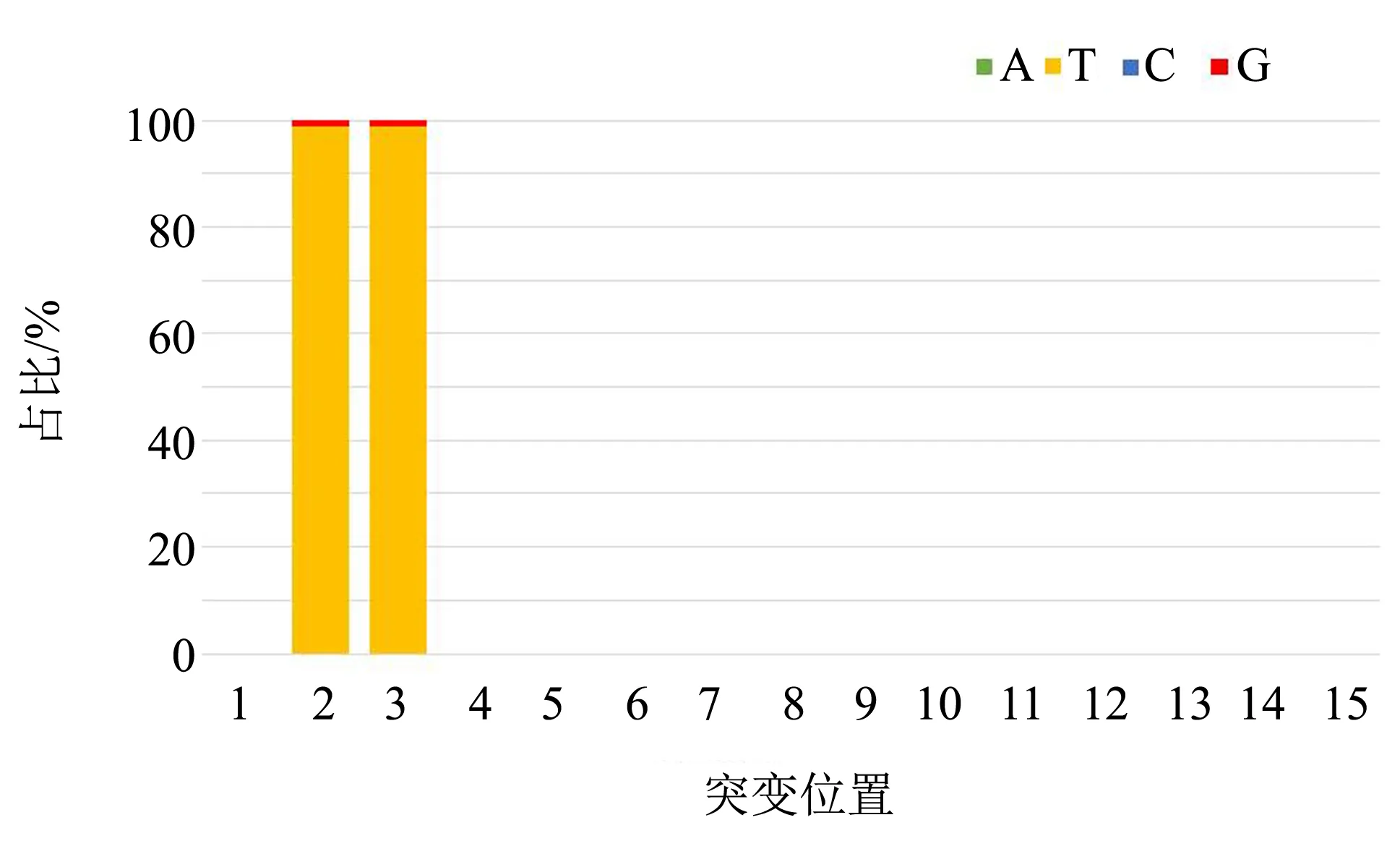
圖12 enCas9-poll5M串聯突變質粒突變高通量測序結果
3 討論
本研究旨在探究EvolvR系統在酶定向進化中的應用, 將原先僅僅用于抗生素耐藥基因進化的EvolvR首次嘗試用于酶基因的隨機誘變, 同時嘗試擴大突變窗口長度, 將4個能高效表達的 sgRNA 串聯表達, 發現其具備同時靶向同一基因的不同靶標位點的潛力. 與單個sgRNA相比, 4個sgRNA串聯表達的突變效率會明顯降低, 4個sgRNA里有3個能檢測到其發揮了作用. 本研究的一個創新點在于選用新型定向進化工具EvolvR對目標酶基因序列產生隨機突變, 嘗試擴大突變窗口個數, 以快速構建一個大型的突變文庫. 本研究揭示了EvolvR技術具有在目標基因的特定區域大范圍制造基因突變潛力, 證明了EvolvR具有重要的應用價值.

