大菱鲆體重和體尺性狀聯合GWAS分析*
高 進 楊潤清
大菱鲆體重和體尺性狀聯合GWAS分析*
高 進1,2楊潤清1,2①
(1. 南京農業大學無錫漁業學院 無錫 214081;2. 中國水產科學研究院生物技術研究中心 北京 100141)
為了揭示大菱鲆()體重和體尺性狀的分子遺傳機制,探尋用于改良目標性狀的分子標記及候選基因,本研究以大菱鲆育種群體為研究對象,分別測量其體重、體長、體寬和尾柄寬性狀的表型值,利用簡化基因組測序技術(2b-RAD)獲得相應基因型數據,進行全基因組關聯研究(Genome-wide association study, GWAS),篩選與大菱鲆體重和體尺性狀顯著關聯的數量性狀核苷酸(Quantitative trait nucleotides, QTNs)遺傳位點。結果顯示,以多性狀線性混合模型(mvLMM)對體重–體長和體長–體寬–尾柄寬2個性狀組合進行多性狀GWAS分析,分別檢測到9個和2個一因多效QTNs;以單一性狀線性混合模型(LMM)對各個性狀進行GWAS分析,在體重性狀中檢測到4個與之顯著關聯的QTNs,在體長和體寬性狀中各檢測到1個QTN,而在尾柄寬性狀中則沒有檢測到顯著的遺傳位點。比較2種模型的結果,發現mvLMM相較于LMM能夠檢測到更多QTNs,且檢測到的QTNs為更具生物學意義的一因多效QTNs。本研究首次利用mvLMM和LMM對大菱鲆體重和體尺性狀進行聯合GWAS分析,共篩選到17個顯著的QTNs,其中,有4個QTNs被重復檢測到。以這些檢測到的QTNs為探針,在大菱鲆全基因組上找到了距離其最近的12個候選基因,它們可能是影響大菱鲆體重和體尺性狀的重要候選標記和功能基因,本研究為大菱鲆體重和體尺性狀的分子標記輔助選育提供了理論素材和參考。
大菱鲆;體重和體尺性狀;全基因組關聯分析;多性狀線性混合模型
大菱鲆(),俗稱“歐洲比目魚”或“多寶魚”,是中國北部沿海地區重要的海水養殖經濟魚類。大菱鲆具有生長速度快、肉質鮮美以及抗逆性強等諸多優點,其養殖區域迅速擴展、產量產值不斷攀升(雷霽霖等, 2002、2005)。然而,隨著工廠化養殖規模和集約化程度的擴大,特別是種質資源的退化,成活率低、病害嚴重和生產緩慢等問題逐漸顯現(李杰等, 2019; 王嵐等, 2017)。因此,開展大菱鲆相關經濟性狀的選育研究,培育具有強抗逆性和高生長性能的新品種對大菱鲆產業發展具有重要意義。傳統的育種方法是基于目標性狀的表型值來開展選育工作(Hulata, 2001),對于高生長性能品種選育,通常挑選符合標準的個體進行群體混養或是建立多個家系并選擇優良的家系作為親本用于進一步選育。然而,分子生物學和基因組學的快速發展則促使魚類育種研究由傳統選育和雜交育種向基于基因組信息的分子育種轉變(Xu, 2015),如以基因定位和數量性狀位點(Quantitative trait locus, QTL)作圖為基礎的分子標記輔助選擇(Marker-assisted selection, MAS)育種(Wang, 2015; 劉曉菲等, 2019)。利用目標性狀與分子標記間的顯著關聯性,依據標記對后代個體進行全基因組選擇或是綜合標記與表型信息構建選擇指數篩選個體,加速育種過程,提高選育效率。
隨著測序成本的下降與高通量基因分型技術的迅猛發展,基于海量單核苷酸多態性(Single-nucleotide polymorphisms, SNP)標記的全基因組關聯分析(Genome-wide association studies, GWAS)已取代利用稀疏遺傳標記的QTL定位,成為鑒定與目標性狀顯著關聯的遺傳變異的主要方法。早期GWAS研究主要針對單一性狀表型(Yu, 2006; Burton, 2007),而在進行復雜性狀的遺傳解析且記錄有多個相關性狀表型值時,考慮多個性狀聯合信息的多性狀GWAS策略通常要優于逐個性狀與遺傳位點間的關聯分析(Kim, 2009; Bolormaa, 2010)。相較單一性狀GWAS分析,表型具有潛在相關性的多性狀GWAS分析更具優勢,主要是因為:當不同性狀間存在遺傳相關時,因考慮了單一性狀分析中忽略的性狀間協方差,多性狀分析提高了檢驗功效(Stephens, 2013)和參數估計的精度(Zhu, 2009);在多性狀GWAS分析過程中,數量性狀核苷酸(Quantitative trait nucleotides, QTNs)的統計推斷只進行一次統計檢驗,與逐個性狀單獨分析相比,降低了多重檢驗造成的誤差(Klei, 2008);存在基因多效性時,單個遺傳變異與多個性狀相關聯,使得多性狀GWAS分析更具生物學意義(Chavali, 2010);Porter等(2017)綜合比較了當下流行的多性狀GWAS分析方法,發現大多數現存的多性狀GWAS分析方法具有明顯相似的統計檢驗功效,與單一性狀GWAS相比,能夠大幅增加顯著遺傳變異的檢測效率。
本研究采用實驗成本較低的簡化基因組(2b- RAD)測序技術進行SNP分型,基于多性狀線性混合模型對體重–體長和體長–體寬–尾柄寬共2種多性狀相關表型組合進行全基因組關聯分析,關聯定位控制這些性狀的一因多效QTNs,為大菱鲆的體重和體尺性狀改良提供理論基礎。此外,對逐個性狀進行了單一性狀線性混合模型GWAS分析,并將其結果與多性狀GWAS分析結果進行比較。
1 材料與方法
1.1 實驗群體及表型測量
實驗用魚共585條,取自29個全同胞家系組成的大菱鲆育種群體,4月齡時,對群體中所有大菱鲆個體注射電子標記,并提取鰭條組織DNA用于后續的2b-RAD測序。隨后,將標記后的大菱鲆隨機混養在2個6 m×6 m×1.5 m具有循環水養殖系統的水泥池中,控制水溫在5℃~24℃,每天定時投喂商用餌料2次至飽食。實驗用魚按孵化日期至表型測量時的生長天數為275~1001 d不等。對實驗用魚進行11次不定期表型測量,每次測量前利用50 mg/ml MS-222魚用安定劑麻醉待測大菱鲆個體,避免應激反應造成的魚體損傷。利用電子秤稱量每個個體的體重(Body weight, g, BM)性狀,同時在統一的參考標尺下,用數碼相機由固定高度向下垂直拍攝相應個體的體尺性狀。根據拍攝圖形,利用ImageJ軟件標定每條大菱鲆的體長(Body length, cm, BL)、體寬(Body width, cm, BW)和尾柄寬(Caudal peduncle width, cm, CPW)共3個體尺性狀的表型值。采用簡化基因組2b-RAD高通量標記分型技術對具有表型測量記錄的個體進行SNP分型,共獲得30049個多態SNP分子標記,參考大菱鲆基因組(ASM318616v1)建立多態標記物理圖譜。挑選生長周期為473 d左右(前后皆不超過5 d)大菱鲆個體的表型觀測值作為多性狀GWAS分析的表型值,使用PLINK v1.9 (http://www.cog- genomics.org/plink2)對相應樣本個體的基因型數據進行質量控制。剔除低于90%最小檢出率的個體以及最小檢出頻率低于95%、最小哈代溫伯格平衡為1.0×10–6、最小等位基因頻率小于3%和方差變異大于0.05的SNPs,最終得到441個樣本的23988個SNP標記用于全基因組關聯分析。
1.2 關聯統計分析
本研究使用GEMMA軟件(Zhou, 2014)中的多性狀線性混合模型(Multivariate linear mixed model, mvLMM)進行多個性狀表型值和SNPs標記的全基因組關聯分析,所用模型為:
=+++
式中,為×維表型矩陣,是樣本個數,是分析性狀個數;為×維協變量(非遺傳固定效應,如性別、年齡等)矩陣,為維相應系數行向量,是包含截距項在內的協變量個數;為維當前檢驗SNP的基因型指示變量列向量,為維當前檢驗SNP的加性遺傳效應行向量;為×維不包括當前檢驗SNP的剩余多基因效應矩陣,~(0, V,是由全基因組SNP標記構建的基因組親緣關系矩陣,V是×維剩余多基因方差–協方差矩陣;為×維誤差項矩陣,~(0,VI),V是×維誤差方差–協方差矩陣,是單位矩陣。
最終,全基因組上每個SNP都能得到1個一因多效Wald統計量和相對應的統計概率值。此外,還利用因子譜分解線性混合模型(Factored spectrally transformed linear mixed model, FaST-LMM) (Lippert, 2011)逐個性狀進行單一性狀的GWAS分析,檢測與各個性狀相關聯的QTNs。使用R語言繪制曼哈頓和Quantile-Quantile (QQ)圖,同時,統計用于判別群體分層影響大小的膨脹系數或稱基因組控制(Genomic control, GC)值,在實際研究中GC值被定義為所有SNP標記的卡方統計量均值(Price, 2010)。
1.3 遺傳參數估計和基因注釋
將質量控制后的基因型數據用PLINK軟件(Chang, 2015)處理為分析所需的格式,再利用單性狀約束最大法估計法(Restricted maximum likelihood, REML)逐個估計各性狀的遺傳參數。查找關聯分析得到的顯著SNP位點在全基因組上所處的物理位置,于大菱鲆全基因組(GCA_003186165.1)上選擇距離其最近的候選基因進行注釋分析。
2 結果
2.1 表型值的描述性統計
選取生長天數為473 d左右的441條大菱鲆的體重、體長、體寬和尾柄寬4個性狀進行聯合GWAS分析。4個性狀的表型頻數分布見圖1。從圖1可以看出,各個性狀的表型值基本都符合正態分布,具有一定的可靠性,適合后續的GWAS分析。各性狀的原始表型的統計分析和遺傳力估計值見表1。此外,還對原始以及校正了性別、池子和測定日期等非遺傳固定效應后的表型值進行了性狀間的相關分析,發現各性狀表型值間具有較強的相關性(表2)。
2.2 GWAS分析結果
根據質量控制結果,441條大菱鲆的23988個SNPs被用于體重和體尺性狀的聯合GWAS分析,分別進行了BM-BL和BL-BW-CPW兩個性狀組合的多性狀線性混合模型以及逐個性狀的單一性狀線性混合模型GWAS分析。對BM-BL兩個性狀的多性狀GWAS分析后(圖2和表3)發現,超過基因組顯著水平(5% Bonferroni校正閾值,即2.084×10–6)的QTN位點共9個,其中,各有1個顯著QTN位于1、10和20號染色體上,即SNP_1_31495825、SNP_10_ 6893888和SNP_20_18773114,分別有2個顯著QTNs位于3 (SNP_3_21601589與SNP_3_24007357)、5(SNP_5_25635891與SNP_5_26888833)和22(SNP_22_ 6190492與SNP_22_6380029)號染色體上。而在相同顯著水準下,BL-BW-CPW 3個體尺性狀的GWAS分析則在10號染色體上檢測到2個顯著的QTN位點SNP_10_2724296和SNP_10_6893888。2組多性狀GWAS分析的GC值分別為BM-BL(1.035)和BL- BW-CPW(1.044)。
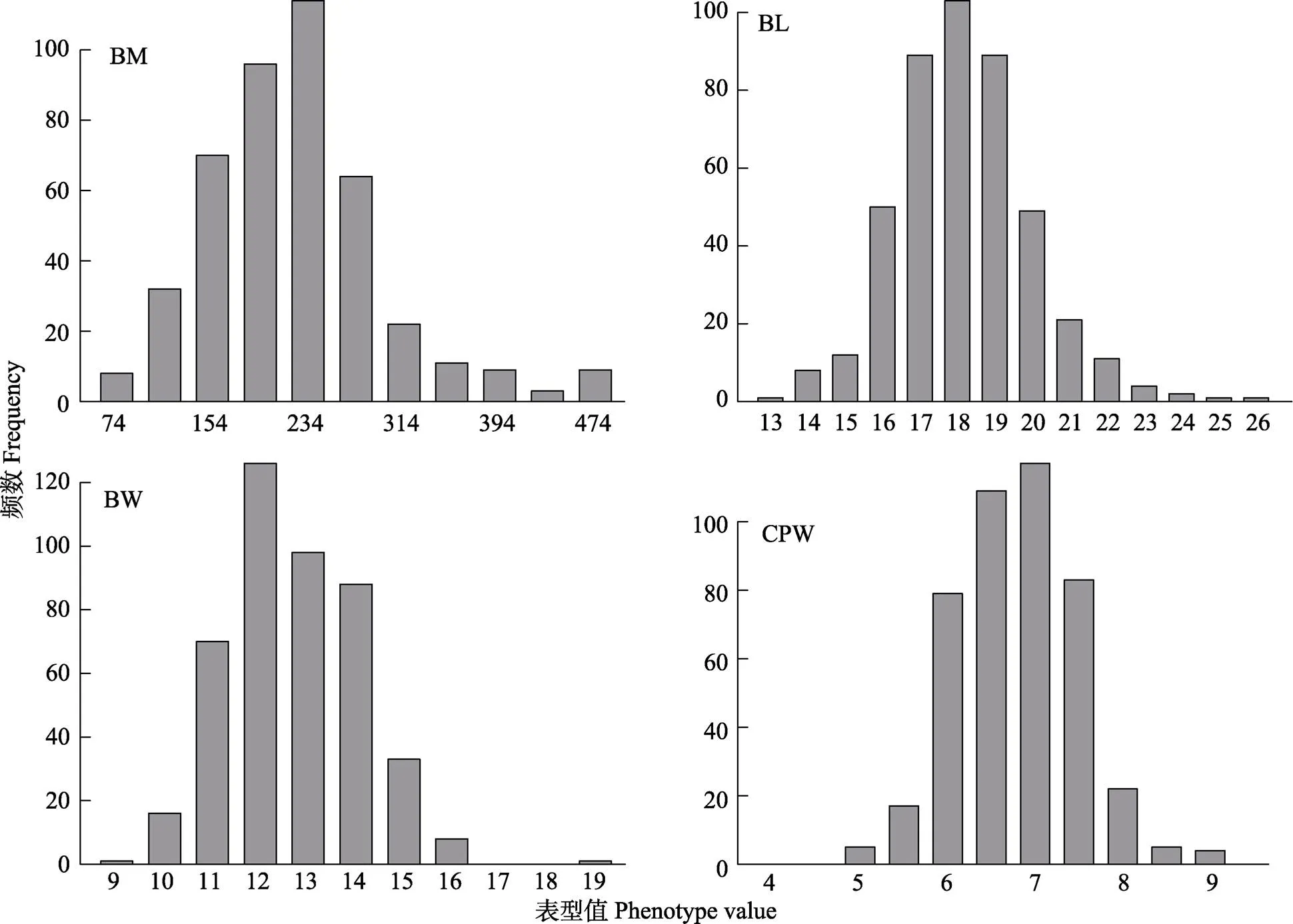
圖1 4個性狀的頻數分布
表1 大菱鲆體重和體尺性狀的描述性統計

Tab.1 Descriptive statistics of body weight and morphological traits in turbot
表2 大菱鲆體重和體尺性狀相關程度
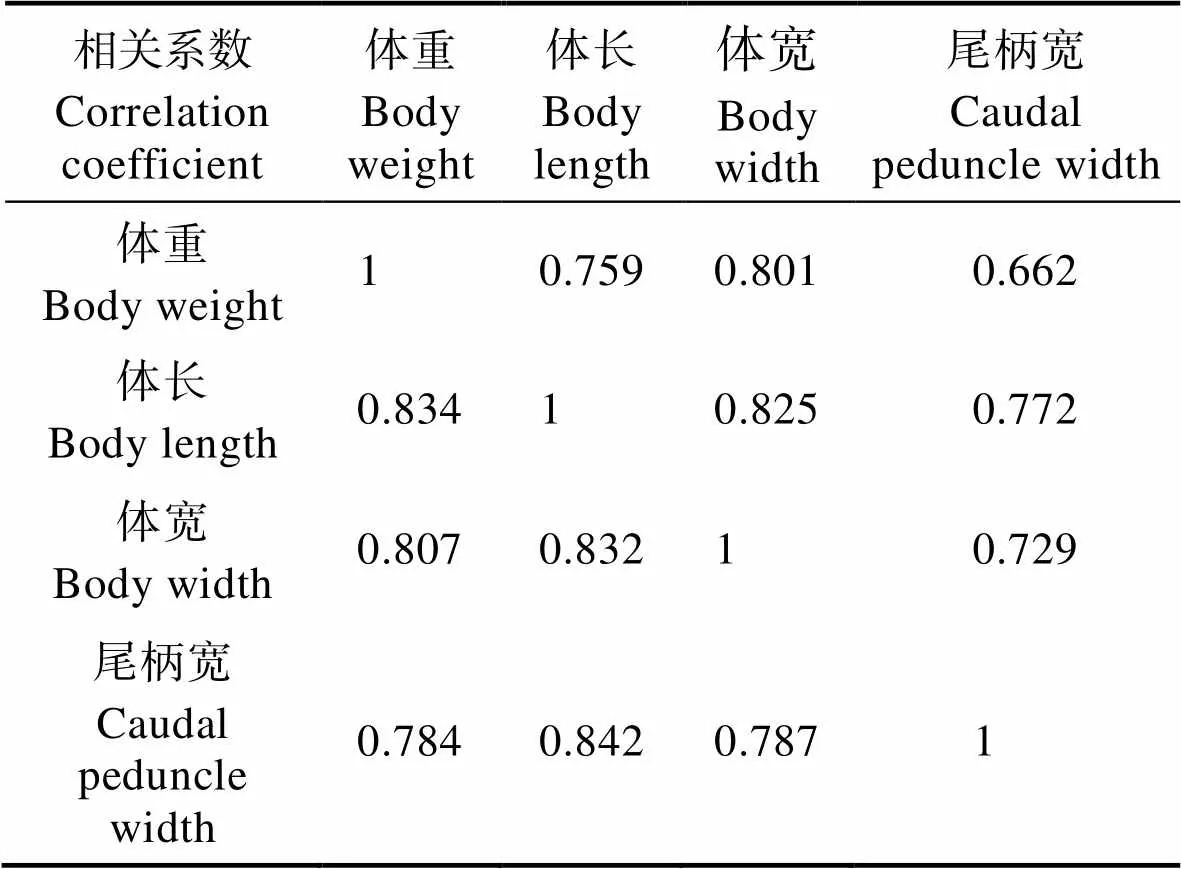
Tab.2 Degree of correlation for body mass and morphological traits in turbot
注:下三角中的值為性狀原始表型間相關程度,上三角中的值為校正了非遺傳固定效應后表型間的相關程度
Note: Values in lower-triangular matrix represent degrees of correlation among original phenotypes, and values in up-triangular matrix mean degrees of correlation among phenotypes which were adjusted by non-genetic fixed effects
對體重和體尺的逐個性狀單一性狀GWAS分析結果(圖3和表3)顯示,在BM性狀中,于3和5號染色體上各檢測到1個顯著的QTN位點,即SNP_3_21601589和SNP_5_26888833,10號染色體上檢測到2個顯著的QTN位點SNP_10_2724296和SNP_10_6893888。BL和BW性狀分別在62和8號染色體上檢測到1個顯著的QTN位點SNP_6_ 6047375和SNP_8_3870447。而在CPW性狀中則沒有檢測到顯著的遺傳變異位點。各性狀單一性狀GWAS分析的GC值分別為1.026 (BM)、1.021 (BL)、1.034 (BW)和1.031 (CPW)。
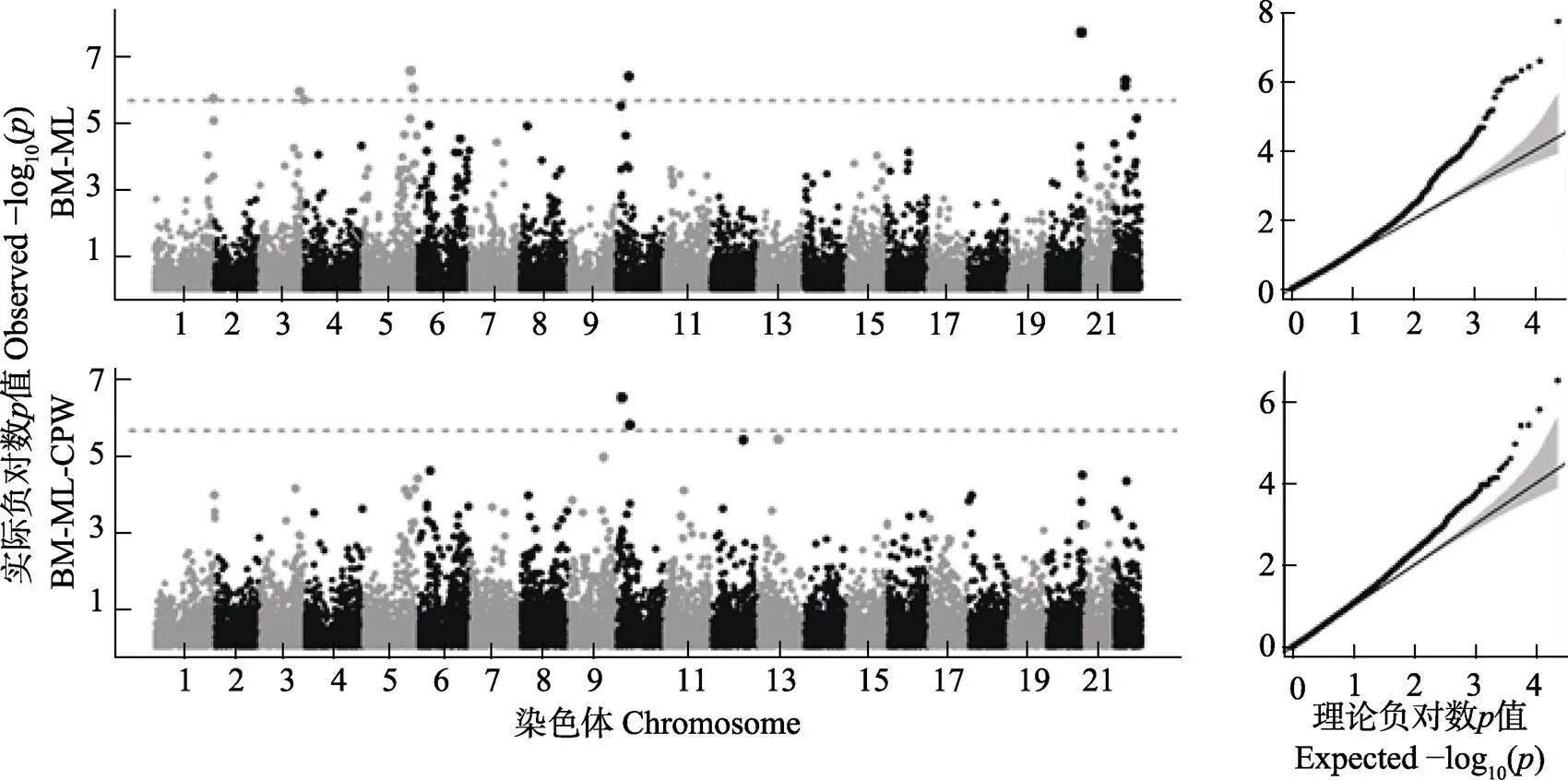
圖2 大菱鲆體重與體尺多性狀GWAS曼哈頓和QQ圖
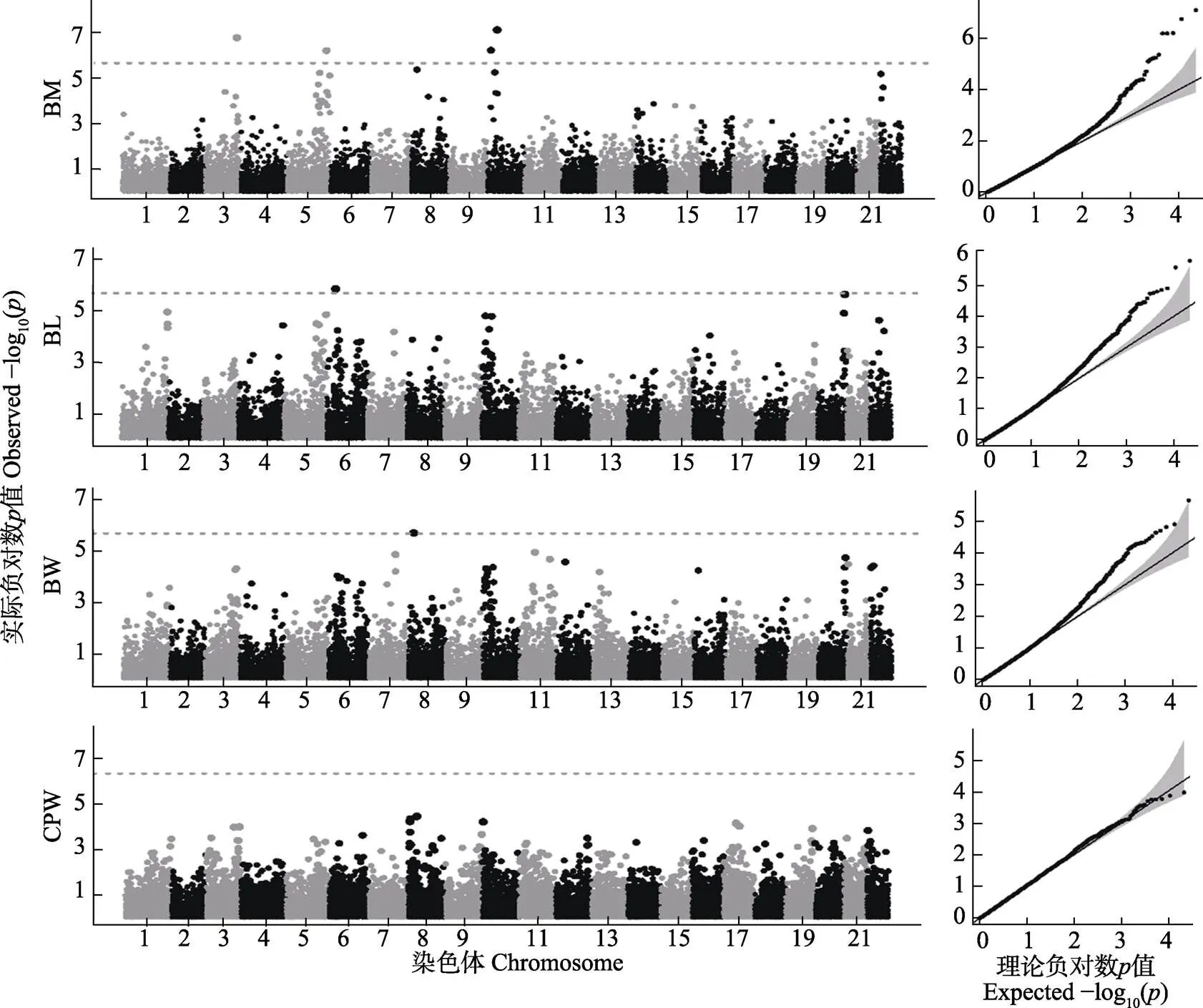
圖3 大菱鲆體重與體尺單一性狀GWAS曼哈頓和QQ圖
表3 大菱鲆體重和體尺性狀顯著關聯的QTNs信息
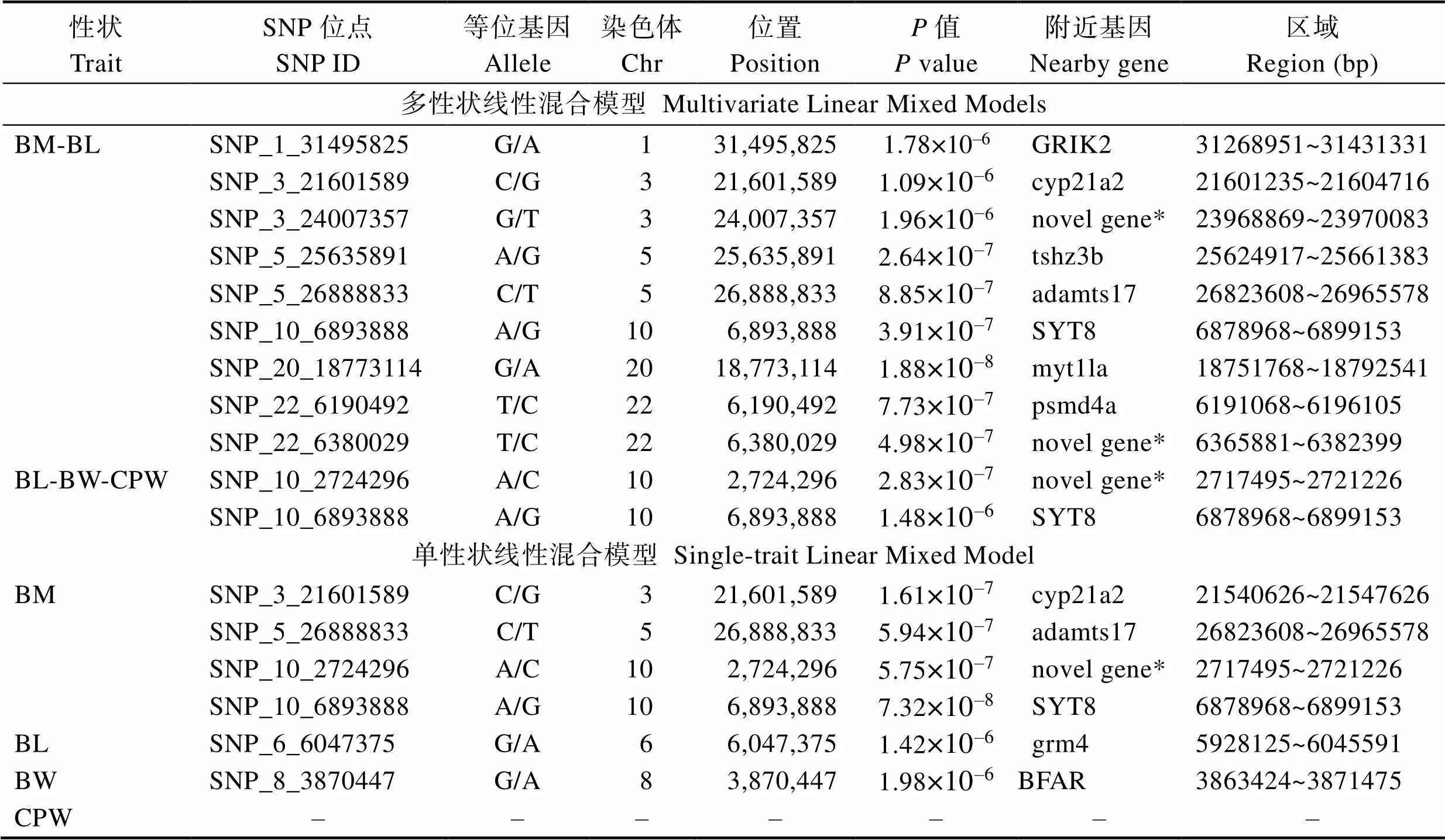
Tab.3 Information of QTNs significantly associated with body mass and morphological traits in turbot
注: *表示未在大菱鲆全基因組中進行功能注釋的新基因
Note: * represents novel gene which has not found any gene function annotation in genome of
2.3 候選基因
以大菱鲆多性狀與單一性狀GWAS獲得的顯著SNP位點為探針,根據其在大菱鲆全基因組的位置,向上下游尋找距離最近的候選基因。在本研究檢測到的17個QTNs附近共找到12個候選基因(表3),分別為GRIK2 (Glutamate receptor, ionotropic, kainate 2)、cyp21a2 (Cytochrome P450, family 21, subfamily A, polypeptide 2)、tshz3b (Teashirt zinc finger homeobox 3b)、adamts17 (ADAM metallopeptidase withthrombo- spondin type 1 motif, 17)、SYT8 (Synaptotagmin 8)、myt1la (Myelin transcription factor 1-like, a)、psmd4a (Proteasome 26S subunit, non-ATPase 4a)、grm4 (Glutamate receptor, metabotropic 4)、BFAR (Bifunctional apoptosis regulator)和3個新基因。對這些候選基因進行GO注釋分析發現,細胞組分主要涉及核膜、細胞質膜及細胞核等;分子功能主要涉及離子通道活性、激素活性、氧化還原酶活性、肽酶活性、核苷酸結合、蛋白結合及G蛋白偶聯受體活性等;而生物學進程則主要涉及離子轉運、糖皮質激素生物合成過程、氧化還原過程、細胞信號轉導、基因表達調控、蛋白酶解、胞外分泌、轉錄調控和G蛋白偶聯受體信號通路等。其中,3個新基因在大菱鲆基因組中未有相關注釋信息。
3 討論
GWAS作為一種定位影響重要經濟性狀分子標記的有效方法,是以全基因組SNPs直接關聯復雜性狀表型,廣泛應用于動植物分子標記開發和輔助選擇育種等方面(Santana, 2015; He, 2017)。隨著諸多水產動物基因組測序工作相繼完成以及測序成本的下降,水產動物生長和抗病等復雜性狀的GWAS分析多有研究報道。Gutierrez等(2015)對大西洋鮭()在性成熟時的生長速率性狀進行GWAS分析,檢測到多個在大西洋鮭代謝和生長發育過程中發揮重要作用的候選基因。Zhou等(2019)利用GWAS揭示了半滑舌鰨()抗病的遺傳機制,共定位了與抗病性狀顯著關聯的33個SNP位點,結合基因表達和甲基化分析,檢測到數個影響半滑舌鰨抗病力的候選基因。Jiang等(2019)則對羅非魚()的耐鹽性狀進行GWAS和QTL定位研究,為羅非魚耐鹽性遺傳機理揭示及進一步的功能分析奠定了基礎。目前,大菱鲆生長和體尺相關性狀的GWAS分析鮮有應用,但QTL定位研究已在大菱鲆生長(Enrique, 2011)和氣單胞菌()耐藥性(Silvia, 2011)等相關性狀中開展。
現有的絕大多數GWAS研究都是關聯全基因組上SNP位點與單個性狀表型值,即使當多個相關表型性狀存在時,往往也會選擇逐個性狀分別進行單一性狀GWAS分析(Stephens, 2013)。在多個相關表型性狀條件下,單一性狀關聯定位方法忽略了QTNs對多個表型性狀的共同影響。相較于單一性狀關聯分析方法,多性狀GWAS方法具有更高的統計功效和參數估計及定位精確度。通過比較大菱鲆體重和體尺多性狀GWAS與各個性狀逐個進行單一性狀GWAS的定位結果發現,在相同的顯著水準下,前者檢測到的QTN數多于后者(表3)。在BM-BL性狀組合的多性狀GWAS分析中,共檢測到9個QTNs,而在相對應的BM和BL單一性狀GWAS分析中,BM中檢測到4個QTNs,BL中僅定位到1個QTN,且BM中檢測到的4個QTNs中有3個與多性狀GWAS分析得到的QTNs重復。同樣,BL-BW-CPW性狀組合多性狀GWAS分析檢測到的QTNs也多于3個性狀各自進行單一性狀GWAS分析得到的QTNs。除了能夠檢測到更多的QTNs,由于多性狀GWAS分析檢測到的QTN是一因多效QTN,相較于單一性狀GWAS分析檢測到的QTN在生物學和實際應用層面更有意義。
在大菱鲆全基因組上尋找距離每個QTN最近的基因,共找到12個候選基因,其中,有9個已知的功能基因及3個新基因。這些影響大菱鲆體重和體尺性狀的候選基因在全基因組分布較為分散,沒有明顯集中區域。本研究為首次對大菱鲆體重和體尺性狀進行全基因組關聯分析,檢測到在大菱鲆相關研究中尚未見報道的顯著的已知功能基因。相關研究表明,GRIK2為離子型受體,它偶聯離子通道并形成受體通道復合物,且與細胞缺血缺氧損傷有關(張冬梅等, 2010)。Eachus等(2017)和Weger等(2018)的研究均表明,cyp21a2在斑馬魚()幼魚的糖皮質激素生物合成中發揮著重要作用。Erickson等(2011)以斑馬魚為實驗動物模型,發現tshz3b可能調控后腦中的Hox功能,而Hox功能則被認為與形態發生和器官形成有關聯(Gair, 2003)。由此可見,該基因可能參與大菱鲆體形態發育過程。Myt1la可能在有絲分裂結束階段發揮重要作用(Nakajima, 2008),BFAR則能與p75NTR的蛋白相互作用并抑制p75NTR信號轉導,而p75NTR可與高親和力受體TrkA協同作用促進細胞增殖或與細胞內配體結合誘導細胞凋亡 (李紅梅等, 2011)。本研究找到的候選基因可能都是大菱鲆體重和體尺性狀的重要候選功能基因,它們在大菱鲆生長發育過程中的影響有待進一步的功能實驗驗證。
Allison DB, Thiel B, St Jean P,. Multiple phenotype modeling in gene-mapping studies of quantitative traits: Power advantages. American Journal of Human Genetics, 1998, 63(4): 1190–1201
Burton PR, Clayton DG, Cardon LR,. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature, 2007, 447(7145): 661–678
Chang CC, Chow CC, Tellier LC,. Second-generation PLINK: Rising to the challenge of larger and richer datasets. GigaScience, 2015, 4(7): 1–16
Chavali S, Barrenas F, Kanduri K,Network properties of human disease genes with pleiotropic effects. BMC Systems Biology, 2010, 4(78): 1–11
Eachus H, Zaucker A, Oakes JA,. Genetic disruption of 21-hydroxylase in zebrafish causes interrenal hyperplasia. Endocrinology, 2017, 158(12): 4165–4173
Enrique SM, Alex C, Miguel AT,. Detection of growth- related QTL in turbot (). BMC Genomics, 2011, 12(1): 473
Erickson T, Pillay LM, Waskiewicz AJ. Zebrafish Tshz3b negatively regulates hox function in the developing hindbrain. Genesis, 2011, 49(9): 725–742
Gair HJC, Lovegrove B. Beyond homeosis-HOX function in morphogenesis and organogenesis. Differentiation, 2003, 71(8): 461–476
在施肥之前,應該結合測土配方施肥結果以及不同整地模式,適當施入農家肥、有機肥和化肥。施肥要堅持以有機肥為主、化肥為輔的施肥原則。此外,確保泥水能夠有效分層,沉淀時間長短應該根據當地水稻田塊的實際情況綜合確定,一般沉淀時間維持在1-3天。沉淀好的秧田,應該確保沉淀不板結,水清澈不渾濁,然后才能栽插秧苗。機械化秧苗殘渣主要采用薄水淺灌,大泥腳深度維持在30 cm以上,水層控制在1-3 cm。
Gutierrez AP, José MY, Fukui S,. Genome-wide association study (GWAS) for growth rate and age at sexual maturation in Atlantic salmon (). PLoS One, 2015, 10(3): e0119730
He JB, Meng S, Zhao TJ,. An innovative procedure of genome-wide association analysis fits studies on germplasm population and plant breeding. Theoretical and Applied Genetics, 2017, 130(11): 2327–2343
Hulata G. Genetic manipulations in aquaculture: A review of stock improvement by classical and modern technologies. Genetica, 2001, 111(1–3): 155–173
Jiang DL, Gu XH, Li BJ,. Identifying a long QTL cluster across chrLG18 associated with salt tolerance in Tilapia using GWAS and QTL-seq. Marine Biotechnology, 2019, 21(2): 250–261
Kim S, Sohn KA, Xing EP. A multivariate regression approach to association analysis of a quantitative trait network. Bioinformatics, 2009, 25(12): 204–212
Klei L, Luca D, Devlin B,. Pleiotropy and principal components of heritability combine to increase power for association analysis. Genetic Epidemiology, 2008, 32(1): 9–19
Lei JL, Ma AJ, Chen C,. The present status and sustainable development of turbot (L.) culture in China. Engineering Sciences, 2005(5): 34–38 [雷霽霖, 馬愛軍, 陳超, 等. 大菱鲆(L.)養殖現狀與可持續發展. 中國工程科學, 2005(5): 34–38]
Lei JL, Men Q, Wang YG,. The factory farming mode of Dalingyu “greenhouse shed + deep well seawater”. Marine Fisheries Research, 2002, 23(4): 1–7 [雷霽霖, 門強, 王印庚, 等. 大菱鲆“溫室大棚+深井海水”工廠化養殖模式. 海洋水產研究, 2002, 23(4): 1–7]
Li HM, Shi HL, Huo KK. p75NTR signal transduction suppressed by BFAR and p75NTR interactions. Scientia Sinica Vitae, 2011, 41(12): 1140–1147 [李紅梅, 施慧莉, 霍克克. BFAR與p75NTR的蛋白相互作用抑制p75NTR信號轉導. 中國科學: 生命科學, 2011, 41(12): 1140–1147]
Li J, Liu YK, Bai L,. Isolation and identification ofassociated with splenic and renal granuloma disease of cultured turbot (). Progress in Fishery Sciences, 2019, 40(5): 195–199 [李杰, 劉耀寬, 白露, 等. 大菱鲆脾腎結節病病原菌的分離和鑒定. 漁業科學進展, 2019, 40(5): 195–199]
Lippert C, Listgarten J, Liu Y,. FaST linear mixed models for genome-wide association studies. Nature Methods, 2011, 8(10): 833–835
Liu XF, Ma AJ, Huang ZH,. Expression characteristics analysis of major QTL candidate genes in response to high temperature stress in turbot (). Journal of Fisheries of China, 2019, 43(6): 1407–1409 [劉曉菲, 馬愛軍, 黃智慧, 等. 大菱鲆高溫脅迫應答主效QTL候選基因的表達特性分析. 水產學報, 2019, 43(6): 1407–1409]
Nakajima H, Yonemura S, Murata M,. Myt1 protein kinase is essential for Golgi and ER assembly during mitotic exit. Journal of Cell Biology, 2008, 181(1): 89–103
Porter HF, O'Reilly PF. Multivariate simulation framework reveals performance of multi-trait GWAS methods. Scientific Reports, 2017, 7: 38837
Price AL, Zaitlen NA, Reich D,. New approaches to population stratification in genome-wide association studies. Nature Reviews Genetics, 2010, 11(7): 459–463
Santana MHA, Ventura RV, Utsunomiya YT,. A genomewide association mapping study using ultrasound-scanned information identifies potential genomic regions and candidate genes affecting carcass traits in Nellore cattle. Journal of Animal Breeding and Genetics, 2015, 132(6): 420–427
Silvia TRR, Toro MA, Bouza C,. QTL detection forresistance related traits in turbot (). BMC Genomics, 2011, 12(1): 541
Stephens M. A unified framework for association analysis with multiple related phenotypes. PLoS One, 2013, 8(7): e65245.
Wang L, Wang YG, Zhang Z,. Diversity and drug resistance of bacterial pathogens isolated from bacterial ascetic disease in cultured turbot. Progress in Fishery Sciences, 2017, 38(4): 17–24 [王嵐, 王印庚, 張正, 等. 養殖大菱鲆()腹水病的病原多樣性及其耐藥性分析. 漁業科學進展, 2017, 38(4): 17–24]
Wang WJ, Hu YL, Ma Y,. High-density genetic linkage mapping in turbot (L.) based on SNP markers and major sex- and growth-related regions detection. PLoS One, 2015, 10(3): e0120410
Weger M, Diotel N, Weger BD,. Expression and activity profiling of the steroidogenic enzymes of glucocorticoid biosynthesis and the FDX1 co-factors in zebrafish. Journal of Neuroendocrinology, 2018, 30(4): e12586
Xu K, Duan W, Xiao J,. Development and application of biological technologies in fish genetic breeding. Science China, Life Sciences, 2015, 58(2): 187–201
Yu JM, Pressoir G, Briggs WH,. A unified mixed-model method for association mapping that accounts for multiple levels of relatedness. Nature Genetics, 2006, 38(2): 203–208
Zhang DM, Wu AM, Lou LX,. Alteration of ion channel gene expression profile in rat model of post-myocardial infarction heart failure. Chinese Journal of Integrated Traditional and West Medicine, 2010, 30(1): 53–57 [張冬梅, 吳愛明, 婁利霞, 等. 心肌梗死后心力衰竭模型大鼠離子通道基因表達譜的變化. 中國中西醫結合雜志, 2010, 30(1): 53–57]
Zhou Q, Su ZC, Li YZ,. Genome-wide association mapping and gene expression analyses reveal genetic mechanisms of disease resistance variations in. Frontiers in Genetics, 2019, 10: 1167
Zhou X, Stephens M. Efficient multivariate linear mixed model algorithms for genome-wide association studies. Nature Methods, 2014, 11(4): 407–409
Zhu W, Zhang H. Why do we test multiple traits in genetic association studies? Journal of Korean Statistics Society, 2009, 38(1): 25–27
Joint Genome-Wide Association Study of Body Mass and Morphological Traits in Turbot ()
GAO Jin1,2, YANG Runqing1,2①
(1. Wuxi Fisheries College, Nanjing Agricultural University, Wuxi 214081; 2. Research Centre for Aquatic Biotechnology, Chinese Academy of Fishery Sciences, Beijing 100141)
To reveal the molecular genetic mechanisms of body mass and morphological traits in turbot () and scan molecular markers and candidate genes, which can be used to improve the target traits, a genome-wide association study (GWAS) was carried out using specific-locus amplified fragment technology (restriction site-associated DNA, 2b-RAD). First, body mass (BM), body length (BL), body width (BW), and caudal peduncle width (CPW) of 441 individuals were measured at about 473 days of growth period in a turbot breeding population. Second, all individuals were genotyped using 2b-RAD, and 23,988 SNPs were obtained after strict quality control. Using a multivariate linear mixed model (mvLMM) for GWAS of traits of BM-BL and BL-BW-CPW, 9 and 2 pleiotropic QTNs were detected for each phenotypic combination, respectively. However, a single-trait linear mixed model (LMM) based on the FaST-LMM algorithm was used for the association analysis of each trait, and the results showed that 4 QTNs were detected in the BM trait, 1 QTN was associated with BL and BW traits, respectively, and no significant locus was found in the CPW trait. A comparison between results of mvLMM and LMM found that mvLMM could detect more QTNs than LMM in GWAS, and the pleiotropic QTNs detected by mvLMM were more biologically meaningful. This study applied mvLMM and LMM to the joint GWAS of body mass and morphological traits in turbot, 17 significant QTNs were detected both using mvLMM and LMM, and 4 of them were detected repeatedly. Furthermore, 12 candidate genes were found by searching the nearest gene of each detected QTN on the whole turbot genome. All of them might be important candidate markers and functional genes, which could influence turbot body mass and morphology. Our study also provided the theory and a reference for marker-assisted selection of body mass and morphological traits in turbot.
Turbot; Body mass and morphological traits; Genome-wide Association Analysis; Multivariate linear mixed models
YANG Runqing, E-mail: runqingyang@cafs.ac.cn
S917.4
A
2095-9869(2021)02-0063-08
10.19663/j.issn2095-9869.20200202001
http://www.yykxjz.cn/
高進, 楊潤清. 大菱鲆體重和體尺性狀聯合GWAS分析. 漁業科學進展, 2021, 42(2): 63–70
Gao J, Yang RQ. Joint genome-wide association study of body mass and morphological traits in turbot (). Progress in Fishery Sciences, 2021, 42(2): 63–70
*中國水產科學研究院基本科研業務費項目“鲆鰈魚分子標記輔助配套選育技術”(2017A001)資助 [This work was supported by the Special Scientific Research Funds for Central Non-Profit Institutes, Chinese Academy of Fishery Sciences (2017A001)].高 進,E-mail: gaojin427@126.com
楊潤清, 研究員, E-mail: runqingyang@cafs.ac.cn
2020-02-02,
2020-02-19
(編輯 馮小花)

