玉米漿發酵產生物丁醇的氨基酸代謝動力學模擬
趙新河,賀壯壯,趙玉斌,馬成業,任曉潔,2
玉米漿發酵產生物丁醇的氨基酸代謝動力學模擬
趙新河1,2,3,賀壯壯1,趙玉斌4,馬成業1,任曉潔1,2※
(1. 山東理工大學農業工程與食品科學學院,淄博 255000;2. 山東理工大學考林臘特列杰微生物脂質國際研究中心,淄博 255000;3. 重慶市科學技術研究院,重慶 401123;4. 魯洲生物科技有限公司,臨沂 276400)
為了深入挖掘利用丙酮丁醇梭菌產生物丁醇過程中氨基酸代謝的動態過程,探究利用廉價氮源玉米漿中的氨基酸用于丙酮丁醇梭菌產生物丁醇的生產策略,尋找生產丁醇的高效率廉價氮源來降低發酵生產成本。該研究首先利用高通量測序技術對玉米漿中微生物多樣性進行分析;同時基于丙酮丁醇梭菌()生產丙酮-丁醇-乙醇(Acetone-Butanol-Ethanol,ABE)碳代謝動態模型的基礎上,構建氨基酸代謝模型,以此模擬15種氨基酸在利用木糖為碳源發酵生產ABE中的氨基酸代謝過程,并對氨基酸的代謝與丙酮丁醇梭菌的生物量以及ABE的合成相關性關系進行冗余分析;通過模型預測實際生產中利用玉米漿發酵時氨基酸的消耗過程。結果表明,梭狀芽胞桿菌屬()占細菌總數的68.76%,是玉米漿中的優勢菌群;最佳參數校正后構建了氨基酸代謝模型,模擬值與試驗值有較好擬合度;11種氨基酸(苯丙氨酸、蘇氨酸、異亮氨酸、亮氨酸、蛋氨酸、纈氨酸、酪氨酸、甘氨酸、絲氨酸、精氨酸、天冬酰胺)在培養過程中迅速消耗用于細胞生長和溶劑生成,3種氨基酸(脯氨酸、組氨酸、天冬氨酸)保持穩定狀態,同時發酵過程中谷氨酰胺積累;冗余分析表明其中5種氨基酸對發酵產物及生物量影響具有相關性,相關性排序從大到小依次為絲氨酸、甘氨酸、亮氨酸、纈氨酸、天冬酰胺;模擬預測玉米漿中纈氨酸、甘氨酸、絲氨酸在發酵過程中基本被消耗,推測其為發酵后期的營養限制性因子。該結論可證實玉米漿可作為丙酮丁醇梭菌發酵丁醇的優勢氮源,為丙酮丁醇梭菌的氨基酸代謝調控及下一步利用并優化玉米漿作為氮源生產生物丁醇提供一定的理論參考和數據支撐。
動力學;模型;丙酮丁醇梭菌;ABE發酵;玉米漿;微生物組分析;氨基酸
0 引 言
近年來,微生物發酵生產生物燃料已成為當前能源研究熱點。而丁醇作為新一代生物燃料,具有能量密度大、燃值高等多種優良特性[1]。梭菌是工業丙酮-丁醇-乙醇(Acetone-Butanol-Ethanol,ABE)發酵模式的主要菌株[2]。然而高成本和低利用率限制了生物丁醇的產業化發展[3]。因此,為了降低生物丁醇生產成本,迫切需要開發及優化更多廉價易降解的原料用于ABE發酵丁醇[4-5]。
農產品加工產業的快速發展,造成眾多農產品加工廢棄物堆棄而未能得到有效的利用。玉米漿(Corn Steep Liquor,CSL)是玉米淀粉加工的副產物,富含多種氨基酸、可溶性糖、礦物質等營養物質,常作為發酵工業的替代氮源[6]。此前有大量研究致力于玉米漿的資源化利用如青霉素合成、生物飼料生產等[6-7]。但玉米漿酸度高、霉菌毒素易超標等問題限制了其在食品生產中高值化的利用。然而,玉米漿作為氮源用于丙酮丁醇梭菌生產ABE,可以為發酵過程提供充足的氨基酸等速效氮源和促營養因子,不但可以有效降低原料成本,也為玉米漿的應用開拓了市場。Zhang等[8]以玉米加工廢棄物(玉米芯水解物為碳源,玉米漿為氮源)用北耶林氏梭狀芽孢桿菌SE-2生產丁醇,優化培養基成分得到總ABE濃度為19.22 g /L,丁醇濃度可達11.65 g /L,比初始培養基高42%。Maddipati等[9]以低成本營養源玉米漿代替酵母抽提液,利用梭狀芽孢桿菌菌株P11發酵生產濃度1.7 g/L的生物乙醇。以上研究均證明了農產品廢棄物玉米漿作為發酵原料的巨大潛力。
微生物動態代謝模型可以模擬微生物對原料中各種營養元素的利用及代謝過程,因此,可以更加精確預測培養條件和營養需求,實現原料的高效利用。Zhao等[10]以中心碳代謝為基礎構建了丙酮丁醇梭菌的ABE發酵模型,該模型模擬了碳源通過糖酵解、戊糖磷酸途徑(Pentose-Phosphate Pathway,PPP)等合成有機酸和溶劑(ABE)的動態代謝過程。該模型引入了與ABE發酵密切相關的丁醇抑制和醋酸鹽激活等代謝調節因子,精確展現了碳源在ABE發酵過程中的代謝流分配及變化。然而該模型并未涉及氮源代謝及其動態分析,例如氨基酸等代謝相關的途徑,以及氮源與溶劑合成的相關性關系。因此為本研究提供了深入挖掘ABE發酵過程中氮源動態代謝過程的思路。
本研究首先通過高通量測序技術對玉米漿中的原始微生物菌群進行了多樣性分析,并在丙酮丁醇梭菌ABE發酵碳代謝模型的基礎上,構建氨基酸代謝模型,以此模擬玉米漿作為氮源生產生物丁醇的氨基酸代謝消耗過程。本研究為廢棄玉米漿為底物發酵生物丁醇提供了優化策略,同時以自然界中廣泛存在的半纖維素的水解物作為碳源進行研究,以期為利用玉米漿—半纖維素水解物(木糖)為碳氮源生產丁醇提供參考。
1 材料與方法
1.1 菌種與培養
研究菌種為丙酮丁醇梭菌ATCC 824,該菌株被認為是具有高產溶劑能力且基因組測序完整的經典種,溶劑產量可達到11~17 g/L[11-14]。使用增強梭狀芽孢桿菌培養基(Reinforced Clostridia Medium,RCM)作為種子培養基[15]。采用改良梭狀芽孢桿菌生長培養基(Modified Clostridia Growth Medium,CGM)作為發酵培養基[16],并在CGM發酵培養基中加入5 g/L CaCO3作為pH緩沖劑。在3.6 L生物反應器(瑞士Infors-HT公司)中分批培養,以5%體積分數接種量接種種子液(37℃血清瓶中厭氧培養),噴入100%氮氣,保證厭氧條件,100 r/min,37 ℃培養。
1.2 氨基酸的測定
氨基酸測定主要針對丙酮丁醇梭菌在CGM發酵培養基中培養時各取樣點的氨基酸含量以及玉米漿中氨基酸的種類及含量測定。通過HPLC/MS/MS系統(1290 超高壓液相色譜/6460 三重串聯四極桿液質聯用儀,美國Agilent科技公司)對上清液進行氨基酸分析。具體操作步驟參考Zhao等[10]的研究。
1.3 玉米漿微生物多樣性分析
玉米漿(Corn Steep Liquor,CSL)由魯洲生物科技有限公司提供。委托上海生工生物有限公司對玉米漿樣品進行預處理并進行16SrDNA高通量測序。基因組DNA提取使用Mag-Bind Soil DNA分離試劑盒(美國Omega Bio-tek公司),按照制造商的說明進行。CSL樣品提取一式三份。每個樣本用瓊脂糖凝膠電泳檢測DNA完整性。使用Qubit2.0 DNA檢測試劑盒對基因組DNA進行精確定量。引物341F(CCTACGGGNGGCWGCAG)和805R(GACTACHVGGGTATCTAATCC)用于細菌16SrDNA基因的擴增,引物ITS1F(CTTGGTCATTTAGAGGAAGTAA)和ITS2R(GCTGCGTTCTTCATCGATGC)用于真菌基因擴增。高通量測序使用Illumina Miseq?平臺進行。
1.4 動態氮源代謝模型建立
基于Zhao等[10]建立的丙酮丁醇梭菌ABE發酵的碳動態代謝模型,增加氨基酸代謝網絡,用以描述玉米漿為底物的氨基酸動態代謝過程。用Michaelis-Menten方程描述了各通量的動力學和細胞的比生長速率。化學反應計量關系、代謝通量動力學方程和質量平衡方程參考Zhao等[10]的研究確定。根據Rizzi等[17]提出的方法確定模型結構和動力學參數,并通過最小二乘法優化模擬結果。通過定義試驗值與模擬值之間殘差的加權平方和為目標函數,在參數變化范圍內優化函數最小值得到參數值。根據在MATLAB中輸入模型的參數值(見表1)和代謝物初始值(見表2)進行動態代謝模型模擬得到氨基酸消耗模擬曲線。運用MATLAB R2018b進行動態代謝模型模擬及數據分析,運用Canoco5對數據進行相關性分析。
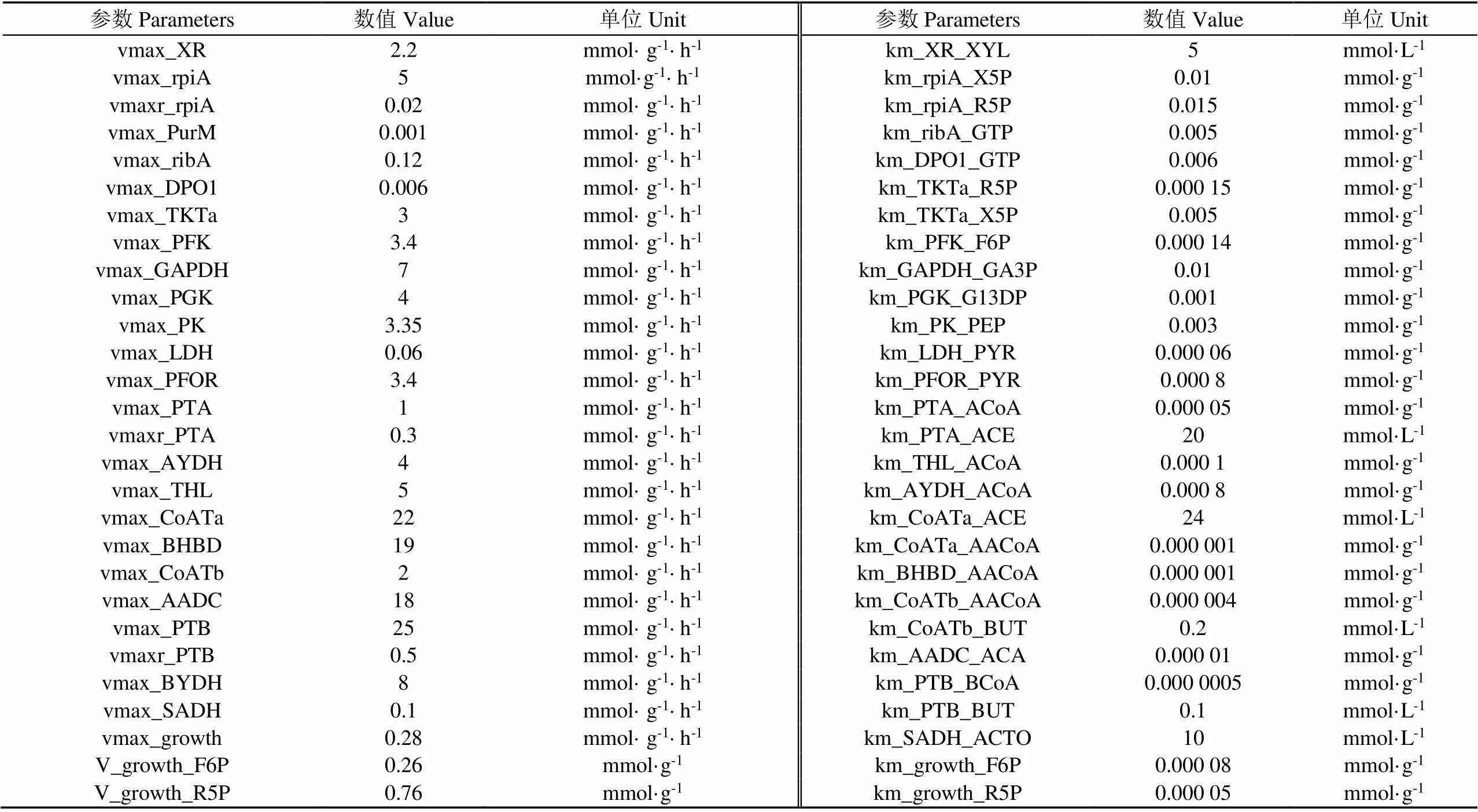
表1 氨基酸代謝模型參數值
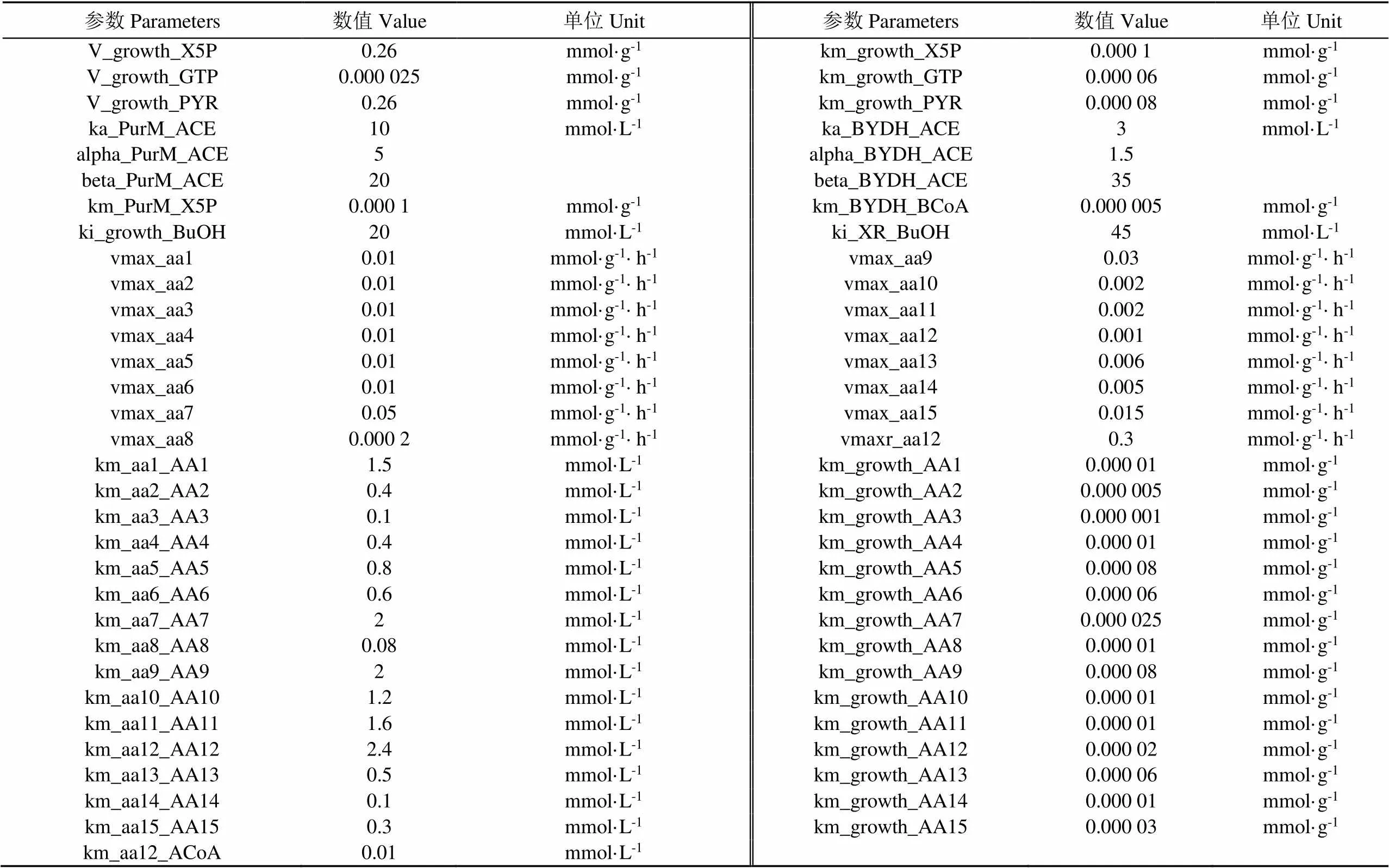
續表
注:XR:木糖還原酶;rpiA :核糖5-磷酸異構酶A;PurM:嘌呤代謝;ribA:環化水解酶II; DPO1:二磷酸核苷激酶;TKTa:轉酮醇酶;PFK:磷酸果糖激酶;GAPDH:甘油醛-3-磷酸脫氫酶;PGK:磷酸甘油酸激酶;PK:丙酮酸激酶;LDH:乳酸脫氫酶;PFOR:鐵氧還蛋白氧化還原酶;PTA:磷酸轉乙酰酶;AYDH:乙醛脫氫酶;THL:硫解酶;CoATa:乙酰基-乙酰輔酶A-乙酸-轉移輔酶A;CoATb:乙酰基-乙酰輔酶A-丁酸-轉移輔酶A;AADC:乙酰乙酰輔酶A;SADH:二級醇脫氫酶;PTB:磷酸轉丁酰酶;BHBD:β-羥丁酰-脫氫輔酶A;BYDH:丁醛脫氫酶;XYL:木糖;X5P:5-磷酸木酮糖;GTP:三磷酸鳥苷;R5P:5-磷酸核糖;RIBO:核黃素;F6P:6 -磷酸果糖;GA3P:3-磷酸甘油醛;G13DP:甘油酸-1,3-二磷酸;PRPO:丙醇;PEP:磷酸烯醇丙酮酸;PYR:丙酮酸;LAC:乳酸;ACoA:乙酰輔酶A;EtOH:乙醇;AACoA:乙酰乙酰輔酶A;BUT:丁酸;ACTO:丙酮;ACA:乙酰乙酸;ACE:乙酸;BCoA:丁酰輔酶A;BuOH:丁醇;AA1:蘇氨酸;AA2:甘氨酸;AA3:絲氨酸;AA4:苯丙氨酸;AA5:酪氨酸;AA6:亮氨酸;AA7:天冬酰胺;AA8:天冬氨酸;AA9:精氨酸;AA10:組氨酸;AA11:脯氨酸;AA12:谷氨酰胺;AA13:蛋氨酸;AA14:異亮氨酸;AA15:纈氨酸。小寫字母“aa”代表相應氨基酸酶促反應,下同。
Note: XR: xylose reductase; rpiA: ribose 5-phosphate isomerase A; PurM: purine metabolism; ribA: GTP cyclohydrolase II; DPO1: nucleoside-diphosphate kinase; TKTa: transketolase; PFK:phosphofructokinase; GAPDH: glyceraldehyde-3-phosphate dehydrogenase; PGK: phosphoglycerate kinase; PK:pyruvate kinase; LDH:lactate dehydrogenase; PFOR: pyruvate-ferredoxin oxidoreductase; PTA: phosphotransacetylase; AYDH: acetaldehyde dehydrogenase; THL: thiolase; CoATa: acetoacetyl-CoA-acetate-CoA-transferase; CoATb:acetoacetyl-CoA-butyrate-CoA-transferase; AADC: acetoacetate decarboxylase; SADH: secondary alcohol dehydrogenase; PTB: phosphotransbutyrylase; BHBD: β-hydroxybutyryl-CoA dehydrogenase; BYDH: butyraldehyde dehydrogenase; XYL: xylose; X5P: xylulose 5-phosphate; GTP: guanosine triphosphate; R5P: ribose 5-phosphate; RIBO: riboflavin; F6P fructose 6-phosphate; GA3P: glyceraldehyde 3-phosphate; G13DP: glycerate-1,3-bisphosphate; PRPO:propanol; PEP: phosphoenolpyruvate; PYR: pyruvate; LAC: lactate; ACoA: acetyl coenzyme A; EtOH : ethanol; AACoA: acetoacetyl coenzyme A; BUT: butyrate; ACTO: acetone; ACA: acetoacetate; ACE: Acetic acid; BCoA: butyryl coenzyme A; BuOH: butanol; AA1: Threonine; AA2: Glycine; AA3: Serine; AA4: Phenylalanine; AA5: Tyrosine; AA6: leucine; AA7: Asparagine; AA8:Aspartic acid; AA9: Arginine; AA10: Histidine;AA11: Proline; AA12: Glutamine; AA13: Methionine; AA14: Isoleucine; AA15: Valine. The lowercase letter “aa” represents the enzymatic reaction of corresponding amino acids, the same as below.
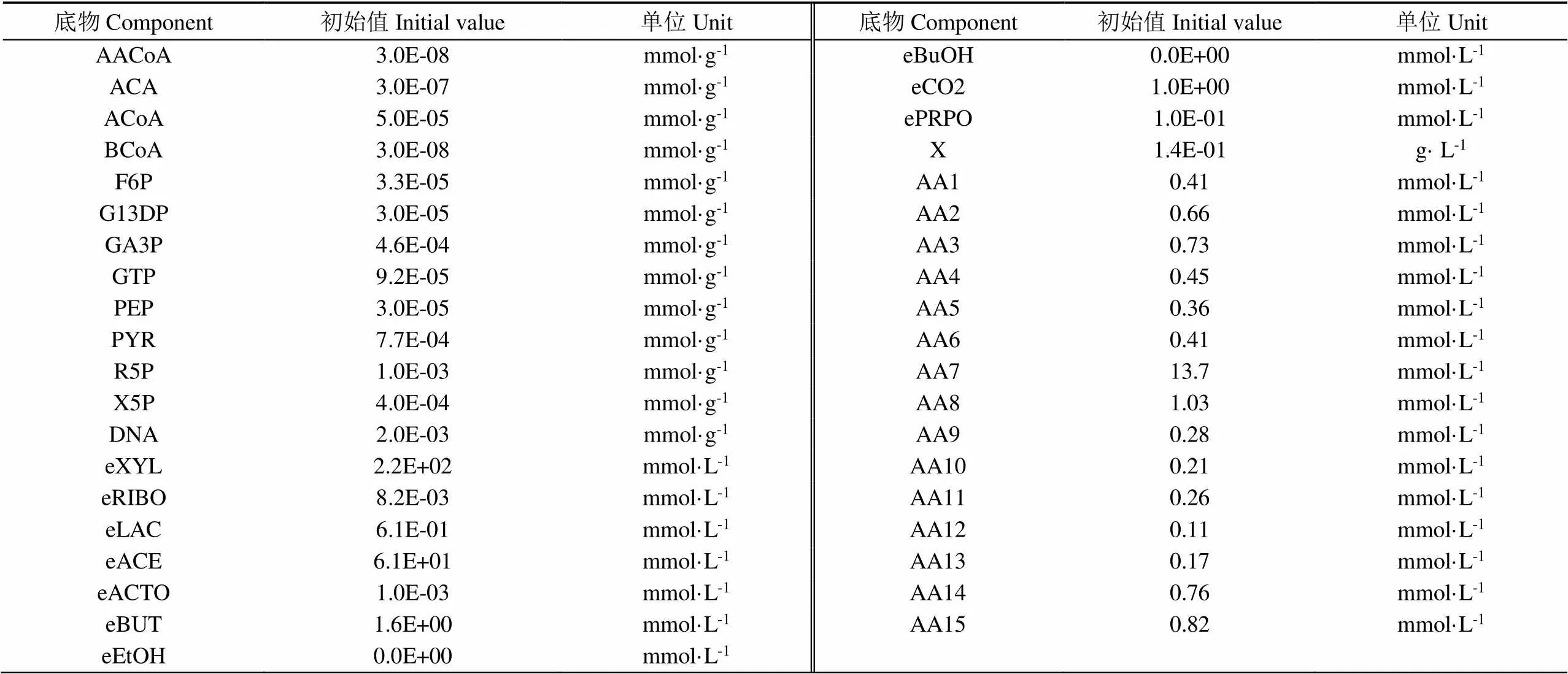
表2 氨基酸代謝模型變量初值
2 結果與分析
2.1 玉米漿微生物多樣性分析
目前并沒有研究報道對玉米漿中微生物多樣性的分析。為了深入了解玉米漿中原始微生物的群落種類與豐度,采用高通量測序技術對玉米漿中原始微生物多樣性進行分析。利用核糖體數據庫工程(Ribosome Database Project,RDP)分類器對每個樣本序列進行分析,將玉米漿中原始菌群的物種信息鑒定在屬水平,并對各菌屬的相對豐度進行了統計(圖 1)。利用ITSrDNA對玉米漿中的真菌進行擴增和測序,結果顯示,真菌目標區域擴增后無明顯條帶,表明玉米漿中真菌群落豐度極低。此外,通過16srDNA技術檢測玉米漿中細菌的豐度及多樣性,結果顯示,玉米漿中的細菌要比真菌豐富得多。其中玉米漿中各主要的細菌菌屬如梭狀芽孢桿菌屬()(68.76%)、芽孢桿菌屬()(3.98%)、短波單胞菌屬()(2.53%)、假單胞菌屬()(1.6%)、乳桿菌屬()(1.45%)、類芽孢桿菌屬()(1.42%)、賴氨酸芽胞桿菌屬()(1.41%)占到所有原始菌屬的70%以上。從各菌屬的占比中可以看出,梭狀芽胞桿菌是玉米漿原始菌群中的絕對優勢菌屬。絕大多數梭狀芽孢桿菌是嚴格厭氧微生物,能夠分解碳水化合物,可以通過Stickland反應分解多種氨基酸,并且可以耐受較高的滲透壓,對不良環境具有極強的抵抗力[18-19]。玉米漿中營養成分、滲透壓等原始環境,更適合梭狀芽孢桿菌等微生物定殖,這對玉米漿在發酵領域的資源化利用提供了理論參考。
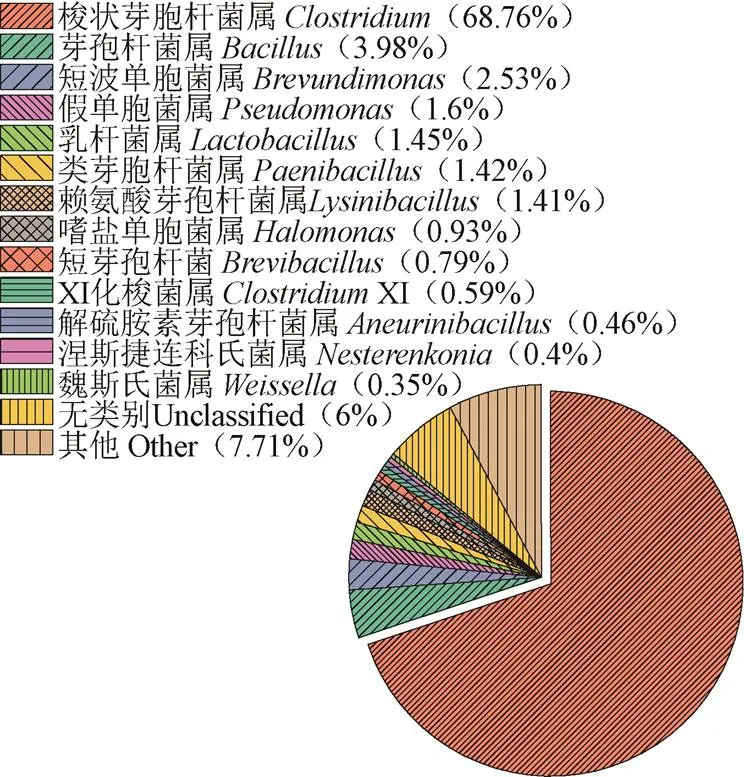
圖1 玉米漿微生物群落豐度餅圖
同時對玉米漿中原始菌群構建了進化樹和相似性分析,并將玉米漿中菌群基因的功能分類按總體豐度進行分布,如圖2所示。圖2a選取總體豐度最高的前50個OUT(操作單元分類)簇的代表性序列和與其相近的數據庫序列在屬水平上的進化樹結果,結果顯示玉米漿原始菌群可分為兩大簇,其中一簇菌屬數量較多,另一簇只有幾株菌,為賴氨酸芽孢桿菌屬和枝芽胞桿菌屬,兩種菌屬親緣關系較近,其與其他菌屬親緣關系較遠。另外梭狀芽孢桿菌屬多種亞種均在系統進化樹的同一大簇,而且與芽孢桿菌科其他菌屬在系統進化樹進化關系上較親近。因此它們在玉米漿中可以呈現較好的共生狀態。

圖2 玉米漿中原始菌群構建的相似進化樹和基因功能分類
圖2b為玉米漿原始菌群基因的功能分類分布,按總體豐度排序。從圖中可以看出,特征注釋的結果分為25個類別。其中大部分的功能都屬于管家基因的功能,例如:[R]一般功能預測、[K]轉錄、[T]信號轉導機制、[M]細胞壁結構、[D]細胞分裂周期控制、[L]核酸復制修復、[V]細胞防御、[O]蛋白分泌、[A]轉錄后修飾、[P][H]各種輔酶及離子的轉運、[C]能量的合成與轉化等。而與代謝相關的功能有只有[E]氨基酸轉運與代謝、[G]碳水化合物轉運與代謝、[I]油脂代謝及[Q]少量次級代謝產物合成等。此外,還有大量功能未知基因待進一步研究。這說明玉米漿中的原始菌群功能主要集中在對碳、氮源的轉化,這與玉米漿中豐富的氨基酸以及可溶性糖含量可能有一定關系。經測定,玉米漿中含有4%的葡萄糖及50%以上的氨基酸,這進一步說明作為優勢群體的梭狀芽孢桿菌對糖類及氨基酸類營養物質具有較高的利用效率。為下一步選擇玉米漿混菌發酵的菌株提供了理論依據。
2.2 氨基酸代謝通量分析
通過對玉米漿中原始菌群的多樣性分析發現梭狀芽孢桿菌是優勢菌,它屬于梭菌屬,該屬的細菌通常是嚴格的厭氧桿菌,能夠在高效利用碳氮源的同時產生乙酸、丁酸、丙酮、乙醇、丁醇等小分子有機酸和醇類[20]。玉米漿中富含多種氨基酸,其含氮量能達到干質量的4.7%。常作為優質發酵氮源。因此,預測梭菌屬微生物對玉米漿中氨基酸的利用效率將對指導玉米漿發酵提供參考與數據支撐。此前Zhao等人利用屬中的丙酮丁醇梭菌發酵生產丙酮-乙醇-丁醇(ABE),并建立了該菌的碳代謝模型,本研究在此基礎上,進一步檢測了該菌在GCM發酵培養基中對氨基酸的消耗情況,并進一步建立和完善了其對氨基酸的氮代謝動力學模型,用于指導利用玉米漿發酵ABE過程中的氨基酸及糖的代謝研究。15種氨基酸在細胞中的代謝途徑如圖 3所示[21]。
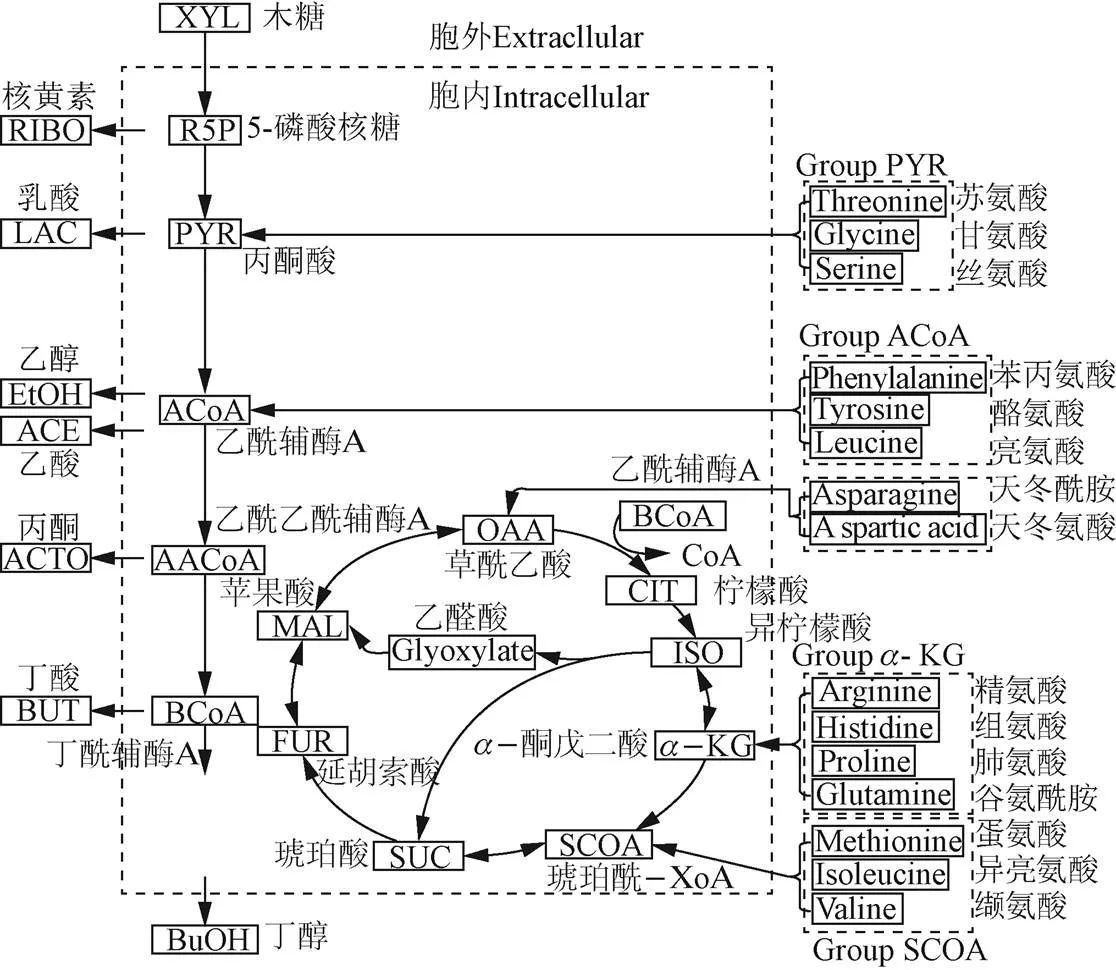
圖3 丁醇梭菌15種氨基酸主要代謝路徑
圖4模擬了丙酮丁醇梭菌培養過程中對15種氨基酸的消耗情況。通過定義試驗值與模擬值之間殘差的加權平方和為目標函數,在參數變化范圍內優化函數最小值得到參數值。參數校正結果顯示,模型模擬曲線與試驗測量值基本吻合,證明建立的氨基酸代謝模型能夠較好地反映培養基中氨基酸的動態變化。結果顯示,其中11種氨基酸(苯丙氨酸、蘇氨酸、異亮氨酸、亮氨酸、蛋氨酸、纈氨酸、酪氨酸、甘氨酸、絲氨酸、精氨酸、天冬酰胺)在代謝過程中消耗較快,尤其在10~30 h氨基酸消耗最快,在50 h后趨于穩定。而另外3種氨基酸(脯氨酸、組氨酸、天冬氨酸)基本不被梭菌利用,其濃度保持在初始值附近。值得注意的是,谷氨酰胺濃度升高,其在0~80 h時從初始的(0.12 ± 0.003)mmol/L積累到(0.24 ± 0.04)mmol/L(圖4l),這與絕大多數氨基酸被消耗的趨勢截然不同。谷氨酰胺的積累說明該菌在合成ABE過程中伴隨著谷氨酰胺的合成。在目前谷氨酰胺合成途徑的研究中發現,谷氨酸在谷氨酰胺酶的催化作用下,被轉化為谷氨酰胺[22]。同時多種氨基酸如天冬酰胺、苯丙氨酸和酪氨酸可通過檸檬酸循環中α-酮戊二酸產生谷氨酸。因此,積累的谷氨酰胺可能是從其他降解的氨基酸轉化而來。同時,有研究發現丁醇會影響細胞膜的流動性和組成,導致細胞膜通透性增加[23],因此30 h后丁醇的積累增加了胞內合成的谷氨酰胺排出胞外的速率。此外,Fonknechten等[21]研究發現梭狀芽胞桿菌()在消耗甘氨酸、絲氨酸、精氨酸和蘇氨酸的同時也有谷氨酸、天門冬氨酸和丙氨酸等氨基酸的積累。
2.3 氨基酸代謝與產物生成通量分析
為了進一步分析丙酮丁醇梭菌氨基酸代謝與產物合成間的關系,以各氨基酸消耗與產物生成比值來表征二者的聯系(圖5)。結果顯示,各氨基酸對乙醇消耗生成比明顯高于對丙酮和丁醇的消耗生成比,這主要是因為培養過程中乙醇生成速率明顯低于其他兩種溶劑。這說明氨基酸合成乙醇的效率相對較低。結果表明,9種氨基酸(甘氨酸、絲氨酸、苯丙氨酸、酪氨酸、亮氨酸、精氨酸、蛋氨酸、纈氨酸、異亮氨酸)消耗速率與3種溶劑產出速率的比率隨著培養時間先增加后減少,除異亮氨酸在60 h比率達到最高值外,其余均在40 h達到最高值。這是由于在0~40 h各氨基酸消耗速率升高,40 h后消耗速率減慢甚至趨近停止,而溶劑在40 h后生成速率升高,開始大量積累,導致呈下降趨勢。尤其是絲氨酸、纈氨酸、苯丙氨酸、亮氨酸、甘氨酸比率明顯高于其他氨基酸,這是因為它們在梭菌從產酸階段轉向產溶劑階段的生產過程中氨基酸被迅速消耗。Amador-Noguez 等[24]研究表明,在梭菌從產酸階段轉向產溶劑階段過程中氨基酸被迅速消耗,尤其是以絲氨酸、亮氨酸、異亮氨酸、纈氨酸變化最明顯,這與本研究的結果一致。后期各氨基酸消耗緩慢,這是由于細胞在從產酸向產溶劑轉變過程中誘導了溶劑合成基因上調[25],使得胞內糖酵解、TCA循環和氨基酸合成過程受到顯著影響,減少了還原性TCA循環和氨基酸合成過程,使得三磷酸腺苷(Adenosine triphosphate,ATP)和還原型煙酰胺腺嘌呤二核苷酸磷酸(Nicotinamide Adenine Dinucleotide Phosphate,NADPH)更多地用于丁醇合成[24]。其他氨基酸(天冬氨酸、組織胺、脯氨酸)與溶劑的比率曲線均保持穩定的狀態。另外可以看出苯丙氨酸與溶劑濃度比值的曲線拐點時間略早于其他氨基酸,這說明苯丙氨酸代謝較快,所以很快進入了較低的濃度水平。苯丙氨酸屬于芳香性氨基酸,大量研究表明其更有利于進入細胞積累,消耗更快,因此其比值拐點時間較早于其他氨基酸,但容易導致溶劑生產滯后[26-27]。
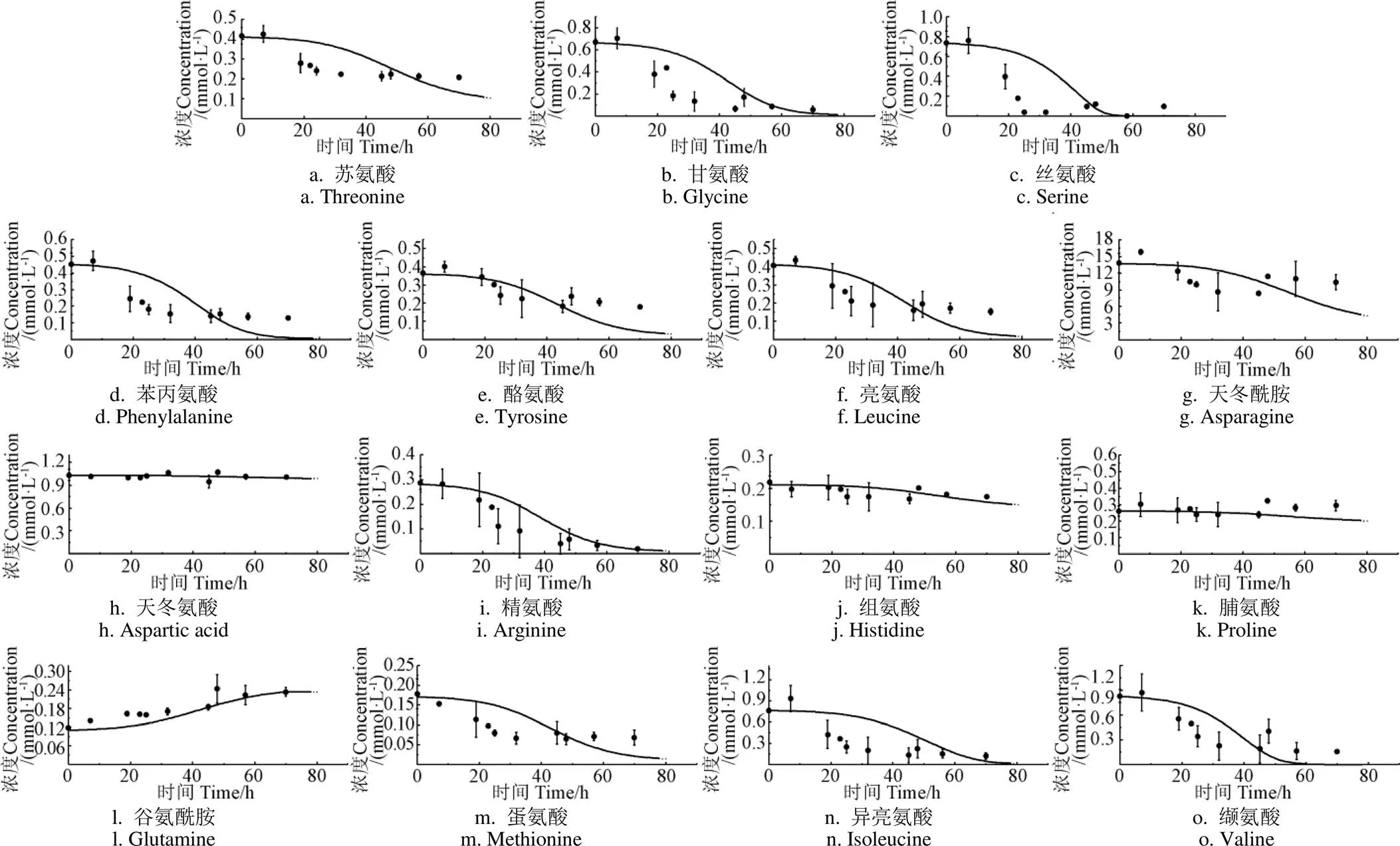
注:實心圓點為氨基酸濃度的試驗測定值;實線為氨基酸濃度的模擬值。
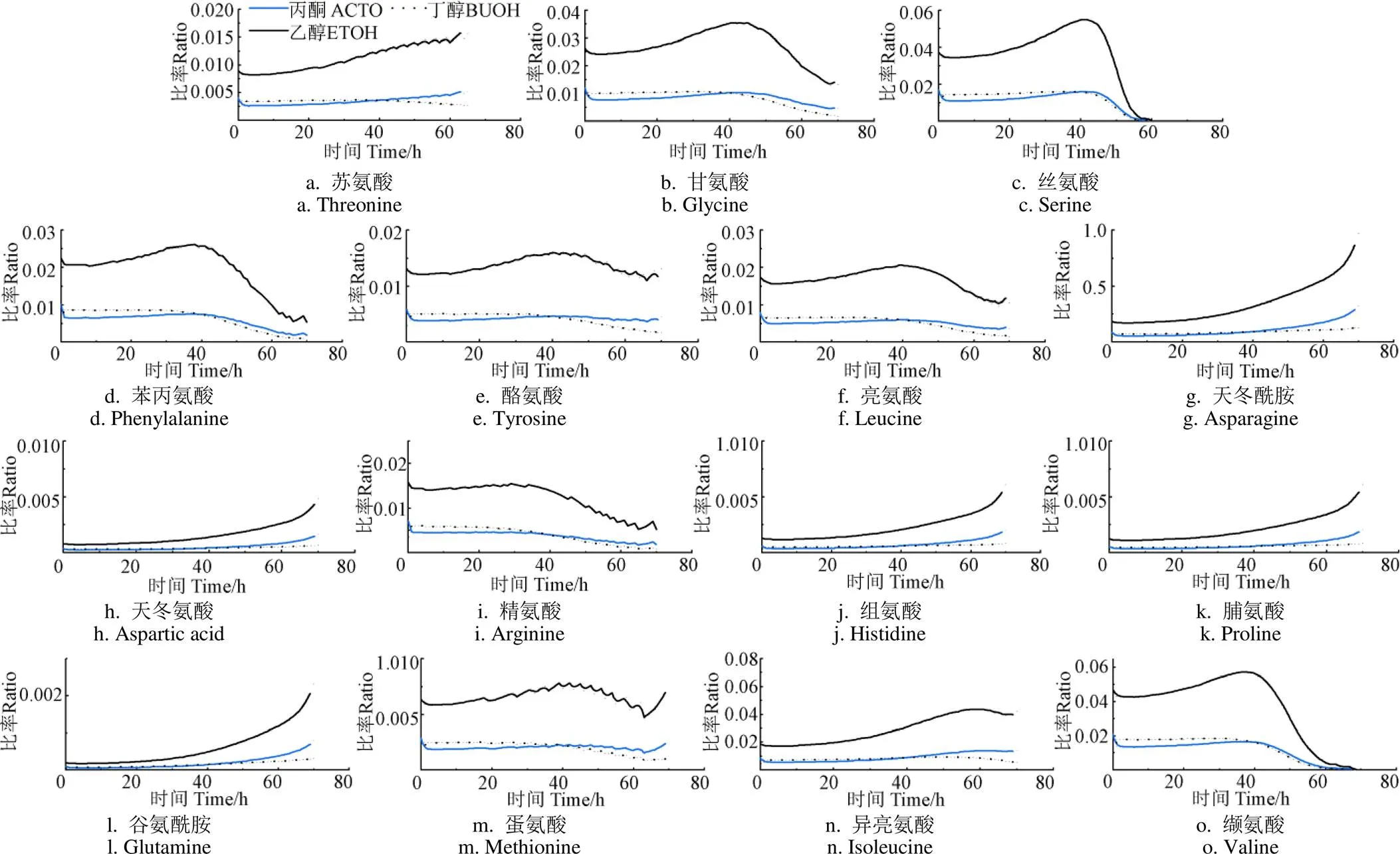
注:深色實線為各氨基酸濃度對乙醇濃度比率的模擬結果;淺色實線為各氨基酸濃度對丙酮濃度比率的模擬結果;深色虛線為各氨基酸濃度對丁醇濃度比率的模擬結果。
在丙酮丁醇梭菌中,除了代謝產生乙酸、丙酸、丁酸及相應的溶劑乙醇、丙酮及丁醇之外,生物量的積累及核黃素的合成也消耗了大量的碳、氮源。圖6表示了各氨基酸濃度與生物量及核黃素濃度之間的比率。初始時各氨基酸與核黃素濃度比率曲線呈現斷崖式下降,這是由于培養初期培養基中核黃素生成速率低,處于延滯狀態,導致比率過高。6種氨基酸(甘氨酸、絲氨酸、苯丙氨酸、亮氨酸、精氨酸、纈氨酸)代謝速率與生長速率的比率總體上隨著培養時間先增加后減少,在50 h出現峰值,這是由于這幾種氨基酸在0~50 h消耗速率增大,50 h后氨基酸基本不消耗,而此時生物量達到最大,達到生長穩定期。因此50 h比率最高,50 h后生物量呈現負增長,導致比率曲線下降。其他氨基酸培養過程中消耗速率很低,而細胞量迅速增長,所以曲線呈增長狀態。前期糖代謝和氨基酸代謝有利于細胞生長,但不利于丁醇合成[28]。氨基酸與生物量比率變化程度最大的是絲氨酸、甘氨酸和纈氨酸。Liu等[29]研究發現添加氨基酸,尤其是甘氨酸與絲氨酸能明顯促進細胞密度增長,而添加組氨酸并不能促進細胞生長或丁醇的生成,推測是由于最終產物丁醇對磷酸戊糖途徑的抑制作用,這與本研究的結果一致。在50 h丁醇生產速率最高,有研究報道丁醇對細胞有一定毒性,溶劑濃度達到抑制水平導致后期細胞裂解[30]。另外丙酮丁醇梭菌內自溶酶在指數期結束后達到最大值,后期菌體自溶,釋放氨基酸和還原力[31],導致氨基酸消耗減少,溶劑生產減少導致部分氨基酸比率呈迅速下降趨勢。Nimbalkar等[32]研究表明纈氨酸和精氨酸組合以及鳥氨酸和苯丙氨酸作為良好的氮源,可以促進細胞生長,提高ABE產量。原因可能是精氨酸通過瓜氨酸快速代謝成鳥氨酸,從而轉化為能量ATP[21,33];另一個原因可能是精氨酸促進了醋酸鹽的形成,進而致使丙酮產量增加[34]。同時這幾種氨基酸消耗速率與核黃素生成速率在50 h時呈現急劇下降的趨勢,這是由于各氨基酸在50 h時消耗速率迅速減低,甚至不被消耗,而核黃素生長速率達到最高,導致比率曲線迅速下降。
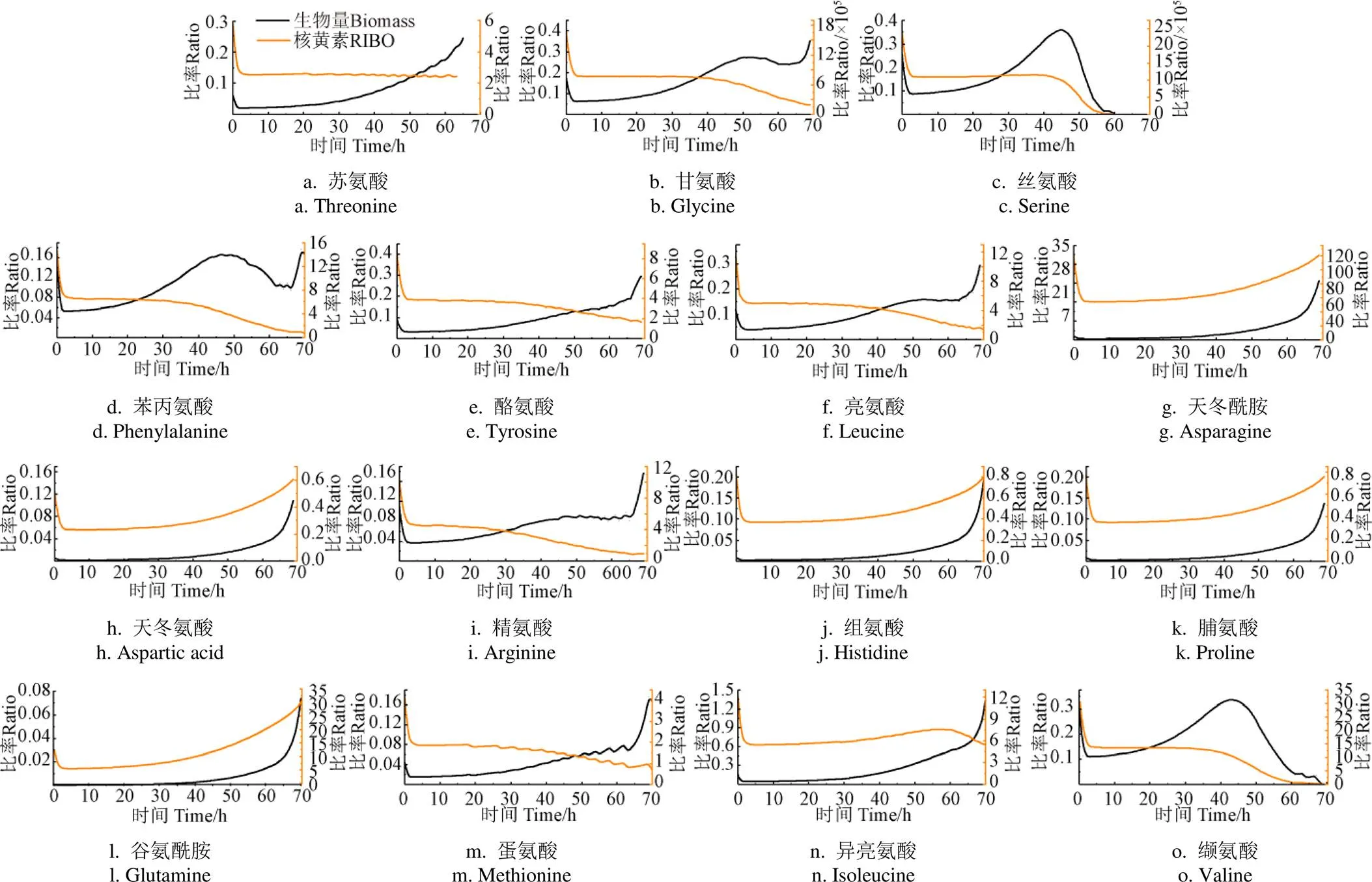
注:淺色實線為各氨基酸濃度對核黃素濃度比率的模擬結果;深色實線為各氨基酸濃度對生物量比率的模擬結果。
2.4 培養過程中氨基酸與合成產物的相關性分析
為了進一步分析氨基酸的代謝與丙酮丁醇梭菌生物量以及ABE、核黃素合成的相關性關系,以各時間段試驗數據對15種氨基酸和產物進行冗余分析,結果如圖7所示。2個排序軸解釋了各氨基酸與各產物的累積變化率分別為95.23%和3.19%,2個排序軸共同解釋了98.42%的樣本信息,說明生物量、溶劑生成、核黃素合成與15種氨基酸中的5種氨基酸(絲氨酸、天冬酰胺、甘氨酸、亮氨酸、纈氨酸)的代謝消耗有關。其中亮氨酸對結果影響解釋度為64.4%,可以認為是對產物影響的主要氨基酸。從圖中可以看出,5種氨基酸對產物影響相關性排序從大到小依次為絲氨酸、甘氨酸、亮氨酸、纈氨酸、天冬酰胺;生物量與各產物對氨基酸消耗影響相關性排序從大到小依次為生物量、丙酮、乙醇、丁醇、核黃素。以上5種氨基酸向量與丁酸向量趨近垂直,說明氨基酸代謝與丁酸合成相關性低。丁酸向量與生物量和丙酮向量趨近垂直,與乙醇、丁醇、核黃素向量夾角呈鈍角,說明丁酸與生物量、丙酮合成相關性較低,與乙醇、丁醇、核黃素合成相關性較高,這與丁酸代謝產生丁醇的代謝理論相對應。乙酸鹽向量與所有產物向量夾角均呈鈍角,說明乙酸鹽有利于產物形成,尤其是與核黃素向量夾角趨近180°,說明乙酸鹽存在可以促進核黃素的合成。這也驗證了Zhao等[10]前期工作中添加乙酸鈉能夠刺激核黃素的產生的結論。
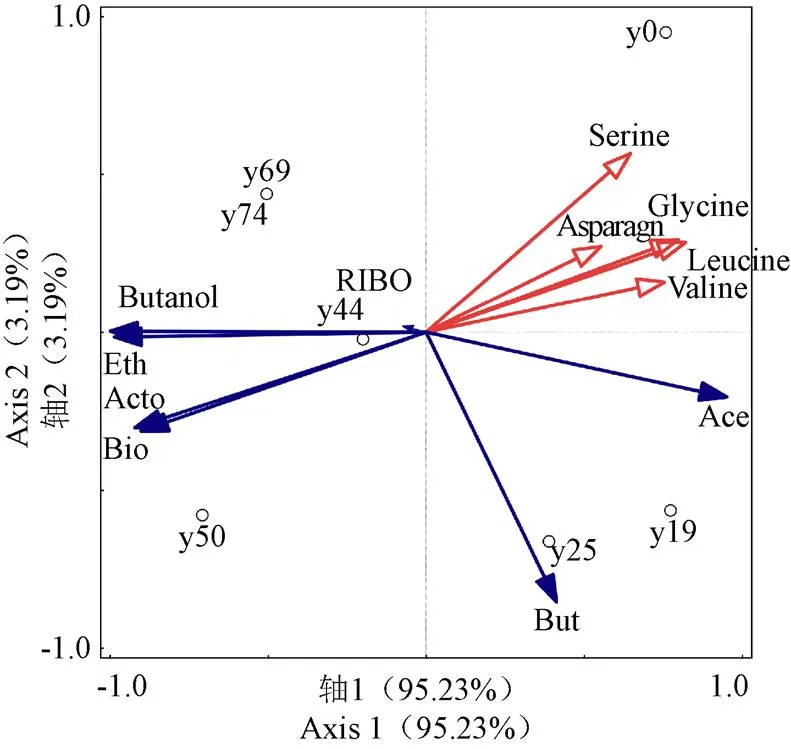
注:But:丁酸;Ace:乙酸;Acto:丙酮;RIBO:核黃素;Bio:生物量;Eth:乙醇。
三磷酸鳥苷(Guanosine triphosphate,GTP)是核黃素產生的重要通量節點。氨基酸通過嘌呤代謝為其從頭合成提供碳氮源。因此,部分氨基酸參與嘌呤代謝時與GTP、核黃素的產生有關。例如,甘氨酸已經被證明可以生成嘌呤環原子中5C和7N[22]。同時,甘氨酸很容易被甘氨酸羥甲基轉移酶催化成絲氨酸,被l-蘇氨酸醛縮酶催化成蘇氨酸。因此,甘氨酸的消耗導致了核黃素的產生,在核黃素的生成中應發揮特殊的作用,這與圖6和圖7所示的結果一致。另一種對核黃素敏感的有機氮源精氨酸(圖6 i)也表現出類似的消耗趨勢。同時,作為主要氮源之一,天冬酰胺在培養中的利用率也較高(圖4g)。因此,本研究中甘氨酸、精氨酸和天冬酰胺對核黃素的積累起到了促進作用。
2.5 氨基酸代謝模型預測玉米漿ABE發酵
在丙酮丁醇梭菌的氨基酸代謝模型基礎上,對利用木糖和玉米漿為主要原料發酵ABE的氨基酸消耗過程進行模擬預測。模型使用玉米漿中氨基酸的含量作為初始值,模擬結果如圖8所示。其中蘇氨酸、甘氨酸、絲氨酸、苯丙氨酸、纈氨酸在發酵過程中被迅速消耗,且在60 h時纈氨酸完全消耗。其中精氨酸消耗量最高,消耗約7 mmol/L,說明培養過程中梭菌大量利用精氨酸用于細胞生長和產物代謝。主要原因為精氨酸通過瓜氨酸快速代謝成鳥氨酸,鳥氨酸可以進一步分解產生ATP[21]。同時,精氨酸的存在也會促進乙酸鹽形成,導致丙酮產量增加[34]。另外,丁醇脅迫也會對精氨酸和嘧啶代謝有調節作用[28]。谷氨酰胺在玉米漿為原料的模擬預測中最終積累濃度達到0.15 mmol/L,但在CGM發酵培養基的實驗中卻增加了約0.10 mmol/L。其他氨基酸,如蛋氨酸、異亮氨酸在發酵培養實驗中被迅速消耗,但在模擬預測中無明顯消耗。分析原因發現,玉米漿中這兩種氨基酸的初始濃度是CGM發酵培養基的10倍左右,說明初始濃度升高并不會大幅增加蛋氨酸、異亮氨酸的消耗。另外纈氨酸、甘氨酸、絲氨酸在玉米漿發酵的預測值都被完全消耗,說明丙酮丁醇梭菌對這3種氨基酸的利用率最高。因此,可以考慮在發酵后期補加消耗較快的氨基酸,以避免造成營養限制因子的缺乏。通過氨基酸代謝模型模擬玉米漿中氨基酸的消耗過程,為下一步利用玉米漿作為氮源生產生物丁醇提供數據支持。
3 討 論
玉米漿中擁有豐富的氨基酸、可溶性糖以及溶磷等營養物質,是作為微生物培養基中廉價氮源的最佳選擇之一。但是,玉米漿中特有的營養物質組成,以及與細胞生長相關的抑制物質的存在,使其對不同微生物有著一定的偏好。但目前,仍未有關于玉米漿中原始菌群的微生物多樣性分析的研究。本研究通過16SrDNA測序技術分析了玉米漿中微生物多樣性及其菌屬的系統進化樹和基因功能預測結果,發現梭狀芽孢桿菌是玉米漿中占有絕對優勢的菌屬。梭狀芽孢桿菌是嚴格厭氧細菌,在生物技術工程中有較多的應用,比如利用其生物代謝丙酮、丁醇、乙醇等有機溶劑,以及有機酸、神經毒素等[35]。本研究在丙酮丁醇梭菌發酵生產ABE,建立該菌的碳代謝模型基礎上完善并建立了氮代謝模型,并模擬了發酵培養基中15種氨基酸的代謝過程。
氨基酸是許多生化反應的前體,梭狀桿菌利用氨基酸具有優先順序,通過Stickland反應利用多種氨基酸[21,33]。氨基酸的代謝需要大量ATP和NADPH,因此溶劑合成過程中,需要減少氨基酸合成用于溶劑生產[24]。草酰乙酸是多種氨基酸的合成前體,可以通過調節丙酮酸羧化酶,降低其活性,減少丙酮酸流向草酰乙酸[36]。大量研究用來表征氨基酸在丙酮丁醇梭菌發酵ABE中的作用。在本研究中,苯丙氨酸、酪氨酸、蛋氨酸在培養液中均明顯被消耗。多個研究表明添加芳香族氨基酸(苯丙氨酸和酪氨酸)以及天冬氨酸族氨基酸(賴氨酸和蛋氨酸)促進了丙酮丁醇梭菌胞內溶劑快速積累,有利于溶劑生產和細胞存活[27,37]。在發酵系統中存在氨基酸能夠促進細菌代謝,提高產品產量和菌株的耐酒精性。Wang等[38]研究表明增加蘇氨酸、甘氨酸、丙氨酸、苯丙氨酸、酪氨酸、色氨酸、天冬氨酸、谷氨酸等氨基酸濃度水平可以提高梭菌對丁醇的耐受性。梭菌能夠調節氨基酸代謝,增加胞內氨基酸積累來適應丁醇脅迫,如Amador-Noguez等[24]檢測到在酸向溶劑轉化過程中胞內支鏈氨基酸(纈氨酸、亮氨酸、異亮氨酸、谷氨酰胺的變化。因此可以通過添加外源調控氨基酸代謝進一步優化溶劑生產和細胞耐性。如Luo等[39]添加了乙酸鹽刺激氨基酸在胞內積累,進而適應丁醇脅迫。Ding等[40]添加Na2SO4/CaSO4作為電子受體刺激苯丙氨酸、蛋氨酸、亮氨酸、纈氨酸胞內積累,丁醇濃度升高至12.96 g/L。但Liu等[29]的研究認為多種氨基酸只與正常細胞代謝相關,并沒有顯著增加丁醇的產量。因此需針對氨基酸在丙酮丁醇梭菌中的代謝途徑及作用作進一步研究。
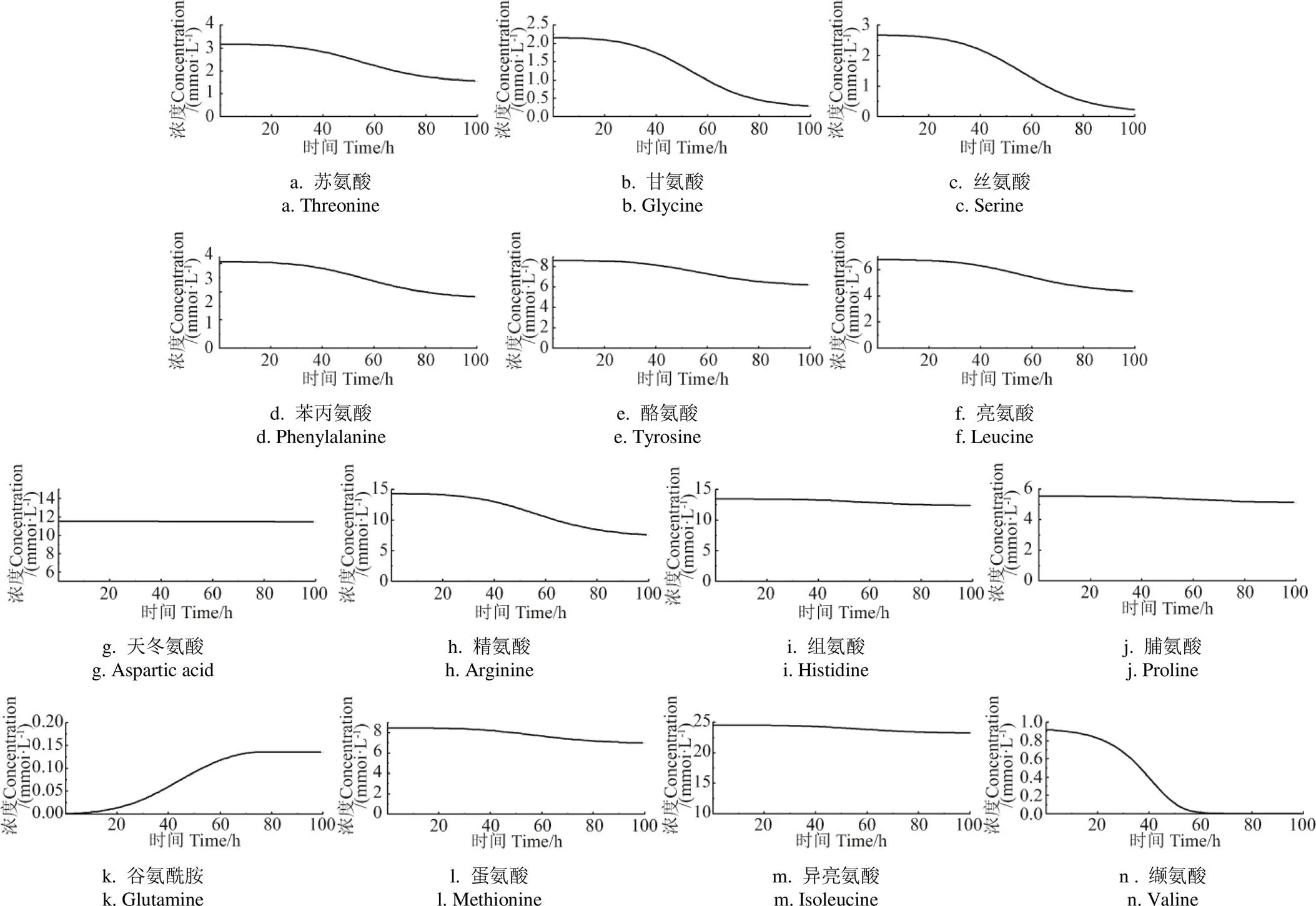
圖8 基于代謝模型的玉米漿ABE發酵的氨基酸代謝模擬預測
大量研究致力于尋求低價碳水化合物和脂類原料用于生產,包括大量蛋白質類廢棄物[41]。本研究致力于利用富含氨基酸的玉米漿為原料生產生物丁醇,以降低生產成本,促進蛋白質廢棄物管理。玉米漿中豐富的氨基酸資源,用于丙酮丁醇梭菌發酵ABE切實可行。玉米漿中含有高水平的乳酸[42]。乳酸可以被梭狀芽孢桿菌同化,進而提高丁醇產量[43-44]。乳酸添加可以提高乳酸向丙酮酸的轉化率,增加了更多NADPH和ATP用于丁醇合成[45]。同時玉米漿中可溶性糖也有利于有機酸生產。Cao等[46]利用造紙廠污泥水解物和5%濃度玉米漿獲得了16.5 g/L濃度的丁醇。Choi等[16]以玉米漿替代酵母膏,在添加6%濃度玉米漿發酵18 h,產生21.4 g/L濃度的ABE。很多研究利用了不同丁酸梭菌對玉米漿發酵丁醇進行了試驗,均證明了玉米漿生產ABE的可行性,但并沒有對玉米漿中氨基酸的利用進行深入研究。本研究通過丙酮丁醇梭菌氨基酸代謝模型的建立,能夠準確模擬玉米漿中氨基酸的消耗情況,為實際生產提供了理論指導作用。
4 結 論
1)玉米漿原始菌群中梭狀芽孢桿菌相對豐度達到68.76%,是玉米漿原始菌群中優勢菌屬。玉米漿中營養成分、滲透壓等原始環境,更適合梭狀芽孢桿菌等微生物定殖。
2)建立了丙酮丁醇梭菌氨基酸代謝模型,該模型能夠較好地擬合試驗數據并能對相關數據進行很好地預測和挖掘。11種氨基酸(苯丙氨酸、蘇氨酸、異亮氨酸、亮氨酸、蛋氨酸、纈氨酸、酪氨酸、甘氨酸、絲氨酸、精氨酸、天冬酰胺)在培養過程中迅速消耗用于細胞生長和溶劑生成,3種氨基酸(脯氨酸、組氨酸、天冬氨酸)保持在穩定的狀態,另外檢測到谷氨酰胺在培養過程中獲得積累。
3)冗余分析發現5種氨基酸(絲氨酸、天冬酰胺、甘氨酸、亮氨酸、纈氨酸)與丙酮丁醇梭菌的生物量、ABE產物、核黃素有顯著相關性,相關性排序從大到小依次為絲氨酸、甘氨酸、亮氨酸、纈氨酸、天冬酰胺。其中亮氨酸對結果影響解釋度為64.4%,被認為是對產物主要影響的氨基酸。
4)氨基酸動態模型模擬預測玉米漿作為氮源發酵ABE時,精氨酸消耗量最高,約7 mmol/L,纈氨酸、甘氨酸、絲氨酸基本被完全消耗,實際生產中應考慮在發酵后期補加消耗較快的氨基酸,以避免缺乏營養限制因子。該結論為丙酮丁醇梭菌的氨基酸代謝調控及下一步以玉米漿—木糖為原料利用梭菌ABE發酵生產進一步提供理論支撐和數據參考。
[1] Ndaba B, Chiyanzu I, Marx S. Direct fermentation of sweet sorghum juice by Clostridium acetobutylicum and Clostridium tetanomorphum to produce bio-butanol and organic acids[J]. Biofuel Research Journal, 2015, 2(2): 248-252.
[2] Jones D T, Woods D R. Acetone-butanol fermentation revisited[J]. Microbiological Reviews, 1986, 50(4): 484-524.
[3] 王風芹,謝媱嬛,蘇增平,等. 酶法復合脫毒提高玉米秸稈水解液丁醇發酵效率[J]. 農業工程學報,2017,33(8):204-210. Wang Fengqin, Xie Yaohuan, Su Zengping, et al. Enzymatic composite detoxification improving effect of butanol fermentation from corn stover hydrolysate[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2017, 33(8): 204-210. (in Chinese with English abstract)
[4] Zhang J, Zhu W, Xu H, et al. Simultaneous glucose and xylose uptake by an acetone/butanol/ethanol producing laboratory Clostridium beijerinckii strain SE-2[J]. Biotechnology Letters, 2016, 38(4): 611-617.
[5] Kumar M, Gayen K. Developments in biobutanol production: New insights[J]. Applied Energy, 2011, 88(6): 1999-2012.
[6] Hofer A, Hauer S, Kroll P, et al. In-depth characterization of the raw material corn steep liquor and its bioavailability in bioprocesses of Penicillium chrysogenum[J]. Process Biochemistry, 2018, 70: 20-28.
[7] Filipovi? S, Risti? M, Saka? M. Technology of corn steep application in animal mashes and their quality[J]. Roumanian Biotechnology Letters, 2001, 7(3): 705-710.
[8] Zhang J, Jia B. Enhanced butanol production using Clostridium beijerinckii SE-2 from the waste of corn processing[J]. Biomass and Bioenergy, 2018, 115: 260-266.
[9] Maddipati P, Atiyeh H K, Bellmer D D, et al. Ethanol production from syngas by Clostridium strain P11 using corn steep liquor as a nutrient replacement to yeast extract[J]. Bioresource Technology, 2011, 102(11): 6494-6501.
[10] Zhao X, Kasbi M, Chen J, et al. A dynamic metabolic flux analysis of ABE (acetone‐butanol‐ethanol) fermentation byATCC 824, with riboflavin as a by-product[J]. Biotechnology and Bioengineering, 2017, 114(12): 2907-2919.
[11] Li X, Li Z, Zheng J, et al. Yeast extract promotes phase shift of bio-butanol fermentation byATCC824 using cassava as substrate[J]. Bioresource Technology, 2012, 125: 43-51.
[12] Sun Z, Liu S. Production of n-butanol from concentrated sugar maple hemicellulosic hydrolysate byATCC824[J]. Biomass and Bioenergy, 2012, 39: 39-47.
[13] Choi S J, Lee J, Jang Y S, et al. Effects of nutritional enrichment on the production of acetone-butanol-ethanol (ABE) by[J]. Journal of Microbiology, 2012, 50(6): 1063-1066.
[14] Harris L M, Welker N E, Papoutsakis E T. Northern, morphological, and fermentation analysis of spo0A inactivation and overexpression inATCC 824[J]. Journal of Bacteriology, 2002, 184(13): 3586-3597.
[15] Zhao X, Condruz S, Chen J, et al. A quantitative metabolomics study of high sodium response inATCC 824 acetone-butanol-ethanol (ABE) fermentation[J]. Scientific Reports, 2016, 6(1): 28307.
[16] Choi J D R, Jang Y S, Cho J H, et al. Characterization and evaluation of corn steep liquid in acetone-butanol-ethanol production by[J]. Biotechnology and Bioprocess Engineering, 2013, 18(2): 266-271.
[17] Rizzi M, Baltes M, Theobald U, et al. In vivo analysis of metabolic dynamics in: II. Mathematical model[J]. Biotechnology and Bioengineering, 1997, 55(4): 592-608.
[18] Tracy B P, Jones S W, Fast A G, et al. Clostridia: The importance of their exceptional substrate and metabolite diversity for biofuel and biorefinery applications[J]. Current Opinion in Biotechnology, 2012, 23(3): 364-381.
[19] Charubin K, Bennett R K, Fast A G, et al. Engineeringorganisms as microbial cell-factories: challenges & opportunities[J]. Metabolic Engineering, 2018, 50: 173-191.
[20] Hu X, Du H, Ren C, et al. Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J]. Applied and Environmental Microbiology, 2016, 82(8): 2506-2515.
[21] Fonknechten N, Chaussonnerie S, Tricot S, et al., a specialist in amino acid degradation: Revisiting its metabolism through its genome sequence[J]. BMC Genomics, 2010, 11(1): 555.
[22] Michal G. Biochemical pathways: An atlas of biochemistry and molecular biology[J]. Journal of Chemical Education, 2012, 77(2): 163.
[23] Baer S H, Blaschek H P, Smith T L. Effect of butanol challenge and temperature on lipid composition and membrane fluidity of butanol-tolerant[J]. Applied and Environmental Microbiology, 1987, 53(12): 2854-2861.
[24] Amador-Noguez D, Brasg I A, Feng X J, et al. Metabolome remodeling during the acidogenic-solventogenic transition in[J]. Applied and Environmental Microbiology, 2011, 77(22): 7984-7997.
[25] Janssen H, D?ring C, Ehrenreich A, et al. A proteomic and transcriptional view of acidogenic and solventogenic steady-state cells ofin a chemostat culture[J]. Applied Microbiology and Biotechnology, 2010, 87(6): 2209-2226.
[26] Masion E, Amine J, Marczak R. Influence of amino acid supplements on the metabolism of[J]. FEMS Microbiology Letters, 1987, 43(3): 269-274.
[27] Wu J, Dong L, Zhou C, et al. Developing a coculture for enhanced butanol production byand Saccharomyces cerevisiae[J]. Bioresource Technology Reports, 2019, 6: 223-228.
[28] Yoo M, Nguyen N P T, Soucaille P. Trends in systems biology for the analysis and engineering of clostridium acetobutylicum metabolism[J]. Trends in Microbiology, 2020, 28(2): 118-140.
[29] Liu H, Huang D, Wen J. Integrated intracellular metabolic profiling and pathway analysis approaches reveal complex metabolic regulation by[J]. Microbial Cell Factories, 2016, 15(1): 1-14.
[30] Janssen H, Grimmler C, Ehrenreich A, et al. A transcriptional study of acidogenic chemostat cells of-solvent stress caused by a transient n-butanol pulse[J]. Journal of Biotechnology, 2012, 161(3): 354-365.
[31] Croux C, Canard B, Goma G, et al. Autolysis ofATCC 824[J]. Microbiology, 1992, 138(5): 861-869.
[32] Nimbalkar P R, Khedkar M A, Kulkarni R K, et al. Strategic intensification in butanol production by exogenous amino acid supplementation: Fermentation kinetics and thermodynamic studies[J]. Bioresource Technology, 2019, 288: 121521.
[33] Sangavai C, Chellapandi P. Amino acid catabolism-directed biofuel production in Clostridium sticklandii: An insight into model-driven systems engineering[J]. Biotechnology Reports, 2017, 16: 32-43.
[34] Grassi M C B, Carazzolle M F, Nakagawa B T, et al. New contributions for industrial n-butanol fermentation: An optimized Clostridium strain and the use of xylooligosaccharides as a fermentation additive[J]. Biomass and Bioenergy, 2018, 119: 304-313.
[35] Gheshlaghi R, Scharer J M, Moo-Young M, et al. Metabolic pathways of clostridia for producing butanol[J]. Biotechnology Advances,2009, 27(6): 764-781.
[36] Daniel A N. Systems-level metabolic flux profiling elucidates a complete, bifurcated tricarboxylic acid cycle in Clostridium acetobutylicum[J]. Journal of Bacteriology, 2010, 17(192): 4452-4461.
[37] Abd Alla M H, Elsadek El-Enany A W. Production of acetone-butanol-ethanol from spoilage date palm (L) fruits by mixed culture ofand Bacillus subtilis[J]. Biomass and Bioenergy, 2012, 42: 172-178.
[38] Wang Y F, Tian J, Ji Z H, et al. Intracellular metabolic changes ofand promotion to butanol tolerance during biobutanol fermentation[J]. The International Journal of Biochemistry & Cell Biology, 2016, 78: 297-306.
[39] Luo H, Ge L, Zhang J, et al. Enhancing acetone biosynthesis and acetone–butanol–ethanol fermentation performance by co-culturing Clostridium acetobutylicum/Saccharomyces cerevisiae integrated with exogenous acetate addition[J]. Bioresource Technology, 2016, 200: 111-120.
[40] Ding J, Luo H, Xie F, et al. Electron receptor addition enhances butanol synthesis in ABE fermentation by Clostridium acetobutylicum[J]. Bioresource Technology, 2018, 247: 1201-1205.
[41] El-Dalatony M M, Saha S, Govindwar S P, et al. Biological conversion of amino acids to higher alcohols[J]. Trends in Biotechnology, 2019, 37(8): 855-869.
[42] Gao Y, Yuan Y J. Comprehensive quality evaluation of corn steep liquor in 2-Keto-l-gulonic acid fermentation[J]. Journal of Agricultural & Food Chemistry, 2011, 59(18): 9845-9853.
[43] Ahn J H, Sang B I, Um Y. Butanol production from thin stillage using Clostridium pasteurianum[J]. Bioresource Technology, 2011, 102(7): 4934-4937.
[44] Zhang J, Zong W, Hong W, et al. Exploiting endogenous CRISPR-Cas system for multiplex genome editing in Clostridium tyrobutyricum and engineer the strain for high-level butanol production[J]. Metabolic Engineering, 2018, 47: 49-59.
[45] Zhou Q, Liu Y, Yuan W. Kinetic modeling of lactic acid and acetic acid effects on butanol fermentation by Clostridium saccharoperbutylacetonicum[J]. Fuel, 2018, 226: 181-189.
[46] Cao X, Chen Z, Liang L, et al. Co-valorization of paper mill sludge and corn steep liquor for enhanced n-butanol production with Clostridium tyrobutyricum Δcat1:: adhE2[J]. Bioresource Technology, 2020, 296: 122347.
Kinetic simulation of amino acid metabolism of butanol produced from the fermentation of corn steep liquor
Zhao Xinhe1,2,3, He Zhuangzhuang1, Zhao Yubin4, Ma Chengye1, Ren Xiaojie1,2※
(1,,255000,; 2.,,255000,;3.,401123,; 4.,276400,)
This study aimsto investigate the dynamic process of amino acid metabolism in thefermentation of biobutanol. In a production strategy, a type of cheap nitrogen source, such as amino acid in the Corn Steep Liquor (CSL), can be used to produce butanol from. The efficient and cheap nitrogen source is seeking for butanol production to reduce the production cost of fermentation. In this study, a high-throughput sequencing technology was used to analyze the microbial diversity in CSL, based on the dynamic model of carbon metabolism for ABE (acetone-butanol-ethanol) produceby. A model of amino acid metabolism was constructed, using the metabolism network of amino acid, chemical reaction flux dynamics, and mass balance measurement. 15 amino acids were selected to simulate the metabolism process in ABE fermentation, using xylose as a carbon source. A redundancy analysis was conducted to establish the correlation between amino acid metabolism, the biomass of, and the synthesis of ABE. The predict model was achieved for the consumption process of amino acid in the CSL fermentation in an actual production. The results showed that theaccounted for 68.76% of the total number of bacteria, indicating the dominant bacterial group in the CSL. The prediction results showed that the gene function of original bacteria was mainly focused on the transformation of carbon and nitrogen sources in the CSL. The optimal parameter correction demonstrated that the simulated values were in good agreement with the experimental ones, indicating that the proposed model can accurately reveal the dynamic metabolic process of amino acid under the utilization by. Both experimental and simulation results indicated that 11 amino acids (phenylalanine, threonine, isoleucine, leucine, methionine, valine, tyrosine, glycine, serine, arginine, asparagine) were rapidly consumed for cell growth and solvent generation during the cultivation process, whereas 3 amino acids (proline, histamine, aspartic acid) remained stable, particularly that the glutamine accumulation was detected during fermentation. In addition, the metabolism of sugar and amino acid can be beneficial to the cell growth in the early stage, but not to the butanol synthesis. Correlation analysis results showed that five types of amino acids (serine, asparagine, glycine, leucine, and valine) were closely related to the fermentation products and biomass accumulation. The correlation sequence was serine, glycine, leucine, valine, asparagine in order from the largest to the smallest. A high consumption rate was obtained for valine, glycine, and serine during the fermentation process, indicating a nutrient limiting factor in the later stage of fermentation. A fully consumption was found in the predicted values of valine, glycine, and serine in the fermentation of CSL, indicating thatthehad the highest utilization rate of three amino acids. Therefore, amino acids can be expected to add in the late period of fermentation, to avoid the deficiency of nutrient limiting factors. The CSL can also serve as the dominant nitrogen source of butanol fermentationbyThe findings can provide a sound theoretical reference and data support for the amino acid metabolism of, and for the subsequent utilization and optimization of CSL as a promising nitrogen source to produce biobutanol.
dynamics; models;; ABE fermentation; corn steep liquor; microbiome analysis; amino acid
趙新河,賀壯壯,趙玉斌,等.玉米漿發酵產生物丁醇的氨基酸代謝動力學模擬[J]. 農業工程學報,2020,36(18):263-274.doi:10.11975/j.issn.1002-6819.2020.18.031 http://www.tcsae.org
Zhao Xinhe, He Zhuangzhuang, Zhao Yubin, et al. Kinetic simulation of amino acid metabolism of butanol produced from the fermentation of corn steep liquor[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2020, 36(18): 263-274. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2020.18.031 http://www.tcsae.org
2020-05-28
2020-08-18
重慶市技術創新與應用發展重點項目(cstc2019jscx-gksbX0113);國家博士后基金面上項目(2019M662362);山東省自然科學基金博士項目(ZR2019BC099);山東省博士后創新項目(201902048)
趙新河,博士,講師,主要從事發酵工程方面的研究。Email:zhaoxinhe@sdut.edu.cn
任曉潔,博士,講師,主要從事發酵工程方面的研究。Email:renxiaojie2020@163.com
10.11975/j.issn.1002-6819.2020.18.031
TQ923
A
1002-6819(2020)-18-0263-12

