基于鹽脅迫轉錄組信息的蠶豆F-box基因家族分析
郝樹琳,陳宏偉,廖芳麗,李莉,劉昌燕,劉良軍,萬正煌,沙愛華
基于鹽脅迫轉錄組信息的蠶豆F-box基因家族分析
郝樹琳1,陳宏偉2,廖芳麗3,李莉2,劉昌燕2,劉良軍2,萬正煌2,沙愛華1
(1長江大學農學院/主要糧食作物產業化湖北省協同創新中心,湖北荊州 434025;2湖北省農業科學院糧食作物研究所/糧食作物種質創新與遺傳改良湖北省重點實驗室,武漢 430064;3荊州市種子管理局,湖北荊州 434020)
【】通過生物信息學方法分析蠶豆F-box基因家族成員的分布、結構及進化,研究家族成員在不同處理時間條件下的表達模式及對鹽脅迫的響應,為該類基因生物學功能和鹽脅迫機制的研究提供參考。基于蠶豆鹽脅迫轉錄組測序(RNA-seq)數據,利用NR、Swiss-prot和PFAM 3個數據庫和NCBI網站,對蠶豆F-box基因進行篩選注釋;利用Web Logo 3、Prot Comp 9.0、MEGA-X和MEME等軟件進行保守結構域、亞細胞定位、系統進化樹和Motif等生物信息學分析。基于鹽脅迫轉錄組數據分析蠶豆(yz17134耐鹽和yz17078不耐鹽)F-box基因家族在鹽脅迫下的差異表達模式,并采用實時熒光定量PCR技術(qRT-PCR)檢測部分家族成員在16和24 h的表達情況。基于鹽脅迫轉錄組測序數據,注釋得到161個蠶豆F-box基因,均含有F-box保守結構域。根據C端結構域的不同,將其分成11個亞族:FBX、FBXFBA、FBXLRR、FBXPP2、FBXKelch、FBXTUB、FBXFBD、FBXDUF、FBXACTIN、FBXWD40和FBO。保守結構域分析表明,F-box保守基序中包含1個極度保守的色氨酸殘基。比較分析蠶豆F-box家族和擬南芥F-box家族共同構建的進化樹,發現同一C端結構域的基因大多聚集在一起。亞細胞定位預測結果顯示,124個F-box基因定位于細胞外,37個定位于細胞核中。基因結構分析表明,蠶豆F-box家族基因的DNA序列中均無內含子,且均由UTR區和CDS區組成。基于鹽脅迫轉錄組數據的F-box差異表達模式分析表明,蠶豆F-box基因在2個不同處理時間點上的表達各不相同,在鹽處理16 h的表達較為明顯。qRT-PCR分析結果表明,在F-box家族成員中,共存在5個差異基因。其中、和在鹽處理16 h的表達量均上調,和在鹽處理16 h的表達量均下調。蠶豆F-box基因家族注釋得到161個蠶豆F-box基因,分為11個亞族。其中5個重要的F-box基因在不同鹽處理時間的表達量存在差異。
蠶豆;轉錄組測序;F-box家族;鹽脅迫;表達模式
0 引言
【研究意義】蠶豆(L)是巢菜屬一年生或越年生草本植物,有生物固氮之王的美譽,具有較高經濟價值[1]。在蠶豆生長發育過程中,生物和非生物脅迫等因素對蠶豆產生影響,最終導致其產量和品質下降,造成經濟損失。鹽脅迫是其中一種主要的非生物脅迫,嚴重危害蠶豆的生長發育。F-box基因廣泛參與植物生長發育及響應非生物脅迫途徑,如細胞蛋白質降解、激素感知和信號傳導等[2-4]。因此,研究蠶豆F-box基因功能和培育蠶豆耐鹽品種具有重要意義。【前人研究進展】自然條件下,植物的生長會受到內部或外部的刺激,為了應對產生的刺激,植物已經發展出多種調節機制來協調它們的生命活動。其中,泛素26s蛋白酶體系統(ubiquitin-26s proteasome system,UPS)降解蛋白是一種重要的翻譯后調控機制,并在真核生物中高度保守[5]。UPS催化多種泛素與靶底物的共價連接,使底物被26S蛋白酶體識別并進行蛋白水解。泛素化主要由泛素激活酶(ubiquitin-activating enzymes,E1s)、泛素偶聯酶(ubiquitin-conjugating enzymes,E2s)和泛素蛋白連接酶(ubiquitin protein ligases,E3s)3種酶來完成。其中,E3s起到關鍵的底物識別功能[6]。F-box蛋白作為E3s的核心成分,即SCF(Skp1-Cul1-F-box)復合物,介導不同底物的特異性降解[7]。F-box基因廣泛存在于各種植物,是植物中最大、進化最快的基因家族之一,越來越多的真核F-box基因被發現[8-11]。F-box基因在不同物種間的數量相差較大。動物體內的F-box基因數量相對較少,秀麗線蟲是目前發現含有F-box基因最多的動物,具有326個,而人體內僅含有38個,果蠅只含有22個[12];植物中F-box基因家族成員個數遠多于動物,模式植物苜蓿和擬南芥分別含有972和694個[13-14],農作物水稻、大豆、玉米和鷹嘴豆分別含有687、509、359和285個[15-16],經濟作物蘋果和梨分別含有517和226個[17-18]。F-box結構域一般位于蛋白的N端,含有40—50個氨基酸,與SKP1相互作用形成SCF復合物,而C端通常是介導底物識別特異性的二級結構[14]。在哺乳動物中,根據C末端二級結構的不同,F-box蛋白可分為3類。具有LRR結構域的FBXL,具有WD40重復結構域的FBXW和具有其他結構域的FBXO[19]。在植物中,通過對擬南芥和水稻F-box蛋白的全基因組分析,發現了大量結構域,如WD40(Tryptophan-aspartic acid 40)、kelch重復序列、LRR(Leucine-rich repeat)、FBD(F-Box and BRCT domain)、環指結構、PAS/PAC(Per Arnt Sim)、PPR(pentatricopeptide repeat)和TUB(tubby)等[15]。因此,F-box蛋白可以識別多種類型的底物,在植物生長發育、生物和非生物脅迫、激素信號轉導等生物學過程中發揮重要作用。UFO是植物中發現的第一個F-box蛋白,在花分生組織鑒定和花器官發育中具有重要作用[20]。隨著深入研究,越來越多的F-box蛋白被發現,并且這些蛋白的功能變得多樣化。在擬南芥中,許多F-box蛋白參與植物激素反應。例如,含有F-box結構域的TIR1是擬南芥的生長素受體[21]。AFB 1-3是一種生長素信號轉導的F-box蛋白,可以結合類似TIR1的Aux/IAA蛋白,促進生長素的應答[22]。F-box蛋白也可調控赤霉素信號通路,例如擬南芥的SLEEPY1(SLY1)和SNEEZY(SNE),水稻中的赤霉素不敏感矮桿2(GID2)[23]。在乙烯信號轉導途徑中,乙烯不敏感3(EIN3)通過結合F-box1/2(EBF1/2)進行降解[24]。COI1也屬F-box蛋白,它是茉莉酸(JA)信號反應的一個重要因子,也是擬南芥JA依賴性反應必需的[25]。此外,F-box蛋白LKP1/ZTL、LKP2/FKL和FKF1參與光形態發生、晝夜節律和開花時間[26]。DOR是擬南芥中與ASK14和CUL1特異性相互作用的F-box蛋白,在耐旱條件下起負調控作用[27]。水稻F-box基因與葡萄糖延遲種子萌發有關[28]。此外,早期研究中也發現了一些植物的S位點F-box基因,證明其參與花粉管伸長,甚至參與自交不親和[29]。【本研究切入點】目前,對于蠶豆的研究多集中在生理層面,在蠶豆中對F-box基因家族的鑒定和系統分析鮮見報道。【擬解決的關鍵問題】本研究利用生物信息學的方法對蠶豆F-box基因家族成員進行鑒定,并對其基因結構、亞細胞定位、蛋白保守結構域及系統進化等進行分析,同時利用qRT-PCR技術對F-box家族成員響應鹽脅迫的差異表達模式進行分析,為后續研究蠶豆F-box基因的功能提供一定的理論依據。
1 材料與方法
1.1 材料
蠶豆(L)種子由湖北省農業科學院雜糧研究工程中心提供。于2018年9月,選取飽滿、大小均等的2個不同品種(Y134耐鹽和Y078不耐鹽)的蠶豆種子進行發芽試驗,用0.8% NaCl處理,分別于16和24 h收集具有胚胎1/4部分子葉的材料,每個處理設置3個生物重復,材料收集后液氮速凍,-80℃保存備用。
試驗于2018年在長江大學農學院耐漬油料種質資源湖北省重點實驗室進行。
1.2 蠶豆總RNA的提取、文庫構建及轉錄組測序
植物總RNA提取采用TRNzol試劑法(TRNzol Universal Reagent,天根生化科技有限公司,北京,中國)分別提取蠶豆芽期胚的總RNA,并用TGem spectrophotometer Plus(天根生化科技有限公司,北京,中國)微量分光光度計檢測RNA純度(OD260/280比值)以及RNA濃度,最后用1%瓊脂糖凝膠電泳檢測。檢測合格的樣品送到廣州賽哲生物科技股份有限公司進行轉錄組測序,并通過De Novo方法[30]組裝得到116 093條Unigene,后續分析基于此測序結果進行。
1.3 蠶豆和擬南芥F-box基因的篩選
以鹽脅迫轉錄組數據為基礎,在NR、Swiss-prot和Pfam 3個數據庫中進行注釋,初步注釋到451個F-box相關基因,并用TBtools軟件[31]批量提取相應的蛋白序列。將這些蛋白序列于NCBI進行Blast搜索比對,同時利用CDD(conserved domain database)在線工具(https://www.ncbi.nlm.nih.gov/Structure/ bwrpsb/bwrpsb.cgi)對序列的保守結構域進行預測,去除重復序列及冗余轉錄本后,最終得到161個具有F-box結構域的蛋白序列。根據Wang等[32]對梨F-box基因家族成員保守結構域不同的方法進行分類,擬南芥蛋白序列從Ensembl Plants(http://plants.ensembl. org/index.html)數據庫中下載獲得。
1.4 蠶豆F-box基因家族保守域及其進化關系分析
利用序列分析軟件Clustal W對161個蠶豆F-box蛋白進行多序列比對,去除保守域之外的區域,通過在線軟件Web Logo 3(https://weblogo.Threeplusone. com/)對蠶豆F-box蛋白進行保守基序分析;利用MEGA-X軟件的Clustal W程序,對161個蠶豆F-box蛋白和部分功能明確的擬南芥F-box蛋白(共15個)進行多序列比對,并用最大似然法(maximum likelihood)構建系統發育樹,采用Jones-Thornton- Taylor(JTT)模型算法進行估計,并進行Bootstrap分析,重復值設置為1 000。通過在線程序iTOL(https:// itol.embl.de/)可視化系統發育樹。
1.5 蠶豆F-box基因家族亞細胞定位預測、基因結構及Motif分析
利用在線軟件Prot Comp 9.0(http://linux.softberry. com)對161個候選F-box基因家族各成員的氨基酸序列進行亞細胞定位預測分析;根據基因組注釋信息,利用TBtools軟件對F-box基因的基因結構進行可視化分析;利用在線軟件MEME(http://meme-suite.org/)分析蠶豆F-box基因家族成員Motif類型和排列順序,獲得基因家族Motif特點。
1.6 蠶豆F-box基因的表達模式分析與驗證
蠶豆轉錄組測序文庫中各基因片段表達量的計算采用FPKM(fragments per kilobase million)法[33]。將數據標準化后,利用TBtools軟件對F-box基因在2個不同處理時間和2個不同品種間的表達繪制熱圖。以蠶豆2個不同處理時間的2個品種為材料,選取了5個在處理16 h上、下調明顯的F-box基因,進行實時熒光定量PCR。各樣品RNA采用第一鏈cDNA合成試劑盒(Promega,Madison,USA)反轉錄cDNA,選用蠶豆作內參[34],采用Primer Premier 5.0設計熒光定量引物(表1),反應體系為模板cDNA 1 μL、正/反向引物各0.5 μL(10 μmol·L-1)、2×RealUniversal PreMix(SYBR Green, blue,FP201,TIANGEN,北京,中國)10 μL和ddH2O 8 μL。PCR反應程序為95℃ 15 min;95℃ 10 s,57℃ 20 s,72℃ 30 s,40個循環。每個樣品設置3次技術重復。所用儀器為美國伯樂(BIO-RAD,CA,USA)CFX Connect實時熒光定量PCR儀,采用2-△△CT方法計算相對表達量的[35]。
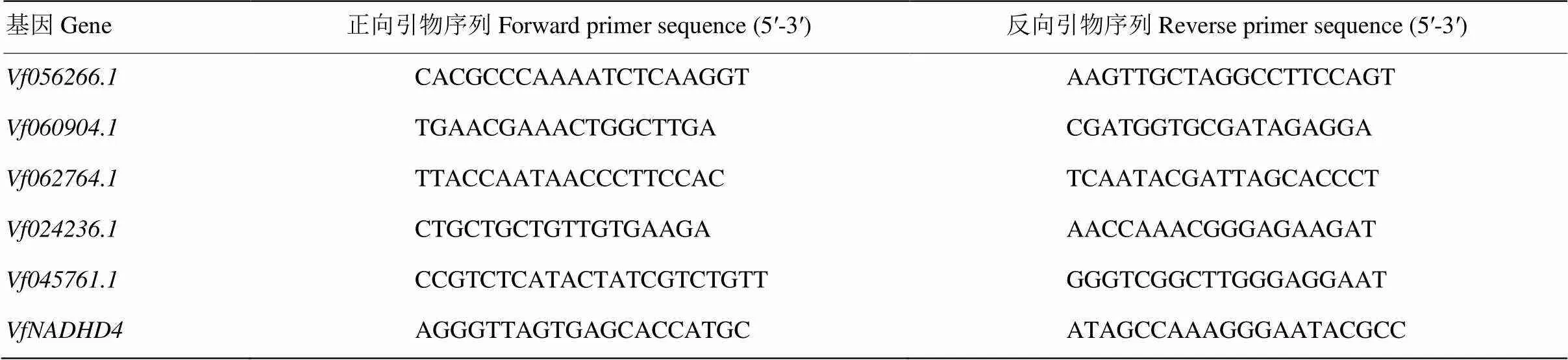
表1 實時熒光定量PCR所用的引物
2 結果
2.1 蠶豆F-box基因家族的鑒定及分類
基于鹽脅迫轉錄組測序數據,在NR、Swiss-prot和Pfam 3個數據庫中進行注釋,初步注釋到451個F-box相關基因,通過NCBI Blast預測,去除重復序列和冗余轉錄本后,最終得到161個蠶豆F-box基因。
利用SMART和CDD在線工具對所得到的161條F-box蛋白序列進行預測,并根據Wang等[18]對梨F-box基因家族的分類方法,按照每個F-box基因所包含的C端保守結構域類型,將其分為FBX、FBXFBA、FBXLRR、FBXPP2、FBXKelch、FBXTUB、FBXFBD、FBXDUF、FBXACTIN、FBXWD40和FBO 11類。其中FBX類基因64個,FBXFBA類基因27個,FBXLRR類基因19個,FBXPP2類基因8個,FBXKelch類基因16個,FBXTUB類基因7個,FBXFBD類基因10個,FBXDUF類基因2個,FBXACTIN類基因2個,FBXWD40類基因1個,FBO類基因5個。
2.2 蠶豆F-box蛋白的保守基序分析
利用序列分析軟件Clustal W對161個蠶豆F-box蛋白進行多序列比對,去除保守域之外的不保守區域,對其保守結構域進行基序分析。通常F-box蛋白在N端含有40—50個氨基酸的F-box基序[18]。由圖1蠶豆F-box蛋白的保守基序圖可知,其約含49個氨基酸殘基,包括1個極度保守的色氨酸殘基(W)。此外,在F-box蛋白的保守基序中還存在其他保守氨基酸殘基,如第4位的亮氨酸殘基(L)、第5位的脯氨酸殘基(P)、第12位的蘇氨酸殘基(T)、第30位的纈氨酸殘基(V)。這些保守的氨基酸殘基可能與色氨酸殘基共同維持蠶豆F-box基序的α螺旋結構[36],進而發揮其特定的功能。
2.3 蠶豆與擬南芥F-box基因的進化關系與功能預測分析
利用MEGA-X軟件構建蠶豆與擬南芥F-box蛋白家族的系統進化樹,根據進化樹(圖2)的拓撲結構將2個物種的F-box家族蛋白進行聚類分析。研究表明,對植物的F-box蛋白進行系統發育分析時,具有相同C端結構域的F-box蛋白傾向于聚集在系統發育樹的同一類群中[11]。因此,根據不同的特異性區域將F-box基因家族成員分為11個不同的亞群。FBX亞族(64)是由只含有F-box結構域的基因組成,不包含其他任何特定結構域,其成員數量多于其他子群。其他10個亞族由包含一個不可缺少的F-box結構域和其他可變結構域的基因組成,如LRR(命名為FBXLRR)、Tub(FBXTUB)、Kelch(FBXKelch)、FBA (FBXFBA)、FBD(FBXFBD)等。例如,FBXFBA和FBXFBD亞群分別表示含有FBA結構域或FBD結構域而沒有其他可變域的基因。為了驗證上述分類,利用蠶豆F-box全長序列與部分功能明確的擬南芥全長序列共同構建了一個系統發育樹,事實上,觀察到一些分組成員聚集在一起,例如FBXFBA、FBXFBD、FBXTUB和FBXPP2,這在一定程度上支持了基于C端結構域分類的方法。
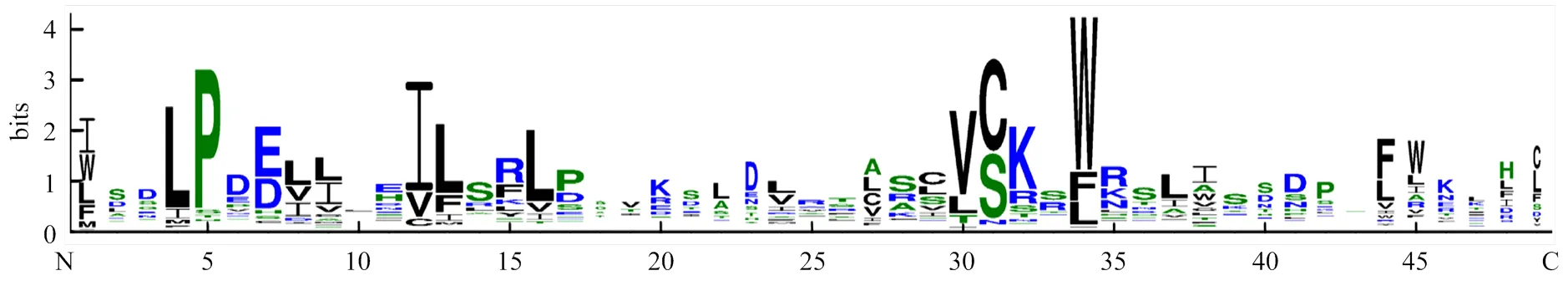
圖1 蠶豆F-box蛋白保守基序
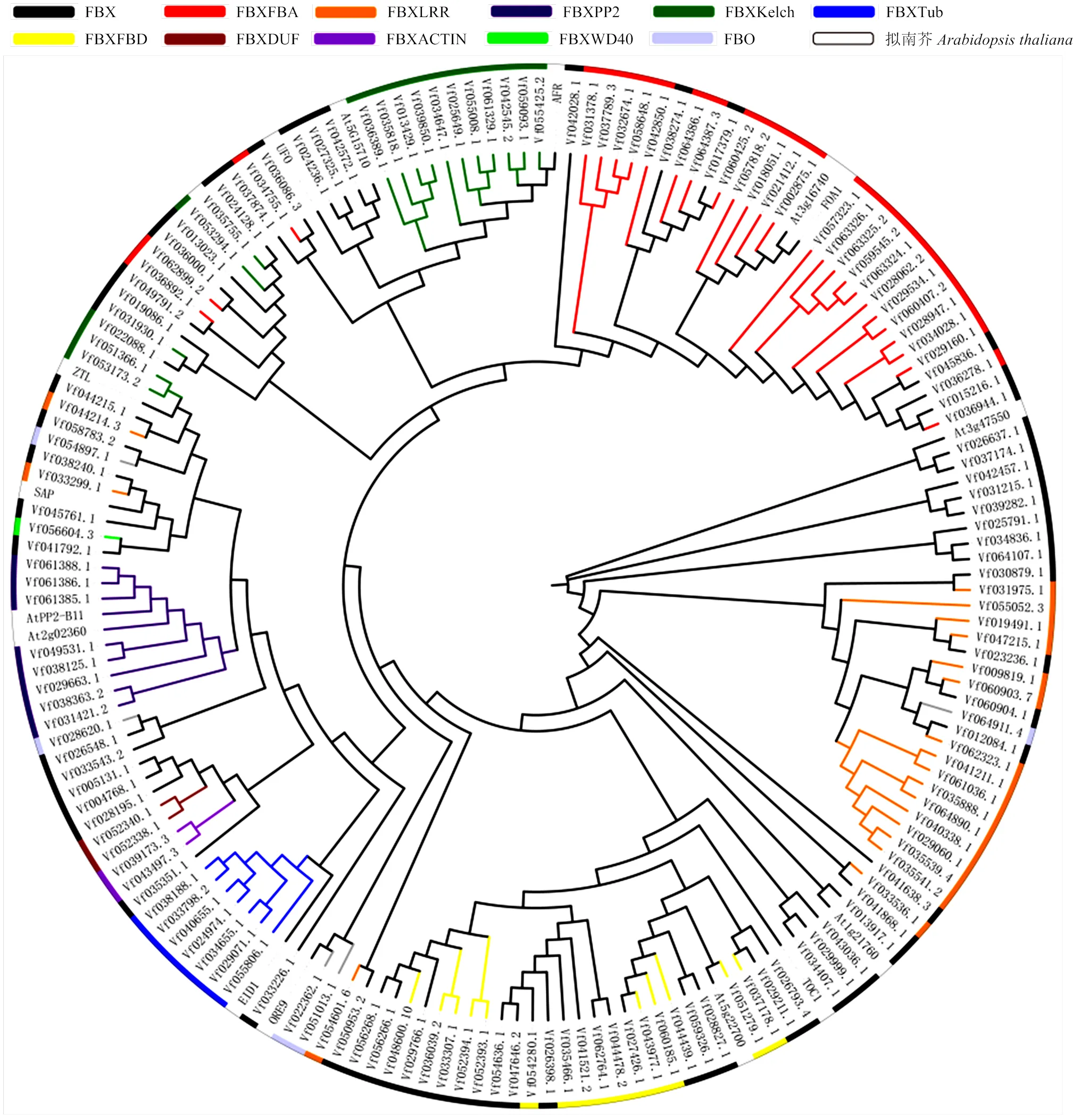
圖2 蠶豆與擬南芥F-box蛋白家族的進化分析
由圖2可知,一些F-box亞族與擬南芥F-box蛋白進化關系較近。例如,FBXFBA亞族中Vf057818.2、Vf018051.2、Vf021412.1、Vf002875.1與擬南芥At3g16740、FOA1位于同一進化亞枝上;FBX亞族中Vf026637.1、Vf037174.1與擬南芥At3g47550w位于同一進化亞枝上,Vf026793.4、Vf029211.1與擬南芥TOC1進化關系較近;FBXFBD亞族中Vf051279.1與擬南芥At5g22700位于同一進化亞枝上;FBXTub亞族與擬南芥EID1位于同一進化亞枝上;FBXPP2亞族與擬南芥AtPP2-B11、At2g02360位于同一進化亞枝上;FBXKelch亞族與擬南芥AFR位于同一進化亞枝,表明它們可能具有相似的功能。
2.4 蠶豆F-box基因的亞細胞定位預測
通過在線軟件Prot Comp 9.0(http://linux.softberry. com)對161個蠶豆F-box個成員的氨基酸序列進行亞細胞定位預測(圖3),結果顯示,共有124個候選F-box基因位于細胞外,其中FBX亞族多達59個,占候選基因總數的47.58%,說明這些F-box蛋白可能參與胞外某些特定的生物學過程;37個位于細胞核中,其中FBXPP2亞族多達8個,占候選基因總數的21.62%,表明其可能通過調控細胞核基因的轉錄和翻譯來發揮特定功能。
2.5 蠶豆F-box基因的基因結構及Motif分析
根據基因組注釋信息,利用TBtools軟件對F-box基因的基因結構進行可視化分析(圖4),發現F-box基因家族成員無內含子結構,且每一個成員都含有UTR區和CDS區。根據F-box家族成員的進化關系可知,同一亞族的編碼區長度大致相同。因此,基因結構分析在一定程度上也支持了基于C端結構域分類的方法。
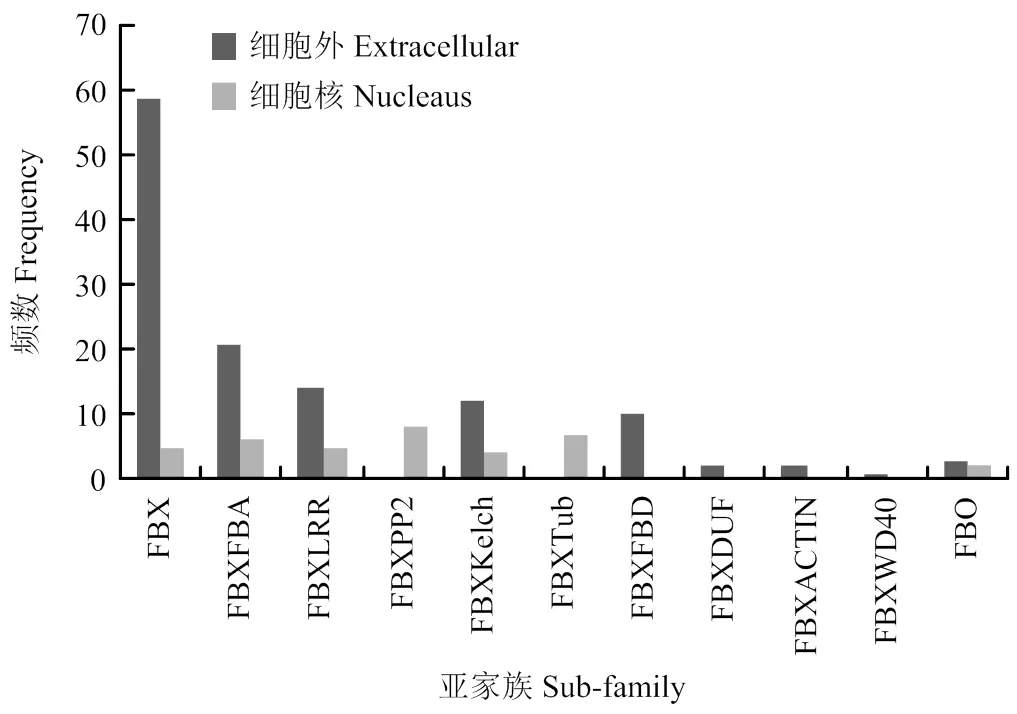
圖3 蠶豆F-box基因家族的亞細胞定位預測
利用在線軟件MEME(http://meme-suite.org/)分析蠶豆F-box基因家族成員基序類型和排列順序。如圖4所示,F-box基因家族預測含有20個Motif,F-box基因家族成員之間所包含的Motif數目及種類存在一定的差異。除了Vf036806.3和Vf033536.1不含Motif1外,其他均含有Motif1,相比Motif1,Motif2在蠶豆F-box基因家族中有60個成員不含該基序,Motif3在蠶豆F-box基因家族中有131個成員不含該基序。Motif4出現29次,Motif5出現17次,Motif6出現21次,Motif7出現35次,Motif8出現5次,Motif9出現11次,Motif10出現7次,Motif11出現51次,Motif12出現40次,Motif13出現39次,Motif14出現40次,Motif15出現42次,Motif16出現6次,Motif17出現6次,Motif18出現22次,Motif19出現25次,Motif20出現7次。結果表明,出現頻率較高的Motif為F-box基因家族中非常重要的保守基序。對于出現頻率較低的Motif,通過Smart在線分析,其功能的相關信息未知,有待日后進一步深入研究。
2.6 蠶豆F-box基因在鹽處理不同時間的表達模式分析
基因的表達模式可以為基因功能提供重要的依據,從蠶豆鹽脅迫轉錄組測序結果的基因表達數據庫中,分別提取161個F-box家族基因在不同品種(Y134 T2-T1為以Y078(不耐鹽)品種16 h鹽處理表達量為參照,Y134(耐鹽)品種16 h鹽處理與之相比較的差異表達量;CK2-CK1為以Y078(不耐鹽)品種16 h對照的表達量為參照,Y134(耐鹽)品種16h對照與之相比較的差異表達量,同理T4-T3為以Y078(不耐鹽)品種24 h鹽處理表達量為參照,Y134(耐鹽)品種24 h鹽處理與之相比較的差異表達量;CK4-CK3為以Y078(不耐鹽)品種24 h鹽處理表達量為參照,Y134(耐鹽)品種24 h鹽處理與之相比較的差異表達量。由圖5可知,隨著鹽處理時間的增加,蠶豆F-box基因的表達模式出現差異化。通過橫向比對每個家族成員的表達差異,發現大部分成員在不同的時間節點均有高表達和低表達,一部分成員在16 h處理表達量明顯升高,還有一些成員在24 h處理表達量明顯降低。說明這些家族成員可能在鹽脅迫前16 h中參與主要作用。為了進一步驗證上述結果,并確定F-box基因在蠶豆鹽脅迫下的參與情況,采用熒光定量PCR,在蠶豆2個品種和2個處理時間中進行差異表達分析(圖6)。選取5個差異表達基因(在鹽脅迫的2個時間點,以至少以一個時間節點的FDR<0.05,且差異倍數2倍及2倍以上的基因為差異表達基因),其中3個上調基因,2個下調基因。結果表明,5個蠶豆F-box基因的表達變化各不相同,其中在鹽處理16和24 h的表達量上調;在鹽處理16 h的表達量下調非常明顯,鹽處理24 h表達量無顯著差異;在鹽處理16 h上調非常明顯,在鹽處理24 h不明顯,后者與轉錄組數據不符;在鹽處理16 h表達量上調,鹽處理24 h表達量上調不明顯;在鹽處理16 h下調非常明顯,鹽處理24 h表達量上調,這與轉錄組數據不符。鹽處理16 h,qRT-PCR檢測到的5個基因,有4個基因相對表達量與轉錄組數據基本一致。
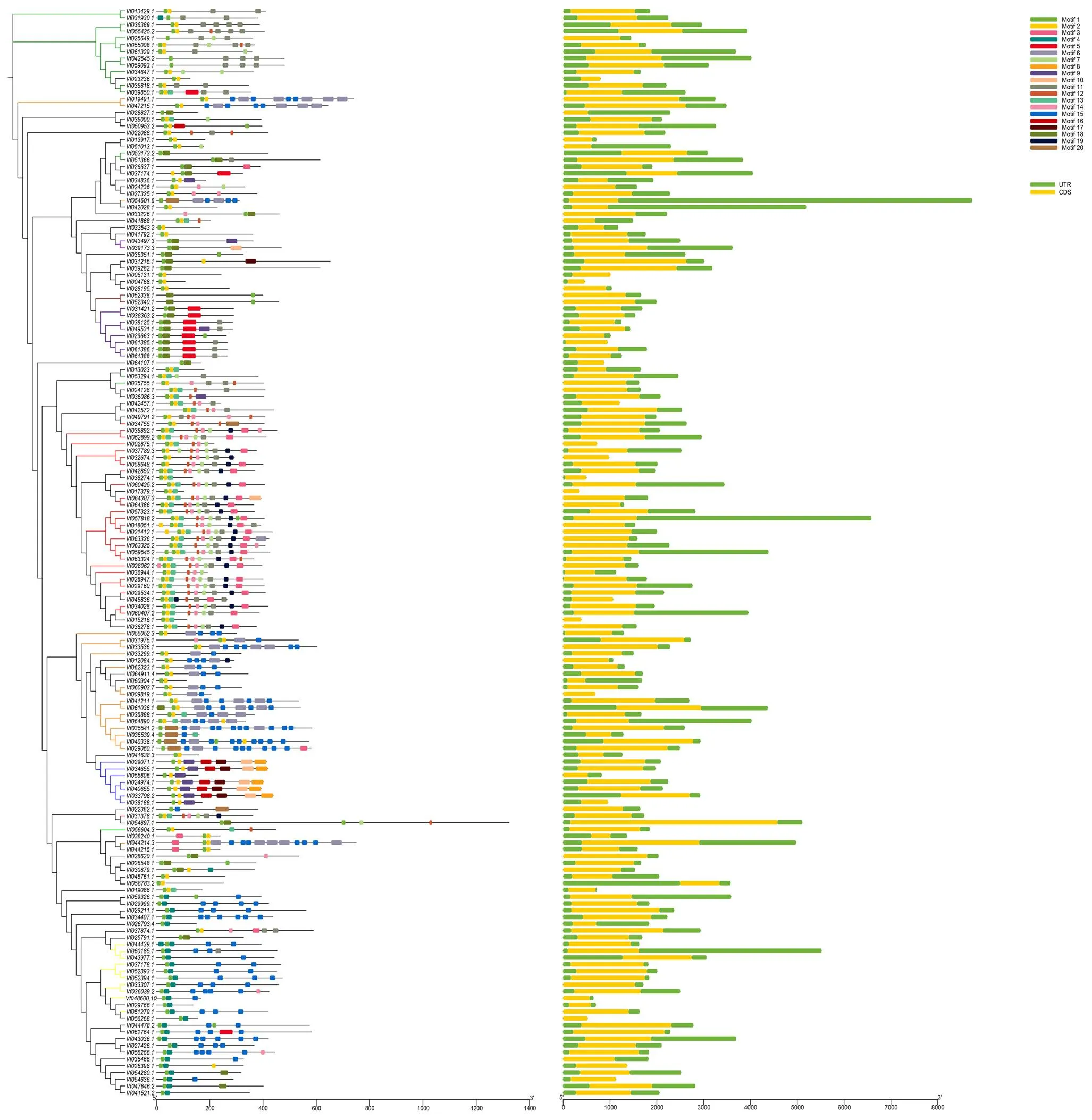
圖4 蠶豆F-box基因家族結構和Motif分析
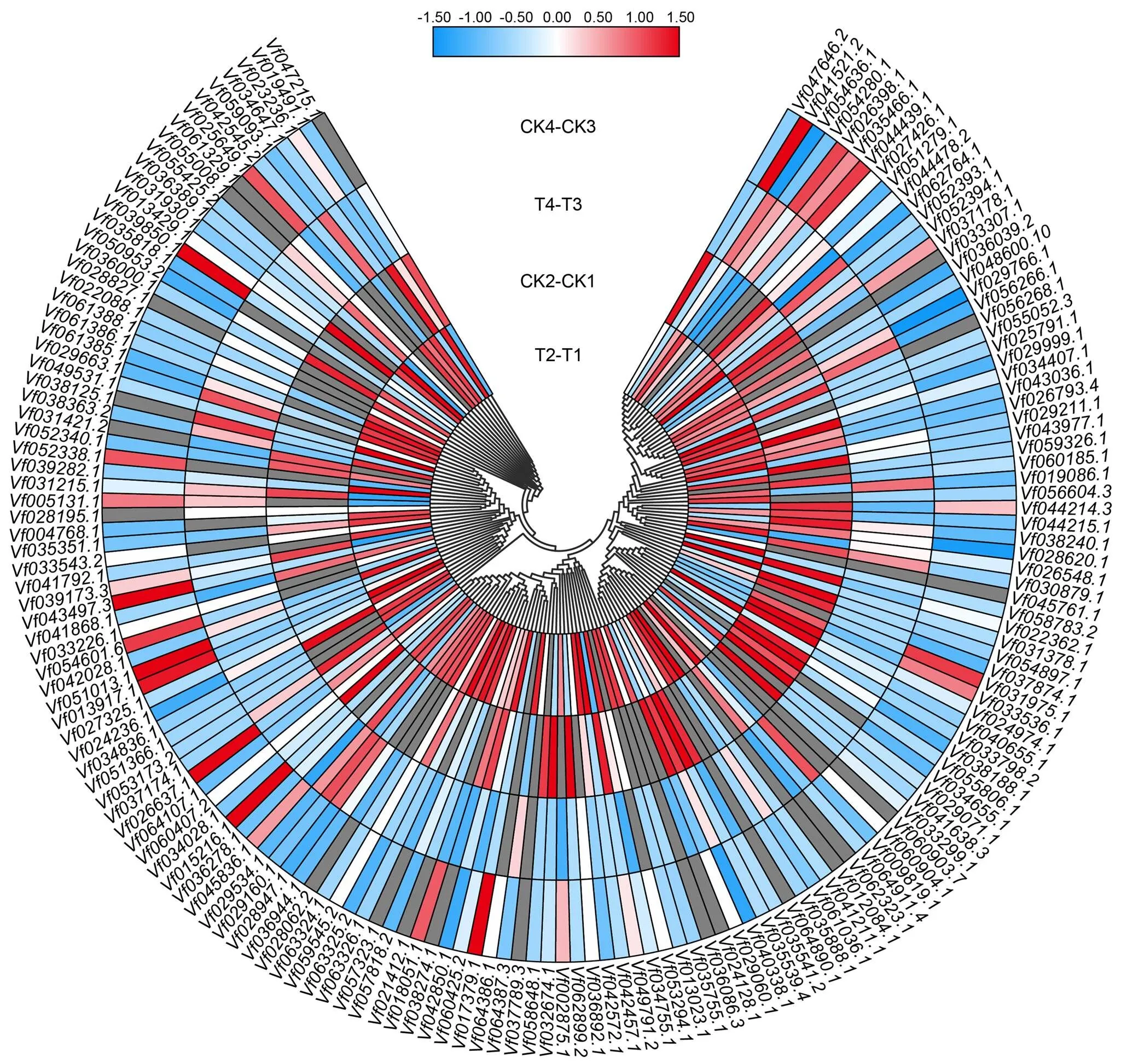
此圖基于蠶豆鹽脅迫轉錄組測序數據的計算得出,每種顏色代表相應表達量的數值,數值越大,表達量越高
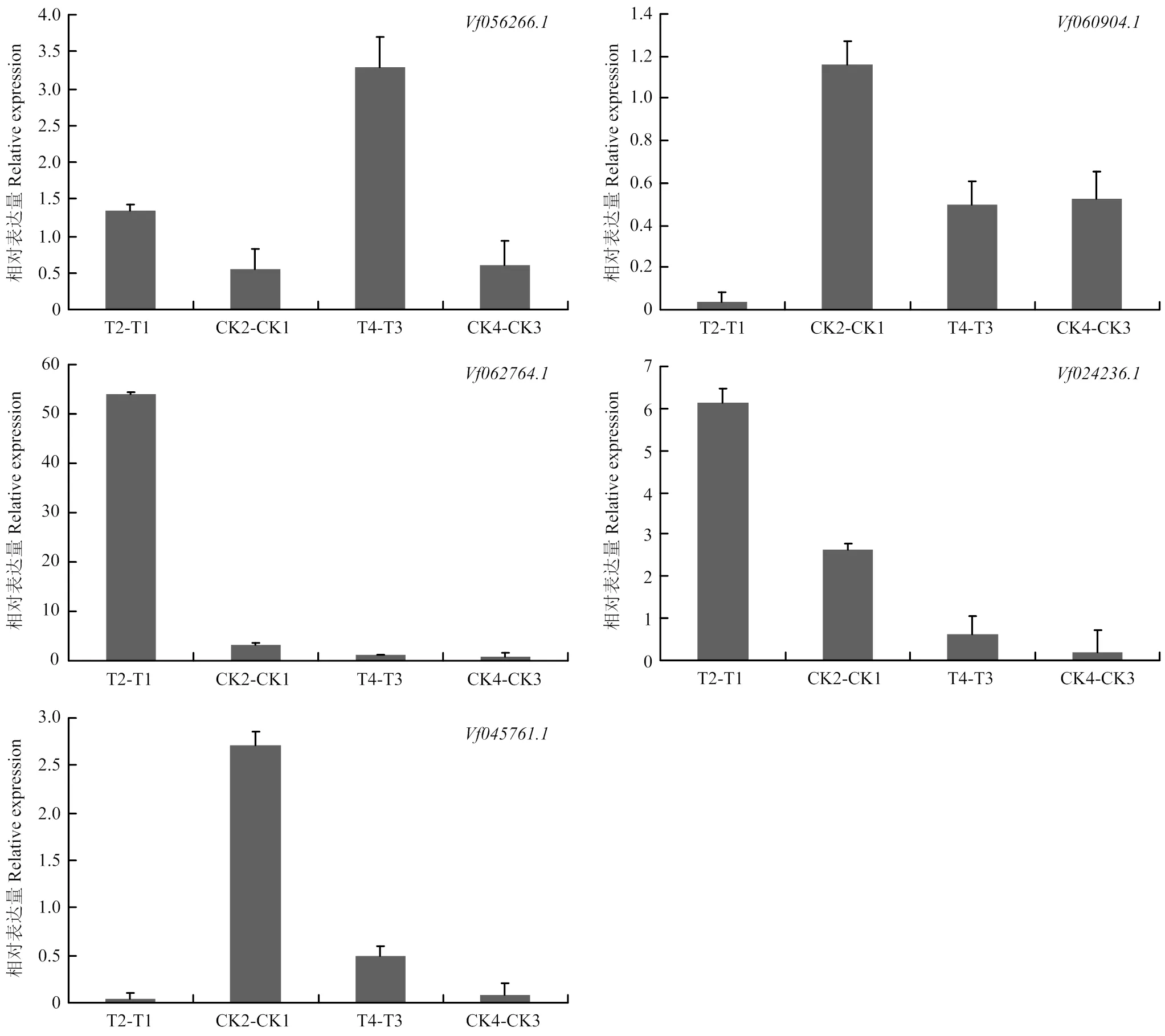
圖6 蠶豆F-box基因的實時熒光定量PCR
3 討論
在植物中,F-box作為一個較大的基因家族,它們參與了多種生物途徑,在整個植物生命周期的一系列生物學過程中發揮重要作用。然而,大多數植物F-box基因的功能仍然未知,特別是在蠶豆中。由于蠶豆的全基因組序列還未公布,在很大程度上限制了其重要性狀基因的研究。本研究基于蠶豆鹽脅迫轉錄組數據,對蠶豆F-box基因家族進行系統分析。根據基因結構、保守基序、系統發育關系將其劃分為不同亞族。在不同品種的不同鹽脅迫下,對其表達模式進行了分析,并通過qRT-PCR進行了驗證。
通過對蠶豆F-box蛋白的保守結構域進行基序分析,得出F-box基序中包含一個極度保守的色氨酸殘基,這與其他植物相類似。因此,蠶豆也有可能普遍形成SCF復合物。此外,大多數已知的蠶豆C端結構域也存在于其他植物中,表明進化基因的保守性以及F-box蛋白在不同植物中的相似性。根據C端結構域的不同,蠶豆F-box蛋白可分為11個亞家族,其中最豐富的F-box蛋白屬于FBX亞家族,含有未知的C端結構域。Motif分析結果表明,大多數基序也存在于其他植物中,而少數基序是豆科或蠶豆特有的。與其他已知的C端保守結構域一樣,這些推測的新基序也可能在F-box蛋白與其底物的相互作用中發揮作用。那些含有豆科或蠶豆特異性基序的蛋白可能具有與豆科特異性細胞過程相關的功能,但具體功能有待深入研究。
本文使用基于C端底物相互作用域的F-box基因分類標準,再次肯定了之前基于N端F-box基序的分類,說明N端F-box基序可能與C端基序共同進化,這一點與其他植物相似[13]。此外,F-box基序生成的系統發育樹與基因結構分析的結果是一致的,說明系統發育樹準確地代表了蠶豆F-box基因家族的系統發育關系。然而,蠶豆F-box家族基因結構中無內含子,無內含子F-box基因在擬南芥、水稻、玉米、鷹嘴豆和胡楊中所占比例較高[16]。由亞細胞定位預測結果可知,蠶豆F-box蛋白主要存在于細胞外和細胞核中,少數在細胞質、細胞質膜和液泡中。大量的豆類F-box蛋白被預測有多個亞細胞定位。據報道,在擬南芥中檢測到17個F-box蛋白大部分定位于胞腔內,單個F-box蛋白可以形成多個SCF復合物[37]。這些都說明了F-box蛋白在調控植物的生物學過程中具有多種作用。
基因表達模式與基因功能關系十分密切,通過研究蠶豆鹽脅迫過程中基因表達的變化,可發現參與鹽脅迫的相關功能基因。F-box基因被證實參與非生物脅迫反應,Zhao等[3]發現過表達的小麥的轉基因煙草各項生理指標均高于野生型。Jia等[38]發現擬南芥F-box基因通過抑制ROS積累和影響體內Na+平衡來提高植株的耐鹽性;An等[39]研究發現超表達能夠提高植株的耐鹽和耐旱性。本文基于蠶豆鹽脅迫轉錄組數據,分析F-box基因在蠶豆鹽脅迫下的差異表達模式,篩選出5個表達差異顯著的基因,用于進一步qRT-PCR分析。qRT-PCR驗證發現,有3個表達上調的差異基因和2個表達下調的差異基因,說明它們可能在蠶豆遭受鹽脅迫過程中起著正向或負向調控作用。為研究蠶豆中參與鹽脅迫過程中的相關F-box基因的功能和機制提供基礎。
擬南芥F-box家族蛋白功能的研究報道很多,UFO參與調節花器官發育[40],AtPP2-B11對耐旱性起負調控作用,提高鹽脅迫抗性,并參與ABA信號轉導過程[38],AtTR1參與鹽脅迫反應[41]。可根據進化樹中位于同一進化亞枝中的擬南芥蛋白功能,預測研究物種蛋白的功能,嚴莉等[42]通過構建擬南芥與黑果枸杞的MYB轉錄因子的系統進化樹,將位于相鄰位置的黑果枸杞和擬南芥MYB轉錄因子列為具有相同或相似結構的轉錄因子,從而推測其功能的一致性。本研究所得的161個蠶豆F-box蛋白與15個功能明確的擬南芥F-box蛋白作系統進化樹,根據15個擬南芥F-box蛋白進化關系的遠近推測蠶豆F-box蛋白的功能。如Vf002875.1與At3g16740相鄰,根據擬南芥At3g16740的功能推測Vf002875.1可能參與影響種子萌發的過程[43];推測Vf061385.1可能與擬南芥AtPP2-B11功能一致,參與鹽脅迫反應[38];Vf026793.4與擬南芥TOC1進化關系較近,可能參與生物鐘調節[44]。通過對擬南芥F-box蛋白保守結構域的搜索發現,含有同一C端結構域的蛋白進化到同一亞枝或進化關系較近,證明相鄰或較近進化關系上的蛋白有可能具有相同或相似的功能,為研究F-box蛋白的功能提供了一種思路。此外,在后續研究中,將對F-box基因家族成員進行轉基因試驗,分析其在鹽脅迫及其他非生物脅迫中的具體表型影響。
4 結論
基于蠶豆鹽脅迫轉錄組測序數據,篩選注釋了161個蠶豆F-box基因,且均含有F-box保守結構域。根據C末端氨基酸序列不同使家族成員多樣化,分為11個亞家族,家族成員均無內含子結構,且家族成員在胞外和細胞核中均存在。、和正調控鹽脅迫,和負調控鹽脅迫,且鹽脅迫下的F-box家族成員在不同處理時間下呈現出不同的響應。
[1] 王海飛, 關建平, 孫雪蓮, 馬鈺, 宗緒曉. 世界蠶豆種質資源遺傳多樣性和相似性的ISSR分析. 中國農業科學, 2011, 44(5): 1056-1062.
Wang H F, Guan J P, Sun X L, Ma Y, Zong X X. Genetic diversity and similarity of global faba bean (L.) germplasm revealed by ISSR markers., 2011, 44(5): 1056-1062. (in Chinese)
[2] Baute J, Polyn S, De Block J, Blomme J, Van Lijsebettens M, Inzé D. F-Box protein FBX92 affects leaf size in., 2017, 58(5): 962-975.
[3] Zhao Z, Zhang G, Zhou S, Ren Y, Wang W. The improvement of salt tolerance in transgenic tobacco by overexpression of wheat F-box gene TaFBA1., 2017, 259: 71-85.
[4] Stefanowicz K, Lannoo N, Zhao Y, Eggermont L, Van Hove J, Al Atalah B, Van Damme E J. Glycan-binding F-box protein fromprotects plants frominfection., 2016, 16(1): 213-226.
[5] Smalle J, Vierstra R D. The ubiquitin 26S proteasome proteolytic pathway., 2004, 55: 555-590.
[6] Sadanandom A, Bailey M, Ewan R, Lee J, Nelis S. The ubiquitin-proteasome system: Central modifier of plant signalling., 2012, 196(1): 13-28.
[7] Ho M S, Ou C, Chan Y R, Chien C T, Pi H. The utility Fbox for protein destruction., 2008, 65(13): 1977-2000.
[8] Somers D E, Fujiwara S. Thinking outside the F-box: Novel ligands for novel receptors., 2009, 14(4): 206-213.
[9] Hua Z, Zou C, Shiu S H, Vierstra R D. Phylogenetic comparison of F-Box (FBX) gene superfamily within the plant kingdom reveals divergent evolutionary histories indicative of genomic drift., 2011, 6(1): e16219.
[10] Navarro-Quezada A, Schumann N, Quint M. Plant F-box protein evolution is determined by lineage-specific timing of major gene family expansion waves., 2013, 8(7): e68672.
[11] Xu G, Ma H, Nei M, Kong H. Evolution of F-box genes in plants: Different modes of sequence divergence and their relationships with functional diversification., 2009, 106(3): 835-840.
[12] Kipreos E T, Pagano M. The F-box protein family., 2000, 1(5): 1-7.
[13] Song J B, Wang Y X, Li H B, Li B W, Zhou Z S, Gao S, Yang Z M. The F-box family genes as key elements in response to salt, heavy mental, and drought stresses in., 2015, 15(4): 495-507.
[14] Jia Q, Xiao Z X, Wong F L, Sun S, Liang K J, Lam H M. Genome-wide analyses of the soybean F-box gene family in response to salt stress., 2017, 18(4): 818-835.
[15] JaIN M, NIJHAWAN A, ARORA R, AGARWAL P, RAY S, SHARMA P, KAPOOR S, TYAGI A K, KHURANA J P. F-Box proteins in rice. genome-wide analysis, classification, temporal and spatial gene expression during panicle and seed development, and regulation by light and abiotic stress., 2007, 143(4): 1467-1483.
[16] Cui H R, Zhang Z R, Lü W, Xu J N, Wang X Y. Genome-wide characterization and analysis of F-box protein-encoding genes in the Malus domestica genome., 2015, 290(4): 1435-1446.
[17] Gupta S, Garg V, Kant C, Bhatia S. Genome-wide survey and expression analysis of F-box genes in chickpea., 2015, 16(1): 1-15.
[18] Wang G M, Yin H, Qiao X, Tan X, Gu C, Wang B H, Cheng R, Wang Y Z, Zhang S L. F-box genes: Genome-wide expansion, evolution and their contribution to pollen growth in pear ()., 2016, 253: 164-175.
[19] 王秀燕, 孫莉萍, 張建鋒, 李輝, 呂文清, 張其清. F-box蛋白家族及其功能. 生命科學, 2008, 20(5): 807-811.
Wang X Y, Sun L P, Zhang J F, Li H, Lü W Q, Zhang Q Q. F-box proteins and their functions., 2008, 20(5): 807-811. (in Chinese)
[20] Hepworth S R, Klenz J E, Haughn G W. UFO in theinflorescence apex is required for floral-meristem identity and bract suppression., 2006, 223(4): 769-778.
[21] Kepinski S, Leyser O. TheF-box protein TIR1 is an auxin receptor., 2005, 435(7041): 446-451.
[22] Walsh T A, Neal R, Merlo A O, Honma M, Hicks G R, Wolff K, Matsumura W, Davies J P. Mutations in an auxin receptor homolog AFB5 and in SGT1b confer resistance to synthetic picolinate auxins and not to 2,4-dichlorophenoxyacetic acid or indole-3-acetic acid in., 2006, 142: 542-552.
[23] Gomi K, Sasaki A, Itoh H, Ueguchi-Tanaka M, Ashikari M, Kitano H, Matsuoka M. GID2, an F-box subunit of the SCF E3 complex, specifically interacts with phosphorylated SLR1 protein and regulates the gibberellin-dependent degradation of SLR1 in rice., 2004, 37(4): 626-634.
[24] Gagne J M, Smalle J, Gingerich D J, Walker J M, Yoo S D, Yanagisawa S, Vierstra R D.EIN3-binding F-box 1 and 2 form ubiquitin-protein ligases that repress ethylene action and promote growth by directing EIN3 degradation., 2004, 101(17): 6803-6808.
[25] Sheard L B, Tan X, Mao H, Withers J, Ben-Nissan G, Hinds T R, Kobayashi Y, Hsu F F, Sharon M, Browse J, He S Y, Rizo J, Howe G A, Zheng N. Jasmonate perception by inositolphosphate-potentiated COI1-JAZ co-receptor., 2010, 468(7322): 400-405.
[26] Imaizumi T, Schultz T F, Harmon F G, Ho L A, Kay S A. FKF1 F-box protein mediates cyclic degradation of a repressor of CONSTANS in., 2005, 309(5732): 293-297.
[27] Zhang Y, Xu W Y, Li Z H, Deng X W, Wu W H, Xue Y B. F-Box protein DOR functions as a novel inhibitory factor for abscisic acid-induced stomatal closure under drought stress in., 2008, 148(4): 2121-2133.
[28] Song S, Dai X, Zhang W H. A rice F-box gene, OsFbx352, is involved in glucose-delayed seed germination in rice., 2012, 63(15): 5559-5568.
[29] Sonneveld T, Tobutt K R, Vaughan S P, Robbins T P. Loss of pollen-S function in two self-compatible selections of Prunus avium is associated with deletion/mutation of an S haplotype-specific F-Box gene., 2005, 17(1): 37-51.
[30] Grabherr M G, Haas B J, Yassour M, Levin J Z, Thompson D A, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q, Chen Z, Mauceli E, Hacohen N, Gnirke A, Rhind N, di Palma F, Birren B W, Nusbaum C, Lindblad- Toh K, Friedman N, Regev A. Full-length transcriptome assembly from RNA-Seq data without a reference genome., 2011, 29(7): 644-652.
[31] Chen C J, Xia R, Chen H, He Y H. TBtools, a Toolkit for biologists integrating various biological data handing tools with a user friendly interface., 2018, 3(27): 1020-1027.
[32] Wang G M, Yin H, Qiao X, Tan X, Gu C, Wang B H, Cheng R, Wang Y Z, Zhang S L. F-box genes: Genome-wide expansion, evolution and their contribution to pollen growth in pear ()., 2016, 253: 164-175.
[33] Kou Y, Qiao L, Wang Q. RETRACTED ARTICLE: Identification of core miRNA based on small RNA-seq and RNA-seq for colorectal cancer by bioinformatics., 2015, 36(4): 2249-2255.
[34] Zhang H M, Wheeler S L, Xia X, Colyvas K, Offler C E, Patrick J W. Transcript profiling identifies gene cohorts controlled by each signal regulating trans-differentiation of epidermal cells ofcotyledons to a transfer cell phenotype., 2017, 8: 2021.
[35] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT-method., 2001, 25(4): 402-408.
[36] Bai C, Sen P, Hofmann K, Ma L, Goebl M, Harper J W, Elledge S J. SKP1 connects cell cycle regulators to the ubiquitin proteolysis machinery through a novel motif, the F-box., 1996, 86(2): 263-274.
[37] Kuroda H, Yanagawa Y, Takahashi N, Horii Y, Matsui M. A comprehensive analysis of interaction and localization ofSKP1-like (ASK) and F-box (FBX) proteins., 2012, 7(11): e50009.
[38] Jia F Y, Wang C Y, Huang J G, Yang G D, Wu C G, Zheng C C. SCF E3 ligase PP2-B11 plays a positive role in response to salt stress in., 2015, 66(15): 4683-4697.
[39] An J P, Li R, Qu F J, You C X, Wang X F, Hao Y J. Apple F-Box protein MdMAX2 regulates plant photomorphogenesis and stress response., 2016, 7: 01685.
[40] Hepworth S R, Klenz J E, Haughn G W. UFO in theinflorescence apex is required for floral-meristem identity and bract suppression., 2006, 223(4): 769-778.
[41] 劉巧紅, 楊亮, 劉志斌, 李旭鋒, 楊毅. 擬南芥AtTR1在鹽脅迫應答中的功能初探. 四川大學學報(自然科學版), 2016, 53(4): 895-901.
Liu Q H, Yang L, Liu Z B, Li X F, Yang Y. First exploration on protein function of Arabidopsis AtTR1 in response to salt stress., 2016, 53(4): 895-901. (in Chinese)
[42] 嚴莉, 王翠平, 陳建偉, 喬改霞, 李健. 基于轉錄組信息的黑果枸杞MYB轉錄因子家族分析. 中國農業科學, 2017, 50(20): 3991-4002.
Yan L, Wang C P, Chen J W, Qiao G X, Li J. Analysis of MYB Transcription Factor Family Based on Transcriptome Sequencing inMurr., 2017, 50(20): 3991-4002. (in Chinese)
[43] 段桂芳, 王立群, 李新梅, 趙福, 羅俊, 趙小英, 劉選明. 擬南芥F-box基因At3g16740的表達分析. 生命科學研究, 2013, 17(6): 486-492.
Duan G F, Wang L Q, Li X M, Zhao F, Luo J, Zhao X Y, Liu X M. Expression Analysis of F-box Gene At3g16740 in., 2013, 17(6): 486-492. (in Chinese)
[44] Más P, Kim W Y, Somers D E, Kay S A. Targeted degradation of TOC1 by ZTL modulates circadian function in., 2003, 426(6966): 567-570.
Analysis of F-Box gene family based on salt-stressed transcriptome sequencing inL
HAO ShuLin1, CHEN HongWei2, LIAO FangLi3, LI Li2, LIU ChangYan2, LIU LiangJun2, WAN ZhengHuang2, SHA Aihua1
(1College of Agriculture, Yangtze University/Hubei Collaborative Innovation Center for Grain Industry, Jingzhou 434025, Hubei;2Institute of Food Crops, Hubei Academy of Agricultural Sciences/Hubei Key Laboratory of Food Crop Germplasm and Genetic Improvement, Wuhan 430064;3Seed Authority of Jingzhou, Jingzhou 434020, Hubei)
【】The distribution structure and evolution of F-box gene family members inwere analyzed by bioinformatics method to study the expression patterns of family members and their responses to salt stress under different treatment times. It can provide a reference for the study of the biological function and the mechanism of F-box genes.【】Based on the salt-stressed transcriptome sequencing (RNA-seq) data, the NR, Swiss-Prot, PFAM and NCBI websites were used at the same time to screen and annotate the F-box genes of. The softwares including Web Logo 3, Prot Comp 9.0, MEGA-X and MEME were also applied to analyze the bioinformatics of conserved domain, subcellular localization, phylogenetic tree and Motif. Based on salt stress transcriptome data, the differential expression patterns of F-box gene family in(yz17134 salt tolerance and yz17078 salt intolerance) under salt stress were analyzed, and real-time fluorescence quantitative PCR (qrt-pcr) was used to verify the specific expression of part family members at 16 h and 24 h.】Based on salt-stressed transcriptome sequencing (RNA-seq) data, 161F-box genes were annotated and all contain F-box conserved domain. According to different C-terminal domains, they were divided into 11 subfamilies (FBX, FBXFBA, FBXLRR, FBXPP2, FBXKelch, FBXTUB, FBXFBD, FBXDUF, FBXACTIN, FBXWD40, and FBO). The analysis of the conserved domain showed that the F-box conserved Motif contained an extremely conserved tryptophan residue. By comparing and analyzing the evolutionary tree constructed by the F-box family ofand the F-box family of, it was found that most of the genes in the same C-terminal domain were clustered together. The results of subcellular localization prediction showed that 124 F-box genes were located outside the cell, and 37 F-box genes were located in the nucleus. The analysis of gene structure showed that there were no introns in the DNA sequences of the F-box family genes of, and all of them were composed of UTR zone and CDS zone. Analysis of F-box differential expression patterns based on salt-stressed transcriptome data showed that F-box gene expression inwas diverse from each other at two different processing time points, the expression was more obvious at 16 hours after salt treatment. The results of qRT-PCR analysis showed that there were five different genes in the F-box family. The expressions of,andwere all up-regulated at 16 hours after salt treatment,andwere both down-regulated at 16 hours after salt treatment.【】161F-box genes were identified by family annotation, and they were identified by family annotation, which were Evolutionarily divided into 11 subfamilies. 5 important genes were obtained through a series of bioinformatics analysis. What’s more, there exist difference among the expressions in diverse salt treatment time.
L; RNA-seq; F-box gene family; salt stress; expression pattern

10.3864/j.issn.0578-1752.2020.17.003
2019-12-19;
2020-03-05
國家重點研發計劃(2019YFD1001303/2019YFD1001300)、國家現代農業產業技術體系建設項目(CARS-09)、國家食用豆產業技術體系建設專項(CARS-08-G13)、湖北省技術創新重大專項(2016ABA087,2018ABA090)
郝樹琳,E-mail:shulinlin6@163.com。通信作者沙愛華,E-mail:aihuasha@163.com。通信作者萬正煌,E-mail:zhwan168@163.com
(責任編輯 李莉)

