梯棱羊肚菌分子鑒別研究
沈千匯 李春紅 謝美霞 李文佳 錢正明,3* 張 霽,*
梯棱羊肚菌分子鑒別研究
沈千匯1李春紅2謝美霞2李文佳2錢正明2,3*張 霽1,2*
(1. 華南師范大學腦科學與康復醫學研究院,廣東 廣州 510631;2. 廣東東陽光藥業有限公司國家中醫藥管理局重點研究室,廣東 東莞 523850;3. 湘南學院,湖南 郴州 423000)
針對羊肚菌品種,特別是產業化培植品種,建立快速準確的品種鑒定方法。通過對來源確切的梯棱羊肚菌樣品及同屬5種其他羊肚菌進行ITS測序,并結合網絡數據庫中的梯棱羊肚菌ITS序列進行分析,獲得梯棱羊肚菌的特異性鑒別引物。進一步對分子鑒別實驗條件進行考察,以確定最終實驗條件。通過對梯棱羊肚菌及羊肚菌近緣品種的分析,發現只有梯棱羊肚菌呈現特征電泳條帶。表明該研究設計的特異性引物和優化的實驗方法,可用于梯棱羊肚菌菌種的快速鑒定及產品的真偽鑒別。
梯棱羊肚菌;分子鑒別;特異性引物;PCR反應
羊肚菌又稱羊肚蘑、草笠竹等,因其菌蓋多褶皺,子實體似羊肚狀凹陷而得名[1,2]。羊肚菌為我國傳統中藥材和珍稀食材,具有很高的藥用及食用價值[3, 4]。一方面由于價格高昂,市場上存在偽品銷售的現象,如皺蓋鐘菌和鹿花菌[5, 6]。偽品若加工處理不當,食用后會出現中毒現象[6, 7],另一方面,羊肚菌人工栽培近年來得到快速發展,但其菌種存在品種銷售混亂等問題。因此,建立快速準確的羊肚菌品種鑒定方法,特別是針對羊肚菌產業化培植品種的真偽鑒別,對保障產業的健康發展具有重要意義。
分子鑒別技術具有準確率高、操作簡便等優點,已被廣泛應用于冬蟲夏草、光慈菇等食藥用真菌的真偽鑒別[8-10]。有文獻報道采用ITS序列分析方法可鑒定梯棱羊肚菌真偽,但需要的樣本量大,測序成本高,分析耗時長,如果樣品DNA有斷裂或降解則有可能無法進行鑒別[11, 12]。特異性引物分子鑒別是一種專屬性強、鑒定速度快的分析方法。本課題組前期采用該技術建立了產業化品種六妹羊肚菌的快速真偽鑒別方法[13]。而另一個大品種梯棱羊肚菌的快速真偽鑒別研究尚未見報道。本試驗對梯棱羊肚菌進行特異性引物分子鑒別研究,可為該品種的質量評價技術提升提供支持。
1 材料與方法
1.1 供試材料
本試驗收集了18批羊肚菌樣品,其中S1~S11均為梯棱羊肚菌,S12~S18為其他品種羊肚菌。相關信息如下:S1采自廣東東莞、S2~S4采自廣東韶關、S5采自貴州遵義、S6采自四川崇州、S7采自四川南充、S8采自云南麗江、S9采自四川成都、S10采自四川綿陽、S11采自四川廣元;S12、S13為六妹羊肚菌,分別采自四川南充和成都;S14、S15為粗柄羊肚菌,來自北京;S16為七妹羊肚菌,來自北京;S17為高羊肚菌,來自北京;S18為小海綿羊肚菌,來自北京。
10條網絡數據庫中的梯棱羊肚菌ITS系列信息如下:MN462953.1和MN462954.1來自伊比利亞半島,MK447933.1來自卡拉布里亞(意大利),MH423881.1和MH468775.1來自秦嶺地區,MK432691.1來自新西蘭,KU865008.1來自塞浦路斯,MK253759.1來自湖南,MH198762.1來自北美西部,MF170632.1來自以色列。
1.2 試劑與儀器
試劑包括:高效植物基因組DNA提取試劑盒(北京天根生化科技有限公司);PrimeSTAR?Max DNA Polymerase(含HS DNA聚合酶),DL500 DNA Marker,10×Loading Buffer(寶日醫生物技術北京有限公司);瓊脂糖,GelRed Nucleic Acid Gelstain(北京全式金生物技術有限公司)。
儀器包括:Vortex Genie 2渦旋振蕩器(Scientific Industries, Inc.),XS-205DR電子天平(梅特勒-托利多儀器有限公司),MM400混合球磨儀(德國萊馳公司),HWS-24電熱恒溫水浴鍋(上海一恒科學儀器有限公司),5425離心機(艾本德中國有限公司),ProFlex 3*32 well PCR儀(美國應用生物系統(ABI)公司),ND-2000核酸蛋白儀(美國賽默飛世爾科技公司),XGLDTED脈動真空滅菌器(山東新華醫療器械股份有限公司),Aqbd Cubee迷你離心機(瑞基海洋生物科技股份有限公司),Tanon 3500凝膠成像系統(上海天能科技有限公司),PJ210C-BF微波爐(美的集團)。
1.3 方法
(1)樣品DNA提取。提取羊肚菌樣品(S1~S18)DNA,取適量樣品采用混合型球磨儀粉碎。取樣品粉末約30 mg至離心管中,使用高效植物基因組DNA試劑盒提取總DNA,并使用核酸蛋白儀測定DNA濃度,將所有樣品DNA濃度分別稀釋至50 ng/μL,分裝放置在-20 ℃下備用。
(2)通用引物PCR擴增。采用真菌通用引物(ITS5F/ITS4R)對所提取的樣品DNA進行PCR擴增并測序,得到樣品ITS序列。引物信息為ITS5F:5'-GGAAGTAAAAGTCGTAACAAGG-3',ITS4R:5'-TCCTCCGCTTATTGATATGC-3。PCR擴增體系總體積25 μL,包含2 × PrimeSTAR Max premix 12.5 μL,ITS5F/ITS4R(10 μmol/L)各1 μL,DNA模板1 μL,雙蒸水補足至25 μL,并設置陰性對照組(等體積雙蒸水代替DNA模板)。PCR反應參數為:94 ℃預變性3 min;94 ℃變性45 s,62 ℃退火45 s,72 ℃延伸1 min,35個循環;72 ℃最后延伸10 min。將PCR產物經過1.5%瓊脂糖凝膠電泳檢測后,采用自動熒光凝膠成像儀檢測目標條帶,之后將PCR產物送至廣州艾基生物有限公司進行雙向測序并拼接。
(3)序列分析和引物設計。以11批梯棱羊肚菌的ITS序列和10條該品種網絡序列為基礎,設計梯棱羊肚菌的特異性引物。所使用的梯棱羊肚菌ITS序列均采用GenBank和MLST數據庫驗證其準確性[12]。應用DNAMAN軟件對11批梯棱羊肚菌的序列和Genbank中下載的10條可靠的梯棱羊肚菌ITS序列進行分析、校對,找出穩定差異的變異位點,并使用Primer Premier 5.0軟件對梯棱羊肚菌的特異位點設計了3對特異性鑒別引物對,命名為TL-1s / TL-1a(TL-1s:GTTTGAT TCTGACGTCGGC / TL-1a:CACCAGGGCTAGTA GCTTTAC,166 bp)、TL-2s / TL-2a(TL-2s:ATGA CGCTCGAACAGGCATG / TL-2a:GCCGACGTC AGA ATCATAAC,191 bp)、TL-3s / TL-3a(TL-3s:ATGACGCTCGAACAGGCATG / TL-3a:CACCAG GGCTAGTAGCTTTAC,337 bp),引物由廣州艾基生物有限公司合成。
(4)引物篩選及特異性引物PCR條件優化。對所設計的引物對進行篩選和擴增反應條件的優化。以梯棱羊肚菌(S5)DNA為模板,用所設計的3對引物進行擴增,PCR反應采用25 μL反應體系:含2 × PrimeSTAR Max premix 12.5 μL,上下游引物各1 μL(濃度為10 μmol/L),雙蒸水 9.5 μL,DNA模板1 μL。PCR參數為:94 ℃預變性3 min;94 ℃變性45 s,58 ℃退火45 s,72 ℃延伸1 min,35個循環;72 ℃最后延伸10 min。PCR反應結束后使用1.5%瓊脂糖凝膠電泳進行檢測。
特異性引物PCR條件優化。對特異性引物TL-1s / TL-1a的PCR條件進行以下參數優化:①退火溫度設62 ℃、64 ℃、66 ℃、68 ℃;②退火時間設10 s、15 s;③延伸時間設10 s、15 s;④反應輪數設30輪、35輪。
(5)方法學考察。分子鑒別研究通常會進行專屬性、靈敏度、耐用性的方法學考察[9, 14]。①專屬性考察:對所有樣品的DNA模板進行PCR擴增,驗證所設計特異性引物的專屬性,PCR反應參數為“1.3(4)”節優化后的條件,擴增產物使用1.5%瓊脂糖凝膠電泳進行檢測。②靈敏度考察:將梯棱羊肚菌(S5)DNA溶液(50 ng/μL)連續稀釋至不同濃度(30 ng/μL、10 ng/μL、1 ng/μL、0.1 ng/μL、0.01 ng/μL),PCR反應參數為“1.3(4)”節優化后的條件,擴增產物使用1.5%瓊脂糖凝膠電泳進行檢測。③耐用性考察:PCR擴增體系中使用不同體積的引物(0.75 μL,1.0 μL,1.25 μL,1.5 μL),PCR反應參數為“1.3(4)”節優化后的條件,擴增產物使用1.5%瓊脂糖凝膠電泳進行檢測。
2 結果與分析
2.1 引物篩選及PCR反應條件優化
將3對特異性引物PCR擴增產物通過瓊脂糖凝膠檢測,結果表明,引物對TL-1s / TL-1a在166 bp左右能成功擴增出單一的目標條帶,另外2對引物顯示出假陽性擴增結果和多條非特異性條帶,因此選擇引物對TL-1s / TL-1a作為梯棱羊肚菌的鑒別引物,并進一步進行PCR反應參數優化。
利用引物TL-1s / TL-1a進行反復試驗,最終確定其在退火溫度為68 ℃,退火時間為15 s,延伸時間為10 s,反應輪數為35個循環(表1)條件下,不僅能保證擴增穩定進行,而且擴增效率最好。

表1 特異性引物TL-1s/TL-1a的PCR反應條件
2.2 方法學考察
(1)引物專屬性考察。利用優化后的反應參數,對11批次的梯棱羊肚菌和7批其他近緣種羊肚菌樣品進行分析,結果顯示所有的梯棱羊肚菌樣品在約166 bp位置均出現唯一目標條帶,而陰性對照和其他樣品在相應位置均無條帶出現(圖1),表明該方法專屬性良好,能準確地鑒別梯棱羊肚菌與羊肚菌近緣種。
(2)引物靈敏度考察。通過將濃度為50 ng/μL梯棱羊肚菌DNA溶液(樣品S5)稀釋至不同濃度進行擴增,當濃度為30 ng/μL和10 ng/μL時,目的條帶沒有明顯的差異;當DNA濃度低至約0.1 ng/μL時,PCR產物顯示出淺淡的單一目的條帶(圖2),表明該方法靈敏度高。
(3)引物耐用性考察。通過使用不同引物量(0.75 μL、1 μL、1.25 μL、1.5 μL)進行擴增,結果顯示樣品在約166 bp位置均出現唯一特征性條帶,表明本方法耐用性良好(圖3)。
3 結論與討論
目前羊肚菌培植種源復雜、菌種市場品種摻假混雜等問題嚴重制約了產業的穩步發展,故羊肚菌品種的鑒定研究對于羊肚菌的產業化栽培和市場產品的質量控制具有重要的意義[15, 16]。近年來,DNA條形碼因能快速準確鑒定物種的優勢逐漸成為鑒定動植物和真菌的熱點技術。其中,ITS序列已被提出可作為真菌遺傳鑒定的條形碼[17, 18]。現有文獻報道了采用ITS序列分析法鑒定梯棱羊肚菌,但用該方法成本高、耗時長,而且若樣品中DNA發生降解,ITS序列的通用引物擴增可能不成功,導致無法進行分析[9, 19]。
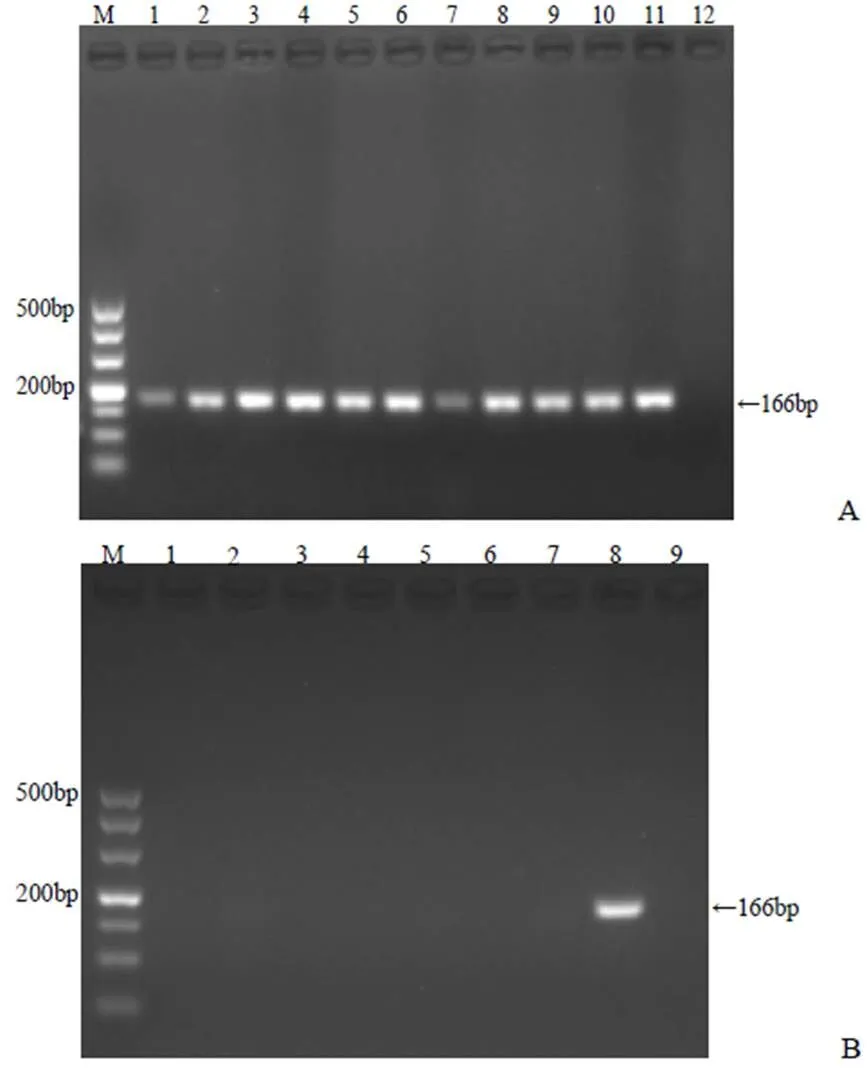
圖A:11批梯棱羊肚菌擴增電泳圖。M:DL 500 Marker;泳道1~11分別為樣品S1~S11;泳道12:陰性對照。
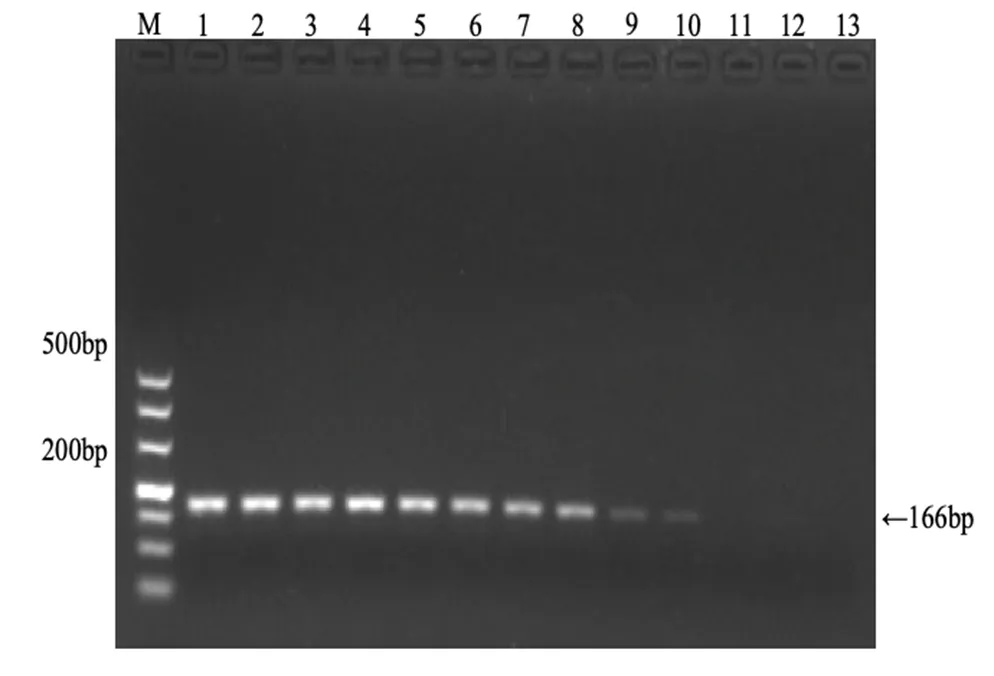
M:DL 500 Marker;泳道1,2:DNA濃度為50 ng/μL;泳道3,4:DNA濃度為30 ng/μL;泳道5,6:DNA濃度為10 ng/μL;泳道7,8:DNA濃度為1 ng/μL;泳道9,10:DNA濃度為0.1 ng/μL;泳道11,12:DNA濃度為0.01 ng/μL;泳道13:陰性對照。
特異性引物是根據物種間序列差異設計的一段特有DNA序列,在適宜條件下通過特異性擴增,便可完成對指定物種的特異性檢驗,具有快速、簡便和特異性強的優點[13, 14]。本研究基于DNA條形碼技術,以梯棱羊肚菌為研究對象,比較序列的差異,在ITS區域內設計了該品種的特異性引物(約166 bp),并確定其最佳PCR反應條件為:98 ℃預變性1 min;98 ℃ 15 s,68 ℃ 15 s,72 ℃ 10 s進行35個循環;72 ℃最后延伸5 min。該PCR方法耗時約30 min,檢測效率高;對不同品種羊肚菌、不同DNA模板濃度、不同引物量的測試結果顯示,其具有良好的專屬性、靈敏度和耐用性。
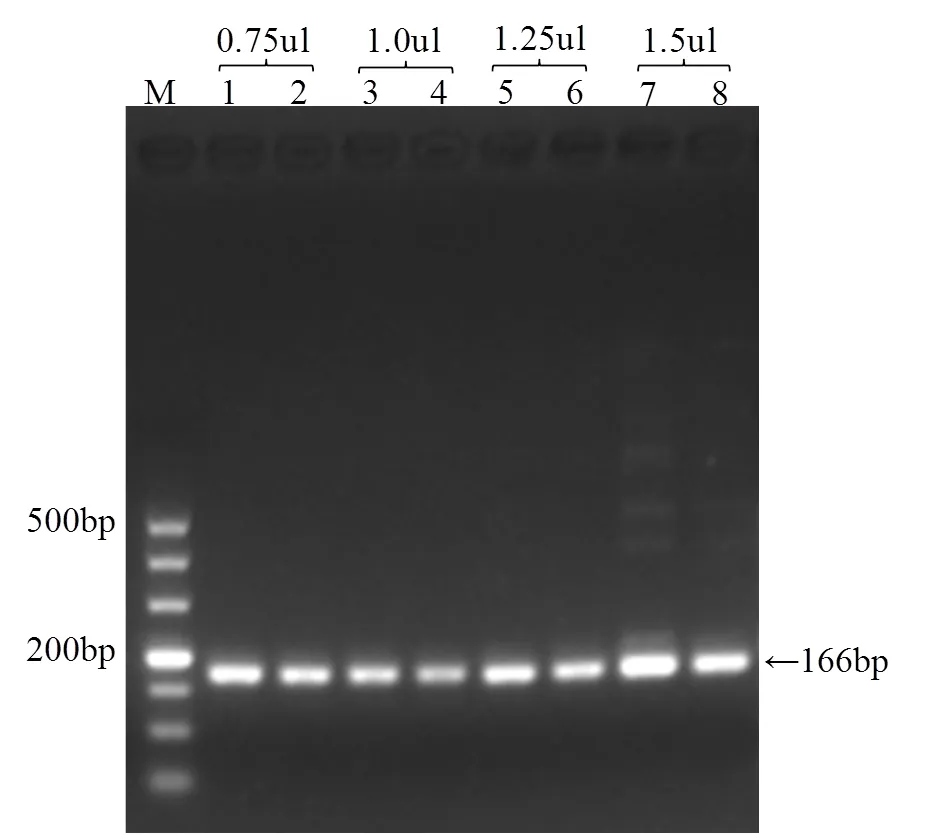
M: DL 500 Marker;泳道1,2:引物量為0.75 μL;泳道3,4:引物量為1.0 μL;泳道5,6:引物量為1.25 μL;泳道7,8:引物量為1.5 μL。
本試驗采用電子PCR驗證所設計引物的應用廣泛性,在使用NCBI的Primer-Blast過程中發現,有非梯棱羊肚菌也可能被擴增出來,如六妹羊肚菌(MH468776.1)。進一步通過MLST數據庫對六妹羊肚菌(MH468776.1)序列進行比對發現,該序列實際為梯棱羊肚菌。此種情況是由于NCBI中部分序列被錯誤記錄導致的,前期已有文獻報道類似情況[4, 12]。
本試驗選取GenBank中粗柄羊肚菌(KR809597.1)六妹羊肚菌(KX809733.1)、高羊肚菌(MK890308.1)、七妹羊肚菌(MG589680.1)、黑脈羊肚菌Morchella angusticeps(KM587987.1)的ITS序列,并經GenBank和MLST數據庫雙庫核對準確后,采用NCBI Primer-Blast進行電子PCR驗證,結果顯示以上5種羊肚菌均未出現特異性片段。說明本試驗設計的特異性引物可有效地鑒別梯棱羊肚菌和其他5種羊肚菌。
綜上,本研究所建立的分子鑒別方法特異性強、靈敏度高、操作簡單、分析速度快,可快速有效鑒別梯棱羊肚菌,為該品種的科學研究、質量評價及菌種市場規范打下了良好的基礎。
[1] Du XH, Zhao Q, O'Donnell K, et al. Multigene molecular phylogenetics reveals true morels (Morchella) are especially species-rich in China[J]. Fungal Genetics and Biology, 2012, 49(6): 455-469.
[2] Tietel Z, Masaphy S. True morels (Morchella) - nutritional and phytochemical composition, health benefits and flavor: A Review[J]. Critical Reviews in Food Science and Nutrition, 2018, 58(11): 1888-1901.
[3] Liu QZ, Ma HS, Zhang Y, et al. Artificial cultivation of true morels: current state, issues and perspectives[J]. Critical Reviews in Biotechnology, 2018, 38(2): 259-271.
[4] 杜習慧, 趙琪, 楊祝良. 羊肚菌的多樣性、演化歷史及栽培研究進展[J]. 菌物學報, 2014, 33(2): 183-197.
[5] 趙琪, 趙永昌, 李榮春, 等. 皺蓋鐘菌生態環境調查[J]. 中國食用菌, 2006, 125(4): 22-24.
[6] Ar?ukowicz-Grabowska M, Wójcicki M, Raszeja-Wyszo-mirska J, et al. Acute liver injury, acute liver failure and acute on chronic liver failure: A clinical spectrum of poisoning due to Gyromitra esculenta[J]. Annals of Hepatology, 2019, 18(3): 514-516.
[7] 李增攀, 黃亮, 曹春水, 等. 毒蕈中毒的分類診治進展(一)—早發型中毒[J]. 嶺南急診醫學雜志, 2008, 13(2): 157-159.
[8] 陳劍山, 鄭服叢. ITS序列分析在真菌分類鑒定中的應用[J]. 安徽農業科學, 2007, 35(13): 3785-3786+3792.
[9] Liu Y, Wang XY, Gao ZT, et al. Detection of Ophiocordyceps sinensis and Its Common Adulterates Using Species-Specific Primers[J]. Front Microbiol, 2017, 8: 1179.
[10] 趙群, 劉楓, 宋向文, 等. 光慈菇藥材的特異性PCR鑒定[J]. 中藥材, 2017, 40(7): 1540-1546.
[11] 王金秋, 葉鴻亮, 梁道崴, 等. 梯棱羊肚菌子實體轉錄組測序及解析[J]. 食品科學, 2018, 39(18): 81-87.
[12] Du XH, Zhao Q, Yang ZL,et al. How well do ITS rDNA sequences differentiate species of true morels (Morchella)?[J]. Mycologia, 2012, 104(6): 1351-1368.
[13] Shen QH, Li CH, Xie MX, et al. A Novel PCR-Based Approach for Rapid Identification of Morchella sextelata Using Species-Specific Primers[J]. Current Microbiology, 2020, 77(2): 232-237.
[14] Yang H, Zhou Y, Yu PT, et al. A novel PCR-based technology for rapid and non-sequencing authentication of Bombyx batryticatus using species-specific primers[J]. Natural Product Research, 2019, 33(9): 1251-1256.
[15] 楊燕, 田鴻, 張小平, 等. 基于ITS和ISSR的羊肚菌種質資源遺傳多樣性分析[J]. 西南農業學報, 2018, 31(10): 2004-2009.
[16] 劉偉, 張亞, 蔡英麗. 我國羊肚菌產業發展的現狀及趨勢[J]. 食藥用菌, 2017, 25(2): 77-83.
[17] Ajmal Ali M, Gyulai G, Hidve?gi N, et al. The changing epitome of species identification – DNA barcoding[J]. Saudi Journal of Biological Sciences, 2014, 21(3): 204-231.
[18] Das S, Deb B. DNA barcoding of fungi using Ribosomal ITS marker for genetic diversity analysis: a review[J]. International Journal of Pure & Applied Bioscience, 2015, 3(3): 160–167.
[19] 劉偉, 蘭阿峰, 張倩倩, 等. 羊肚菌栽培菌株遺傳多樣性分析及種特異性RAPD-SCAR標記開發[J]. 菌物學報, 2018, 37(12): 1650-1660.
Molecular identification of
Shen Qianhui1Li Chunhong2Xie Meixia2Li Wenjia2Qian Zhengming2, 3*Zhang Ji1, 2*
(1. Institute for Brain Research and Rehabilitation, South China Normal University, Guangzhou, Guangdong 510631, China; 2. Key Laboratory of State Administration of Traditional Chinese Medicine, Sunshine Lake Pharma Co., LTD, Dongguan, Guangdong 523850, China; 3. Xiangnan University, Chenzhou, Hunan 423000, China)
It is important to establish a rapid and accurate method for the identification of species for the cultivation morels varieties. The ITS (internal transcribed spacer) sequences was obtained from all ofand 5 other species of morels collected from different location in China. Combined with reliable online-sequences for homology alignment, the specific-species primers ofwere designed according to the variation sites. And the optimal experimental conditions were obtained by optimizing conditions such as annealing temperature, annealing time and extension time, etc. The developed PCR method was applied in all samples.showed unique characteristic band, and other species of morels had no band. The results showed that the developed specific primer and PCR approach can be used for rapid identification of.
; molecular identification; species-specific primer; PCR
S646
A
2095-0934(2020)03-172-06
工信部年度中藥材提升與保障領域項目(2017020)
沈千匯(1995—),女,在讀碩士,研究方向為天然與生化藥物。E-mail:455230573@qq.com。
錢正明(1982—),男,博士,高級工程師,研究方向為中藥質量評價。E-mail:qianzhengming1982@126.com。張霽(1962—),男,博士,藥物研發/工藝研發首席科學家。E-mail:zhangjipaper@163.com。

