利用EST-SSR評估糜子資源遺傳差異
石甜甜,何杰麗,高志軍,陳凌,王海崗,喬治軍,王瑞云,
利用EST-SSR評估糜子資源遺傳差異
石甜甜1,何杰麗2,高志軍3,陳凌4,王海崗4,喬治軍4,王瑞云1,4
(1山西農業大學農學院,山西太谷 030801;2山西農業大學文理學院,山西太谷 030801;3內蒙古鄂爾多斯市農牧業科學研究院,內蒙古鄂爾多斯 017000;4山西省農業科學院農作物品種資源研究所/農業部黃土高原作物基因資源與種質創制重點實驗室/雜糧種質資源發掘與遺傳改良山西省重點實驗室,太原 030031)
【】用微衛星標記分析糜子種質資源(國內外6個不同生態區)的遺傳多樣性水平,揭示不同來源糜子種質資源的親緣關系和遺傳結構差異,便于對糜子資源分類和優異種質的篩選利用。用144個(高、低堿基序列重復分別為64和80個)SSR標記評估96份國內外(國內、國外分別為71和25份)糜子資源;用PowerMarker 3.25和PopGen1.32計算遺傳多樣性參數,用MEGA 5.0和Structure 2.2進行遺傳距離和結構聚類,用Ntsys 2.11進行主成分分析。144個EST-SSR標記共檢測出368個觀測等位變異(),每個位點檢測到等位變異2—3個,平均為2.5556個;觀測雜合度()為0.4070(RYW15)—0.9789(RYW85),平均為0.8288;期望雜合度()為0.4369(RYW59)—0.6693(RYW58),平均為0.5535;Nei's基因多樣性指數()為0.4344(RYW59)—0.6653(RYW58),平均為0.5505;多態性信息含量()為0.1811(RYW68)—0.7508(RYW58),平均為0.4279。Shannon多樣性指數()為0.6474—1.0956,平均為0.8415。就6個生態區材料的遺傳多樣性參數而言,北方春糜子區材料的值和Shannon多樣性指數最高,西北春夏糜子區材料最低。就不同生態區糜子種質間的遺傳距離和遺傳一致度而言,不同生態區糜子種質間的遺傳距離為 0.0111—0.1425,遺傳一致度為 0.8672—0.9889,北方春糜子區和黃土高原春夏糜子區間遺傳距離最小和遺傳一致度最高,西北春夏糜子區和華北夏糜子區間遺傳距離最大。基于UPGMA聚類將試驗材料劃歸3個類群(Ⅰ、Ⅱ、Ⅲ)。類群Ⅰ主要為北方春糜子區材料;類群Ⅱ主要為國外材料;類群Ⅲ主要為北方春糜子區和黃土高原春夏糜子區材料;基于Structure聚類將糜子資源劃歸4個群組,紅色群組,主要為北方春糜子區和黃土高原春夏糜子區材料,代表北方和黃土高原基因庫;綠色群組,主要為北方春糜子區材料,代表北方基因庫;藍色群組,主要包括黃土高原春夏糜子區材料,代表黃土高原基因庫;黃色群組,代表國外基因庫。就各分類群的遺傳多樣性參數而言,群組Ⅱ的值最大(0.4606),群組Ⅳ最小(0.3539);主成分分析將試材劃歸6類,與其地理來源一致。144個SSR標記可以準確評估96份糜子資源的遺傳變異,基于不同依據劃分的類群與6個生態區材料的地理來源基本一致,北方春糜子區材料的遺傳多樣性較豐富。
糜子;SSR;聚類分析;遺傳結構;主成分分析
0 引言
【研究意義】糜子(L.)屬禾本科黍屬,又稱黍稷或糜黍,抗旱耐瘠,在中國、俄羅斯、印度和美國主要用作糧食和飼料[1-3]。隨著全球氣溫日益變暖、未來環境漸趨干旱,作為哺育中華民族的載體作物和戰略儲備作物,糜子在保障國際糧食安全中起重要作用[4]。了解糜子種質現狀,準確評估資源遺傳背景,有助于加速糜子優異種質的發掘,并為其高效利用提供理論基礎。【前人研究進展】糜子分布廣,種植歷史超過一萬年,是中國北方冷涼地區的主糧,也是美國大平原小麥旱作農業系統中的輪作作物[5-6]。在印度國際半干旱熱帶作物研究所(International Crops Research Institute for the Semi-Arid Tropics,ICRISAT)和中國農業科學院種質資源研究所國家種質資源庫(Institute of Crop Germplasm Resources, Chinese Academy of Agricultural Sciences,ICGR- CAAS)分別保存有29 000余份和9 885份糜子[2-3]。首先,基于表型性狀調查,發現糜子農藝性狀遺傳多樣性豐富。2008年,對8 016份(來自ICGR-CAAS)糜子資源的11個農藝性狀進行主成分和聚類分析,結果歸納為8個主成分,聚為5個群組,群組4綜合性狀表現較好[7]。2016年,調查8 515份糜子資源(來自ICGR-CAAS)的7個農藝性狀,發現株高為80—200 cm,野生型偏低,農家種偏高;落粒性程度(輕度<5%,中度6%—10%,重度>11%)的分布呈區域性差異,輕度落粒性和株高較高(高產潛勢)存在協同選擇效應(植株越矮,落粒程度越嚴重)[8]。2019年,調查849份糜子資源(來自ICRISAT)的18個農藝性狀,發現播種至開花的時間為26—50 d,株高為20—133 cm(平均62 cm),穗長為22—400 mm(平均209 mm)[2]。第二,基于基因型檢測,鑒定到糜子資源存在諸多遺傳差異。簡單重復序列(simple sequence repeat,SSR)在基因組中含量豐富、多態性高、重復性好,是研究群體遺傳變異常用的分子檢測工具,已廣泛用于高粱等作物的遺傳多樣性評估[9-13]。2009年,用46個糜子非特異性SSR分析118份糜子資源(中國110份、美國6份和俄羅斯2份),發現黃土高原生態栽培區材料的遺傳多樣性最豐富[14]。2010年,基于基因組DNA富集SSR文庫,25個糜子特異性微衛星標記首次被開發[15]。2011年,用其中16個標記分析98份歐亞大陸糜子資源,發現基于遺傳距離劃分的東部和西部2個群組與地理來源相符,東部類群進一步劃分為4個亞類群或基因庫,而西部類群劃分為2個亞類群。東部群組的材料來源于中國、蒙古、尼泊爾、印度東北部、俄羅斯遠東地區、韓國和日本,西部群組的材料來源于烏克蘭、俄羅斯北高加索、俄羅斯歐洲大陸部分、中亞、印度西北部、巴基斯坦、中國和蒙古[16]。2015年,用其中19個標記分析來自7個地理群組的96份(95份來自中國6個省,1份來自俄羅斯)糜子資源,發現山西資源的遺傳多樣性最豐富[17]。2017年,對其中的15個標記標注熒光后,檢測來源于中國11個省/自治區的132份糜子資源,獲得107個等位變異,平均每個位點為7個,中國資源來自4個基因庫(東北地區、黃土高原、北方地區和西北地區),其豐富的遺傳變異主要存在于材料間[18]。2018年,用其中16個SSR和2個GBSSI標記分析341份歐亞大陸糜子資源(195份來自中國),發現黃土高原西部為糜子的主要馴化地[19]。近年來,利用轉錄組測序手段,構建出一批糜子中開發的EST-SSR,基于這些標記進行了一系列糜子遺傳多樣性研究。2016年,利用67個SSR分析來自中國7個糜子主產區的88份糜子資源,檢測到179個等位變異,基于遺傳距離將試材聚為4個群組[20]。同年,利用63個SSR評估來自國內外10個不同地理來源群體的192份糜子資源,檢測到161個等位變異,平均值為0.4855,發現內蒙、東北和黃土高原地區種質資源遺傳多樣性最豐富[21]。2017年,基于85個高基元SSR標記擴增中國6個糜子生態栽培區的96份糜子資源,檢測到232個等位變異,發現黃土高原春夏糜子區和北方春糜子區資源遺傳多樣性最豐富[22]。2018年,用103個SSR評估國內外12個群體的146份糜子資源(85份國內野生資源和61份國外地方品種),檢測到308個等位變異,為0.5544,發現中國河北群體的遺傳多樣性最豐富[23];對雁黍5號進行高通量測序,挖掘到8 139個全基因組EST-SSR,這些標記有望用于農藝性狀定位、連鎖圖譜構建和分子育種等遺傳研究[24]。【本研究切入點】豐富的糜子資源在旱作生態農業建設中起重要作用,而搞清其遺傳背景是合理利用的前提。但作為小宗作物,其遺傳多樣性評估研究偏少,亟待深入開展。【擬解決的關鍵問題】本研究利用144個糜子中開發的EST-SSR標記評估96份國內外糜子資源,搞清不同生態區資源的遺傳差異,為糜子優異種質的鑒定和篩選提供理論依據。
1 材料與方法
1.1 試驗材料
試材為來自國內5個生態區和國外的96份糜子資源(表1,電子附表1)。
1.2 方法
1.2.1 DNA提取及其質量檢測 剪取三葉期糜子葉片,提取基因組DNA,用瓊脂糖凝膠電泳檢測DNA質量,用核酸檢測儀測定DNA的純度。
1.2.2 PCR反應體系及擴增程序 用山西農業大學糜子分子育種實驗室構建的SSR引物(電子附表2)在BIO-GENER基因擴增儀(GT9612)上擴增糜子材料,反應總體積為20 μL:DNA模板1 μL、2×工員MasterMix(中科瑞泰2×Taq PCR MasterMix)10 μL、前后引物各0.8 μL和ddH2O 7.4 μL。PCR程序為94℃ 5 min;94℃ 40 s,退火40 s,72℃1 min,36個循環;72℃10 min。銀染檢測PCR產物。
1.2.3 數據統計與分析 讀取電泳結果,同一位置有擴增條帶的記為1,否則記為0,在Excel中將整理好的(0,1)原始矩陣數據轉換成不同軟件相應格式。
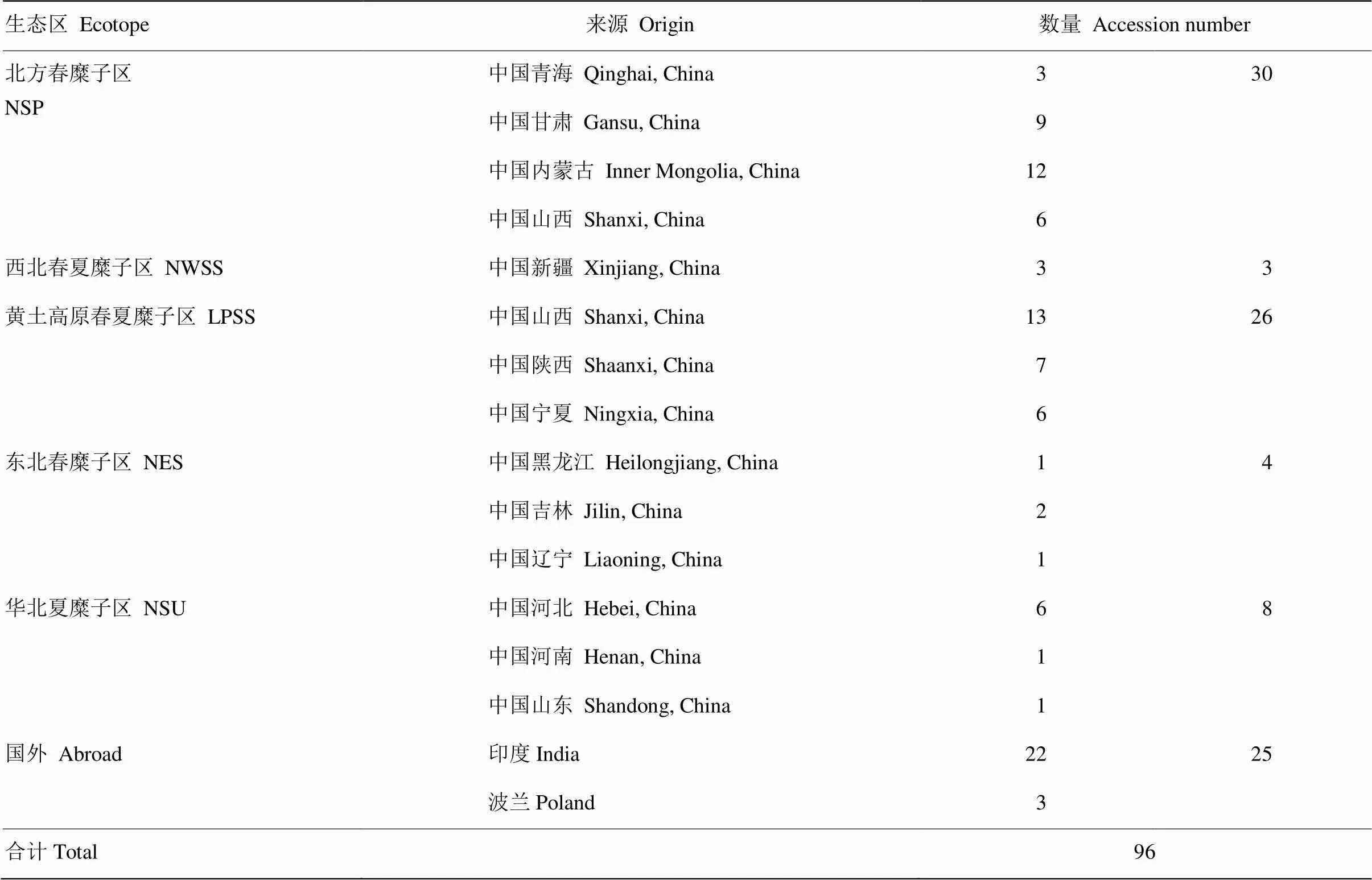
表1 糜子資源的生態區分布
NSP:北方春糜子區;NWSS:西北春夏糜子區;LPSS:黃土高原春夏糜子區;NES:東北春糜子區;NSU:華北夏糜子區。下同
NSP: Northern spring-sowing ecotope; NWSS: Northwest spring & summer-sowing ecotope; LPSS: Loess plateau spring & summer-sowing ecotope; NES: Northeast Spring-sowing ecotope; NSU: Northern Summer-sowing ecotope. The same as below
用PowerMarker 3.25[25]和PopGen1.32[26]計算遺傳多樣性衡量指標,用MEGA5.0[27]和Structure2.2[28]分別基于遺傳距離和結構構建聚類圖。用Ntsys2.11[29]進行主成分分析。
2 結果
2.1 SSR標記的多態性分析
用144對糜子中開發的引物評估糜子資源的遺傳多樣性(電子附表3),結果發現96份材料在144個位點共檢出368個觀測等位變異,每個位點檢測到2—3個,平均為2.5556個;Shannon多樣性指數為0.6474(RYW92)—1.0956(RYW58),平均為0.8415;觀測雜合度為0.4070(RYW15)—0.9789(RYW85),平均為0.8288;期望雜合度為0.4369(RYW59)—0.6693(RYW58),平均為0.5535;Nei's基因多樣性指數為0.4344(RYW59)—0.6653(RYW58),平均為0.5505;多態性信息含量為0.1811(RYW68)—0.7508(RYW58),平均為0.4279。
分析不同生態區糜子資源的遺傳多樣性參數(表2),發現就值和Shannon多樣性指數而言,北方春糜子區材料最高,西北春夏糜子區材料最低,說明前者遺傳多樣性豐富。分析不同生態區糜子資源的遺傳距離與遺傳一致度參數(表3),發現北方春糜子區和黃土高原春夏糜子區間的遺傳距離最小、遺傳一致度最高,西北春夏糜子區和華北夏糜子區間的遺傳距離最大、遺傳一致度最低,表明地理位置越近,親緣關系越近,遺傳相似性越高;地理位置越遠,親緣關系越遠,遺傳相似性越低。
2.2 不同糜子資源的聚類分析
基于UPGMA對材料進行聚類分析(圖1)。可以看出,96份糜子劃分為3個類群(類群Ⅰ、Ⅱ、Ⅲ)。類群Ⅰ共24份,主要為北方春糜子區材料(12份);類群Ⅱ共38份,主要為國外材料;類群Ⅲ共34份,主要為北方春糜子區和黃土高原春夏糜子區材料。

表2 不同生態區糜子的遺傳多樣性參數
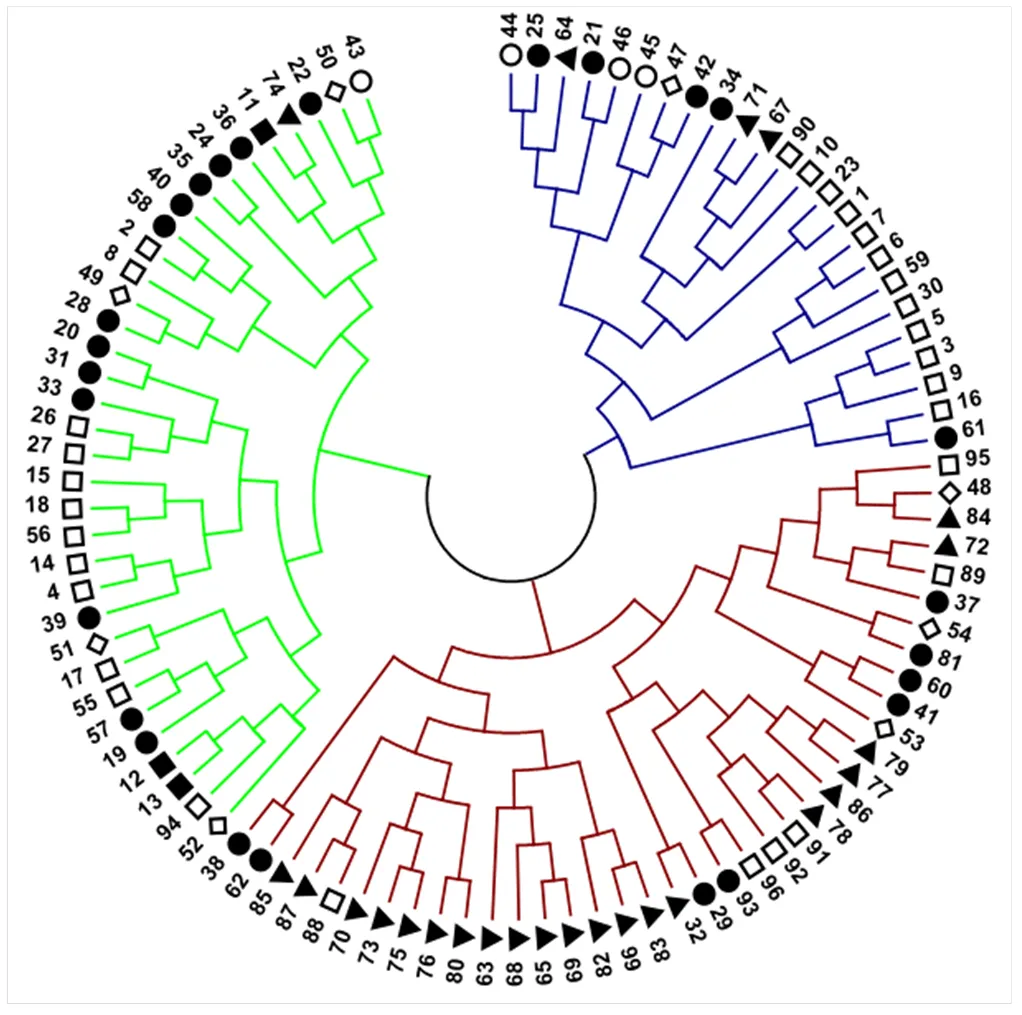
▲國外Abroad;◇華北夏糜子區Northern Summer-sowing ecotope;▉西北春夏糜子區Northwest spring & summer-sowing ecotope;□北方春糜子區Northern spring-sowing ecotope;●黃土高原春夏糜子區Loess plateau spring & summer-sowing ecotope;○東北春糜子區Northeast Spring-sowing ecotope
2.3 基于模型的糜子遺傳群體結構分析
通過對96份供試材料進行遺傳群體結構分析(圖2),發現Delta K在=4處出現峰值,將供試材料分為4個群組(圖3)。紅色群組(26份),主要為北方春糜子區和黃土高原春夏糜子區材料,代表北方和黃土高原基因庫;綠色群組(12份),主要為北方春糜子區材料,代表北方基因庫;藍色群組(18份),主要包括黃土高原春夏糜子區材料,代表黃土高原基因庫;黃色群組(40份),代表國外基因庫。
分析=4遺傳結構圖各分類群的遺傳多樣性參數(表4),發現就值而言,群組Ⅱ最大(0.4606),群組Ⅳ最小(0.3539)。
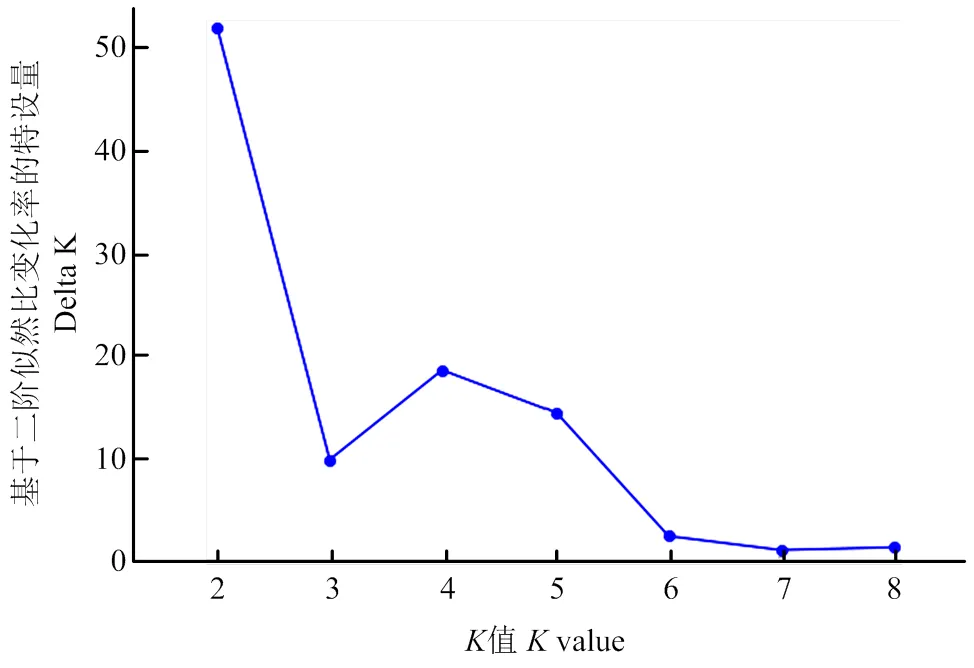
圖2 運用 Structure對糜子基因庫數目建模
2.4 主成分分析
對供試材料進行PCA分析(圖4),可以看出,96份資源劃歸6個類群(G1—G6),G1有30份,均來自北方春糜子區;G2有3份,均來自西北春夏糜子區;G3有26份,均來自黃土高原春夏糜子區;G4有4份,均來自東北春糜子區;G5有8份,均來自華北夏糜子區;G6有25份,全為國外材料。
前3個主成分PC1、PC2和PC3分別解釋總變異的21.21%、3.17%和2.35%,總變異26.73%。

表3 不同群體糜子資源的遺傳距離與遺傳一致度
Nei氏遺傳一致度(對角線以上)和遺傳距離(對角線以下) Nei’s genetic identity (above diagonal) and genetic distance (below diagonal)

橫坐標的數字代表糜子材料序號 Numbers in the horizontal axis represent serial number of accession

圖4 96份糜子資源遺傳多樣性的主成分分析
3 討論
在作物資源的遺傳多樣性評估中,基于遺傳距離和結構劃分的類群,常發生交叉現象或出現個別例外,這在三大糧食作物中均有體現,而運用PCA分析則可以得到合理解釋[30-32]。2010年,基于359個SSR評估1 537份優良玉米自交系種質的群體結構和遺傳多樣性,發現基于遺傳結構與遺傳距離的聚類結果基本一致,但也出現個別例外。如遺傳結構群組G3主要由Flint組成,但1份Stiff Stalk材料也劃歸其中;而Lancaster材料則在4個群組(G1—G4)中均有分布。主成分分析則將上述特例劃歸相應群組。2019年,王舒婷等[33]基于27個微衛星標記分析57份山西糜子資源的遺傳特性,基于UPGMA聚類,類群Ⅱ主要為黃土高原春夏糜子區材料,但北方春糜子區材料(13號忻州定襄大紅黍)也劃歸其中;而黃土高原春夏糜子區的材料則在3個類群中均有分布。基于Structure聚類,紅色群組主要為黃土高原春夏糜子區基因庫,但北方春糜子區材料(7號朔州白糜子)也劃歸其中;而北方春糜子區材料(如7號、1號和3號)在紅、綠和藍色群組均有分布。但利用PCA分析卻將上述個例劃歸相應類群,如13、7、1和3號材料均歸于PC1中,黃土高原春夏糜子區材料均歸于PC2中。本研究也得到了類似的結果,基于UPGMA聚類,群組Ⅱ主要為國外材料,但一份山東資源(54號山東益都黑黍子)也劃歸其中;而山西材料則分布在3個群組,并未歸于同一群組。基于Structure聚類,黃色群組主要為黃土高原夏糜子區基因庫,但2份新疆資源(12號紅糜和13號黃糜)也劃歸其中;而陜西材料在4個群組中均有分布。而主成分分析則將試材劃分為6個類群,合理解釋了上述資源劃分出現交叉的現象,聚類群組中的特例在PCA中均劃歸到相應類群,如國外材料都歸于G6中,山東資源(54號山東益都黑黍子)劃分到G5中,山西資源均劃歸在G3中,新疆、陜西材料則分別G2和G3中。

表4 各遺傳結構群體的多樣性參數
作為簡單序列重復的DNA標記,基于序列來源,SSR分為基因組SSR(genomic SSR/G-SSR)和表達序列標簽SSR(expressed sequence tag-SSR/EST-SSR)。與G-SSR相比,EST-SSR具可在物種間進行轉移的優勢,已廣泛應用于遺傳連鎖圖譜構建、種質資源鑒定和遺傳多樣性分析等方面[21-23, 34-36]。2011年,用G-SSR與EST-SSR評估5個楊樹不同種的15份資源的遺傳差異,結果發現EST-SSR比G-SSR在楊樹種間通用性更好、揭示的遺傳多樣度更高,聚類分析表明EST-SSR能更準確反映楊樹不同種間的遺傳差異和親緣關系[35]。對小麥(L.)、水稻(L.)、毛果楊(s)和木豆((L.)Millspaugh)的研究發現,SSR開發中多態性得率介于10%—83.8%[37-40]。2010年,CHO等[15]運用改進的生物素-鏈霉親和素捕捉法,經過酶消化、雜交和磁珠吸附等一系列程序,從糜子基因組DNA中構建了含有504個推定重組克隆的SSR富集文庫;去除24個冗余序列后,發現480個特異克隆中有157個為SSR克隆,設計143對引物進行多態性篩選獲得25個多態性G-SSR(2、3、4核苷酸序列重復SSR分別為9、13和3個),多態性得率為17.4%。2017年,劉笑瑜[34]基于糜子(黃糜子(00005272)、鎮原大糜子(00002942)、287(00002661)、老糜子(00005435))轉錄組測序結果,從151對引物中篩選出有擴增條帶的126對,其中85對具多態性EST-SSR,多態性得率56.3%。2018年,Jiang等[24]從8 734對引物中設計了8 139對,隨機選取24對,發現21對有擴增條帶,3對具多態性,多態性得率12.5%。本研究所用標記的多態性得率為47%(高、低基元堿基重復多態性得率分別為56.3%和40%),高于Jiang等[24]、低于劉笑瑜[34]的研究結果,這與前者所用標記基本為低基元(2、3堿基重復分別為11.1%和83.6%)、多態性得率低,而后者所用標記全部為高基元、多態性得率高有關。
4 結論
144個EST-SSR可以準確評估96份國內外糜子資源,基于不同依據劃分的類群與6個生態區材料的地理來源基本一致,北方春糜子區材料的遺傳多樣性較為豐富。
[1] 張麗珍, 冀佩雙, 羅旭梟, 穆虹彰, 秦雁鵬, 牛宇, 喬治軍. 糜黍淀粉理化性質及消化特性. 食品科學, 2016, 37(19): 76-81.
ZHANG L Z, JI P S, LUO X X, MU H Z, QIN Y P, NIU Y, QIAO Z J. Physicochemical properties and digestive properties of millet starch., 2016, 37(19): 76-81. (in Chinese)
[2] Vetriventhan M, Azevedo V C R, Upadhyaya H D, Naresh D. Variability in the global proso millet (L.) germplasm collection conserved at the ICRISAT genbank., 2019, 9(5): 112
[3] 王瑞云. 糜子遺傳多樣性及進化研究進展. 北京: 中國農業出版社, 2017: 2-3.
WANG R Y.L.. Beijing: China Agriculture Press, 2017: 2-3. (in Chinese)
[4] 刁現民, 程汝宏. 十五年區試數據分析展示谷子糜子育種現狀. 中國農業科學, 2017, 50(23): 4469-4474.
Diao X M, cheng R H. Current breeding situation of foxtail millet and common millet in China as revealed by exploitation of 15 years regional adaptation test data., 2017, 50(23): 4469-4474. (in Chinese)
[5] LU H Y, ZHANG J P, LIU K B, WU N Q, LI Y M, ZHOU K S, YE M L, ZHANG T Y, ZHANG H J, YANG X Y, SHEN L C, XU D K, LI Q. Earliest domestication of common millet () in East Asia extended to 10,000 years ago., 2009, 106(18): 7367-7372.
[6] RAJPUT S G, TAMMY P H, DIPAK K S. Development and characterization of SSR markers in proso millet based on switchgrass genomics., 2014, 5(1): 175-186.
[7] 胡興雨, 陸平, 賀建波, 王綸, 王玉星, 張紅生, 張宗文, 吳斌. 黍稷農藝性狀的主成分分析與聚類分析. 植物遺傳資源學報, 2008, 9(4): 492-496. HU X Y, LU P, HE J B, WANG L, WANG Y X, ZHANG H S, ZHANG Z W, WU B. Principal components and cluster analysis of agronomic traits of proso millet ()., 2008, 9(4): 492-496. (in Chinese)
[8] WANG R Y, HUNT H V, QIAO Z J, Wang L, HAN Y H. Diversity and cultivation of broomcorn millet (L.) in China: A Review., 2016, 70(3): 332-342.
[9] ELEUCH L, JILAL A, GRANDO S, CECCARELLI S, SCHMISING M V K, TSUJIMOTO H, Hajer A, Daaloul A, Baum M. Genetic diversity and association analysis for salinity tolerance, heading date and plant height of barley germplasm using simple sequence repeat markers., 2008(8): 1004-1014.
[10] Wen Z X, DING Y L, ZHAO T N, Gai J Y. Genetic diversity and peculiarity of annual wild soybean (Sieb. et Zucc.) from various eco-regions in China., 2009, 119(2): 371-381.
[11] Li R Y, ZHANG H, ZHOU X C, GUAN Y A, YAO F X, SONG G A, Wang J C, ZHANG C Q. Genetic diversity in Chinese sorghum landraces revealed by chloroplast simple sequence repeats., 2010, 57(1): 1-15.
[12] Badiane F A, Gowda B S, Ciss N, Diouf D, Sadio O, Timko M P. Genetic relationship of cowpea () varieties from Senegal based on SSR markers., 2012, 11(1): 292-304.
[13] Wang C F, Jia G Q, Zhi H, Niu Z G, Chai Y, Li W, Wang Y F, Li H Q, Lu P, Zhao B H, DIAO X M. Genetic diversity and population structure of Chinese foxtail millet [(L.) Beauv.] landraces., 2012, 2(7): 769-777.
[14] HU X Y, WANG J F, LU P, ZHANG H S. Assessment of genetic diversity in broomcorn millet (L.) using SSR markers., 2009, 36(8): 491-500.
[15] Cho Y I, Chung J W, Lee G A, Ma K H, Dixit A, Gwag J G, Park Y J. Development and characterization of twenty-five new polymorphic microsatellite markers in proso millet (L.)., 2010, 32(3): 267-273.
[16] Hunt H V, Campana M G, Lawes M C, Park Y J, Bower M A, Howe C J, Jones M K. Genetic diversity and phylogeography of broomcorn millet (L.) across Eurasia., 2011, 20(22): 4756-4771.
[17] 董俊麗, 王海崗, 陳凌, 王君杰, 曹曉寧, 王綸, 喬治軍. 糜子骨干種質遺傳多樣性和遺傳結構分析. 中國農業科學, 2015, 48(16): 3121-3131.
DONG J L, WANG H G, CHEN L, WANG J J, CAO X N, WANG L, QIAO Z J. Analysis of genetic diversity and structure of proso millet core germplasm., 2015, 48(16): 3121-3131. (in Chinese)
[18] 王瑞云, 季煦, 陸平, 劉敏軒, 許月, 王綸, 王海崗, 喬治軍. 利用熒光SSR分析中國糜子遺傳多樣性. 作物學報, 2017, 43(4): 530-548.
Wang R y, Ji x, Lu P, Liu M x, Xu Y, Wang L, Wang H g, qiao z j. Analysis of genetic diversity in common millet () using fluorescent SSR in China., 2017, 43(4): 530-548. (in Chinese)
[19] HUNT H V, RUDZINSKI A, JIANG H E, WANG R Y, THOMAS M G, JONES M K. Genetic evidence for a western Chinese origin of broomcorn millet ()., 2018, 28(12): 1968-1978.
[20] Liu M X, Xu Y, He J H, Zhang S, Wang Y Y, Lu P. Genetic diversity and population structure of broomcorn millet (L.) cultivars and landraces in China based on microsatellite markers., 2016, 17(3): 370.
[21] 連帥, 陸平, 喬治軍, 張琦, 張茜, 劉敏軒, 王瑞云. 利用SSR分子標記研究國內外黍稷地方品種和野生資源的遺傳多樣性. 中國農業科學, 2016, 49(17): 3264-3275.
LIAN S, LU P, QIAO Z J, ZHANG Q, ZHANG Q, LIU M X, WANG R Y. Genetic diversity in broomcorn millet (L.) from China and abroad by using SSR markers., 2016, 49(17): 3264-3275. (in Chinese)
[22] 王瑞云, 劉笑瑜, 王海崗, 陸平, 劉敏軒, 陳凌, 喬治軍. 用高基元微衛星標記分析中國糜子遺傳多樣性. 中國農業科學, 2017, 50(20): 3848-3859.
Wang R y, Liu X y, Wang H g, Lu P, Liu M x, Chen L, qiao z j. Evaluation of genetic diversity of common millet () germplasm available in China using high motif nucleotide repeat SSR markers., 2017, 50(20): 3848-3859. (in Chinese)
[23] 薛延桃, 陸平, 喬治軍, 劉敏軒, 王瑞云. 基于SSR標記的黍稷種質資源遺傳多樣性及親緣關系研究. 中國農業科學, 2018, 51(15): 2846-2859.
XUE Y T, LU P, QIAO Z J, LIU M X, WANG R Y. Genetic diversity and genetic relationship of broomcorn millet (L.) germplasm based on SSR markers., 2018, 51(15): 2846-2859. (in Chinese)
[24] Jiang Y m, Li H q, Zhang J N, Xiang J y, Cheng R h, Liu G q. Whole genomic EST-SSR development based on high-throughput transcript sequencing in proso millet ()., 2018, 20(3): 617-620.
[25] Liu K, Muse S V. PowerMarker: Integrated analysis environment for genetic marker data., 2005, 21: 2128-2129.
[26] Yeh F C, Boyle T J. Population genetic analysis of co-dominant and dominant markers and quantitative traits., 1997, 130: 129-157.
[27] Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance and maximum parsimony methods., 2011, 28(10): 2731-2739.
[28] Falush D, Stephens M, Pritchard J K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies., 2003, 164(4): 1567-1587.
[29] Nei M, Li W H. Mathematical model for studying genetic variation in terms of restriction endonucleases., 1979, 76(10): 5269-5273.
[30] Bonman J M, Babiker E M, Cuesta-Marcos A, Esvelt-Klos K, Brown-Guedira G, Chao S, See D, Chen J, Akhunov E, Zhang J, Bockelman H E, Gordon T C. Genetic diversity among wheat accessions from the USDA national small grains collection., 2015, 55(3): 1243-1253.
[31] Courtois B, Frouin J, Greco R, Bruschi G, Droc G, Hamelin C, Ruiz M, Clément G, Evrard J C, Van Coppenole S, Katsantonis D, Oliveira M, Negr?o S, Matos C, Cavigiolo S, Lupotto E, Piffanelli P, Ahmadi N. Genetic diversity and population structure in a European collection of rice., 2012, 52(4): 1663-1675.
[32] Van Inghelandt D, Melchinger A E, Lebreton C, Stich B. Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers., 2010, 120(7): 1289-1299.
[33] 王舒婷, 何杰麗, 石甜甜, 陳凌, 王海崗, 王瑞云, 喬治軍. 利用微衛星標記分析山西糜子的遺傳多樣性. 植物遺傳資源學報, 2019, 20(1): 69-78.
wang S T, he J L, shi T T, Chen L, wang H G, wang R Y, QIAO Z J. Genetic diversity analysis of broomcorn millet (L.) of Shanxi Province using microsatellite markers., 2019, 20(1): 69-78. (in Chinese)
[34] 劉笑瑜. 利用高基元SSR分析中國糜子資源的遺傳多樣性[D]. 太谷: 山西農業大學, 2017.
LIU X Y. Analysis of genetic diversity of Chinese proso millet resources by high substrate SSR[D]. Taigu: Shanxi Agricultural University, 2017. (in Chinese)
[35] 張亞東, 彭嬋, 李振芳, 楊彥伶, 胡興宜. 基因組SSR與EST-SSR標記在楊樹不同種間的遺傳差異. 東北林業大學學報, 2011, 39(12): 8-11, 117.
ZHANG Y D,PENG C,LI Z F,YANG Y L,HU X Y. Genetic diversity of genomic-SSR and EST-SSR markers in interspecies of poplar., 2011 39(12): 8-11, 117. (in Chinese)
[36] 徐陽, 陳金慧, 李亞, 洪舟, 王穎, 趙亞琦, 王新民, 施季森. 杉木EST-SSR與基因組SSR引物開發. 南京林業大學學報(自然科學版), 2014, 38(1): 9-14.
XU Y, CHEN J H, LI Y, HONG Z, WANG Y, ZHAO Y Q, WANG X M, SHI J S. Development of EST-SSR and genomic-SSR in Chinese fir., 2014, 38(1): 9-14. (in Chinese)
[37] CHO Y G, ISHII T, TEMNYKH S, CHEN X, LIPOVICH L, MCCOUCH S R, PARK W D, AYRES N, CARTINHOUR S. Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice ()., 2000, 100(5): 713-722.
[38] EUJAYL I, SORRELLS M, BAUM M, WOLTERS P, POWELL W. Assessment of genotypic variation among cultivated durum wheat based on EST-SSRsand genomic SSRs., 2001, 119(1): 39-43.
[39] TUSKAN G A, GUNTER L E, YANG, YANG Z K, YIN T M, SEWELL M M, DIFAZIO S P. Characterization of microsatellites revealed by genomic sequencing ofs, 2004, 34(1): 85-93.
[40] DUTTA S, KUMAWAT G, SINGH B P, GUPTA D K, SINGH S, DOGRA V, GAIKWAD K, SHARMA T R, RAJE R S, BANDHOPADHYA T K, DATTA S, SINGH M N, BASHASAB F, KULWAL P, WANJARI K B, VARSHNEY R K, COOK D R, SINGH N K. Development of genic-SSR markers by deep transcriptome sequencing in pigeonpea [( L.) Millspaugh]., 2011, 11(1): 17.
Genetic Diversity of Common Millet Resources Assessed with Est-ssr Markers
SHI TianTian1, HE JieLi2, GAO ZhiJun3, CHEN Ling4, WANG HaiGang4, QIAO ZhiJun4, WANG RuiYun1,4
(1College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi;2College of Arts and Sciences, Shanxi Agricultural University, Taigu 030801, Shanxi;3Erdos institute of agriculture and animal husbandry, erdos 017000, Inner Mongolia;4Institute of Crop Germplasms Resources of Shanxi Academy of Agricultural Sciences/Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau, Ministry of Agriculture, Taiyuan 030031)
The objective of this study is to analyze the genetic diversity and relationship of common millet accessions (six different ecotopes at home and abroad) by microsatellite markers, to provide available data for classification, selection and utilization of elite germplasm resources.One hundred and forty-four SSRs (64 high motif nucleotide sequence repeat and 80 low ones) are used to identify polymorphisms in ninety-six common millet accessions (71 home accessions and 25 abroad accessions). Genetic diversity parameters were calculated using software PowerMarker 3.25 and PopGen 1.32. Genetic distance and Structure on accessions were classified with software MEGA 5.0 and Structure 2.2, respectively. PCA (principal component analysis) was conducted by software Ntsys 2.11.Using 144 EST-SSR markers, a total of 368 observed alleles () were detected with 2-3 alleles (mean = 2.5556) per locus. The observed heterozygosity () ranged from 0.4070 (RYW15) to 0.9789 ( RYW85) with an average of 0.8288. The expected heterozygosity () ranged from 0.4369 ( RYW59) to 0.6693 ( RYW58) with an average of 0.5535. The Nei's gene diversity index ranged from 0.4344 (RYW59) to 0.6653 (RYW58) with a mean of 0.5505. The polymorphism information content () ranged from 0.1811 (RYW68) to 0.7508 (RYW58) with an average of 0.4279. The shannon diversity index () range was 0.6474 to 1.0956 with an average of 0.8415. In the case of genetic diversity parameters, theandof common millet accessions from Northern spring-sowing region were the most abundant than that of others. On the contrary, the accessions from Northwest spring & summer-sowing region were the lowest. For the different geographical regions accessions, the range of genetic distance was 0.0111 to 0.1425 and the scope of genetic consistency was 0.8672 to 0.9889. The genetic distance of accessions between Loess Plateau spring & summer-sowing region and Northern spring-sowing region was the least and their genetic consistency was the highest. The genetic distance between Northwest spring & summer-sowing region and North of China summer-sowing region was the largest and their genetic consistency was the lowest. UPGMA analysis divided 96 accessions into three groups (GroupⅠ, Ⅱ and Ⅲ). Group I were mainly Northern spring-sowing region common millet, Group Ⅱwere mainly foreign accessions, and Group Ⅲ were mainly Loess Plateau spring & summer-sowing region and Northern spring-sowing region accessions. Structure cluster divided resources into four groups. The red group contained Loess Plateau spring & summer-sowing region and Northern spring-sowing region accessions, which represented the gene pool of Loess Plateau and North. The green group included Northern spring-sowing region accessions, which represented the North gene pool. The blue group were Loess Plateau spring & summer-sowing region accessions, which represented the gene pool of Loess Plateau. The yellow group represented foreign gene pool. In terms of diversity parameters, thevalue of GroupⅡwas the highest at 0.4606 and the Group Ⅳ was the lowest at 0.3539. The PCA analysis classified all accessions into six clusters, which are related to their geographical region.144 SSRs can evaluate 96 common millet resources accurately. The genetic relationships are related to their geographical region. Genetic diversity of accessions from Northern spring-sowing region are more abundant.
common millet; SSR; cluster analysis; genetic structure; principal component analysis
10.3864/j.issn.0578-1752.2019.22.014

2019-06-13;
2019-08-17
現代農業產業技術體系建設專項(CARS-06-13.5-A16)、國家自然科學基金(31271791)、山西省回國留學人員科研資助項目(2016-066)、山西省重點研發計劃(一般項目)(農業)項目(201803D221008-5)、2019山西省研究生創新項目(2019SY202)
石甜甜,tel:15234420893;E-mail:tt15234420893@163.com。通信作者王瑞云,Tel:15234420135;E-mail:wry925@126.com。通信作者喬治軍,Tel:0351-7065530;E-mail:nkypzs@126.com
(責任編輯 李莉)

