貝類中GI、GII諾如病毒快速檢測與分群方法的建立
蘇來金,趙 峰,馬麗萍,周德慶
諾如病毒(Noroviruses, NoVs)屬于杯狀病毒科(Calicivirida),諾如病毒屬(Norovirus),是一種重要的食源性病原菌,引發了許多傳播與暴發[1-3]。NoVs可分為5個遺傳組(genogroups, G),分別被命名為GI到GVII,其中GI、GII和GIV感染人類[4-5]。貝類通過濾食作用可將諾如病毒持續富集在體內,成為NoVs傳播的重要載體[6],在貝類中經常污染到GI、GII 2種基因群的NoVs[7-10],目前檢測NoVs的分子生物學方法包括:RT-PCR[11],巢式RT-PCR[12],熒光定量RT-PCR[13]等方法,也有同時檢測GI、GII NoVs的檢測方法[14-16],然而不同基因群的NoVs鑒定一般需要有多對引物分別檢測,甚至還需要對擴增產物回收、測序鑒定、操作繁瑣、耗時較長。
本研究通過對比分析GI、GII NoVs流行株的基因序列,篩選一對引物對GI、GII NoVs同時檢測,根據Synergy brands(SYBR) Green I 熒光定量中熔解曲線特性[17-18],建立了基于SYBR Green I熒光定量熔解曲線分析的快速分群方法,旨在為貝類中單基因群或混合型基因群NoVs的快速檢測與分群分型提供技術支撐。
1 材料與方法
1.1試驗材料 6株陽性NoVs分別為57397毒株(GI.1型,GenBank:M87661.2),54108毒株(GI.3型, GenBank: GQ856470.1), 55063毒株(GI.6型,GenBank: GQ856464.), 57395毒株(GII.4型,GenBank:KC752514),F278毒株(GII.12型,GenBank NO:JQ899442), GZ2013-L20毒株(GII.17型,KR869038)為中國疾病預防控制中心提供,為感染病人的糞便樣品,經濃縮冷凍保存,基因型別均經過擴增測序后,使用美國CaliciNet諾如病毒在線分型工具(https://norovirus.phirearchlab.org/)進行分型確認;輪狀病毒(Rotavirus,RV)、星狀病毒(Astrovirus,AstV)為實驗室保存的病毒cDNA,由青島市疾控中心提供。用于人工污染的貝類樣品為青島城陽水產品批發市場采集的鮮活牡蠣,經過檢測未發現NoVs檢出。貝類檢測樣品為2015年1-12月從青島城陽水產品批發市場采集的牡蠣、扇貝、毛蚶、菲律賓蛤仔、中國蛤等5種貝類,每月采集2次,全年共采集120份樣品。
1.2設備與試劑 NanoPhotometerTM微量核酸蛋白測定儀,購自德國IMPLEN公司;羅氏Light Cycler? 2.0全自動熒光定量PCR儀,購自德國Roche公司;Sigma 1-14冷凍離心機,購自德國SIGMA公司;Biometra PCR儀,購自德國Biometra公司;DYCP-31E型電泳儀,購自北京六一儀器廠;凝膠成像系統,購自法國Vilber Lourmat公司; High Pure Viral RNA Kit 、蛋白酶K購自德國羅氏(Roche)公司;瓊脂糖凝膠回收試劑盒、質粒小提試劑盒等購自北京天根(TIANGEN)公司,DNaseI(RNase Free)、SYBR? Premix Ex TaqTM II試劑盒等購于大連寶生物(TaKaRa)公司。
1.3引物篩選 從GenBank下載目前用于分群分型的主要毒株GI(GeneBank NO:M87661、L07418、AB447414、AB042808、KJ402295、AB081723、MH130046、AF538679、KF586507、M0127530)、GII(GeneBank NO. U07611、DQ366347、JQ751034、AB220924、AB220921、 AY032605、EF126965、HM802544、AB294779、AB933767、AB434770、JX459908、DQ078829、 AY682550、AB684664、JX846926、AB039780、AY038599、AB126320、 AJ277618、 JN899242、 GU017903、 AY502010、 LC037415、AY823304、 AY823306、 EU275779、 JN899245、AB233471、MG495080、MG495085、 GQ856469、 KU306738)NoVs的基因序列,利用DNA star進行序列比對,篩選GI、GII同時保守區域的引物,最終選擇同時檢測GI、GII NoVs的引物為:上游引物P290: 5′-GATTACTCCAGGTGGGAYTCMAC-3′(引物位置:4568-4 590),下游引物P289:5′-TGACGATTTCATCATCACCRTM-3′(引物位置:4865-4886),擴增片段大小319 bp。
1.4 實驗方法
1.4.1病毒人工污染、回收、RNA提取 取2 g牡蠣消化道樣品,加入10 μL的病毒原液,加入2 mL PBS緩沖液,勻漿2 min;NoVs回收按照實驗室前期建立的方法[19],RNA提取使用High Pure Viral RNA Kit(Roche)試劑盒說明方法進行。
1.4.2普通RT-PCR與熒光定量反應體系優化 cDNA合成擴增體系為50 μL,5 μL 10×Buffer,8 μL 25 mmol/L MgCl2,2.5 μL 10 mmol/L dNTPs,2 μL P289引物,0.7 μL 10 U/μL AMV-RT 反轉錄酶,0.2 μL 50 U/μL RNasin,2 μL病毒RNA模板,補足去離子水,42 ℃反應1 h。
PCR反應:5 μL 10×Buffer,4 μL 25 mmol/L MgCl2,1 μL 10 mmol/L dNTPs,上下游引物各1 μL,0.5 μL 5 U/μL Taq酶,8 μL cDNA,補足去離子水,反應條件:94 ℃ 3 min,然后94℃ 1 min,49℃ 1 min 20 s,72℃ 1 min,40個循環,最后72 ℃ 10 min。產物電泳后經測序進行鑒定。
熒光定量的PCR采用20 μL的體系進行擴增,SYBR? premix Ex TaqTM II (2×) 15 μL,2條引物(10 μmol/L)各1 μL,cDNA模板100 ng左右,補足去離子水。熒光定量PCR擴增: 95 ℃預變性30 s,1個循環;然后95 ℃變性5 s,49 ℃退火15 s,72 ℃延伸10 s,35個循環。
1.4.3熒光定量標準品制備 使用GII.4型NoV的RNA為擴增對象,RT-PCR產物電泳切取特異性條帶,用膠回收試劑盒回收后,根據實驗室方法制備質粒[20],并提取DNA,用生物分光光度計測定質粒DNA的濃度和純度,換算成拷貝數,并進行梯度稀釋,作為系列標準品進行方法建立。
1.4.4SYBR Green I熒光定量PCR的TM曲線 熔解曲線分析按照熒光定量試劑盒說明書中推薦的程序,以0.1℃/s變化速度從65℃升高至95℃繪制熔解曲線。
1.4.5特異性與準確性試驗 使用GI.1、GI.3、GI.6、GII.4、GII.12、GII.17等6株陽性NoVs,以輪狀病毒(RV)、星狀病毒(AsTV)為陰性對照,以二次水為空白對照,對建立的方法進行驗證,確定檢測方法的特異性和準確性。
1.4.6穩定性與重復性 將不同型別的NoVs,人工污染貝類后,分3個時間批次利用熒光定量PCR熔解曲線法測定Tm值,然后,計算3個批次樣品的Tm平均值、標準偏差、變異系數以及對Tm值進行方差分析,確定方法的穩定性和重復性。
1.4.7驗證性試驗 對采集的120個貝類樣品,利用建立的方法,結合普通RT-PCR,對檢測出的陽性NoVs,進行PCR擴增,片段回收、測序分析,與熔解曲線檢測結果對比,驗證方法的實用性。
1.5數據處理 所有實驗重復3次,數據結果用平均值±標準誤差表示,數據處理使用SPSS 19.0軟件進行單因素方差分析,采用LSD法進行兩兩比較,以P<0.05為差異有統計學意義。熒光定量標準曲線、擴增、熔解曲線使用羅氏Light Cycler? 2.0全自動熒光定量PCR儀自帶軟件自動生成。
2 結 果
2.1普通RT-PCR結果 使用P289和P290引物,對6株NoVs陽性樣品進行普通RT-PCR擴增。由圖1擴增產物電泳圖可知:P289/P290引物可以特異性擴增GI和GII NoVs陽性毒株,不能擴增RV和AstV,說明引物特異性良好,RT-PCR擴增產物大小與預期319 bp相符,反應干擾少,說明擴增反應準確。

備注:從左到右M為DL2000 maker,1-2為 GI.1;3-4為GI.3;5-6為GI.6;7-8為GII.4;9-10為GII.12;11-12為GII.17;13-14為RV;15-16為AstV;17為空白對照圖1 不同型別諾如病毒的普通RT-PCR結果Fig.1 RT-PCR results of different genotype NoVs
2.2熒光定量檢測 從圖2可以看出,在病毒標準品為102~109copies范圍內,熒光定量擴增曲線具有良好的擴增,RV、AstV和空白對照均沒有擴增,說明反應特異性良好;從圖3可以看出,在病毒標準品為103~109copies范圍內的標準曲線性關系良好(R2=0.993,P<0.01),說明定量反應準確。
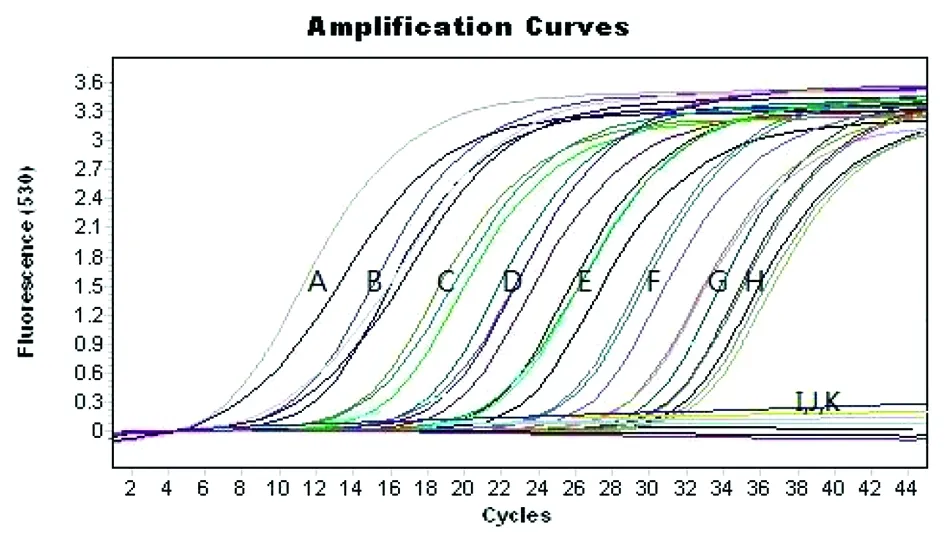
從左到右A-H分別為3.5×109,3.5×108,3.5×107,3.5×106,3.5×105,3.5×104,3.5×103,3.5×102 copies的諾如病毒質粒標準品,I,J,K分別為RV,AstV和空白對照圖2 熒光定量反應擴增曲線Fig.2 Amplification curve of fluorescence quantitative reaction
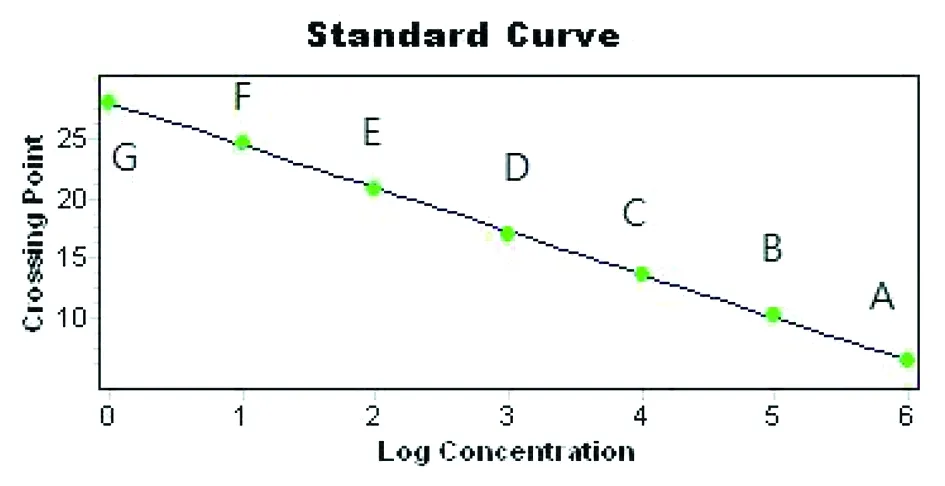
從左到右A-G分別為3.5×109,3.5×108,3.5×107,3.5×106,3.5×105,3.5×104,3.5×103 copies的NoVs質粒標準品圖3 熒光定量標準曲線Fig.3 Standard curve of fluorescence quantitative
2.3準確性和特異性 6株不同型別的NoVs陽性樣品經過SYBR Green I法熒光定量PCR擴增后,進行熔解曲線分析。由圖4可以看出,GI諾如病毒和GII諾如病毒擴增曲線可以明顯區分,其中GI諾如病毒對應的Tm值在85 ℃左右,GII諾如病毒對應的Tm值在86 ℃左右,區分度明顯,型別鑒別與實際樣品型別相符,輪狀病毒、星狀病毒和空白對照均無擴增曲線,也無Tm值,說明反應特異性良好。6株不同型別的NoVs陽性樣品的熔解曲線Tm值與區分度情況見表1。可以看出,GI諾如病毒的Tm值兩兩比較,差異無統計學意義(t1=0.35,t2=0.59,t3=0.23,P>0.05),GII諾如病毒的Tm值兩兩比較,差異無統計學意義(t1=0.35,t2=0.18,t3=0.18,P>0.05),GI諾如病毒的Tm值為85.15±0.02,GII諾如病毒的Tm值為86.06±0.01,兩組基因群之間進行方差分析,差異有統計學意義(F=7507.60,P<0.05 ),因此,通過Tm值不能區分GI或GII同一基因群內的不同型別,但可以用來區分GI和GII基因群間的NoVs。

圖4 不同型別諾如病毒的熔解曲線分析Fig.4 Analysis of dissolution curve of Norovirus different genotype NoVs
表1 不同型別諾如病毒Tm值分析
Tab.1 Analysis of Tm value of different genotype NoVs

基因型Tm 值GI.185.13±0.11aGI.385.15±0.08aGI.685.16±0.05aGII.486.07±0.04bGII.1286.05±0.06bGII.1786.06±0.05b
注:Tm值兩兩比較,同列數字后字母不同表示比較有統計學差異(P<0.05),字母相同表示比較后無統計學差異(P>0.05)。
2.4方法的穩定性 將6株不同型別的陽性NoVs人工污染牡蠣后,分3個時間批次利用熒光定量PCR熔解曲線法測定Tm值,計算3個批次樣品的Tm平均值、標準偏差、變異系數。從表2中可以看出,對3個檢驗批次NoVs的Tm值均沒有較大變化,RSD均小于0.1%,GI和GII NoVs的Tm平均值方差分析,差異有統計學意義(F=2162.25,P<0.05),GI.1組內不同批次的Tm值兩兩比較,差異無統計學意義(t1=0.08,t2=0.40,t3=0.32,P<0.05),GI.3組內不同批次的Tm值兩兩比較,差異無統計學意義(t1=0.34,t2=1.80,t3=1.46,P<0.05),GI.6組內不同批次的Tm值兩兩比較,差異無統計學意義(t1=0.43,t2=0.22,t3=0.22,P<0.05),GII.4組內不同批次的Tm值兩兩比較,差異無統計學意義(t1=0.81,t2=2.00,t3=0.49,P<0.05), GII.12組內不同批次的Tm值兩兩比較,差異無統計學意義(t1=0.10,t2=0.28,t3=0.38,P<0.05), GII.17組內不同批次的Tm值兩兩比較,差異無統計學意義(t1=0.59,t2=1.33,t3=0.74,P<0.05),上述結果表明建立的鑒別方法穩定性強,具有良好的重現性。
2.5樣品中諾如病毒檢測與鑒定 對2015年1-12月采集的不同貝類樣品中NoVs的檢測,并利用本研究建立的方法、RT-PCR擴增后測序鑒定方法進行比較。從表3中可以看出,120份貝類樣品中共有7個樣品檢出了NoVs陽性結果,檢出率為5.83%,其中有2份樣品(牡蠣和毛蚶)中出現了2個Tm值,說明這兩份樣品同時存在GI和GII NoVs,其他樣品均出現1個Tm值,說明這些樣品存在單一類型的NoVs。經過測序鑒定與本方法的結論進行比對,7個樣品的判定結論均一致,說明本研究建立的方法適合貝類中GI和GII NoVs的快速檢測與鑒定分群。
3 討 論
國際上按照NoVs基因組聚合酶區和衣殼蛋白區的序列,將NoVs被分為5個基因組(Genogroups)[21],其中GI和GII以及GIV NoVs感染人類[22-23]。根據NoVs的核苷酸序列,GI和GII NoVs可進一步分為8和17個基因型(Genetic Cluster 或Genotype)[24]。近年來,NoVs的基因重組和變異的不斷出現,GI和GII NoVs 進一步分為9個和29個基因型。對幾個NoVs全長基因組序列的分析表明,同一基因群內不同病毒株的核苷酸序列同源性為69%~97%,而不同基因群的病毒株核苷酸序列同源性只有51%~56%[25],這為本研究利用核苷酸序列開展不同基因群間的快速鑒定奠定了基礎。
貝類引起的NoVs疫情暴發中,有1個顯著的特點是在感染者和受污染的貝類中可以檢測出多個型別的NoVs,盡管針對混合基因型NoVs的多重檢測方法已經建立[26-27],但卻無法快速分群分型,傳統的分群分型方法仍然以基因擴增、測序、比對分析為主,耗時較長,尤其在貝類中NoVs含量低、型別復雜、存在樣品成分干擾等情況下,NoVs快速檢測和分群分型就顯得尤為迫切。因此,如何快速地確定貝類中NoVs污染的基因群類別,對于追溯污染的貝類、發布NoVs疫情預警等具有重要的意義。本研究探索分析了目前流行的NoVs毒株及參考分群分型毒株的基因序列,篩選了可以同時檢測GI、GII基因群的引物,建立了基于SYBR Green I 熒光定量檢測及擴增熔解曲線分析方法,用陽性樣品和實際貝類樣品進行檢測驗證,均與測序方法得到的分型結果一致,該方法既可以實現定量檢測,又可以快速分群,為貝類中NoVs快速檢測和分群分型提供了技術支撐。
表2 不同檢驗批次諾如病毒的Tm值
Tab.2 Results of different test batches for Norovirus’ Tm

樣品Tm值 GI.1GI.3GI.6GII.4GII.12GII.17A185.2385.0985.2286.0486.0386.08A285.1285.1285.0186.0386.0686.05A385.1585.1485.1986.0986.0586.13B185.0985.1185.0886.1186.0286.08B285.2185.0885.1286.0886.0586.09B385.1985.1985.1486.1286.0886.05C185.1385.1885.1986.0185.9886.04C285.1585.1685.1186.1386.0286.06C385.1785.1185.0886.2686.1186.07平均值85.16b85.13b85.13b86.10a86.04a86.07a標準差0.080.050.040.070.040.03相對標準偏差(%)0.090.050.050.080.040.03
注:A、B、C分別代表不同的檢驗批次,對不同型別NoVs的Tm值的平均值進行兩兩比較,同行數字后字母不同表示差異有統計學意義(P<0.05),字母相同表示比較后差異無統計學意義(P>0.05)。
表3 陽性樣品的檢測與基因群鑒定
Tab.3 Detection and genogroups identification of positive samples

月份樣品Tm1 Tm2 本方法測序方法一致性1月牡蠣85.1486.09GI,GIIGI,GII是2月扇貝85.12-GIGI是2月牡蠣-86.07GIIGII是3月毛蚶85.1186.07GI,GIIGI,GII是11月菲律賓蛤仔-86.08GIIGII是12月牡蠣85.16-GIGI是12月扇貝-86.07GIIGII是
備注:-表示未檢出。
利益沖突:無

