中國蘭中幾種常見DNA條形碼標準序列的PCR擴增方法
巫偉峰,陳孝丑,陳發興,黃曉慧,陳 春,張毅智,汪長水
(1.福建省林業科技試驗中心,福建 南靖 363600;2.福建農林大學園藝學院,福建 福州 350000;3.福州植物園,福建 福州 350000;4.貴州省植物園,貴州 貴陽 550000)
【研究意義】中國蘭是國人對中國傳統蘭花的統稱,簡稱國蘭,屬蘭科(Orchidaceae)蘭屬(Cymbidium)植物,主要有春蘭、建蘭、墨蘭、蕙蘭、寒蘭、春劍、蓮瓣蘭和豆瓣蘭等八大類[1]。從古至今,國蘭一直為我國文人雅客所稱頌,觀其態、聞其香、品其韻,素有“花中君子”、“王者香”等美譽,具有極高的觀賞及經濟價值。我國具有豐富的野生國蘭種質資源,但由于過度盜采或不合理開發,許多珍貴資源正逐漸趨于瀕危。近年,隨著我國對國蘭遺傳資源保護與開發利用的日益重視,我國學者陸續開展了國蘭種質資源調查[2]、形態學標記[3]、細胞學標記[4]、生化標記[5]及分子標記[6-8]等方面的研究,在國蘭的系統發育學、物種鑒定、遺傳多樣性及親緣關系等方面已然具有一定的技術積累,但在分析效率、準確性、專業性要求等方面仍然存在諸多局限。因此,探尋一種快速、容易上手、結果可靠的遺傳鑒定策略十分必要。【前人研究進展】DNA條形碼(DNA barcoding)技術是利用基因組中一段公認標準的 DNA 片段來進行物種鑒定的分子診斷新技術,該概念自加拿大分類學家Paul Hebert(2003)首次提出后,便受到各國科學家的廣泛關注,是近年來植物分子系統學研究的熱點[9],已成功應用于生物物種分類和鑒定、生態學調查及生物多樣性評估等研究領域[10]。目前被提議用于DNA條形碼技術的標準DNA片段主要有rpoB、rpoC1、matK、rbcL、psbA-trnH、atpF-atpH、psbK-psbI、ITS、ITS2等核酸片段[11]。其中葉綠體基因片段matK和rbcL在2009年被國際條形碼協會植物工作組提出作為陸地植物通用的條形碼序列,并建議將葉綠體基因片段psbA-trnH和核糖體DNA ITS作為補充條形碼[12]。2011 年,中國植物條形碼研究組通過對1757種植物的psbA-trnH、ITS/ITS2及rbcL+matK序列或序列組合進行鑒別能力評價,提出將ITS/ITS2序列作為種子植物的核心條形碼,psbA-trnH作為輔助條形碼序列[13]。為了實現國蘭鑒定的自動化和標準化,突破對經驗及專業的局限,建立一套有效的國蘭DNA條形碼鑒定技術體系。【本研究切入點】本文建立國蘭中幾種常見DNA條形碼的PCR擴增方法,并利用電泳成像、BLASTN等技術驗證方法的有效性。【擬解決的關鍵問題】為進一步研究國蘭的DNA條形碼技術提供參考和借鑒。
1 材料與方法
1.1 試驗材料
試驗樣品取自福建省南靖縣,樣品信息見表1。采取健康、無病蟲害新鮮嫩葉,使用75 %酒精對葉片表面進行清潔,做好樣品相關信息標記,液氮速凍,置于-80 ℃超低溫冰箱保存備用。
1.2 試驗方法
參照本課題組黃曉慧等文獻報道[14]中的研磨儀法提取樣品DNA。PCR擴增反應體系在高連明等方法的基礎上進行適當優化[10]。修改后方法如下,PCR反應液組成:總20 μl 體系,10 μl 2 ×TaqMaster Mix(廈門泰京生物技術有限公司),1 μl 模板DNA,上下游引物各0.5 μl,無菌水8 μl。引物信息見表2,委托北京六合華大基因科技股份有限公司進行合成。PCR反應程序(BIO-RAD T100TM PCR儀):94 ℃預變性 4 min,94 ℃變性45s,退火 1 min(溫度見表2),72 ℃延伸 1 min,循環數35,72 ℃終延伸 10 min。PCR產物通過經天根DNA純化回收試劑盒(DP214-03)純化后,委托北京六合華大基因科技股份有限公司進行雙向測序。

表1 樣品信息表

表2 引物信息列表
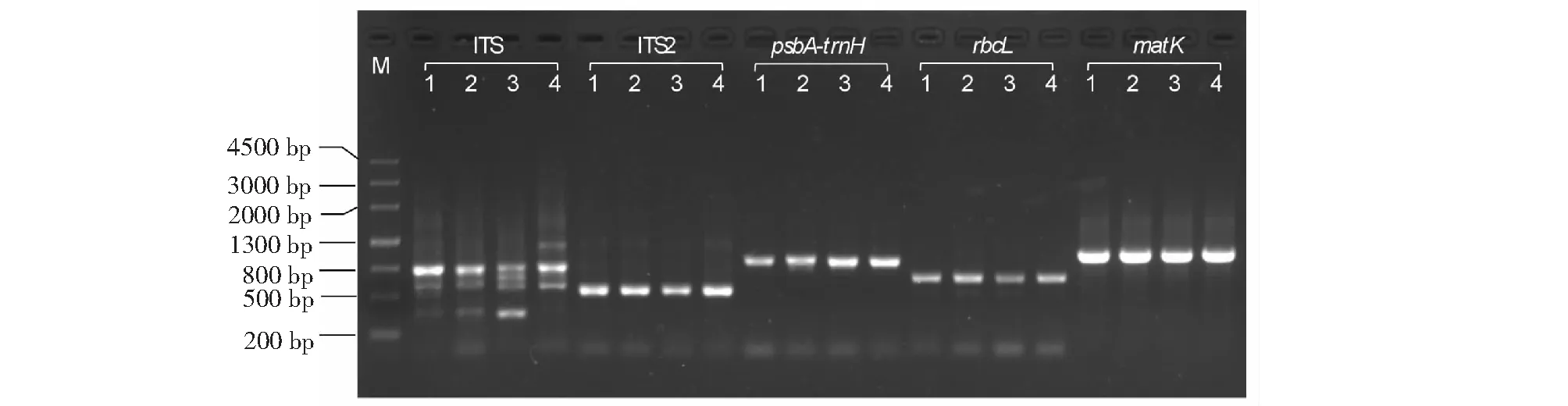
M 表示標準分子量,數字編號對應表1樣品M represents the standard molecular weight, and the numerical number corresponds to the samples of table 1圖1 國蘭中ITS2、psbA-trnH、rbcL、matK的PCR擴增產物電泳圖Fig.1 Electrophoretic map of PCR amplification products of ITS2, psbA-trnH, rbcL and matK in Chinese orchid
1.3 數據分析
測序結果拼接和質量評估使用CodonCode Aligner V2.06(CodonCode, USA)軟件進行分析。序列質量評價判斷方法參照國際DNA 條形碼協會(CBOL)的通用標準[11]。ITS和ITS2序列采用隱馬爾可夫模型HMMer真核生物的注釋方法,去除序列兩端5.8S和26S區段獲得ITS2間隔區序列[22]。psbA-trnH、rbcL、matK等3個條形碼序列直接利用CodonCode Aligner V2.06(CodonCode,USA)軟件除去引物區獲得相應序列。序列驗證使用NCBI網站的BLASTN相似序列檢索在線工具。
2 結果與分析
2.1 PCR擴增產物電泳檢測
由圖1可知,除ITS存在非特異擴增外,ITS2、psbA-trnH、rbcL、matK均在相應分子量位置獲得單一特異條帶。結果表明White等[15]設計的ITS通用引物不適用于國蘭,ITS2、psbA-trnH、rbcL、matK等幾種條形碼序列通用引物可以用于國蘭的DNA條形碼分析。
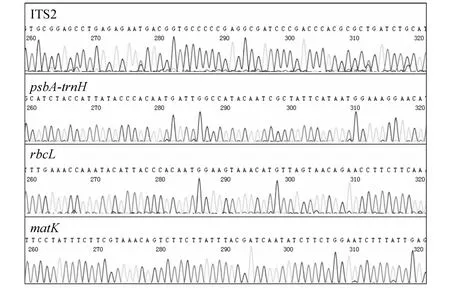
圖2 ‘復興奇蝶’的 ITS2、psbA-trnH、rbcL、matK 條形碼序列測序局部峰圖Fig.2 Local peak map of ITS2, psbA-trnH, rbcL and matK sequences of ‘Fuxingqiedie’

表3 序列的BLASTN驗證結果
注:C.為Cymbidium.縮寫。
2.2 序列驗證
通過對PCR擴增獲得的ITS2、psbA-trnH、rbcL、matK產物進行雙向測序,測序成功率為100 %,成功獲得16條條形碼序列如圖2所示。采用NCBI的BLASTN核苷酸序列相似檢驗在線工具對獲得16條序列進行相似序列檢驗如表3所示,列出每個供試樣品相應條形碼序列相似總得分排名前5的物種名稱,結果顯示所獲16條序列均與蘭屬物種具有極高相似度,表明通過本研究PCR擴增方法可以有效獲得相應條形碼序列。
3 小 結
本研究通過電泳成像、BLASTN核苷酸序列相似度檢驗等分析手段,驗證了國蘭中常見幾種DNA條形碼PCR擴增方法的有效性,結果表明文中所報道的PCR擴增方法可以用于國蘭ITS2、psbA-trnH、rbcL、matK等幾種條形碼的分析研究。ITS序列由于引物存在非特異擴增,需要設計或選擇新的引物進行嘗試,這可能與ITS序列兩端保守性較低有關,針對不同類群需要相適宜的引物,通用性較差。高連明等針對ITS通用性較差的問題,列出了多個目前種子植物ITS通用性較高的引物組合,3個正向引物(ITS5、ITS5a、ITS1)均可以與反向引物ITS4組合進行PCR反應[10]。
相比前人的DNA條形碼技術PCR反應體系[10, 23],本研究的PCR反應溶液操作體系更為簡單,這得益于混合酶試劑的使用。傳統的PCR反應溶液需要加入DNA聚合酶(Taq酶)、反應緩沖液(10 × buffer)、dNTPs混合物(甚至dATP、dTTP、dCTP、dGTP)、上游引物和下游引物、MgCl2、無菌水等試劑,加樣操作繁瑣。而混合酶試劑是由TaqDNA聚合酶、dNTP混合物、MgCl2以及反應緩沖液制成的混合試劑,試劑可以直接用于PCR擴增,且不影響反應效果和后續測序,使用混合酶只需在PCR反應溶液中加入混合酶、上游引物和下游引物、無菌水及DNA模板即可進行PCR反應,大大簡化了PCR反應操作流程。本研究結果顯示,該混合酶可以有效用于國蘭ITS2、psbA-trnH、rbcL、matK等幾種條形碼的PCR反應,是提高國蘭DNA條形碼鑒定效率的一種有益嘗試。

