高通量測序技術在釀酒微生物多樣性研究中的應用
吳 成,王春曉*,王曉丹,周鴻翔,黃永光,邱樹毅*
(貴州大學釀酒與食品工程學院,貴州省發酵工程與生物制藥重點實驗室,貴州 貴陽 550025)
中國酒類產品種類豐富,包括白酒、米酒、黃酒、啤酒及葡萄酒等,均具有悠久的釀造歷史,各具獨特的風味。其中,茅臺酒、紹興黃酒及煙臺張裕葡萄酒等產品更是以其獨特的風味聞名于世。據報道,2009年,中國白酒年消費量已超過40億 t[1],深受國內外消費者的喜愛。中國酒類產品的獨特風味與其釀造過程中微生物的消長演替相關,細菌、酵母菌、霉菌等微生物在其生產及風味形成過程中發揮了至關重要的作用。釀酒原料、釀造工藝、地理位置及氣候條件等因素會影響釀酒微生物群落結構,因此,釀酒微生物多樣性與中國各酒類產品的風味特征息息相關[2]。
自1904年日本學者齊藤研究中國紹興酒中釀酒微生物的多樣性至今已有100多年的歷史[3]。起初對釀酒微生物多樣性的研究主要依賴于傳統的技術手段,得到的研究結果具有一定的局限性。現代分子生物技術的發展促進了越來越多不依賴于培養的技術的發展與應用。對葡萄酒釀造過程中微生物多樣性的相關研究發現,原位雜交技術[4]、實時定量聚合酶鏈式反應(real-time?quantitative?polymerase chain reaction,qPCR)[5]、基因芯片技術[6]、變性凝膠梯度電泳技術[7-8]及高通量測序技術[9]等不依賴于培養的技術,的確有助于對處于存活但不可培養狀態的菌群或細胞濃度較低菌群的檢測、鑒定及定量分析[10-11]。近年來,由于高通量測序技術具有測序時間短、測序通量高的優勢,國內外研究者將該技術應用于釀酒微生物多樣性的研究中,促進了研究者對于釀酒微生物多樣性的認識。本文在介紹高通量測序技術的基礎上,分析其在釀酒微生物多樣性研究中的應用情況,旨在為中國酒類產品的發酵機理、質量與風味控制等相關領域的深入研究提供理論指導。
1 高通量測序技術簡介
高通量測序又稱下一代測序、大規模測序等,由第一代測序技術即Maxam-Gilbert和Sanger測序發展而來[12]。與第一代測序技術相比,高通量測序技術可以在較短的時間內對數以千計甚至百萬計的序列進行檢測分析。因此,高通量測序技術不僅有利于復雜微生物群落多樣性的快速分析,而且可以檢測到低存在率的微生物以及存活但不可培養的微生物,從而提供更為全面的微生物群落信息。
近年來,結合本課題組采用高通量測序技術對酒樣(如白酒釀造過程中的大曲、葡萄酒釀造過程中的發酵液等)中微生物群落多樣性進行的研究可知,高通量測序技術流程一般包括采樣、提取及純化核酸序列、構建文庫、測序、原始序列信息比對注釋、數據分析等步驟(圖1)[10,12-15]。整個技術流程中,關于高通量測序平臺的選擇和數據分析方法的使用兩方面的報道較少,而其對微生物多樣性的研究十分重要,因此本文在技術部分將著重介紹這兩方面的內容。
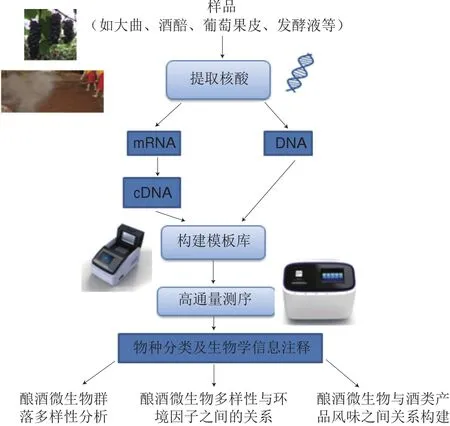
圖1 高通量測序技術流程圖(針對釀酒微生物多樣性研究)[10,12-15]Fig.1 Flow chart of high-throughput sequencing technology applied to analyze the diversity of microbial communities involved in the fermentation of alcoholic beverages[10,12-15]
1.1 高通量測序平臺
目前可采用的高通量測序平臺主要包括Roche 454焦磷酸測序平臺、Illumina測序平臺、SOLiD測序平臺、Ion Torrent測序平臺和PacBio SMRT測序平臺(第三代測序平臺)。這五大主要測序平臺在測序原理、測序試劑、序列讀取長度、測序數量等方面都存在差異(表1)[9,16-20]。由于釀酒微生物特定核酸片段序列長度一般在1 000 bp以內,因此在微生物多樣性研究中較常采用的為Roche 454和Illumina測序平臺。
1.2 高通量測序技術常用的數據分析方法
高通量測序平臺輸出的是原始序列數據reads,研究者一般將其分類鑒定為可操控分類單元(operational taxonomic units,OTUs)后再進行統計分析。OTUs的獲取主要通過與已建立的基因數據庫比對以獲得分類學注釋。但是,數據庫中未儲備信息的菌種無法得到分類鑒定。目前,常用的基因數據庫有GenBank[21]、RDP[22]、SILVA[15]、Greengenes[23]和UNITE[24]等。近年來研究者廣泛使用Mothur[25]和QIIME[26]進行分類學注釋及后續統計分析。在后續的分析中,常用的多元統計學方法有聚類分析、判別分析、排序分析、相關性分析和差異性分析等[27]。采取合適的數據分析策略可以準確反映釀酒微生物群落的多態性,溯源酒類產品的風味物質,把握釀酒微生物與環境因子的相互作用以及調控其中的健康因子和有害物種等[28]。在釀酒微生物多樣性分析研究中常用的數據分析方法包括主成分分析(principal component analysis,PCA)、典型相關分析(canonical correlation analysis,CCA)及相對豐度熱圖。
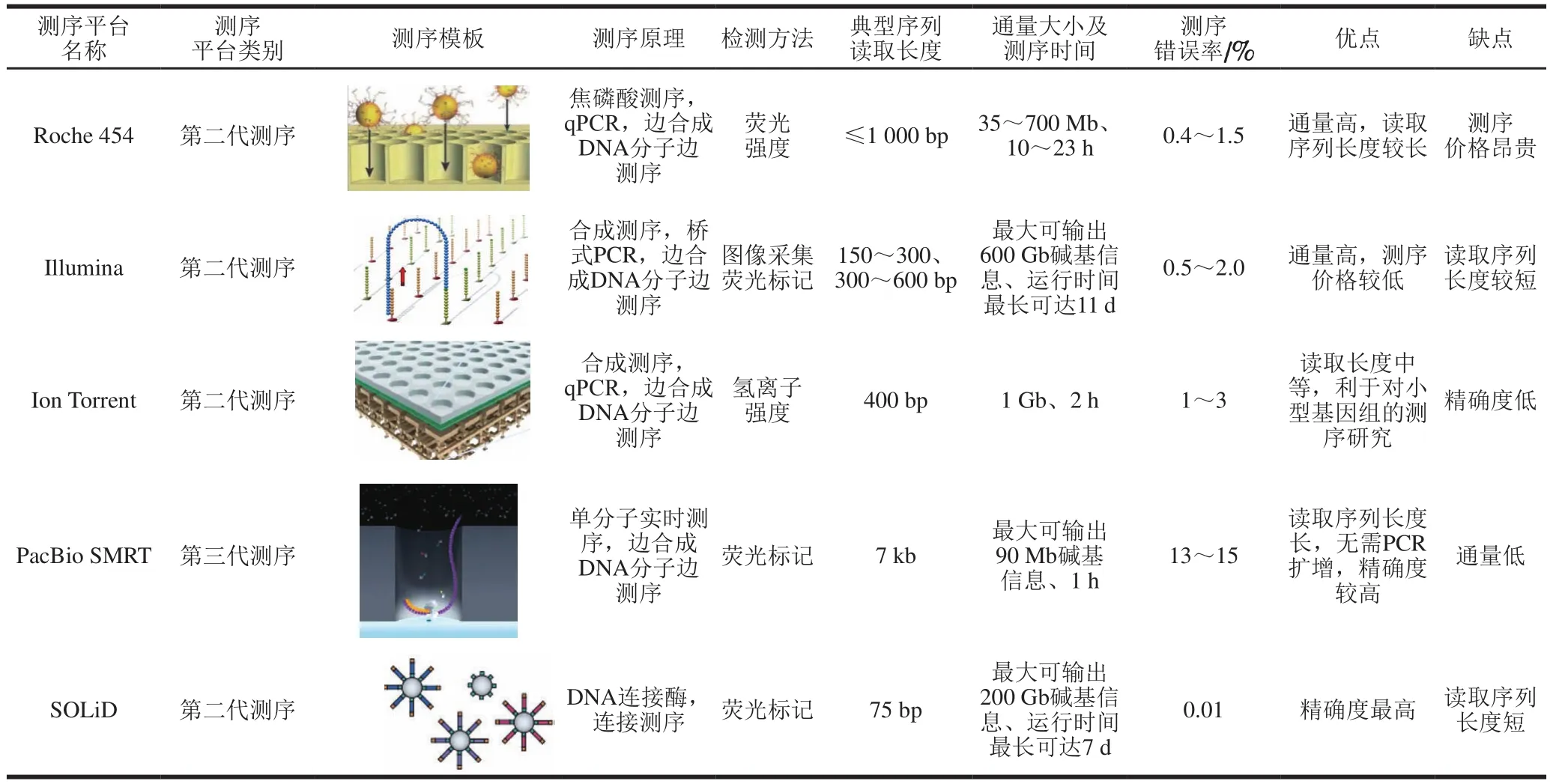
表1 5 種高通量測序平臺的測序原理、讀取序列長度和優缺點[9,16-20]Table1 Principle, sequencing length, merits and drawbacks of fi ve different high-throughput sequencing platforms[9,16-20]
1.2.1 PCA
PCA是多元統計學分析方法的一種。主要是通過對大量的樣本原始數據進行降維處理,從而分析樣本之間差異性,并且可以對不同樣本進行歸類分析。PCA可使用SPSS、CANOCO等軟件進行分析,其分析結果一般可通過得分圖和載荷圖體現。前者可以反映出各樣本之間的差異性,而后者則可以反映出導致樣本間產生差異性的具體原因[29-32]。PCA將測序數據反映在坐標軸行與行、行與列及列與列之間,直接反映其獲得的主成分得分及變量得分,可為其他分析提供基礎。但是,PCA中各樣本間關系僅通過散點間距衡量,結果判斷帶有較強的主觀性。本課題組采用高通量測序方法對3 種醬香型白酒中細菌微生物群落結構進行研究,采用PCA可以區分出3 種醬香型白酒大曲,并指出了產生差異的原因[33]。
1.2.2 CCA
CCA是利用綜合變量之間的相關關系來反映兩組指標間整體相關性的多元統計分析方法。其基本原理是:基于從總體上需要把握兩組指標之間的相關關系,分別從兩組變量中提取有代表性的兩個綜合變量U1和V1(分別為兩個變量組中各變量的線性組合),利用這兩個綜合變量之間的相關關系來反映兩組指標之間的整體相關性。CCA可以通過CANOCO軟件對數據進行處理分析。借由CCA能夠反映出釀酒微生物與環境因子之間的響應關系[34-35]。該方法不僅可以通過雙標圖反映物種與樣本、樣本與環境、物種與環境之間的關系,而且還可以直接通過三標圖反映這三者之間的關系,降低干擾因素產生的方差。但是,CCA僅表明了其三者之間的一種趨勢關系,結果判定主觀性較強。Zheng Xiaowei等[34]運用CCA闡述了汾酒大曲中微生物多樣性與環境因子(酸度、水分含量及溫度等)之間的關系,其分析結果有助于控制汾酒釀造過程。
1.2.3 相對豐度熱圖
相對豐度熱圖可以反映樣品之間的相似性以及群落結構的相似性[36]。在相對豐度熱圖中,每一個色塊代表一個樣品的物種分類學信息(相對豐度)。一般而言,樣品橫向排列,物種分類學信息縱向排列。相對豐度熱圖可以采用R軟件(http://www.r-projict.org/)或HemI軟件(http://hemi.biocuckoo.org/down.php)進行繪制。除此之外,不同測序公司也會基于高通量測序所得的原始序列數據,為研究者免費提供某些數據分析服務,其中也包括相對豐度熱圖。相對豐度熱圖不僅能夠對物種進行聚類分析,并且注釋有詳細的物種豐度信息,研究者借此即可直觀地對樣本中的物種進行分析。Tang Jing等[36]采用相對豐度熱圖反映出了兩種醬香型白酒大曲中細菌群落多樣性的異同,并在此基礎上進一步分析了其與醬香型白酒風味物質間的關系。
2 高通量測序技術在釀酒微生物多樣性研究中的應用現狀
近年來,高通量測序技術在酒類產品釀酒微生物多樣性的研究中得到了越來越多的關注和應用,涵蓋了白酒、葡萄酒和其他酒類(表2)。其應用主要集中在對釀酒微生物進行分類鑒定、監測發酵過程中釀酒微生物群落動態變化、追溯酒類風味物質以及辨別酒類產品是否摻假等方面[2]。
2.1 高通量測序技術在白酒微生物多樣性研究中的應用
在白酒領域,高通量測序技術多用于研究酒曲中釀酒微生物的多樣性,對于窖泥和酒醅也有相關報道。高通量測序技術在白酒領域主要集中于對細菌多樣性的分析,酒樣主要為醬香、清香、濃香3 種香型。本課題組主要針對醬香型白酒大曲中微生物多樣性進行研究,采用Roche 454焦磷酸測序平臺對3 種醬香型白酒大曲中細菌菌群進行了測序,發現在3 種醬香型大曲中共檢測到35 種細菌科,并且首次在醬香型大曲中報道了芽孢乳桿菌科、鏈霉菌科、假單胞菌科、多孢放線菌科及柄桿菌科等細菌。同時,通過采用PCA作圖,進一步分析出3 種醬香型大曲差異性的原因[33]。運用高通量測序技術對醬香型白酒不同發酵輪次酒中微生物多樣性的研究也有報道[37-38]。本課題組采用高通量測序技術比較了不同輪次酒醅中的細菌與真菌群落結構的異同:發現醬香型白酒發酵過程3 輪次酒醅中細菌優勢菌屬為乳桿菌屬和芽孢桿菌屬,真菌優勢菌屬為嗜熱真菌屬和嗜熱子囊菌屬;而發酵7 輪次酒醅中細菌優勢菌屬為鹽單胞菌屬,真菌優勢菌屬為隱球菌屬[37]。此外,Zhang Xiuhong等[13]采用Roche 454 GS FLX測序平臺,對清香型白酒不同大曲中細菌群落結構進行了研究,結果表明清香型白酒不同大曲中細菌群落結構有較大差異。Zheng Qi等[39]采用Illumina測序平臺探究了濃香型白酒不同年份窖池中窖泥微生物群落多樣性及其與風味成分間的關系。
2.2 高通量測序技術在葡萄酒微生物多樣性研究中的應用
在葡萄酒領域,高通量測序多用于研究發酵過程中微生物的多樣性及動態變化,對葡萄表面的微生物多樣性也有相關報道。Portillo等[40]采用Ion Torrent PGM測序平臺對西班牙Priorat地區不同葡萄園中歌海娜和佳麗釀葡萄表皮上細菌群落多樣性進行了研究,并研究其與地理環境間的關系。結果表明厚壁菌門(Firmicutes)、變形桿菌門(Proteobacteria)和放線菌門(Actinobacteria)為主要的優勢菌,且地理環境分布對發酵過程中的細菌群落結構影響明顯。Wang Chunxiao等[10]采用傳統的分離鑒定技術、高通量測序技術、變性梯度凝膠電泳技術和qPCR技術等不依賴于培養的技術,對產自西班牙Priorat地區3 個葡萄園中的歌海娜和佳麗釀葡萄發酵過程中的酵母菌群落結構進行了相關研究。結果表明,與其他方法相比,高通量測序技術雖無法進行定量檢測,但可檢測到絕大多數的非釀酒酵母菌屬。
2.3 高通量測序技術在其他酒類微生物多樣性研究中的應用
高通量測序技術還被應用于黃酒、米酒、安第斯吉開酒(chicha)等其余酒類領域中,已成功揭示了其各自發酵液或酒曲中的微生物多樣性。Xie Guangfa等[41]采用Illumina?HiSeq?2000測序平臺從門、屬分類學水平對不同發酵時間段紹興黃酒釀酒微生物多樣性進行了研究,結果表明發酵初期和后期其微生物群落結構截然不同,優勢微生物的種類和數量發生明顯變化。Mendoza等[21]采用Roche 454 GS Junior測序平臺從安第斯吉開酒發酵過程中檢測出超過100 種酵母菌,其中超過一半的酵母菌歸屬于絲狀真菌屬。
可見,雖然高通量測序技術被應用于不同的研究領域和樣本,但目前針對釀酒微生物多樣性分析相關研究所使用的測序目標片段、方法和平臺具有相似性。而且,對于相同的釀酒微生物,采取不同的測序目標片段或方法都會影響測序結果的準確度及精確度[10,42-44]。一般對于原核微生物常選用的測序片段為16S rDNA V1~V9各變異區片段,而對于真核微生物而言,常選取的測序片段為18S rDNA、26S rDNA D1/D2區和內部轉錄間隔區(internal transcribed spacer,ITS)等。數據庫的選用也會影響微生物鑒定的準確性,本課題組利用RDP、SILVA和GenBank數據庫對葡萄酒中的真核微生物鑒定分析的對比結果表明,GenBank數據庫更適用于真核微生物尤其是酵母菌的鑒定:其可以實現種水平上的鑒定;并且具備較高的菌種鑒定置信度,利于準確的種水平上菌株的鑒定[10]。目前,原核微生物的相關研究中主要選用的數據庫為RDP和SILVA,但是尚鮮有相關報道對這兩個數據庫進行比對,本課題組擬在后期工作中開展相關研究。
2.4 不同酒類發酵過程中的微生物組成差異
聚焦至不同酒類的發酵過程,高通量測序的結果表明了更明顯的微生物組成差異。目前,采用高通量測序技術對醬香型白酒微生物多樣性的研究主要集中于揭示其細菌群落結構方面,測序發現主要的微生物為芽孢桿菌(Bacillaceae)和乳酸菌屬(Lactobacillus)等。而對醬香型白酒中真核微生物群落結構也有相關報道,發現了嗜熱真菌屬(Thermomyces)、熱子囊菌屬(Thermascus)、隱球酵母屬(Cryptococcus)、青霉菌屬(Penicillium)等[14,33,36-38]。在濃香型白酒發酵過程中,發現厚壁菌屬(Firmicutes)、放線菌屬(Actinobacteria)、醋酸桿菌屬(Acetobacter)、乳酸菌屬(Lactobacillus)、甲烷桿菌屬(Methanobacterium)等為主要的微生物[12,39,45-47]。在清香型白酒發酵過程中報道的主要微生物為厚壁菌屬(Firmicutes)和放線菌屬(Actinabacteria)[13,48]。不同白酒中微生物群落結構的差異是賦予白酒不同香型特征的重要原因。葡萄酒本土風味的形成與其在發酵過程中相關酵母菌屬的消長演替密切相關。研究發現葡萄酒發酵過程中主要的酵母菌屬包括釀酒酵母屬(Saccharomyces)、有孢漢遜酵母屬(Hansenispora)、漢生酵母屬(Hansenula)、畢赤酵母屬(Pichia)、假絲酵母屬(Candida)等。而乳桿菌屬(Lactobacillus)是葡萄酒發酵過程中主要的細菌微生物[10,49-53]。米酒作為中國傳統的發酵食品之一,通過對其微生物多樣性的研究發現,米酒中主要的微生物為芽孢桿菌屬(Bacillaceae)、乳桿菌屬(Lactobacillus)、乳球菌屬(Lactococcus)、高溫放線菌屬(Thermoactinomyces)等[54-55]。高通量測序技術揭示,不同酒類的微生物組成差異主要表現在兩個方面:1)主要微生物菌屬(種)的種類和比例差異;2)小類微生物菌屬(種)及未知菌屬(種)的種類和比例差異。如具有復雜工藝的醬香型白酒釀造過程中發現了很多小類微生物菌屬和未知菌屬,雖然它們在微生物群落結構中所占比例較小,通過其他技術未必能分離出來,但其構成了醬香型白酒獨特微生物組成特征不可忽視的一面。微生物的組成差異受到多種因素的影響,首先是酒類工藝過程,其次是同種酒類工藝在不同地區的釀造過程中發現的微生物組成差異,體現了微生物分布的地域性。
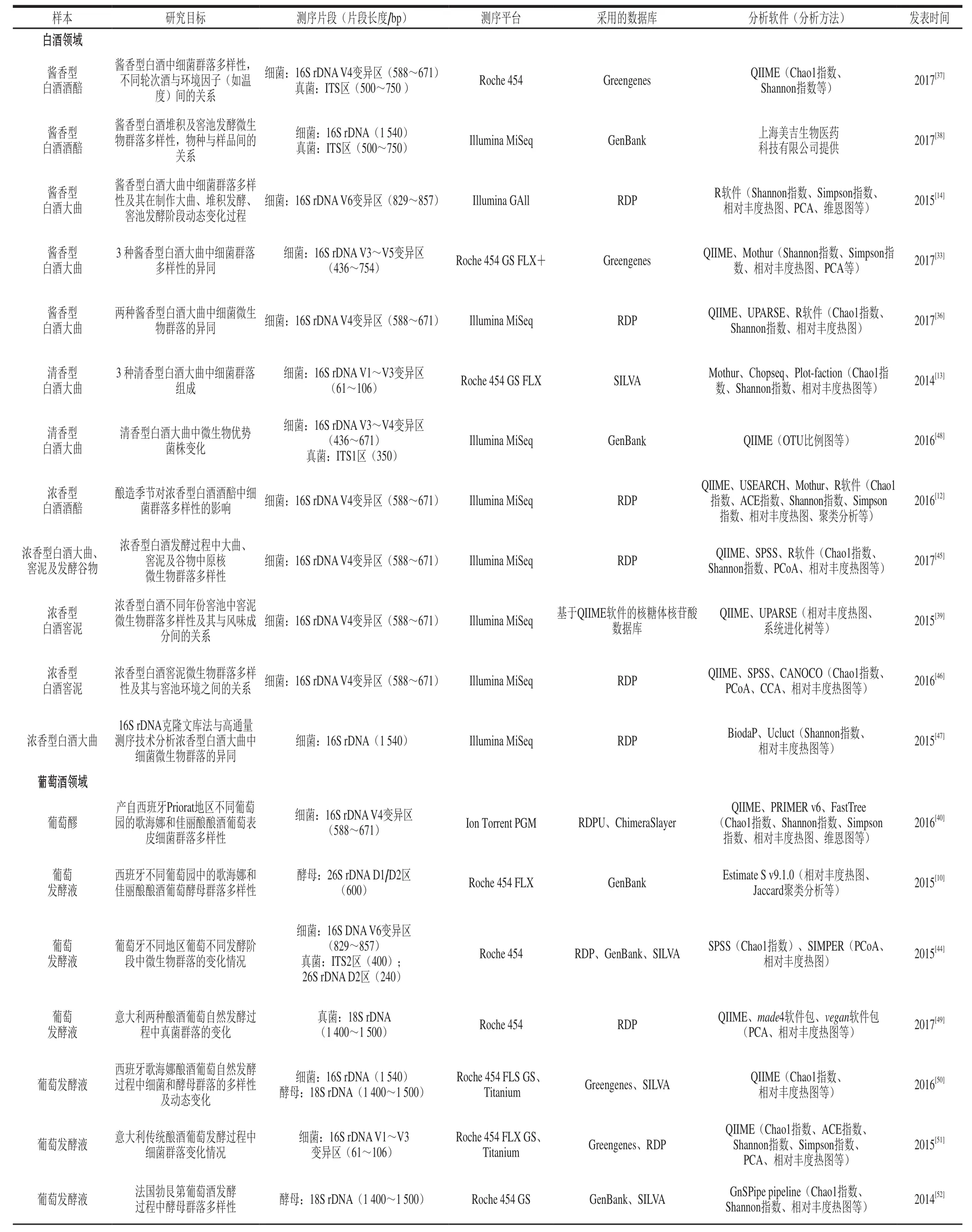
表2 高通量測序技術在釀酒微生物多樣性研究中的應用情況Table2 Recent applications of high-throughout sequencing technology to analyze the diversity of microbial communities involved in the fermentation of alcoholic beverages
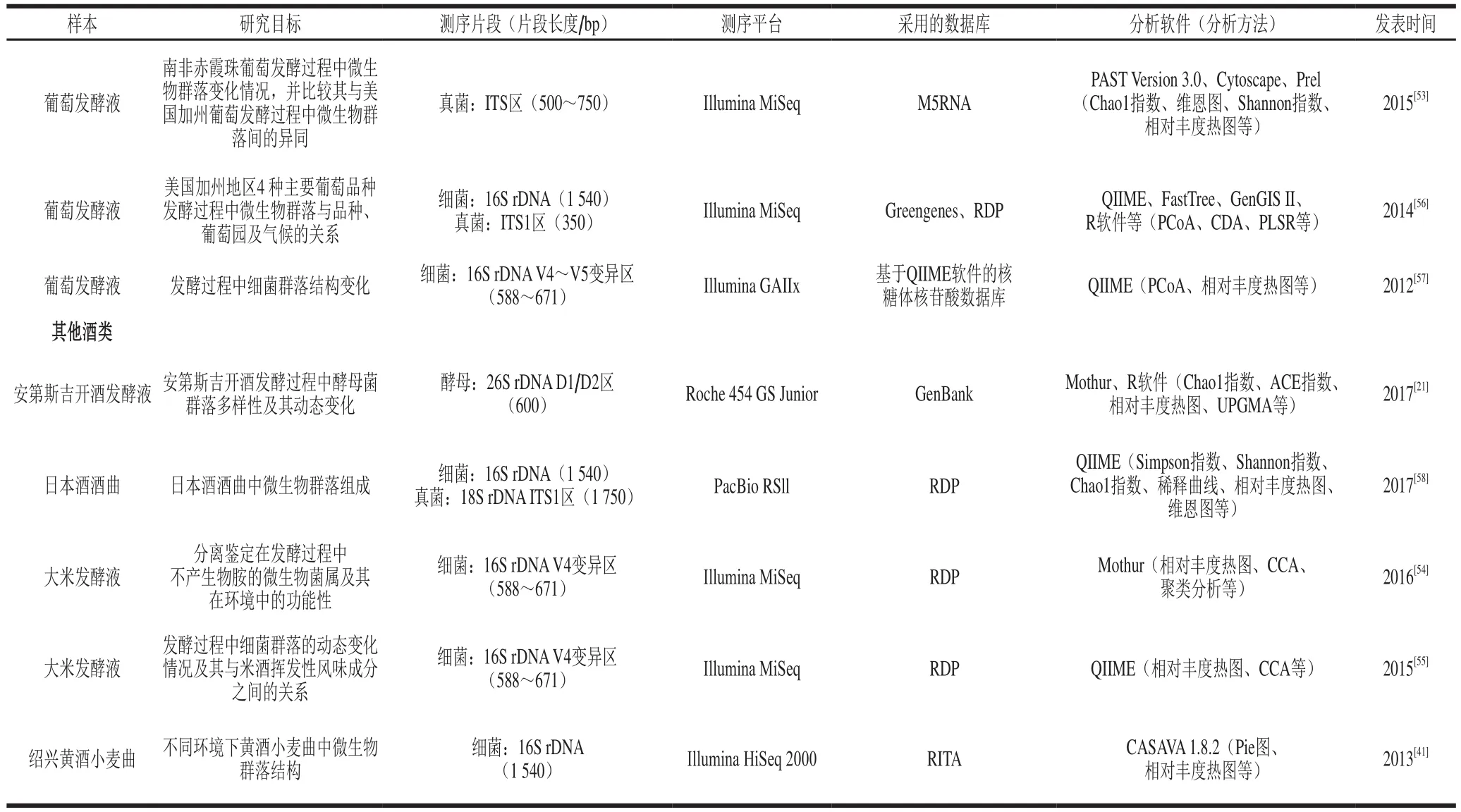
續表2
3 結 語
從高通量測序技術誕生之日起,由于其具有測序方法簡單、通量高、測序速度快和準確度較高等特點,在醫學、食品、環境等研究領域受到廣泛關注。隨著高通量測序技術和測序平臺的不斷發展,該技術的精確度不斷提高而成本逐漸降低,并且測序目標序列讀取長度也得到提高。因此,目前高通量測序技術已被國內外研究者作為研究釀酒微生物多樣性的重要手段。研究者提取不同酒樣中釀酒微生物核酸后,可通過高通量測序技術直接進行測序分析。雖然測序過程簡單,但其獲取的測序數據信息量極為龐大,如何快速從中分析出有效的信息將成為限制研究者采用此方法分析釀酒微生物多樣性的首要因素。如前文所述,數據分析方法如PCA、CCA等的使用,有助于對釀酒微生物多樣性的分析,在此基礎上可進一步建立起微生物多樣性與功能代謝產物之間的關聯[39]。
除此之外,高通量測序技術在應用中還面臨著測序文庫的建立等方面的挑戰。在分析釀酒微生物多樣性時,測序文庫的建立及測序方法需依賴PCR技術。而在PCR過程中,若出現引物退火溫度不適宜、模板DNA代表性不強等情況,則會在一定程度上引入系統偏差和隨機性,從而導致樣本中核酸分子含量與真實含量存在偏差。因此,依賴于PCR技術的高通量測序技術對樣本中目標微生物定量分析的準確度有待提升[59]。建議結合使用其他不依賴于培養的技術實現目標微生物的準確鑒定和定量分析,如qPCR技術通過建立微生物濃度與PCR循環次數之間的標準曲線,可以準確進行菌群濃度的定量分析[60]。高通量測序技術若在建立測序文庫時成功引入類似標準曲線,則將可以初步實現準確的定量分析。
近年來,第三代測序[61]技術逐漸發展,如DNA納米球測序、Heliscope單分子測序、納米孔測序、隧道電流DNA測序、質譜測序及基于顯微鏡測序技術等[59]。就SMRT測序技術而言,其在DNA聚合酶合成DNA分子的同時進行測序,克服了PCR帶來的不確定性和偏差。與以Roche 454焦磷酸測序為代表的第二代測序技術相比,第三代測序技術在測序速度和序列長度上得到了大幅度的提升,SMRT測序技術每秒可測750 個堿基,序列長度最長可達1萬 個堿基[19]。并且第三代測序平臺PacBio(P4/C2)已在PacBio(XL/C2)平臺的基礎上大幅度提高了精確度[20]。盡管其價格昂貴,目前第三代測序技術已開始用于釀酒微生物多樣性方面的分析[20]。相信隨著高通量測序技術的不斷完善,釀酒微生物的多樣性將得到更深入、更準確、更全面的分析與研究。

