二化螟實時熒光定量PCR內參基因篩選和表達穩定性評價
徐紅星 王國榮 魯艷輝,* 楊亞軍 鄭許松 田俊策 呂仲賢,*
?
二化螟實時熒光定量PCR內參基因篩選和表達穩定性評價
徐紅星1王國榮2魯艷輝1,*楊亞軍1鄭許松1田俊策1呂仲賢1,*
(1浙江省農業科學院 農業部農產品信息溯源重點實驗室/省部共建國家重點實驗室培育基地, 杭州 310021;2杭州市蕭山區農業技術推廣中心, 杭州 311202;*通訊聯系人: luyanhui4321@126.com; luzxmh@163.com)
【目的】篩選特定試驗條件下二化螟()穩定表達的內參基因,為二化螟基因表達研究奠定基礎。【方法】根據二化螟轉錄組測序結果挑選出11個候選基因,利用實時熒光定量PCR測定其在二化螟不同發育歷期、不同組織、溫度處理、殺蟲劑處理、取食不同飼料、取食不同水稻、dsRNA處理和混合樣品的表達量,利用RefFinder、BestKeeper、GeNorm和NormFinder等軟件和ΔC值對11個候選基因的穩定性進行評估。【結果】5種分析方法表明,在二化螟不同發育歷期穩定性較高的內參基因是、和,不同組織中穩定性較高的內參基因是、和,不同溫度下較穩定的內參基因為、和,殺蟲劑處理樣品中較穩定的是和,取食不同飼料較穩定的是、、和,取食不同品種水稻較穩定的是和,dsRNA處理樣品中較穩定的是、、和,混合樣品中較穩定的是和。【結論】為不同試驗條件下選擇合適的內參基因提供了參考,也有利于在二化螟基因表達研究中獲得更加可靠準確的數據。
二化螟;內參基因;熒光定量PCR;基因表達
實時熒光定量PCR(qRT-PCR)是目前檢測生物樣品中基因表達差異最靈敏、便捷的方法之一[1]。在利用qRT-PCR進行目的基因的表達水平研究時,需要使用合適的內參基因對實驗樣品進行校正和標準化,以減少轉錄效率、cDNA濃度差異等對結果的影響。這些內源性參照基因(簡稱內參或看家基因)應不受外部環境的影響,在各種組織或細胞中以及各種試驗條件下都能穩定表達[2]。常用的看家基因包括18S核糖體RNA基因(18S rRNA)、微管蛋白基因(tubulin)、甘油醛-3-磷酸脫氫酶基因(glyceral dehyde-3-phosphate dehydrogenase, GAPDH)、核糖體蛋白基因(ribosomal protein)和β-肌動蛋白基因(β-actin)。然而越來越多的研究表明,這些看家基因在用作內參基因時,因為選用的試驗條件不同,穩定性不一[3]。如Gutierrez等[4]報道在用微管蛋白基因(tubulin6)校正發育動態表達時,與已經驗證的內參基因相比, 使用未驗證的內參基因進行校正將對目標基因表達水平的定量出現100倍的偏差。盲目地使用一種看家基因作為內參, 一方面可能導致基因表達的微小差異難以發現;另一方面可能出現錯誤甚至相反的結論[5-6]。因此,篩選和評價在不同試驗條件下穩定表達的內參基因對獲得更準確的試驗結果十分重要。
二化螟[(Walker)],是我國水稻上的常發性害蟲,食性較雜,除為害水稻外,還為害茭白、玉米等。隨著對研究的不斷深入,基因表達水平分析對于研究二化螟的寄主選擇、適應性、抗藥性、滯育和神經調節等相關基因的調控機理方面具有重要作用。因此,篩選穩定的內參基因在二化螟基因表達研究中起著關鍵作用。在之前的研究中已有對多種鱗翅目昆蟲篩選穩定的內參基因的報道,如小菜蛾[7]、斜紋夜蛾[8]、棉鈴蟲[9-11]、甜菜夜蛾[12]、褐蝶[13]、大螟[14, 15]、大斑蝶[16]、蝙蝠蛾[17]、草原毛蟲[18]和桃蛀螟[19]等。盡管之前有二化螟內參基因評價的相關研究[20, 21],探討了在不同組織、不同發育歷期和不同溫度處理下內參基因的穩定性,但本研究還對殺蟲劑處理、不同喂食、取食不同品種水稻和dsRNA處理下內參基因的表達穩定性進行了評估,為不同試驗條件下選擇合適的二化螟內參基因提供依據。
1 材料與方法
1.1 供試昆蟲和不同的處理
二化螟種群于2014年采自浙江省杭州市蕭山區義橋鎮(N 30o04', E 120o12')常規水稻田,置于人工氣候室飼養并維持種群。溫度為(25±1)℃,相對濕度為75%,光周期為16 h光照/8 h黑暗。幼蟲采用人工飼料飼養[22],羽化后的成蟲以10%的蔗糖水補充營養。
不同組織樣本:選擇個體大小一致、健康的3齡幼蟲,饑餓3 h,在預冷的生理鹽水中解剖,獲取頭、中腸、脂肪體和表皮四部分組織。
不同發育時期樣本:收集不同發育階段的樣本,包括第5天的卵、1~6 齡幼蟲(蛻皮后第1天的幼蟲)、早期蛹(第1天的蛹)、晚期蛹(第7天的蛹)、雌雄成蟲(第1天羽化的成蟲)。
溫度處理:分別采用5℃,10℃,15℃,20℃,25℃,30℃,35℃,40℃處理二化螟3齡幼蟲3 h,收集樣品,液氮速凍,置于?80℃超低溫冰箱保存,以備提取RNA用,每個處理3個生物學重復,每個重復收集5頭幼蟲。
藥劑處理:選取三種常用于防治二化螟的藥劑氯蟲苯甲酰胺、阿維菌素、甲氧蟲酰肼(原藥由中國農業大學高希武教授提供),采用稻苗浸漬法進行毒力測定[23]。每種藥劑用含0.1% TritonX-100的清水稀釋5個濃度梯度,0.1% TritonX-100的清水作為空白對照。將分蘗期的水稻莖稈基部放入各處理藥液中浸漬10 s,取出晾干后將稻莖齊根剪下,留下5~6 cm的莖稈。事先準備6.5 cm直徑的培養皿若干,每皿內鋪4層濾紙并加入無菌水保濕。將莖稈放入培養皿中,每個培養皿放置15根莖稈并用毛筆接入10頭3齡幼蟲,每組重復3次。然后用兩層黑布覆蓋。48 h后觀察幼蟲死亡情況,計算LC25。然后以每種藥劑的LC25分別處理二化螟3齡幼蟲,48 h后收集存活的試蟲,每個重復收集5頭,共收集3個重復,液氮凍存置于?80℃下供提取總RNA用。
不同寄主植物飼喂:利用人工飼料、水稻品種TN1(臺中本地1號)的莖稈、茭白和香根草飼喂二化螟3齡幼蟲3 d后,收集樣品,提取總RNA。
不同水稻品種飼喂:用不同水稻品種(TN1和轉基因的明恢63)莖稈飼喂二化螟3齡幼蟲3 d后,收集樣品,提取總RNA。
dsRNA處理:選取大小一致的3齡幼蟲,利用以二化螟乙酰膽堿酯酶基因為靶標的dsRNA(利用TranscriptAid? T7 High Yield Transcription Kit按照說明書操作合成)進行顯微注射(儀器:Nanoject II,Drummond Scientific Company;注射部位:第三和第四節間膜[24]),單頭注射量為1 μL(1.0 μg/μL)的dsRNA,對照采用緩沖液(dsRNA合成試劑盒中提供的溶解dsRNA的緩沖液)。每組注射30頭,注射完3 d后,收集存活的試蟲,每個重復收集5頭,共收集3個重復,?80℃下保存供提取總RNA用。
1.2 總RNA提取和cDNA合成
所有樣本總RNA提取采用Trizol試劑,具體步驟參照Trizol試劑說明書(Invitrogen)。取1 μL RNA樣品測定260/280(Nanodrop 2000,Thermo Fisher Scientific),比值在1.8~2.2之間的樣品用于cDNA合成。
cDNA合成:取3.0 μg總RNA,利用Oligo dT進行cDNA合成,操作步驟參照Thermo Fisher反轉錄試劑盒說明書。cDNA置于?20℃下保存以供實時PCR用。
1.3 qRT-PCR檢測候選內參基因引物特異性和擴增效率
利用Primer 3.0設計二化螟備選基因(表1)。實時熒光定量PCR反應按照iTaqTMUniversal SYBR Green Supermix (Bio-Rad,USA)說明書進行,使用ABI 7300定量PCR儀。反應體系共20 μL,包含1 μL的cDNA,10 μL iTaq Universal SYBR Green Supermix (2×),正反向引物各1 μL,雙蒸水7 μL;反應條件為95℃下30 s,95℃下10 s,55℃下30 s,共40個循環。融解曲線:55℃到95℃,每0.5℃收集一次熒光信號。每個cDNA樣本設置2個技術重復。cDNA按5倍等比稀釋成5個不同濃度,獲得引物擴增效率。
1.5 基因表達穩定性分析
分別利用RefFinder (http://www.leonxie.com/ referencegene.php?type=reference)、BestKeeper[25]、GeNorm[26]、NormFinder[6]等分析軟件和ΔC值[27],評估候選內參基因在不同條件下的表達穩定性。每個樣品重復3次。
2 結果與分析
2.1 引物的擴增效率及特異性評價
qRT-PCR數據顯示,每個候選內參基因的溶解曲線均為單一峰,表明所選的定量引物均具有很好的特異性。同時,基于內參基因不同濃度的cDNA獲得的標準曲線具有良好的決定系數(R>0.987),PCR擴增效率也在97.6%~107.0% (表1)。由此可見,候選內參基因的定量引物設計合理,可以用于相應基因的定量檢測。
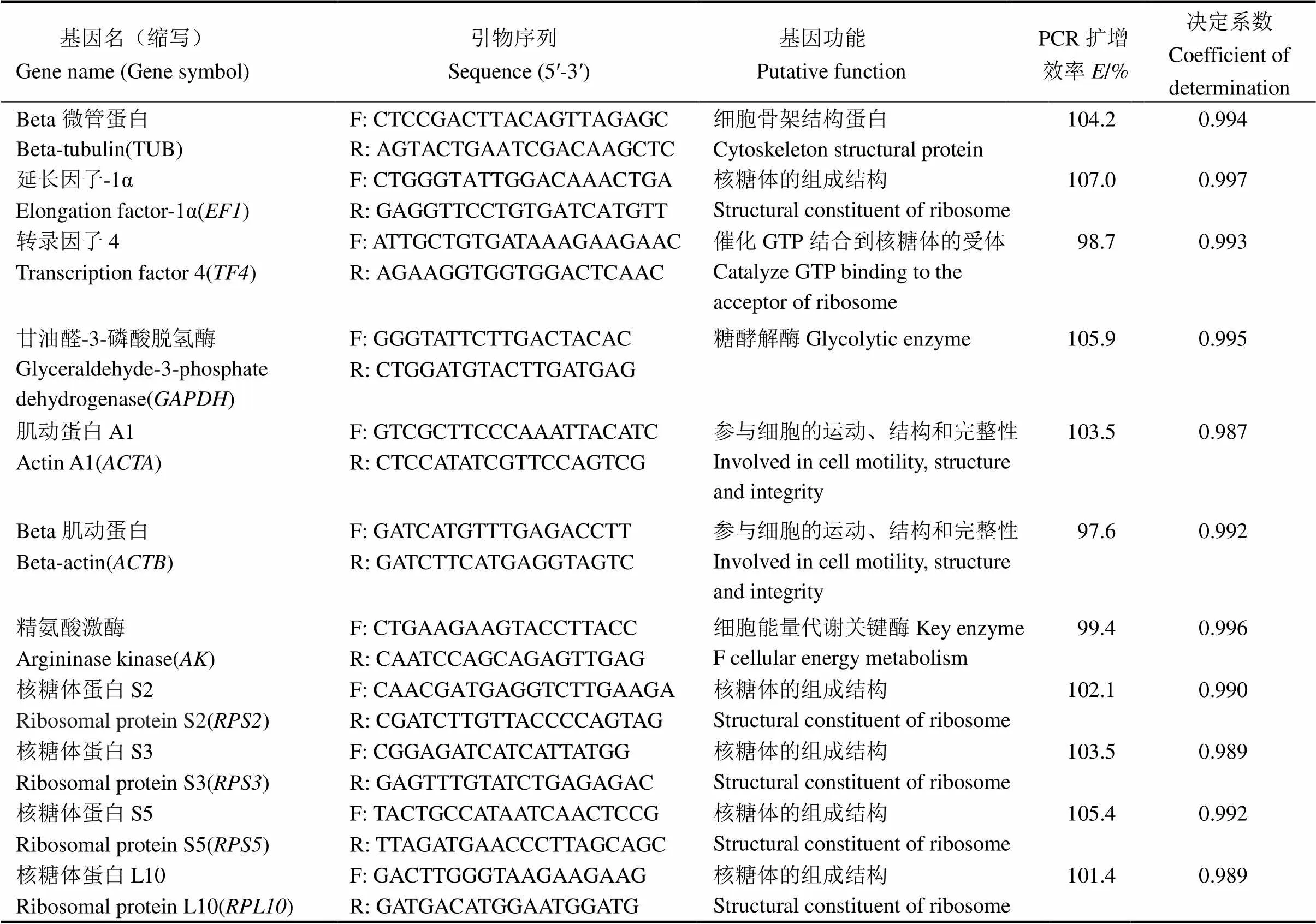
表1 備選基因的功能、引物序列和產物特性
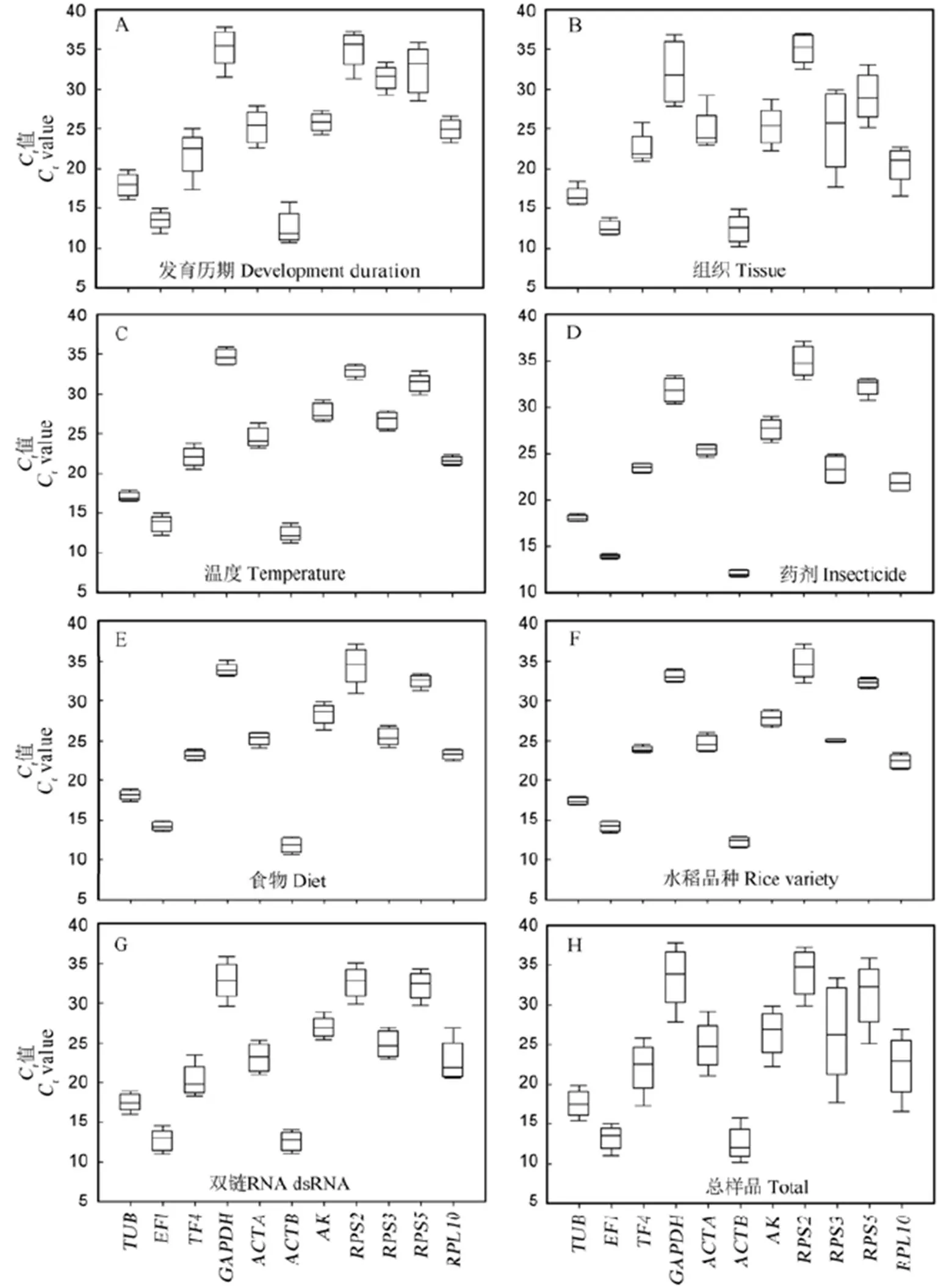
圖1 候選內參基因的表達水平(Ct值)
Fig. 1. Expression levels of candidate reference genes (Cvalues).
2.2 候選內參基因的表達水平分析
檢測了每個候選內參基因在不同條件下的C值,如不同發育歷期(圖1-A)、不同組織(圖1-B)、不同溫度處理(圖1-C)、殺蟲劑處理(圖1-D)、喂食不同飼料(圖1-E)、取食不同水稻(圖1-F)和dsRNA處理(圖1-G)。所有11個候選內參基因的C值中位數為12.08()~34.73()(圖1-H)。其中的表達水平最高,C值為10.22~15.84,其他依次為(11.0~14.99)、(15.50~19.90)、(17.36~ 25.86)、(16.57~26.87)、(21.06~29.19)、(17.76~33.45)、(22.26~29.87)、(25.14~ 35.91)、(27.90 ~37.83)、(29.88~37.29)。在不同樣品間的表達差異最大,而在不同樣品間的表達差異最小(圖1-H)。因此,沒有一種內參基因在所有的試驗條件下都穩定表達。
2.3 候選內參基因在不同發育歷期的表達穩定性分析
在不同發育歷期樣品中,利用RefFinder、GeNorm和NormFinder分析均顯示是穩定性最高的基因。BestKeeper和比較t值分析分別表明和是最穩定的基因(圖2)。另外,5種分析方法均表明是最不穩定的內參基因(圖2)。

Fig. 2. Comparison of the rankings in candidate reference genes expression stability in development durations of, across different tissues ofexposed to different temperatures or insecticides.
根據RefFinder分析,表達穩定性排名為、、、、、、、、、和(圖2)。利用GeNorm軟件判斷配對變異值(V/V+1)來確定內參基因的最佳數目,即當V/V+1<0.15時,該條件下的內參基因最佳數目為個。GeNorm分析顯示在不同發育歷期中所有配對變異值均高于0.15(圖2),說明通過V/V+1不能確定內參基因的最佳數目。此時,我們應選表達穩定性最高的3個基因作為最適內參基因。綜合分析,在不同發育歷期,表達最穩定的3個內參基因為、和(表2)。
2.4 候選內參基因在不同組織的表達穩定性分析
在不同組織,RefFinder、ΔC值和NormFinder分析表明是最穩定的內參基因,而BestKeeper和GeNorm分析顯示是穩定性最高的(圖2)。另外,5種分析方法均顯示是最不穩定的內參基因(圖2)。根據RefFinder分析,表達穩定性排名為、、、、、、、、和(圖2)。GeNorm分析顯示V/V+1均高于0.15(圖2)。綜合分析,在不同組織樣品中,內參基因表達穩定性排名依次為、和(表2)。
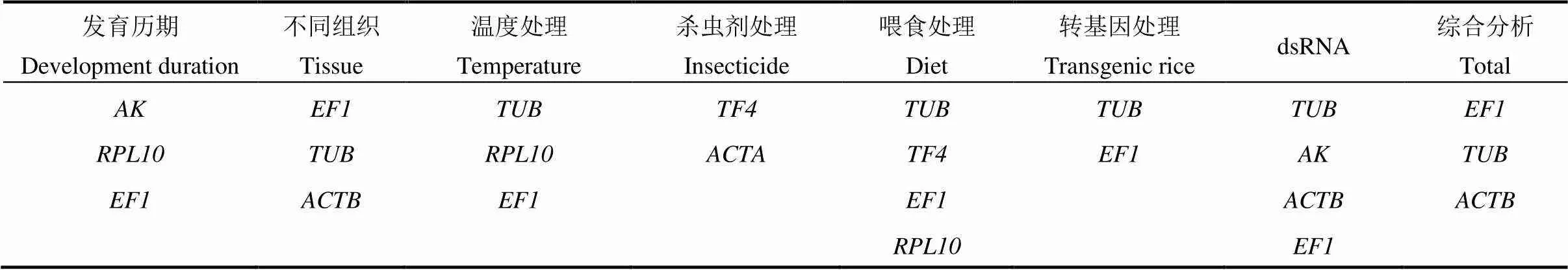
表2 不同實驗條件下的理想內參基因
2.5 候選內參基因在不同溫度處理下的表達穩定性分析
在不同溫度處理下,RefFinder、ΔC值、GeNorm和NormFinder分析均表明是最穩定的內參基因,是最不穩定的,而BestKeeper分析顯示是穩定性最高的,是最低的(圖2)。根據RefFinder分析,穩定性排名為、、、、、、、、和(圖2)。GeNorm分析顯示4/5低于0.15(圖4),因此,需要4個內參基因,然而的平均C值大于30,不適合作為不同溫度處理條件下的內參基因(圖1-C)。綜合分析,在不同溫度處理樣品中,3個表達穩定的內參基因分別為、和(表2)。
2.6 候選內參基因在殺蟲劑處理下的表達穩定性
分別利用氯蟲苯甲酰胺、阿維菌素、甲氧蟲酰肼處理二化螟3齡幼蟲,RefFinder、ΔC值、GeNorm和NormFinder分析均表明是最穩定的內參基因,是最不穩定的,而BestKeeper分析顯示的穩定性最高,最低(圖2)。根據RefFinder分析,穩定性排名為、、、、、、、、和(圖2)。GeNorm分析,2/3低于0.15(圖4),表明需要2個內參基因。因此,在氯蟲苯甲酰胺處理樣品中,2個需要的內參基因為和(表2)。
2.7 候選內參基因在不同喂食處理下的表達穩定性分析
在取食人工飼料、水稻莖稈和茭白條件下,RefFinder和BestKeeper分析顯示是最穩定的內參基因。ΔC值和NormFinder分析表明是穩定性最高的內參基因,GeNorm分析顯示是最穩定的(圖3)。另外,5種分析方法都顯示是最不穩定的內參基因(圖3)。RefFinder分析顯示內參基因排名為、、、、、、、、和(圖3)。GeNorm分析顯示4/5低于0.15(圖4),表明需要4個內參基因,它們分別是、、和(表2)。
2.8 候選內參基因在取食不同水稻品種下的表達穩定性分析
在取食兩種不同品種水稻條件下,RefFinder、ΔC值和NormFinder分析顯示是穩定性最高的內參基因。BestKeeper分析表明是最穩定的內參基因,GeNorm分析顯示是最穩定的(圖3)。5種分析方法均顯示是最不穩定的內參基因(圖3)。RefFinder分析表明內參基因排名為、、、、、、、、和(圖3)。GeNorm分析顯示2/3低于0.15(圖4),表明需要2個內參基因,它們是和(表2)。
2.9 候選內參基因在dsRNA處理下的表達穩定性
在注射dsRNA處理的條件下,RefFinder、ΔC值、GeNorm和NormFinder分析均表明是最穩定的內參基因。BestKeeper分析顯示是穩定性最高的(圖3)。根據RefFinder分析,內參基因表達穩定性排名為、、、、、、、、和(圖3)。根據GeNorm分析,9/10和10/11低于0.15(圖4)。綜合分析,4個最穩定的內參基因是、、和(表2)。
2.10 候選內參基因在所有供試樣品中的表達穩定性分析
為選擇可以通用的內參基因,將所有供試樣品的qRT-PCR的原始數據放在一起進行評估。根據RefFinder、ΔC值和BestKeeper分析,二化螟中最穩定的內參基因是、和;根據GeNorm和NormFinder分析,、和是最穩定的。根據RefFinder綜合分析,內參基因穩定性排名為、、、、、、、和(圖3)。根據GeNorm分析,V/V+1均高于0.15(圖4)。因此,在所有樣品可以穩定使用的內參基因為和(表2)。

Fig. 3. Comparison of the rankings in candidate reference gene expression stability offed on different diets, reared on different rice varietiesunder dsRNA treatments or in all the tested samples of.
3 討論
qRT-PCR是研究基因表達水平的重要技術,采用相對定量的方法對基因的表達量進行檢測,即以內參基因作為標準,校準目的基因的表達量,具有靈敏度高、準確性好的特點,目前已成為研究基因表達中檢測mRNA水平的標準技術[1,28]。為了保證基因表達分析的可靠性,內參基因應該在不同試驗條件下穩定表達[6]。因此,篩選合適的內參基因是研究基因表達的關鍵。之前關于二化螟內參基因篩選的研究,對不同發育時期、不同組織和不同溫度處理的樣品中內參基因的穩定性進行了評估[20-21]。而本研究通過RefFinder、BestKeeper、GeNorm、NormFinder和ΔC值這5種分析方法,在不同發育時期、不同組織、不同溫度、殺蟲劑處理、取食不同飼料、取食不同水稻、dsRNA處理和所有的供試樣品中評估合適的內參基因用于mRNA水平的表達分析。

圖4 二化螟最優內參基因綜合分析
Fig. 4. Determination of the optimal number of reference genes in
候選內參基因的穩定性分析,首先使用BestKeeper、GeNorm、NormFinder和t值依據相關參數分析各基因的穩定性,并對穩定性高低進行排序,然后進一步使用在線程序RefFinder給出候選內參基因的綜合排名。研究結果表明在不同溫度、取食不同飼料、取食不同品系水稻和dsRNA處理的二化螟樣品中是最穩定的內參基因。在很多基因表達研究被作為內參基因[29-31],它屬于真核生物結構基因家族,參與形成微管和骨架的基礎組分,調控細胞分裂、形狀、運動和胞內活動等[32]。在一些昆蟲內參基因評價研究中,被認為是最穩定表達的內參基因,如在褐飛虱的不同地理種群[33]、白背飛虱不同發育時期和溫度處理[34]、松墨天牛不同發育時期和不同組織[31]、蝙蝠蛾的微生物感染[17]和煙粉虱溫度處理樣品中[35]等。通過催化氨酰基轉運RNA的依賴型GTP結合到核糖體的受體位點,在翻譯過程中起著重要作用[36]。在很多研究中,被認為是合適的內參基因,如斜紋夜蛾的溫度處理樣品[8]、東亞飛蝗的不同發育歷期[37]、西花薊馬的不同歷期和溫度處理[38]、大螟的不同組織和不同發育歷期[15]和瓢蟲的不同發育時期和溫度處理[39]等。在本研究中,在二化螟不同組織和混合供試樣品中,是最穩定的內參基因。在之前一些二化螟基因表達研究中[40-43],通常被用作內參基因,表現非常穩定。
本研究通過5種方法評估了內參基因的穩定性,ΔC值和NormFinder分析得到的結果在所有實驗條件中都非常相似。因此,為了確定合適的內參基因數量,GeNorm分析被用來計算配對變異值(V/V+1);當配對變異值小于0.15,意味著不需要增加額外的內參基因來提高準確性[6]。本研究中,配對變異值在不同發育時期、不同組織和混合樣品中,都高于0.15,意味著通過配對變異值不能確定內參基因的最佳數目,根據GeNorm使用手冊,當配對變異值大于0.15時,可以根據配對變異值趨勢選擇2~3個最穩定的內參基因。因此,在這3種條件下需要選擇3個表達穩定的內參基因最為合理,以獲得更加準確可靠的結果。
本研究中,我們利用5種分析方法評估了、、、、、、、、和這11種候選內參基因在8種不同試驗條件下的穩定性。在溫度處理、取食不同飼料、取食不同品系水稻和dsRNA處理的二化螟樣品中是最穩定的,在不同組織和混合樣品中是第二穩定的。在不同組織和混合樣品中是最穩定的,在取食不同品系水稻中是第二穩定的,在不同發育時期、溫度處理和取食不同飼料中被認為是第三穩定的。另外,和在不同發育時期和殺蟲劑處理的樣品中分別是最穩定的內參基因。這些結果表明沒有一個內參基因適用于任何試驗條件,因此我們認為更多的內參基因需要在特定的試驗條件下進行評估。本研究結果為不同試驗條件下選擇合適的內參基因提供了參考,也有利于在二化螟基因表達研究中獲得更加可靠準確的數據。
[1] Bustin S A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays., 2000, 25: 169-193.
[2] Thellin O, Zorzi W, Lakaye B, De Borman B, Coumans B, Hennen G, Grisar T, Igout A, Heinen E. Housekeeping genes as internal standards: Use and limits., 1999, 75: 291-295.
[3] de Jonge H J, Fehrmann R S, de Bont E S, Hofstra R M, Gerbens F, Kamps W A, de Vries E G, van der Zee A G, te Meerman G J, ter Elst A. Evidence based selection of housekeeping genes., 2007, 2: e898.
[4] Gutierrez L, Mauriat M, Pelloux J, Bellini C, Van Wuytswinkel O. Towards a systematic validation of references in real-time RT-PCR., 2008, 20: 1734-1735.
[5] Huggett J, Dheda K, Bustin S, Zumla A. Real-time RT-PCR normalisation; strategies and considerations., 2005, 6: 279-284.
[6] Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes., 2002, 3: Research0034.
[7] 符偉, 謝文, 張卓, 吳青君, 王少麗, 張友軍. Bt毒素誘導下小菜蛾實時定量PCR 內參基因的篩選. 昆蟲學報, 2012, 55(12): 1406-1412.
Fu W, Xie W, Zhang Z, Wu Q J, Wang S L, Zhang Y J. Selection of valid reference genes for gene expression studies by quantitative real-time PCR in(Lepidoptera: Plutellidae) after exposure to Bt toxin., 2012, 55(12): 1406-1412. (in Chinese with English abstract)
[8] Lu Y H, Yuan M, Gao X W, Kang T H, Zhan S, Wan H, Li J H. Identification and validation of reference genes for gene expression analysis using quantitative PCR in(Lepidoptera: Noctuidae)., 2013, 8: e68059.
[9] Chandra GS, Asokan R, Manamohan M, Kumar N K, Sita T. Evaluation of reference genes for quantitative real-time PCR normalization in cotton bollworm,., 2014, 48: 927-938.
[10] Shakeel M, Zhu X, Kang T, Wan H, Li J. Selection and evaluation of reference genes for quantitative gene expression studies in cotton bollworm,(Lepidoptera: Noctuidae)., 2015, 18: 123-130.
[11] Zhang S D, An S H, Li Z, Wu F M, Yang Q P, Liu Y C, Cao J J, Zhang H J, Zhang Q W, Liu X X. Identification and validation of reference genes for normalization of gene expression analysis using qRT-PCR in(Lepidoptera: Noctuidae)., 2015, 555: 393-402.
[12] Zhu X, Yuan M, Shakeel M, Zhang Y J, Wang S L, Wang X, Zhan S, Kang T H, Li J H. Selection and evaluation of reference genes for expression analysis using qRT-PCR in the beet armyworm(Hubner) (Lepidoptera: Noctuidae)., 2014, 9: e84730.
[13] Arun A, Baumle V, Amelot G, Nieberding C M. Selection and validation of reference genes for qRT-PCR expression analysis of candidate genes involved in olfactory communication in the butterfly., 2015, 10: e0120401.
[14] Lu Y H, Zheng X S, Liang Q, Xu H X, Yang Y J, Tian J C, He X C, Lu Z X. Evaluation and validation of reference genes for SYBR Green qRT-PCR normalization in(Lepidoptera: Noctuidae)., 2015, 18: 669-675.
[15] Sun M, Lu M X, Tang X T, Du Y Z. Exploring valid reference genes for quantitative real-time PCR analysis in(Lepidoptera: Noctuidae)., 2015, 10: e0115979.
[16] Pan H P, Yang X W, Bidne K, Hellmich R L, Siegfried B D, Zhou X G. Selection of reference genes for RT-qPCR analysis in the monarch butterfly,(L.), a migrating bio-Indicator., 2015, 10: e0129482.
[17] Liu G Q, Qiu X H, Cao L, Zhang Y, Zhan Z B, Han R C. Evaluation of reference genes for reverse transcription quantitative PCR studies of physiological responses in the ghost moth,(Lepidoptera, Hepialidae)., 2016, 11: e0159060.
[18] Zhang L, Zhang Q L, Wang X T, Yang X Z, Li X P, Yuan M L. Selection of reference genes for qRT-PCR and expression analysis of high-altitude-related genes in grassland caterpillars (Lepidoptera: Erebidae: Gynaephora) along an altitude gradient., 2017, 7: 9054-9065.
[19] 楊苓, 胡曉靜, 徐志峰, 何林, 肖偉. 桃蛀螟實時熒光定量PCR內參基因的篩選. 昆蟲學報, 2017, 60(11): 1266-1277.
Yang L, Hu X J, Xu Z F, He L, Xiao W. Screening of reference genes for qRT-PCR in(Lepidoptera: Crambidae)., 2017, 60(11): 1266-1277. (in Chinese with English abstract)
[20] Teng X L, Zhang Z, He G L, Yang L W, Li F. Validation of reference genes for quantitative expression analysis by real-time RT-PCR in four lepidopteran insects., 2011, 12: 60.
[21] Xu J, Lu M X, Cui Y D, Du Y Z. Selection and evaluation of reference genes for expression analysis using qRT-PCR in(Lepidoptera: Pyralidae)., 2017, 110: 683-691.
[22] 胡陽, 鄭永利, 曹國連, 傅強. 利用半人工飼料大規模簡便化飼養二化螟中國水稻科學, 2013,27(5): 535-538. Hu Y, Zheng Y L, Cao G L, Fu Q. A technique for rearingin the large scale with an oligidic diet in laboratory., 2013, 27(5): 535-538. (in Chinese with English abstract)
[23] 吳敏, 張真真, 高聰芬. 水稻二化螟抗藥性監測方法. 應用昆蟲學報, 2013, 50(2): 548-552.
Wu M, Zhang Z Z, Gao C F. Methods of insecticides resistance monitoring of the striped stem borer, Chilo suppressalis., 2013, 50(2):548-552. (in Chinese with English abstract)
[24] Hui X M, Yang W, He G L, Yang Q P, Han Z, Li F. RNA interference ofandinreveals their different contributions to motor ability and larval growth., 2011, 4: 507-518.
[25] Pfaffl M W, Tichopad A, Prgomet C, Neuvians T P. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity. BestKeeper: Excel-based tool using pair-wise correlations., 2004, 26: 509-515.
[26] Andersen C L, Jensen J L, Orntoft T F. Normalization of real-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets., 2004, 64: 5245-5250.
[27] Schmittgen T D, Livak K J. Analyzing real-time PCR data by the comparative CT method., 2008, 3: 1101-1108.
[28] Radonic A, Thulke S, Mackay I M, Landt O, Siegert W, Nitsche A. Guideline to reference gene selection for quantitative real-time PCR., 2004, 313: 856-862.
[29] Huis R, Hawkins S, Neutelings G. Selection of reference genes for quantitative gene expression normalization in flax (L.)., 2010, 10: 71.
[30] Kozera B, Rapacz M. Reference genes in real-time PCR., 2013, 54: 391-406.
[31] 馮波, 郭前爽, 毛必鵬, 杜永均. 松墨天牛化學感受組織熒光定量PCR內參基因的鑒定與篩選. 昆蟲學報, 2016, 59(4): 427-437.
Feng B, Guo Q, Mao B P, Du Y J. Identification and selection of valid reference genes for assaying gene expression in the chemosensory tissues of(Coleoptera: Cerambycidae) by RT-PCR., 2016, 59(4): 427-437. (in Chinese with English abstract)
[32] Nielsen M G, Gadagkar S R, Gutzwiller L. Tubulin evolution in insects: Gene duplication and subfunction- alization provide specialized isoforms in a functionally constrained gene family., 2010, 10: 113.
[33] Yuan M, Lu Y H, Zhu X, Wan H, Shakeel M, Zhan S, Jin B R, Li J H. Selection and evaluation of potential reference genes for gene expression analysis in the brown planthopper,(Hemiptera: Delphacidae) using reverse-transcription quantitative PCR., 2014, 9: e86503.
[34] An X K, Hou M L, Liu Y D. Reference gene selection and evaluation for gene expression studies using qRT-PCR in the white-backed planthopper, Sogatella furcifera (Hemiptera: Delphacidae)., 2016, 109: 879-886.
[35] Dai T M, Lu Z C, Liu W X, Wan F H. Selection and validation of reference genes for qRT-PCR analysis during biological invasions: The thermal adaptability ofMED., 2017, 12: e0173821.
[36] Ponton F, Chapuis M P, Pernice M, Sword GA, Simpson S J. Evaluation of potential reference genes for reverse transcription-qPCR studies of physiological responses in Drosophila melanogaster., 2011, 57: 840-850.
[37] Yang Q P, Li Z, Cao J J, Zhang S D, Zhang H J, Wu X Y, Zhang Q W, Liu X X. Selection and assessment of reference genes for quantitative PCR normalization in migratory locust Locusta migratoria (Orthoptera: Acrididae)., 2014, 9: e98164.
[38] Zheng Y T, Li H B, Lu M X, Du Y Z. Evaluation and validation of reference genes for qRT-PCR normalization in(Thysanoptera: Thripidae)., 2014, 9: e111369.
[39] Pan H P, Yang X W, Siegfried B D, Zhou X G. A comprehensive selection of reference genes for RT-qPCR analysis in a predatory lady beetle,(Coleoptera: Coccinellidae)., 2015, 10: e0125868.
[40] Qiu L, Wang P, Wu T, Li B, Wang X, Lei C, Lin Y, Zhao J, Ma W. Downregulation ofalkaline phosphatase genes associated with resistance to three transgenicrice lines., 2018, 27: 83-89.
[41] Xu G, Gu G X, Teng Z W, Wu S F, Huang J, Song Q S, Ye G Y, Fang Q. Identification and expression profiles of neuropeptides and their G protein-coupled receptors in the rice stem borer., 2016, 6: 28976.
[42] Xu G, Wu S F, Teng Z W, Yao H W, Fang Q, Huang J, Ye G Y. Molecular characterization and expression profiles of nicotinic acetylcholine receptors in the rice striped stem borer,(Lepidoptera: Crambidae)., 2017, 24: 371-384.
[43] Xu G, Wu S F, Wu Y S, Gu G X, Fang Q, Ye G Y. De novo assembly and characterization of central nervous system transcriptome reveals neurotransmitter signaling systems in the rice striped stem borer,., 2015, 16: 525.
Screening Reference Genes and Evaluating of Their Expression Stability for qRT-PCR Normalization in(Lepidoptera: Pyralididae)
XU Hongxing1, WANG Guorong2, LU Yanhui1,*, YANG Yajun1, ZHENG Xusong1, TIAN Junce1, Lü Zhongxian1,*
(Key Laboratory of Information Traceability for Agricultural Products, Ministry of Agriculture, P. R. of China/Key Laboratory Breeding Base for Zhejiang Sustainable Pest and Disease Control, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China;Xiaoshan Agricultural Technology Extension Center,,;Corresponding author,
【Objective】We aim to screen reference genes stably expressed in striped stem borers (SSB),,under certain conditions, to lay a foundation for the study on the gene expression in SSB【Method】According to the results of SSB transcriptome data, 11 candidate genes from SSB were selected and their expression stability was detected under different treatmentconditions (including development duration, tissues, feeding on different diets and rice varieties, dsRNA and mixed samples under temperature stress, insecticide treatment), by using real-time fluorescence quantitative PCR. The stability of the 11 candidate genes was evaluated by RefFinder, BestKeeper, GeNorm, NormFinder andΔC. 【Result】,andwere highly stable in different development durations;,andwere highly stable in different tissues.,andwere relatively stable under temperature stress.andwere stable in insecticide treated samples.,,andwere stable in samples fed on different diets.andwere stable reference genes in samples fed on different rice varieties.,,andwere stable in the sample treated with dsRNA.,andwere stable in mixed samples. 【Conclusion】It provide a reference for selection of suitable reference gene under different test conditions, and also provide more reliable and accurate data for research in gene expression in riceSSB.
; reference gene; qRT-PCR; gene expression
10.16819/j.1001-7216.2019.8035
Q755; S435.112+.1
A
1001-7216(2019)01-0075-10
2018-03-27;
2018-05-15。
國家自然科學基金資助項目(31672050);浙江省植物有害生物防控省部共建國家重點實驗室培育基地自主設計項目(2010DS700124- ZZ1601)。

