3種碳源條件下香蕉枯萎病菌次生代謝物合成基因的表達譜分析
趙艷娟 趙亞娟 劉駿麒 金天 程卯 黃立鈺 王振中 秦世雯
摘? 要? 在前期獲得的香蕉枯萎病菌(Fusarium oxysporum f. sp. cubense, Foc)轉錄組數據基礎上,本研究利用antiSMASH、PHI-base數據庫對3種碳源條件下香蕉枯萎病1號小種(Foc 1)和4號小種(Foc 4)表達的次生代謝物合成基因進行了預測和表達譜分析。結果表明:3種碳源條件下,Foc共有486個候選的次生代謝物合成基因表達,并分類到32個次生代謝產物合成基因簇,其中13個基因可能與Foc的致病性相關。此外,Foc在3種碳源條件下次生代謝物合成基因的表達模式存在明顯差異,特別是在寄主細胞壁條件下Foc差異表達的次生代謝物合成基因最多;而且Foc 4相較于Foc 1在侵染寄主細胞壁時有更多的次生代謝物合成基因特異表達或高表達。以上研究結果將為進一步明確次生代謝產物在Foc致病過程中的作用機理提供理論依據。
關鍵詞? 香蕉枯萎病;尖孢鐮刀菌古巴專化型;次生代謝物;基因表達
中圖分類號? S432.1; S668.1? ? ? ?文獻標識碼? A
Expression Analysis of Genes Involved in Biosynthesis of Secondary Metabolites for Fusarium oxysporum f. sp. cubense in Response to Three Carbon Sources
ZHAO Yanjuan1, ZHAO Yajuan2, LIU Junqi1, JIN Tian1, CHENG Mao3, HUANG Liyu1, WANG Zhenzhong2*, QIN Shiwen1*
1. School of Agriculture, Yunnan University / Research Center of Perennial Rice Engineering and Technology in Yunnan, Kunming, Yunnan 650500, China; 2. College of Agriculture, South China Agricultural University / Guangdong Provincial Key Laboratory of Microbial Signals and Disease Control, Guangzhou, Guangdong 510642, China; 3. Jinghong Center of Agricultural Technology Extension, Jinghong, Yunnan 666101, China
Abstract? To understand the pathogenesis mechanism of secondary metabolites for Fusarium oxysporum f. sp. cubense (Foc) during infection in banana, genes involved in the biosynthesis of secondary metabolites were predicted based on the transcriptome of Foc 1 and Foc 4 in response to different carbon sources, using antiSMASH and PHI-base database. 486 genes involved in the biosynthesis of secondary metabolites in the transcriptome were identified and sorted to 32 gene clusters, including 13 virulence-associated genes. Additionally, the gene expression pro?les involved in the biosynthesis of secondary metabolites of Foc 1 and Foc 4 in response to different carbon sources appeared remarkably different. A great majority of genes were differentially expressed in Foc when induced by host cell wall. Besides, more specific differentially expressed genes with high transcription level were identified in Foc 4 as comparison with Foc 1 during the degradation of banana cell wall. The results would provide a theoretical basis for understanding and studying the molecular pathogenesis mechanism of Foc.
Keywords? Fusarium wilt; Fusarium oxysporum f. sp. cubense; secondary metabolites; gene expression
DOI? 10.3969/j.issn.1000-2561.2019.12.018
香蕉枯萎病是由尖孢鐮刀菌古巴專化型(Fusarium oxysporum f. sp. cubense, Foc)引起的香蕉維管束病害,給全世界香蕉產業帶來了嚴重的危害[1]。該病菌1號小種(Foc 1)曾導致香蕉主栽品種‘大蜜哈的毀滅,直到抗1號小種的Cavendish香蕉品種應用,世界香蕉生產才得以峰回路轉。但是,近半個世紀來,侵染Cavendish香蕉的4號小種(Foc 4)出現迅速蔓延,使香蕉產業重新面臨嚴重的困境[2]。因此,研究Foc的致病機理及其2個小種的致病性分化機制,將為香蕉枯萎病防治技術研發提供新的途徑。
毒素是香蕉枯萎病菌的主要致病因子之一[3-5]。Foc分泌細胞壁降解酶突破寄主細胞壁,隨后在寄主根部維管束組織內大量增殖,并沿著維管束向根莖和假莖部分蔓延,堵塞木質部導管,阻礙水分和營養的運輸,同時產生真菌毒素(如鐮刀菌酸和脫氫鐮刀菌酸等)造成維管束壞死和葉片黃化,最終導致植株枯萎死亡[6]。
大部分真菌毒素屬于次生代謝產物(secondary metabolites, SMs),如白僵菌素(beauvericin, BEA)和鐮刀菌酸(fusaric acid, FA)[7-8]。真菌毒素能夠破壞寄主細胞膜,影響寄主正常的代謝過程,導致寄主生理失調和細胞死亡,在死體營養型真菌的致病過程中起著決定性作用[9]。毒素的生物合成是由一系列基因簇(secondary metabolite gene clusters, SMC)控制,包括非核糖體多肽合成酶(nonribosomal peptide synthetase, NRPS)和聚酮合成酶(polyketide synthase, PKS)基因,其他酶類(如氫化酶、氧化酶和轉運蛋白等)基因以及相關調控基因[10]。例如,白僵菌素主要是由BEA合成酶(NRPS)催化合成,并且鐮刀菌屬真菌BEA生物合成還需要一系列不同功能的SMC參與催化代謝[11];鐮刀菌酸的生物合成主要由FA基因簇中的聚酮合成酶Fub 1控制,敲除Fub1能夠使F. oxysporum f. sp. lycopersici不產生FA,并且顯著降低其對番茄的致病力[12]。
本研究前期通過RNA-seq測序分析,成功獲得了3種碳源(寄主細胞壁、果膠和葡萄糖)條件下Foc 1和Foc 4的轉錄組數據,并發現在寄主細胞壁多糖條件下能有效模擬Foc侵染寄主的基因表達情況[13]。本研究將利用antiSMASH數據庫對該轉錄組數據進行次生代謝物合成(secondary metabolism biosynthesis, SMB)基因的預測和表達分析,挖掘Foc致病相關的SMB基因,分析SMB基因在2個小種致病過程中的表達差異,為Foc致病基因的篩選和致病性分化的分子機理研究提供參考。
1? 材料與方法
1.1? 材料
本研究所用3種碳源條件下Foc 1和Foc 4轉錄組數據由華南農業大學植物病理生理研究室提供。該轉錄組測序所用菌株為香蕉枯萎病菌1號生理小種菌株C2和4號生理小種菌株DZ1。3種碳源分別為寄主細胞壁多糖的主要成分:寄主細胞壁(粉蕉細胞壁用于Foc 1菌株的培養,以FCW表示;巴西蕉細胞壁用于Foc 4菌株的培養,以BCW表示)、果膠(以P表示)和葡萄糖(以G表示)。該轉錄組raw reads序列已提交至NCBI,登錄號為SRA486974。
1.2? 方法
1.2.1? 次生代謝物合成基因的預測? 本研究利用antiSMASH數據庫對轉錄組23 147個unigene序列進行SMB基因預測,比對參數為默認值。
1.2.2? 次生代謝物合成基因的致病相關性分析 本研究利用病原物與寄主互作數據庫PHI-base(http://www.phi-base.org/)對發掘的SMB基因的致病功能進行預測,比對閾值(e-value)為1e-5。
1.2.3? 不同寄主細胞壁多糖條件下次生代謝物合成基因的表達分析? 對轉錄組數據進行參考序列比對,基因表達水平進行FPKM(fragments per kilobase of exon model per million reads mapped)換算,隨后將基因差異表達數據進行標準化處理,最后進行基因差異表達水平DEGseq分析,差異表達基因(differentially expressed gene, DEG)的篩選閾值為q-value<0.005且|log2 (Fold Change)| >1。
差異基因維恩圖的制作使用Draw Venn Diagrum軟件(http://bioinformatics.psb.ugent.be/ web t ools/Venn/);差異基因表達水平熱圖制作使用OmicShare Tools Heat map軟件(https://www.o micshare.com/ tools)。
2? 結果與分析
2.1? 次生代謝物合成基因的預測
本研究以不同的寄主細胞壁多糖條件下Foc 1和Foc 4的轉錄組數據進行SMB基因分析,通過antiSMASH數據庫的注釋,共預測到486個unigene。其中,83個unigene被預測為SMC的主要合成基因,并分類到32個SMC中(表1)。32個SMC包括3個T1pks基因簇,1個T3pks基因簇,9個Nrps基因簇,1個Nrps-T1pks基因簇,10個Terpene基因簇,3個Indole基因簇和5個其他基因簇。根據上述SMB基因分析推測,3種碳源條件下Foc主要次生代謝物類型為I型聚酮、III型聚酮、T1pks-nrps聚酮、萜類和吲哚類。
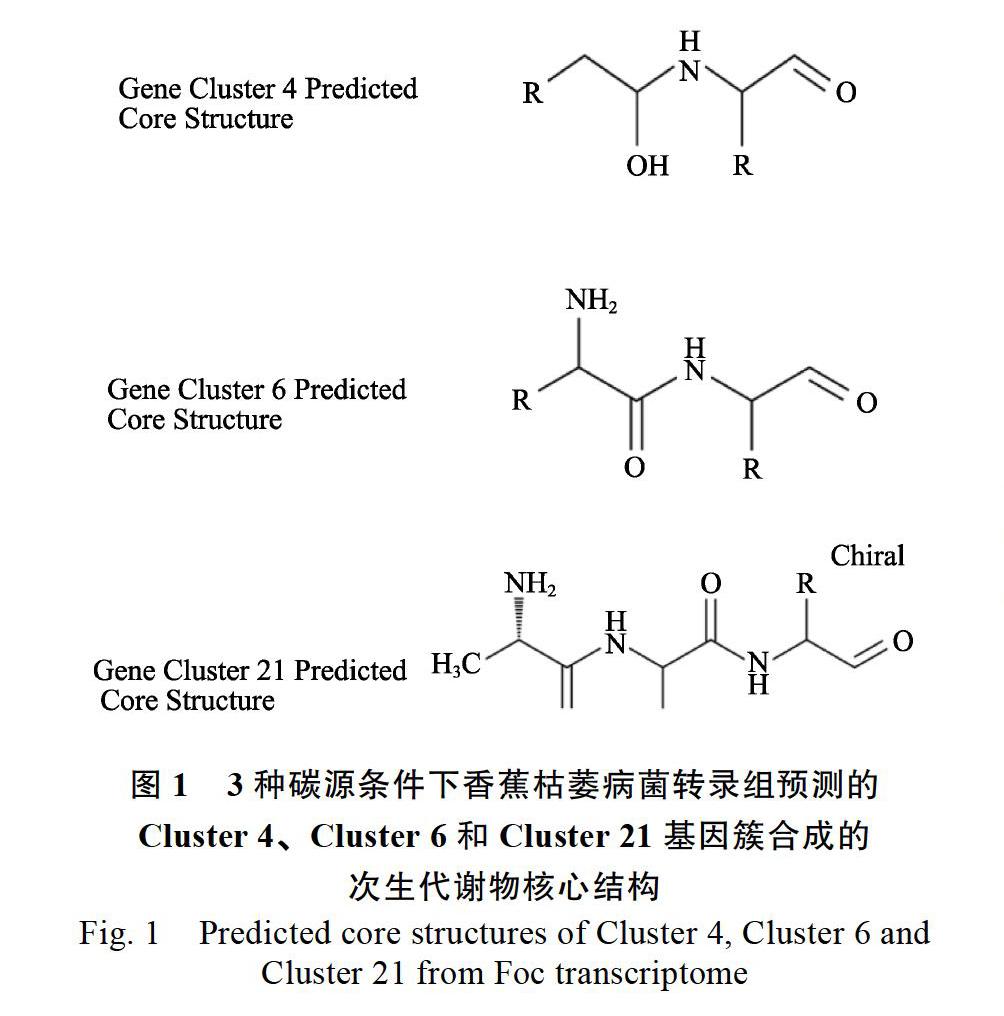
通過antiSMASH數據庫的預測,3個SMC(Cluster 4、Cluster 6和Cluster 21)預測到合成的次生代謝物核心結構,同時還發現Cluster 6和Cluster 21雖同為Nrps基因簇,但合成的次生代謝物組成單位存在明顯差異(圖1)。
2.2? 次生代謝物合成基因的致病相關性分析
通過PHI-base對486個SMB基因的致病功能進行分析預測(e-value≤105),結果發現有37個基因可能與Foc和寄主的互作相關(即PHI基因),其中13個SMB基因缺失后可能導致Foc致病性喪失或致病力減弱。結果還顯示,83個SMC主要合成基因在PHI-base中并無致病相關功能注釋。
2.3? 3種碳源條件下次生代謝物合成基因的差異表達分析
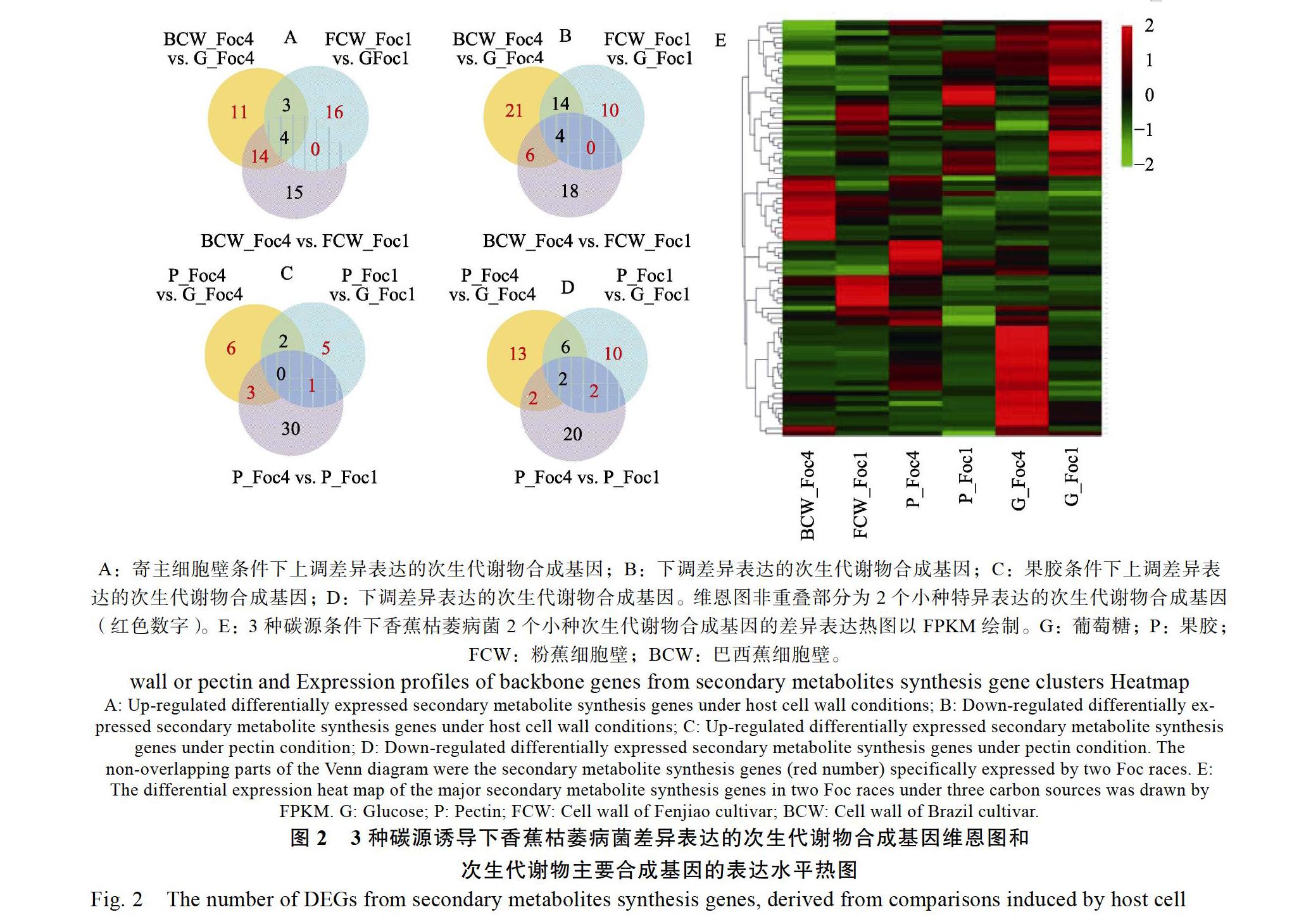
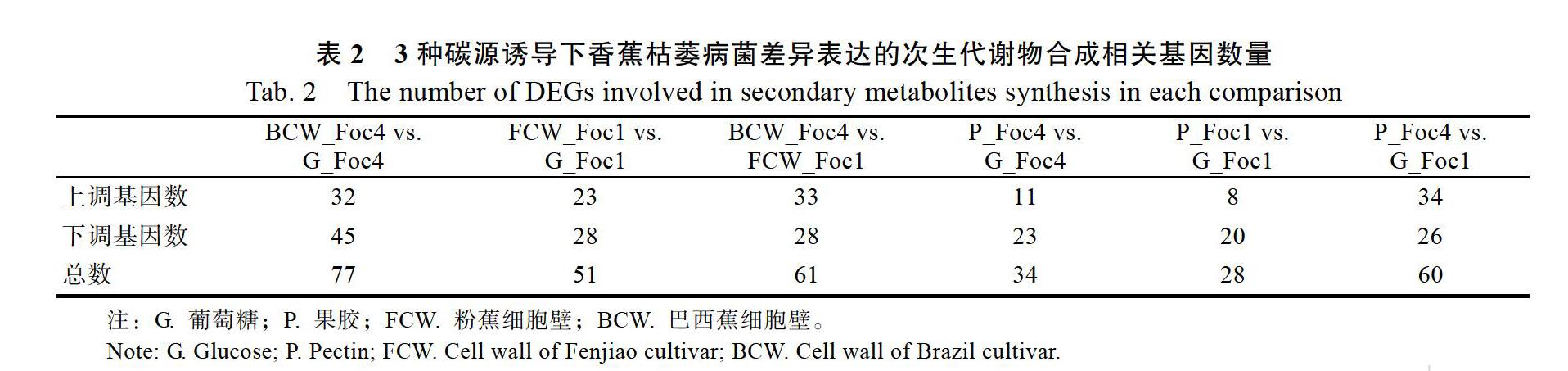
為了解次生代謝物在2個小種致病性分化中的作用,本研究對3種碳源條件下Foc 1和Foc 4的SMB基因表達情況進行分析,總共獲得6個比較組(FCW_Foc1 vs. G_Foc1、BCW_Foc4 vs. G_Foc4、BCW_Foc4 vs. FCW_Foc1、P_Foc4 vs. G_Foc4、P_Foc1 vs. G_Foc1和P_Foc4 vs. P_Foc1)的差異表達基因。結果表明,在3種碳源條件下Foc 1或Foc 4表達的SMB基因均存在明顯差異(圖2)。同時,相較于Foc 1,Foc 4在降解寄主細胞壁多糖時SMB基因的差異表達數量較多(表2)。而在同種碳源條件下,2個小種SMB基因的表達模式也存在明顯差異(圖2),具體表現在:Foc 4特異表達的SMB基因數量相較于Foc 1更多(圖2A、圖2B、圖2C和圖2D);在Foc 4中高表達的SMB基因在Foc 1中基本都低表達(圖2E)。
上述結果表明,這些在Foc 4中特異表達或高表達的SMB基因可能參與Foc 4對Cavendish香蕉的侵染過程。
A:寄主細胞壁條件下上調差異表達的次生代謝物合成基因;B:下調差異表達的次生代謝物合成基因;C:果膠條件下上調差異表達的次生代謝物合成基因;D:下調差異表達的次生代謝物合成基因。維恩圖非重疊部分為2個小種特異表達的次生代謝物合成基因(紅色數字)。E:3種碳源條件下香蕉枯萎病菌2個小種次生代謝物合成基因的差異表達熱圖以FPKM繪制。G:葡萄糖;P:果膠;FCW:粉蕉細胞壁;BCW:巴西蕉細胞壁。
wall or pectin and Expression profiles of backbone genes from secondary metabolites synthesis gene clusters Heatmap
A: Up-regulated differentially expressed secondary metabolite synthesis genes under host cell wall conditions; B: Down-regulated differentially expressed secondary metabolite synthesis genes under host cell wall conditions; C: Up-regulated differentially expressed secondary metabolite synthesis genes under pectin condition; D: Down-regulated differentially expressed secondary metabolite synthesis genes under pectin condition. The non-overlapping parts of the Venn diagram were the secondary metabolite synthesis genes (red number) specifically expressed by two Foc races. E: The differential expression heat map of the major secondary metabolite synthesis genes in two Foc races under three carbon sources was drawn by FPKM. G: Glucose; P: Pectin; FCW: Cell wall of Fenjiao cultivar; BCW: Cell wall of Brazil cultivar.
3? 討論
antiSMASH數據庫是預測微生物次生代謝物合成基因簇的綜合性數據庫,可實現基因組與基因組之間的相關天然產物合成基因簇的查詢[14]。PHI-base數據庫整合了大量病原真菌和卵菌在侵染寄主時具有互作功能的基因研究數據[15]。目前,這2種數據庫已廣泛利用于植物病原菌基因組和轉錄組的致病基因預測。通過antiSMASH和PHI-base數據庫對引起多種植物土傳病害的菜豆殼球孢(Macrophmina phaseolina)基因組分析發現,該病原菌相對于F. oxysporum和F. graminearum擁有更多的致病酶類和毒素摧毀寄主防御,其中包括75個主要SMB基因和537個致病基因[16]。在小麥赤霉病菌(F. graminearum)與非致病型鐮刀菌(F. venenatum)的比較基因組研究中,利用antiSMASH、PHI-base等數據庫揭示了F. graminearum中5個特有的致病相關SMC參與了該菌侵染小麥的病程,另外2個PHI轉錄因子敲除后使其對小麥的致病力顯著降低[17-18]。
本研究利用antiSMASH和PHI-base數據庫對3種碳源條件下Foc 1和Foc 4表達的次生代謝物合成基因進行了預測分析,發現了32個SMC,總共獲得486個候選的SMB基因(包含83個主要合成基因),但大部分SMB基因的功能未知。該結果與Foc 1和Foc 4基因組的SMC注釋結果基本相符[5]。Foc基因組中存在4個合成二甲基丙烯基色氨酸(DAMT)的基因簇,但在本研究中未發現合成DAMT的基因簇,可能是在突破寄主細胞壁防御時,DAMT合成途徑并不參與Foc與寄主互作過程。
本研究發現3種碳源條件下Foc 1和Foc 4表達的13個SMB基因可能與其致病性相關,但83個主要合成基因致病功能未獲得預測信息。這可能是由于SMB基因的功能在植物病原真菌中研究還較少,利用同源比對分析,從PHI-base收錄的致病基因中不能找到Foc主要SMB基因的同源基因。因此,進一步開展SMB基因功能驗證,將有助于深入了解次生代謝物在Foc致病過程中的作用。
本研究發現在3種碳源條件下,Foc的SMB基因具有不同的表達模式,說明不同營養條件能夠影響Foc次生代謝物的合成。已有研究證明,Foc在PDA培養條件(in vitro)和侵染巴西蕉(in vivo)時SMB基因的表達存在明顯差異[5];而在水稻惡苗病菌(F. fujikuroi)中也發現了相似結果,該病菌次生代謝物的合成受不同氮源和植物信號的影響[19]。本研究還發現,相較于Foc 1,Foc 4在侵染寄主細胞壁時有更多的次生代謝物合成基因特異表達或表達量升高,說明Foc 4能夠產生更多的次生代謝物來適應不同的寄主環境,并成功侵染大部分香蕉主栽品種,這可能與2個小種寄主專化性和致病性分化密切相關。本研究結果將為進一步明確次生代謝產物在Foc致病性分化的作用提供參考依據。
參考文獻
Ploetz R C, Churchill A C L. Fusarium wilt: the banana disease that refuses to go away[J]. Acta Horticulturae, 2011, 897: 519-526.
Ploetz R C. Management of Fusarium wilt of banana: A review with special reference to tropical race 4[J]. Crop Protection, 2015, 73: 7-15.
[3]王振中. 香蕉枯萎病及其防治研究進展[J]. 植物檢疫, 2006, 20(3): 198-200..
Bacon C W, Porter J K, Norred W P, et al. Production of fusaric acid by Fusarium species[J]. Applied & Environmental Microbiology, 1996, 62(11): 4039-4043.
Guo L, Han L, Yang L, et al. Genome and transcriptome analysis of the fungal pathogen Fusarium oxysporum f. sp. cubense causing banana vascular wilt disease[J]. PLoS One, 2014, 9(4): e95543.
Ploetz R C. Fusarium wilt of banana[J]. Phytopathology, 2015, 105(12): 1512-1521.
Friesen T L, Faris J D, Solomon P S, et al. Host-specific toxins: effectors of necrotrophic pathogenicity[J]. Cellular Microbiology, 2008, 10(7): 1421-1428.
Friesen T L, Stukenbrock E H, Liu Z, et al. Emergence of a new disease as a result of interspecific virulence gene transfer[J]. Nature Genetics, 2006, 38(8): 953-956.
Desjardins A E, Hohn T M. Mycotoxins in plant pathogenesis[J]. Molecular Plant-Microbe Interactions, 1997, 10(2): 147-152.
Keller N P, Hohn T M. Metabolic pathway gene clusters in filamentous fungi[J]. Fungal Genetics & Biology, 1997, 21(1): 17-29.
Zhang T, Zhuo Y, Jia X P, et al. Cloning and characterization of the gene cluster required for beauvericin biosynthesis in Fusarium proliferatum[J]. Science China Life Sciences, 2013, 56(7): 628-637.
López-Díaz C, Rahjoo V, Sulyok M, et al. Fusaric acid contributes to virulence of Fusarium oxysporum on plant and mammalian hosts: Fusaric acid promotes virulence of F. oxysporum[J]. Molecular Plant Pathology, 2018, 19(2): 440-453.
Qin S, Ji C, Li Y, et al. Comparative Transcriptomic Analysis of Race 1 and Race 4 of Fusarium oxysporum f. sp. cubense Induced with Different Carbon Sources[J]. G3-Genes Genomes Genetics, 2017, 7(7): 2125-2138.
Weber T, Kai B, Duddela S, et al. antiSMASH 3.0—a comprehensive resource for the genome mining of biosynthetic gene clusters[J]. Nucleic Acids Research, 2015, 43(W1): W237-W243.
Urban M, Cuzick A, Rutherford K, et al. PHI-base: a new interface and further additions for the multi-species pathogen–host interactions database[J]. Nucleic Acids Research, 2017, 45(Database issue): D604-D610.
Islam M S, Haque M S, Islam M M, et al. Tools to kill: Genome of one of the most destructive plant pathogenic fungi Macrophomina phaseolina[J]. BMC Genomics, 2012, 13(1): 493.
King R, Brown N A, Urban M, et al. Inter-genome comparison of the Quorn fungus Fusarium venenatum and the closely related plant infecting pathogen Fusarium graminearum[J]. BMC Genomics, 2018, 19(1): 269.
[18]Son H, Seo YS, Min K, et al. A Phenome-based functional analysis of transcription factors in the cereal head blight fungus, Fusarium graminearum[J]. PLoS Pathogens, 2011, 7(10): e1002310.
Wiemann P, Sieber C M K, Bargen K W V, et al. Deciphering the cryptic genome: genome-wide analyses of the rice pathogen Fusarium fujikuroi reveal complex regulation of secondary metabolism and novel metabolites[J]. PLoS Pathogens, 2013, 9(6): e1003475.

