改進(jìn)的無(wú)需比對(duì)的DNA序列分類算法
張文影,昌 攀,鐘 誠(chéng)
(廣西大學(xué) 計(jì)算機(jī)與電子信息學(xué)院,廣西 南寧 530004)
0 引 言
目前主流的DNA序列分類方法[1,2]有基于序列比對(duì)的分類方法[3-5]和無(wú)需比對(duì)的分類方法[6-8]。盡管基于比對(duì)的分類方法可以獲得較高的分類精度,但仍存在對(duì)功能相似結(jié)構(gòu)差異較大的DNA序列分類時(shí)精度較低、比對(duì)大量序列增加了計(jì)算復(fù)雜度[9]等弊端。
為了克服基于比對(duì)的序列分類方法的局限性,學(xué)者們提出了無(wú)需比對(duì)的DNA序列分類方法。常用的無(wú)需比對(duì)的分類使用了K元組[10]方法,文獻(xiàn)[11]通過(guò)實(shí)驗(yàn)表明K元組較適用于核小體DNA序列的分類,在對(duì)核小體的分類中取得了較高的精度和敏感度。然而,基于K元組方法的計(jì)算結(jié)果很大程度上取決于參數(shù)K的設(shè)定,此外,當(dāng)K值較大時(shí),這些方法也面臨著特征向量維數(shù)較高和計(jì)算復(fù)雜等問(wèn)題。另一些DNA序列分類研究則使用有效的壓縮算法對(duì)序列進(jìn)行壓縮,進(jìn)而分類DNA序列。文獻(xiàn)[12]介紹了3種DNA壓縮算法,并通過(guò)實(shí)驗(yàn)闡明DNA壓縮算法在序列分類領(lǐng)域的應(yīng)用。文獻(xiàn)[13]利用Voss映射將DNA序列映射成二進(jìn)制指示序列,然后利用離散傅里葉變換(discrete fourier transformation,DFT)處理該指示序列,進(jìn)而分析DNA序列之間的進(jìn)化關(guān)系。這種方法只考慮了單個(gè)堿基,而忽略了DNA序列堿基化學(xué)性質(zhì)、出現(xiàn)頻率和位置等重要生物學(xué)信息。本文結(jié)合K元組方法和離散傅里葉變換DFT提取序列堿基化學(xué)性質(zhì)、出現(xiàn)頻率和位置的特征值,將任意長(zhǎng)度的DNA序列映射成等長(zhǎng)的特征向量,從而更準(zhǔn)確地分類DNA序列。
1 分類算法
1.1 DFT與功率譜
DFT變換可以將數(shù)值序列從時(shí)域序列轉(zhuǎn)化為頻域序列,其中序列的信息也就相應(yīng)地轉(zhuǎn)化為不同頻率下的幅度和相位,這樣就可以挖掘出序列中隱藏的重要信息。DFT已被應(yīng)用于DNA序列分析研究中[14]。
利用DFT變換,長(zhǎng)度為N的時(shí)間序列f(n)在特定的k頻率上的映射可表示為F(k)
(1)
F(k)在頻率k上的功率譜PS(k)[15]
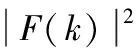
(2)
當(dāng)k=0時(shí),頻率為0時(shí)的特定功率譜PS(0)為
(3)
1.2 提取DNA序列的特征向量
DNA序列是由4種堿基:腺嘌呤A、鳥(niǎo)嘌呤G、胞嘧啶C和胸腺嘧啶T組成的一維線性序列。任一條DNA序列可以被唯一描述為3種獨(dú)立的嘌呤(R)和嘧啶(Y)的分布、氨基(M)和羰基(K)的分布、強(qiáng)氫鍵(S)和弱氫鍵(W)的分布[16]。依據(jù)4種堿基的不同化學(xué)性質(zhì)可以將堿基分為以下3類:
(1)嘌呤R=A、G,嘧啶Y=C、T;
(2)氨基M=A、C,羰基K=G、T;
(3)強(qiáng)氫鍵S=G、C,弱氫鍵W=A、T。
依據(jù)這3類堿基分類,可將任一條DNA序列映射成3條特定堿基組成的符號(hào)序列[17]。例如,DNA序列x=GTCCACTGGCATGGT可映射為3條符號(hào)序列φRY(x)=RYYYRYYRRYRYRRY,φMK(x)=KKMMMMKKKMMKKKK,φWS(x)=SWSSWSWSSSWWSSW。
參照文獻(xiàn)[17]的方法,將φβ(x)(β∈{RY,MK,WS})轉(zhuǎn)換成如下12條二進(jìn)制指示序列H(x)={hRR(x),hRY(x),hYY(x),hYR(x),hMM(x),hMK(x),hKK(x),hKM(x),hWW(x),hWS(x),hSS(x),hSW(x)}
(4)
其中,α∈{RR,RY,YY,YR,MM,MK,KK,KM,WW,WS,SS,SW},φβ(n)為序列φβ(x)的第n個(gè)符號(hào),N為DNA序列的長(zhǎng)度。
例如,對(duì)于序列φRY(x)、φMK(x)和φWS(x)可生成如下12條二進(jìn)制指示序列:hRR(x)=00000001000010,hRY(x)=10001000101001,hYY(x)=01100100000000,hYR(x)=00010010010100,hMM(x)=00111000010000,hMK(x)=00000100001000,hKK(x)=10000011000111,hKM(x)=01000000100000,hWW(x)=00000000001000,hWS(x)=01001010000100,hSS(x)=00100001100010,hSW(x)=10010100010001。
設(shè)Sα為hα(x)中子序列α的數(shù)量,則可計(jì)算子序列α在二進(jìn)制指示序列hα(x)中的出現(xiàn)頻率T(α)
T(α)=Sα/(N-1)
(5)
依據(jù)式(1),將二進(jìn)制指示序列hα(x)進(jìn)行DFT變換后得到序列Fα(k)
(6)
其中,L=N-1為二進(jìn)制指示序列hα(x)的長(zhǎng)度。根據(jù)式(2),F(xiàn)α(k)在特定頻率k上的DFT功率譜PSα(k)為
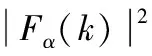
(7)
將帕賽瓦爾定理運(yùn)用在DFT變換中得到
(8)
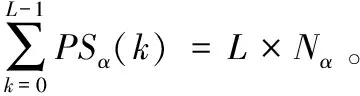
(9)

(10)
其中,E(k)是每個(gè)功率譜PSα(k)的加權(quán)值
(11)

于是,可提取得到的二進(jìn)制指示序列hα(x)的離散傅里葉功率譜的特征值Mα。
式(10)充分體現(xiàn)了二進(jìn)制指示序列hα(x)子序列種類、出現(xiàn)頻率和位置的序列特征信息。這樣,就可以將DNA序列x轉(zhuǎn)換為一個(gè)12維的特征向量(MRR,MRY,…,MSW)。
1.3 分類算法描述
為了完成DNA序列的分類,將提取的DNA序列特征值利用式(12)分別計(jì)算出待分類DNA序列對(duì)應(yīng)特征向量與每個(gè)樣本DNA序列特征向量的距離,給定近鄰數(shù)目K,通過(guò)K-NN分類器[18],選取K個(gè)距離最小的樣本DNA序列組成K-最近鄰樣本集合S,統(tǒng)計(jì)S中每種DNA序列類別出現(xiàn)的次數(shù),然后選擇出現(xiàn)頻率最大的DNA序列的類別作為待分類DNA序列的類別。


(12)
算法流程如圖1所示。

圖1 IAF-DNASC算法流程
算法1形式描述了改進(jìn)的無(wú)需比對(duì)的分類算法(簡(jiǎn)記為IAF-DNASC算法)。
算法1:IAF-DNASC
Input:待分類DNA序列集X={x1,x2,…,xm},樣本DNA序列集Y={y1,y2,…,yn},近鄰數(shù)目K
Output:輸出DNA序列xi的類標(biāo)簽
Begin
(1)按式(4)將DNA序列xi映射為12條二進(jìn)制指示序列H(xi),i=1,2,…,m;
(2)按式(4)將DNA序列yj映射為12條二進(jìn)制指示序列H(yj),j=1,2,…,n;


(5)按式(10)對(duì)H(xi)的功率譜xi_PSα(k)進(jìn)行特征提取,得到一個(gè)12維特征向量,記為xi_Mα,i=1,2,…,m;
(6)按式(10)對(duì)H(yj)的功率譜yj_PSα(k)進(jìn)行特征提取,得到一個(gè)12維特征向量,記為yj_Mα,j=1,2,…,n;
(7)對(duì)每個(gè)待分類序列xi,i=1,2,…,m,執(zhí)行以下步驟:
1)用式(12)計(jì)算xi與yj之間的距離:
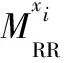
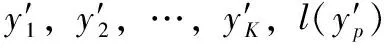
3)輸出DNA序列xi的類標(biāo)簽Label(xi)
其中,c表示所有可能的類標(biāo)簽。
End
IAF-DNASC算法依據(jù)DNA序列不同堿基化學(xué)性質(zhì),結(jié)合K元組方法將DNA序列映射成12條二進(jìn)制指示序列,二進(jìn)制指示序列中保存了DNA序列的堿基化學(xué)性質(zhì),能充分表征DNA序列;對(duì)映射生成的二進(jìn)制指示序列進(jìn)行DFT變換得到離散傅里葉功率譜,綜合利用二進(jìn)制指示序列的不同子序列出現(xiàn)頻率和位置信息,對(duì)功率譜采取匹配加權(quán)值的方法提取特征值,解決不等長(zhǎng)離散傅里葉功率譜的比較問(wèn)題,使得可采用歐氏距離準(zhǔn)確計(jì)算出不同類別DNA序列各特征向量之間的距離,進(jìn)而利用計(jì)算得到的距離值通過(guò)K-NN分類器準(zhǔn)確分類DNA序列。
2 實(shí) 驗(yàn)
2.1 實(shí)驗(yàn)數(shù)據(jù)和環(huán)境
實(shí)驗(yàn)選取了3個(gè)核小體DNA序列的數(shù)據(jù)庫(kù):Caenorhabditis elegans(CE)、Homo sapiens(HM)和Drosophila melanogaster(DM)。這些數(shù)據(jù)可以在網(wǎng)站http://lin.uestc.edu.cn/server/iNucPseKNC/dataset下載。HM數(shù)據(jù)庫(kù)包含2273條核小體形成DNA序列和2300條核小體抑制DNA序列;CE數(shù)據(jù)庫(kù)包含2567條核小體形成DNA序列和2608條核小體抑制DNA序列;DM數(shù)據(jù)庫(kù)包含2900條核小體形成DNA序列和2850條核小體抑制DNA序列。核小體是染色體的基本組成單位,也是染色體中的最重要的重復(fù)單元。有關(guān)這3個(gè)核小體DNA序列的實(shí)驗(yàn)數(shù)據(jù)提取和篩選的具體方法可參見(jiàn)文獻(xiàn)[19]。每個(gè)核小體DNA序列數(shù)據(jù)庫(kù)中包含了兩類核小體DNA序列:一類是核小體形成DNA序列樣本(positive data),另一類是核小體抑制DNA序列樣本(negative data)。實(shí)驗(yàn)利用精度(A),敏感度(Se)和特異性(Sp)3個(gè)指標(biāo)評(píng)價(jià)DNA序列分類性能
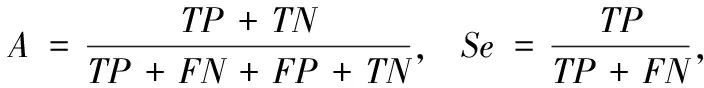
其中,TP表示核小體形成DNA序列正確分類的序列數(shù),F(xiàn)P為核小體形成DNA序列錯(cuò)誤分類的序列數(shù),TN表示核小體抑制DNA序列正確分類的序列數(shù),F(xiàn)N為核小體抑制DNA序列錯(cuò)誤分類的序列數(shù)。
實(shí)驗(yàn)使用的計(jì)算機(jī)為8核Intel(R) Core(TM) i7-6700K CPU @ 4.00 GHz 4.00 GHz處理器、內(nèi)存容量為32.0 GB。運(yùn)行操作系統(tǒng)為Windows 10。軟件運(yùn)行環(huán)境為Visual studio 2012,采用C++編程實(shí)現(xiàn)算法。實(shí)驗(yàn)將本文的IAF-DNASC算法與已有的同類代表性算法correlation,UCD-gzip和UCD-deflate的分類性能進(jìn)行比對(duì)分析。
2.2 實(shí)驗(yàn)結(jié)果
在實(shí)驗(yàn)過(guò)程中,我們利用十折交叉驗(yàn)證法,通過(guò)隨機(jī)抽樣將每個(gè)數(shù)據(jù)集劃分為10個(gè)子集,每次選擇其中的9個(gè)子集作為訓(xùn)練數(shù)據(jù),剩余的1個(gè)子集作為測(cè)試數(shù)據(jù),重復(fù)10次實(shí)驗(yàn),取實(shí)驗(yàn)結(jié)果的平均值。K-NN分類算法中參數(shù)K的最優(yōu)取值取決于測(cè)試數(shù)據(jù)集,參照文獻(xiàn)[11],實(shí)驗(yàn)選取K=1,3,5,7,…,21,L=2。
圖2、圖3和圖4分別給出了4種算法在3個(gè)數(shù)據(jù)集CE、DM和HM上取不同K值時(shí)獲得的DNA序列分類精度、敏感度和特異性。
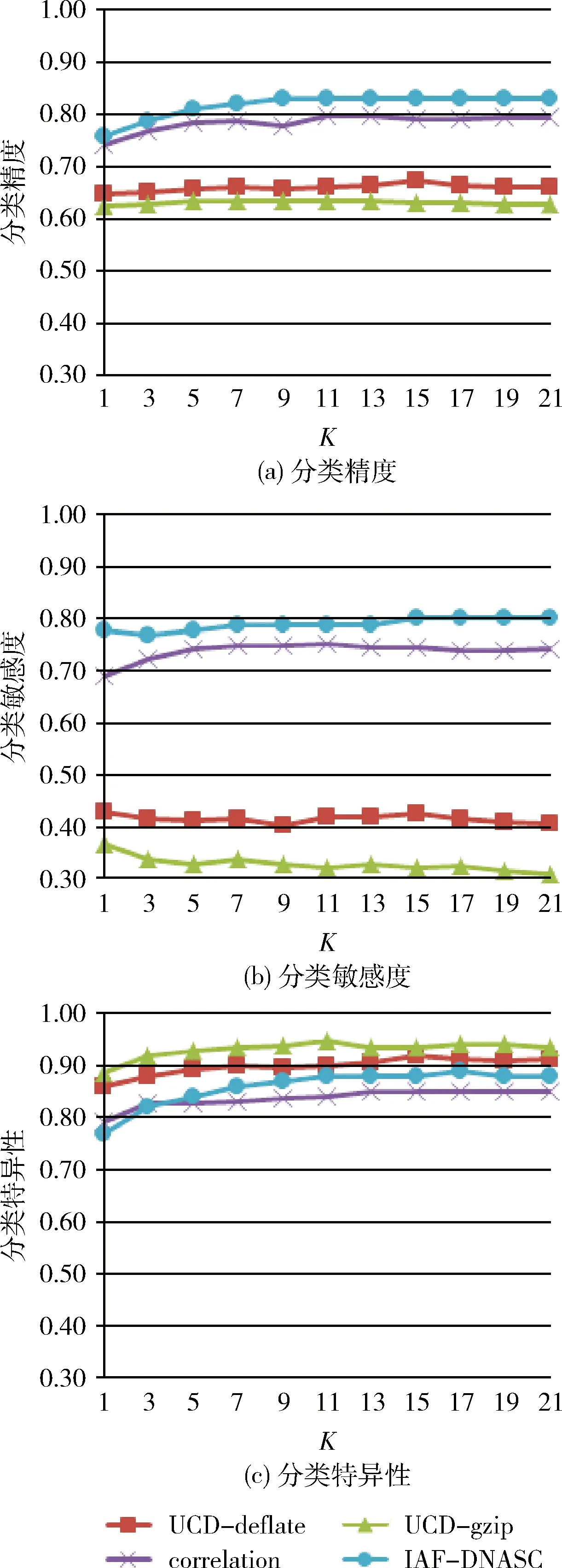
圖2 4種算法在CE數(shù)據(jù)集上的分類精度、敏感度和特異性
圖2的結(jié)果表明:在數(shù)據(jù)集CE上,對(duì)于不同的K值,IAF-DNASC的平均分類精度和敏感度均高于算法UCD-deflate、UCD-gzip和correlation的平均分類精度和敏感度,尤其是遠(yuǎn)高于UCD-deflate和UCD-gzip的分類精度和敏感度;IAF-DNASC的特異性略低于UCD-gzip和UCD-deflate,但高于correlation的值。
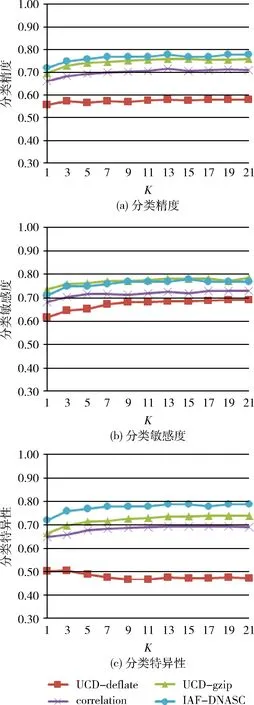
圖3 4種算法在DM數(shù)據(jù)集上的分類精度、敏感度和特異性
由圖3可看出:在數(shù)據(jù)集DM上,對(duì)于不同的K值,IAF-DNASC算法的分類精度和特異性均高于算法UCD-deflate、UCD-gzip和correlation,IAF-DNASC算法的分類敏感度與UCD-gzip幾乎一樣,高于UCD-deflate和correlation。

圖4 4種算法在HM數(shù)據(jù)集上的分類精度、敏感度和特異性
由圖4可知,在數(shù)據(jù)集HM上,IAF-DNASC算法的分類精度和特異性在4種算法中都是最高的,IAF-DNASC算法分類時(shí)取得敏感度略低于correlation,但高于UCD-gzip和UCD-deflate。
上述這些結(jié)果表明,在數(shù)據(jù)集CE、DM和HM上,整體上,IAF-DNASC算法的分類性能優(yōu)于correlation、UCD-gzip和UCD-deflate,這是因?yàn)镮AF-DNASC算法綜合利用堿基化學(xué)性質(zhì)、出現(xiàn)頻率和位置信息,采用匹配加權(quán)值方法提取DNA序列DFT功率譜特征值,能夠充分挖掘和表征DNA序列的重要特征,利用這些特征信息可以更準(zhǔn)確地計(jì)算出不等長(zhǎng)DNA序列間的距離,從而可以更為準(zhǔn)確地完成DNA序列的分類。
3 結(jié)束語(yǔ)
本文結(jié)合K元組方法和離散傅里葉變換,提出了改進(jìn)的無(wú)需比對(duì)的DNA序列分類算法,該算法有效地提取了DNA序列堿基化學(xué)性質(zhì)、出現(xiàn)頻率和位置特征信息,通過(guò)這些提取的特征信息可以更加準(zhǔn)確地計(jì)算出不等長(zhǎng)DNA序列之間的距離,利用計(jì)算得到的距離值通過(guò)K-NN分類器實(shí)現(xiàn)更準(zhǔn)確地分類DNA序列,從而整體上獲得更高的分類精度、敏感度和特異性。RNA序列、蛋白質(zhì)序列舉有不同的生化性質(zhì),下一步工作將研究將本文方法拓廣到RNA序列、蛋白質(zhì)序列的序列挖掘中。

