基于歷史增強型植被指數時序的農作物類型早期識別
郝鵬宇,唐華俊,陳仲新,牛 錚
?
基于歷史增強型植被指數時序的農作物類型早期識別
郝鵬宇1,唐華俊1※,陳仲新1,牛 錚2
(1. 中國農業科學院農業資源與農業區劃研究所農業部農業遙感重點實驗室,北京 100081; 2. 中國科學院遙感與數字地球研究所遙感科學國家重點實驗室,北京 100101)
快速準確地獲取農作物分布數據對作物估產、災害預警具有重要意義。該文針對目前農情遙感監測業務中普遍存在的缺乏地面數據和分類時效性較低的問題,以美國堪薩斯州為研究區,提出了基于參考時間序列獲得訓練樣本的方法。首先,基于2006到2013年的MODIS EVI時間序列數據和cropland data layer(CDL)數據,使用免疫系統網絡方法建立苜蓿、玉米、高粱和冬小麥的參考EVI時間序列;根據2006年到2013年作物分布情況,將作物超過總記錄年數一半的象元作為2014年“潛在”訓練樣本;通過計算參考EVI時間序列和“潛在”樣本的MODIS EVI時間序列的歐氏距離確認2014年訓練樣本;最后使用這些樣本和2014年Landsat NDVI月合成數據進行30 m作物識別,并且評價時間序列長度對作物識別結果的影響。試驗結果表明,時間序列長度為4-8月時,獲得2014年樣本10 183個,樣本正確率為96.32%,總體分類精度為94.02%,接近使用完整時間序列數據的結果(總體分類精度94.89%);提取的苜蓿、玉米、高粱和冬小麥的面積分別為549.5、1 999.5、2 851.5和6 415.3 km2,與CDL數據相比誤差低于20%,說明基于參考時間序列方法獲得的訓練樣本具有較高的正確率,具備進行30 m作物早期制圖的潛力。該研究可為提高農作物遙感制圖工作效率提供參考。
作物;遙感;識別;參考EVI時間序列;作物識別;樣本;免疫系統網絡;CDL數據
0 引 言
快速獲取與分析農作物的種植分布是農作物長勢監測的基礎,遙感技術以其快速、無損和客觀等特點,已廣泛應用于農作物種植面積提取[1]。由于多時相遙感數據可以在整個生長期內反映不同作物生長狀況的差異,具有更強的區分不同作物的能力[2-3],大多數農作物分類產品需要使用全年的多時相遙感數據,因而需要在年底或者第二年才能獲得農作物分類的結果,時效性較低[4]。如果能使用較短的時間序列提前獲得作物分類的結果,將使作物提取的結果更有意義[5]。例如,在農作物夏季灌溉之前獲得農作物分類圖,可以輔助決策灌溉用水量的分配,提高灌溉的用水效率;在收獲之前獲得作物分類結果,可以更有效地管理農作物的收割。另外,目前大多數農作物遙感識別工作均采用監督分類的方法,因而分類的訓練樣本是農作物精確識別的關鍵。但是,通過野外試驗的方法獲得訓練樣本的方案會造成極大的人力、物力消耗,因而每年獲得地面訓練樣本存在較大困難[6-7]。所以,在不能獲得地面調查數據的情況下,探討使用較短的時間序列數據進行作物提前識別的方法,在農作物識別的業務化運行工作中具有重要意義。
由于多年間同種作物物候的差異小于不同作物的物候差異,一些學者根據先驗知識,直接使用遙感數據描述特定作物的特征,從而實現作物提取。如Dong等[8]發現中國東北的水稻在移栽期時,主要體現為水體特征,而在移栽期之后與其他作物的特征類似,提出了基于先驗知識的水稻自動提取方法。Zhong等[6]使用MODIS EVI數據計算作物的物候期,使用線性差值的方法獲得物候期的作物光譜,并根據作物關鍵物候期的光譜人為定義決策樹進行作物識別。這些方法針對特定作物提出,不能實現多種類的作物識別;而且需要首先使用遙感數據提取物候,而物候的提取會引入誤差,影響作物識別的精度。
另一種方法是使用相同研究區其他年份的訓練樣本訓練分類器,再使用這些分類器進行作物識別[9]。例如,Muhammad等[10-11]使用多年歷史數據訓練分類器,并在此基礎上分別在美國堪薩斯州和中國新疆進行作物識別,其總體分類精度達80%以上。Howard等[12]使用5 a歷史數據訓練決策樹分類器,并進行作物識別,其分類精度也在80%左右。Friesz等[13]將該方法進行擴展,實現了2000年到2013年的全美250 m連續作物分布制圖,總體分類精度達到90%。Hao等[14]使用2006到2010年的歷史地面數據和MODIS數據建立作物的參考曲線,并嘗試使用參考曲線在新疆博樂市和瑪納斯縣進行2011年30 m作物識別,其識別精度達到85%左右。但是,由于使用MODIS計算的植被指數與使用Landsat和HJ數據計算的植被指數存在差異,作物識別過程中需要使用線性轉換的方法將Landsat和HJ數據的植被指數轉換為MODIS植被指數,轉換過程中引進的誤差會影響分類精度。所以如何改進植被指數線性轉換的過程有待進一步探討。
本文針對農作物遙感識別時效性較低和缺乏訓練樣本的問題,通過作物歷史EVI時間序列直接獲得分類年需要的訓練樣本,并使用這些樣本進行作物識別。另外,評價時間序列長度較短時,使用該方法進行作物早期識別的適用性,為提高業務運行的效率提供新的思路。
1 研究區和數據
1.1 研究區概況
堪薩斯州(37°N—40°N,94°W—102°W)位于美國大平原,其主要土地利用類型為農田,面積占全州的46.6%[15],主要農作物包括玉米、高粱和冬小麥等[16]。由于堪薩斯州西南ASD 30 (Agricultural Statistics Districts 30)區域包括了堪薩斯州主要的農作物,本文選擇堪薩斯州西南的ASD30區域作為研究區(圖1)。根據Shroyer等[17],研究區在堪薩斯州種植分區中屬于“Zone 2”和“Zone 3”;作物的播種期早于堪薩斯州東南的ASD 90區域,晚于堪薩斯州西北的ASD 10區域。最主要的作物為冬小麥,種植面積占該區域農田面積的53.16%,玉米和高粱也為該區域的主要作物,其種植面積占該區域農田面積的21.24%和21.19%[18]。
1.2 遙感數據
1.2.1 數據及預處理
本文使用的遙感數據包括2部分:1)2006年至2014年的MODIS EVI時間序列數據;2)2014年Landsat數據。MODIS數據為16 d合成的植被指數產品MOD13Q1,空間分辨率為250 m,包括NDVI、EVI、紅、藍、近紅、中紅和VI 質量文件等數據[3]。本文使用的MODIS數據的條帶號為h09v05的數據,數據時相為2006年到2014年每年1月1日至12月31日,2006年至2013 年的數據用于建立參考EVI曲線,2014年的數據用于識別2014年作物。本文使用MRT(MODIS reprojection tool)對MODIS數據進行鑲嵌和投影轉換,投影為UTM(universal transverse mercator projection)投影,投影帶號為14N,地理坐標系為WGS84 坐標系。使用試驗區域對MODIS數據進行裁剪,并使用Savizky-Golay濾波的方法進行EVI時間序列重構[19]。另外,本文獲得2014 年4月至10月所有Landsat數據(包括Landsat-7 ETM+數據和Landsat-8 OLI數據),空間分辨率為30 m。所有Landsat數據均為經過幾何精校正的Landsat地表反射率數據,其幾何校正誤差小于12 m[20]。使用地表反射率計算NDVI,再使用月最大合成的方法對NDVI進行數據合成,取每個月NDVI中的最大值代表這個月的NDVI。最后,使用試驗區的邊界對月合成的Landsat NDVI時間序列進行裁剪,并進行投影轉換,地理坐標系為WGS84,投影為UTM投影,投影帶號為14 N。

注:圖中影像為2014年7月MODIS EVI數據。
1.2.2 樣本數據
本文使用美國農業部提供的CDL(cropland data layer)數據作為地面數據,涉及的作物類型為堪薩斯州的4種主要的作物,分別為苜蓿、玉米、高粱和冬小麥[21],4種作物CDL數據的用戶精度高于85%[18]。
在獲得2014年的驗證樣本時,同時使用CDL數據的作物類型數據和作物識別置信度數據[18]。分類置信度是分類器在進行分類時輸出的分類可靠性,代表分類器對分類結果的確定性。首先,使用90%的分類置信度作為閾值獲得“高置信度”掩膜;接著使用“高置信度”掩膜刪除CDL分類置信度較低的象元。隨后,根據2014年CDL數據計算每個250 m×250 m的MODIS象元內主要作物所占的面積比例。如果在一個象元內,一種作物所占的比例大于80%,將這個象元定義為相應作物的“純網格”,將這個網格中心的坐標定義為該作物的驗證樣本。
本文使用類似的方法,基于CDL數據獲得2006年至2013年的歷史參考樣本。由于在這些年份,CDL數據未提供作物識別置信度產品,在獲得歷史訓練樣本時,省略了“高置信度”掩膜的過程。2006至2013年的訓練樣本和2014年的驗證樣本的數量如表1所示。

表1 2006年至2014年樣本數量
2 研究方法
本文的技術流程如圖2所示。首先,使用免疫系統網絡方法[22],基于2006到2013年的MODIS EVI數據和CDL數據生成的訓練樣本構建4種典型作物的參考EVI時間序列曲線。再根據2006到2013年的CDL數據獲得2014年的“潛在”訓練樣本。由于本文嘗試進行作物早期識別,共嘗試使用7種時間序列長度(4月、4-5月、4-6月等,從4月開始,以月為單位延長時間序列長度)的數據提取作物。針對每種時間序列長度,通過比較參考EVI時間序列和2014年“潛在”訓練樣本的MODIS EVI時間序列獲得2014年訓練樣本。再使用這些訓練樣本和對應的Landsat NDVI數據進行30 m分辨率農作物識別。最后,通過比較本文獲得的2014年訓練樣本與對應的CDL數據和使用這些訓練樣本進行作物識別的精度,對本文所提出的方法進行評估。

圖2 基于歷史增強型植被指數時序的農作物類型早期識別流程圖
2.1 建立參考EVI時間序列曲線
本研究使用免疫系統網絡算法(ABNet)建立作物的參考EVI時間序列曲線。該算法的基礎單元是“抗體”,而“免疫系統”模型由多個抗體組成。每個抗體由3個屬性組成,分別是該抗體對應的類別、中心向量和識別半徑。中心向量即分類的特征序列。與之相對應的“抗原”由2個屬性組成,分別是中心向量和所屬類別。每個“抗體”可以識別其識別半徑內的“抗原”。ABNet主要包括訓練和分類2個過程,本研究使用訓練過程獲得參考EVI時間序列。例如,在獲得作物A的參考EVI時間序列時,具體過程為:1)使用2006年至2013年的EVI時間序列數據和訓練樣本獲得“抗原”,并對所有“抗原”的“中心向量”進行歸一化,使不同的特征具備可比性;2)通過“抗原”篩選,獲得代表性最強的“代表抗原”;3)通過克隆,獲得大量“代表抗原”;4)使用遺傳算法對第3)步結果進行變異,產生作物A的隨機“抗體”;5)分別使用所有隨機“抗體”識別作物A的抗原和其他作物的抗原,將識別作物A“抗原”數量最多的隨機“抗體”作為“抗體”記錄,被這個抗體識別的作物A“抗原”從“抗原集”中刪除。重復該過程直到所有作物A的“抗原”均被識別,作物A的訓練過程結束。最后,本研究把獲得的所有“抗體”的中心向量作為EVI參考時間序列。因ABNet方法的優點是每個類別可以擁有多個“抗體”,可以解決“同物異譜”的現象,尤其適合本研究基于多年EVI時間序列建立參考曲線的問題。本研究使用IDL語言實現ABNet算法,在使用“抗體”識別“抗原”的過程中,涉及到相似度計算的方法,由于歐氏距離計算簡便,本文使用歐氏距離法作為相似度評價方法[23],其計算方法如式(1)所示,式中ED()指向量和的歐氏距離。a和b分別為時間序列和在時相的EVI值,為時間序列中時相的數量。

2.2 基于參考EVI曲線獲得2014年訓練樣本
基于2006到2013年歷史數據獲得2014年分類樣本時,首先使用ABNet方法,根據2006到2013年的歷史MODIS EVI時間序列數據建立作物的參考MODIS EVI時間序列;在2006到2013年的作物類型記錄中,如果一個象元為作物A的次數超過總記錄年數的一半,則假設在2014年,該MODIS象元可以被標記為作物A。隨后計算該象元的MODIS EVI曲線與所有參考MODIS EVI曲線的歐氏距離,如果任意一個歐式距離小于對應抗體的識別半徑,則確認該象元屬于作物A,將該象元的中心坐標作為2014年作物A的訓練樣本,否則不記錄該象元。
2.3 作物識別和精度驗證
本研究的作物識別工作僅針對CDL數據中苜蓿、玉米、高粱和小麥4種作物的地塊。針對不同的時間序列長度,使用對應的訓練樣本和Landsat NDVI時間序列進行作物識別。例如,使用4月的訓練樣本和4月的Landsat時間序列獲得4月的作物分布結果,使用4-5月數據和4-5月Landsat NDVI時間序列獲得5月作物識別結果。以此類推,本研究可以獲得7種時間序列長度的作物識別結果。
識別作物時,本研究選用的分類器為隨機森林算法[24],由于隨機森林在處理大量訓練樣本時效率較高,且對于訓練樣本的容錯能力強,已被廣泛應用于遙感分類中。隨機森林法有2個需要用戶設按定的參數:決策樹的數量(ntree)和選擇的分裂屬性個數(mtry)。本研究將ntree設置為1 000(數值較大,增加隨機森林中決策樹的數量),mtry設置為分特征數量的平方根(大多數隨機森林的默認設置)。本研究使用基于混淆矩陣的精度評價方法,計算分類結果的制圖精度、用戶精度、整體精度和Kappa系數評價作物識別結果的精度。
3 試驗結果
3.1 作物的參考EVI時間序列曲線
由于每種作物有多種生長狀況,所以本研究共獲得53條參考EVI時間序列曲線,其中苜蓿1條、玉米32條,高粱17條,冬小麥3條。圖3為本研究中幾種主要農作物的參考EVI時間序列曲線。其中,玉米EVI曲線的峰值介于0.6到0.8,出現在第175到190天(每年日期排序DOY)左右(6月下旬到7月上旬)。高粱的EVI峰值介于0.5到0.7,略低于玉米,且EVI峰值期為第209天到225天(7月中下旬),比玉米略晚。苜蓿的EVI參考時間序列與其他幾種作物有明顯差異,EVI在第100天達到峰值,峰值為0.7,隨后EVI持續較高,直到第270天開始下降。另外,苜蓿只有1條參考曲線,這也說明苜蓿與其他幾種作物分離度較高。冬小麥是冬季作物,其EVI峰值期為第100到30天,EVI峰值為0.4到0.8。總體而言,本研究所涉及4種作物的參考EVI時間序列曲線具備可分離度。
3.2 驗證本文獲得的2014年訓練樣本
本文使用不同時間序列長度的EVI時間序列,基于各種作物的參考EVI時間序列獲得2014年的訓練樣本,并將這些訓練樣本與2014年CDL數據進行比較,結果如表2所示。由于本研究將CDL數據作為地面真實數據,所以本研究識別的樣本與CDL數據的作物類型相同時,認為該樣本識別正確。

注:玉米和高粱的參考EVI時間序列數量較多,圖中用這2類作物參考曲線的平均值和標準差作為代表;日序DOY從1月1日計。
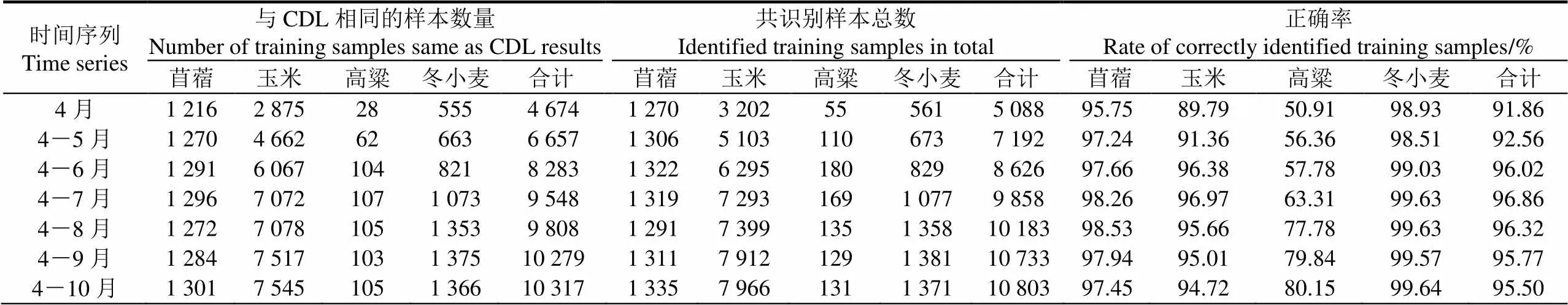
表2 本文識別的2014年訓練樣本與CDL數據的比較
由表2可知,僅使用4月的數據時,識別樣本的正確率最低,為91.86%;其中高粱樣本的識別錯誤較大,識別的55個樣本中僅有28個與CDL數據相同。玉米樣本的正確率相對較低,為89.79%。苜蓿和冬小麥樣本的正確率較高,分別為95.8%和98.9%。隨著延長時間序列長度,識別出的樣本數量增加。只使用4月的數據時,共識別樣本5 088個,而使用4-10月數據時,共識別樣本10 803個,且有95.5%的樣本與CDL數據的作物類型相同。錯誤識別的樣本主要為玉米和高粱樣本,7 966個玉米樣本中,有421個與CDL數據不同,而131個高粱樣本中,有26個與CDL數據不同。本研究識別的高粱樣本較少,可能的原因是高粱僅有1 558個“潛在”高粱樣本,且高粱樣本與玉米樣本容易混淆。
雖然識別的樣本數量和識別樣本正確率隨著時間序列長度增加而提高,當時間序列為4-8月時,獲得樣本10 183個,其中9 808個與CDL數據相同,且高粱樣本的正確識別率為77.78%。使用更長的時間序列不能明顯提高獲得的樣本的數量和正確率。這說明,在分類年不能獲得地面數據的情況下,可以基于歷史數據參考曲線的方法在8月獲得用于識別作物的分類訓練樣本。但是,高粱樣本中與CDL類型相同的樣本數量在時間序列長度為2個月(4-5月時間序列)至7個月(4-10月時間序列)中沒有明顯變化,這些都說明了高粱與其他作物容易混淆。
3.3 作物識別精度驗證
針對不同時間序列長度,本文使用相應的訓練樣本和對應長度的Landsat NDVI時間序列進行30 m作物提取,并使用2014年的驗證樣本(表1)進行作物識別精度驗證,結果如表3所示。在進行2014年作物識別時,本文使用3.2小節中所有樣本(不只是與CDL數據相同的樣本),這樣可以評估基于歷史數據和參考曲線的方法,在分類年不能獲得地面數據時識別作物的精度。

表3 不同長度Landsat NDVI時間序列的作物識別精度
注:表中PA(%)為制圖精度,UA(%)為用戶精度。
Notes: UA (%) means user’s accuracy and PA (%) means producer’s accuracy.
由表3可知,時間序列為4月和4-5月時,作物識別的精度較低,總體精度分別為66.12%和52.51%。僅有冬小麥的用戶精度高于90%,其他作物的制圖精度和用戶精度均較低,例如時間序列為4月時,苜蓿的用戶精度為43.29%。作物識別精度隨所用時間序列長度延長而升高,當時間序列達到4-8月時,總體分類精度為94.02%,更長的時間序列不能明顯提高分類精度,當時間序列為4-10月時,總體分類精度為94.89%。
圖4為局部CDL數據與不同時間序列長度的Landsat NDVI數據識別作物結果的對比。當時間序列為4月和4-5月時,部分冬小麥被識別成苜蓿,而且分類結果中出現明顯的“椒鹽噪聲”。識別作物使用的時間序列較長時,幾種主要作物的分布基本與CDL數據相同。本研究試驗區的特點為地塊面積較大,在地塊邊緣出現錯分。但在分類結果驗證的混淆矩陣中沒能體現出地塊邊緣的錯分。由于本文中使用的“假設樣本”均為MODIS“純”象元,但農田地塊邊界處的混合象元是由農作物和周圍非農作物構成,因而混合的象元易被錯分。
表4為本研究提取的研究區內主要作物的種植面積,其中包括了CDL數據和不同時間序列長度情況下,使用本文獲得的訓練樣本識別的作物種植面積。

圖4 不同長度時間序列數據的作物識別結果(亞區)
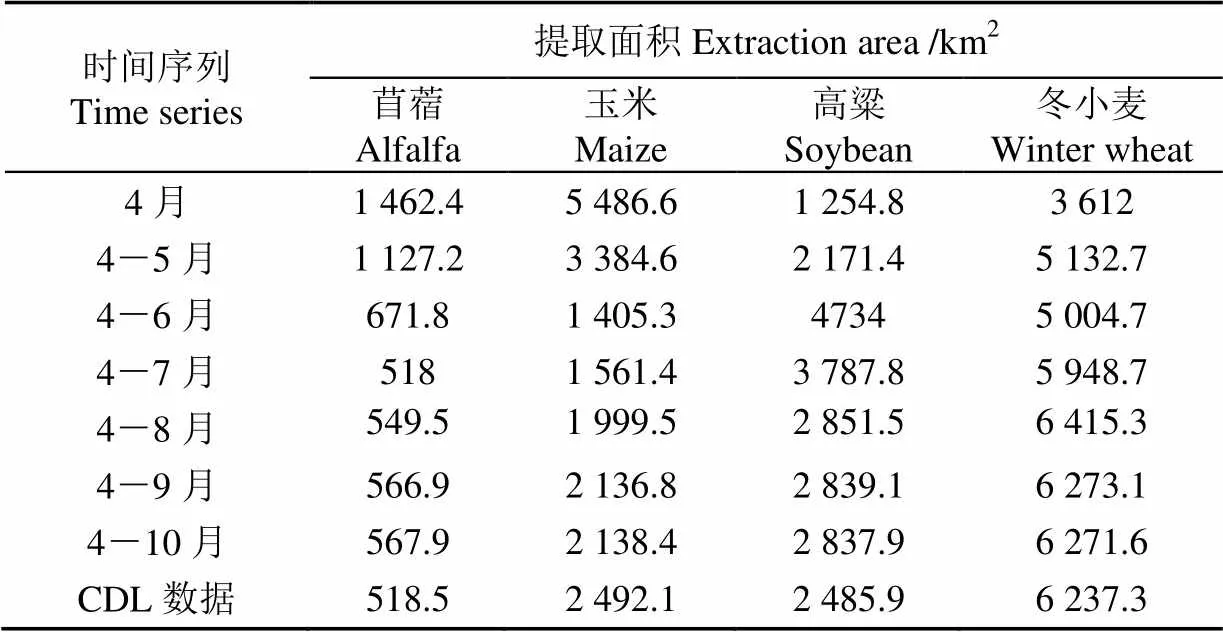
表4 研究區不同長度時間序列數據主要作物種植面積提取結果
當時間序列較短時,作物面積與CDL數據差異較大,苜蓿和玉米的面積被高估,而冬小麥和高粱的面積被低估。例如僅使用4月的數據時,苜蓿的面積為1 462.4 km2,比CDL數據的苜蓿面積高943.9 km2。隨時間序列延長,作物面積逐漸接近CDL數據的作物面積,例如使用4-8月數據時,苜蓿和冬小麥的面積分別為549.5和6 415.3 km2。
4 討 論
本研究中,使用MODIS EVI為媒介,將2006到2013年的作物特征轉換到2014年,這樣引起的問題是同種作物的物候特征在多年間存在差異[23,25]。對于每種作物,2006到2013年的EVI時間序列曲線基本相同,但不同的天氣情況和作物管理狀況仍然引起了作物生長情況的不同,導致MODIS EVI時間序列的變化(圖5)。例如在2013和2014年的日序為100 d時,苜蓿的EVI為0.7左右,明顯低于其他年份。另外,可能是由于2012年嚴重干旱的原因[26],高粱的EVI峰值為在2012年為0.5,也明顯低于其他年。因此,本研究使用多年EVI時間序列,包含了多種農作物的生長情況,這減小了多年間作物生長差異對訓練樣本的影響。

圖5 研究區主要農作物的MODIS EVI曲線年際變化
針對Hao等[14]方法中需要進行MODIS NDVI和Landsat NDVI轉換的問題,本研究直接使用歷史(2006到2013年)MODIS EVI曲線獲得分類年(2014年)的分類樣本,避免了MODIS EVI和Landsat EVI間的轉換。但是,本方法仍然存在缺點:1)本研究使用2006到2013年的CDL數據作為地面真實數據,并選擇了CDL數據中用戶精度較高的農作物種類,但CDL數據的錯分會給參考EVI曲線帶來誤差。2)由于本研究定義2006到2013年的MODIS“純象元”樣本的閾值為80%,這也會引起2006到2013年MODIS EVI曲線的誤差,從而影響各種作物的參考曲線。
在今后的農作物識別工作中有可能消除這2方面的缺陷。首先,從2014年起CDL數據開始提供分類置信度數據,可以挑選分類置信度較高的樣本建立參考時間序列曲線。第二,定義MODIS“純網格”的閾值是樣本質量和樣本數量的平衡,較高的閾值增加了網格內作物的純度,但降低了樣本的數量。所以,“純網格”的閾值應根據不同試驗區的情況由用戶定義。最后,在建立參考曲線時,還需要提高算法對樣本的容錯能力,以降低不準確的樣本對參考曲線的影響。
本文還嘗試了基于參考MODIS EVI時間序列和Landsat NDVI短時間序列的30 m作物提早識別,結果表明基于參考時間序列獲得的訓練樣本和Landsat NDVI時間序列可以在8月時獲得精度較高的作物識別結果。Hao等[25]使用500 m分辨率的MODIS地表反射率數據進行堪薩斯州作物提前識別試驗,其結果也表明作物識別精度和分類確定性在8月達到飽和。總之,在堪薩斯研究區,目前多光譜數據和植被指數數據可以在8月實現作物識別,加入更多的分類特征有可能進一步提高作物分類的時效性。
本文的試驗區是美國堪薩斯州,研究區的特點是地塊較大,在MODIS影像上存在較多純象元。但在其他地區,例如中國大部分地區、東南亞等,地塊破碎,田塊面積較小,MODIS數據多為混合象元數據,因而不能直接使用MODIS數據作為媒介將作物的歷史知識傳遞到分類年,從而獲得訓練樣本。所以,需要考慮使用更高分辨率的遙感數據建立時間序列,進行作物分類知識的傳遞。例如,已有多種中分辨率的多光譜遙感衛星,如美國的Landsat-8[27]、歐洲的Sentinel-2[28]、中國的環境星、高分一號和高分二號衛星等[28-29]。在地塊破碎地區,可以嘗試綜合應用這些中分辨率衛星數據建立類似MODIS時間序列的中分辨率時間序列數據,并以中分辨率時間序列數據作為媒介,進行作物知識的傳遞、在分類年不能獲得野外調查數據時生成分類的訓練樣本。
另外,本文從CDL數據產品中提取驗證樣本進行分類精度驗證。雖然CDL數據是遙感產品,存在不確定性,但本文通過選擇分類精度高于85%的作物、建立CDL分類置信度掩膜和“純”MODIS象元篩選,盡可能使用可信度高的樣本作為驗證樣本。所以本文雖然沒有使用地面數據驗證分類結果,但其驗證精度具有較強的說服力[30-31]。由于美國農業部提供了連續多年的農作物分布歷史數據,可以支持本研究的內容,而且這是能在研究區獲得的唯一數據;所以本文選用堪薩斯州作為研究區。在今后會繼續嘗試在國內獲得多年歷史作物分布數據,并將這個方法應用于國內作物分布提取業務。
5 結 論
針對目前農作物遙感識別工作中存在的缺乏地面數據和識別作物時效性較低的問題,本研究提出了使用多年歷史樣本直接獲得分類年訓練樣本的方法。在堪薩斯州東南(ASD30區域)試驗區,基于2006到2013年數據獲得2014年的訓練樣本,使用這些樣本和Landat NDVI數據進行了2014年30 m農作物提取,并探討了使用短時間序列數據提前獲得作物識別結果的可行性。試驗結果表明:
1)使用完整的時間序列時,基于歷史數據獲得的分類訓練樣本數量為10 803,其中有10 317個樣本的作物類型與CDL數據相同,使用這些樣本識別作物時,總體精度為94.89%。
2)使用4-8月數據時,獲得2014年樣本的數量(10183個)、正確率(96.32%)、分類精度(94.02%)和作物面積(苜蓿549.5 km2、玉米1 999.5 km2、大豆2 839.1 km2和冬小麥6 415.3 km2)都已接近使用完整時間序列所獲得的結果,這表明基于參考時間序列獲得的訓練樣本可以在8月進行高精度的作物分布制圖,在今后的工作中可以使用CDL置信度數據進一步提高參與建立參考曲線的樣本質量。
[1] 唐華俊,吳文斌,余強毅,等. 農業土地系統研究及其關鍵科學問題[J]. 中國農業科學,2015,48(5):900-910.
Tang Huajun, Wu Wenbin, Yu Qiangyi, et al. Key research priorities for agricultural land system studies[J]. ScientiaAgriculturaSinica, 2015, 48(5): 900-910. (in Chinese with English abstract)
[2] Atzberger Clement. Advances in remote sensing of agriculture: Context description, existing operational monitoring systems and major information needs[J]. Remote Sensing, 2013, 5(2): 949-981.
[3] Pan Yaozhong, Li Le, Zhang Jinshui, et al. Winter wheat area estimation from MODIS-EVI time series data using the Crop Proportion Phenology Index[J]. Remote Sensing of Environment, 2012, 119(3): 232-242.
[4] 陳仲新,任建強,唐華俊,等. 農業遙感研究應用進展與展望[J]. 遙感學報,2016,20(5):748-767.
Chen Zhongxin, Ren Jianqiang, Tang Huajun, et al. Progress and perspectives on agricultural remote sensing research and applications in China[J]. Journal of Remote Sensing, 2016, 20(5): 748-767. (in Chinese with English abstract)
[5] Gallego J, Craig M, Michaelsen J, et al. Best practices for crop area estimation with remote sensing [C]. Ispra: GEOSS Community of Practice Ag 0703a. 2008.
[6] Zhong L H, Gong P, Biging G S. Efficient corn and soybean mapping with temporal extendability: A multi-year experiment using Landsat imagery[J]. Remote Sensing of Environment, 2014, 140(1): 1-13.
[7] 黃亞博,廖順寶. 多源數據的土地覆被樣本自動提取[J]. 遙感學報,2017,21(5):757-766
Huang Yabo, Liao Shunbao. An automatic method of samples collection for land cover classification based on multisource datasets[J]. Journal of Remote Sensing, 2017, 21(5): 757-766. (in Chinese with English abstract)
[8] Dong J, Xiao X, Kou W, et al. Tracking the dynamics of paddy rice planting area in 1986-2010 through time series Landsat images and phenology-based algorithms[J]. Remote Sensing of Environment, 2015, 160(4): 99-113.
[9] 郝鵬宇,牛錚,王力, 等. 基于歷史時序植被指數庫的多源數據作物面積自動提取方法[J]. 農業工程學報,2012,28(23):123-131.
Hao Pengyu, Niu Zheng, Wang Li, et al. Multi-source automatic crop pattern mapping based on historical vegetation index profiles [J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2012, 28(23): 123-131. (in Chinese with English abstract)
[10] Muhammad S, Niu Z, Wang L, et al. Crop classification based on time series MODIS EVI and ground observation for three adjoining years in Xinjiang[J]. Spectroscopy and Spectral Analysis, 2015, 35(5): 1345-1350 .
[11] Muhammad S, Zhan Y L, Wang L, et al. Major crops classification using time series MODIS EVI with adjacent years of ground reference data in the US state of Kansas[J]. Optik-International Journal for Light and Electron Optics, 2016, 127(1): 1071-1077.
[12] Howard D M, Bruce K W. Annual crop type classification of the US Great Plains for 2000 to 2011[J]. Photogrammetric Engineering and Remote Sensing. 2014, 80(6): 537-549.
[13] Friesz A M, Wylie B K, Howard D M. Temporal expansion of annual crop classification layers for the CONUS using the C5 decision tree classifier[J]. Remote Sensing Letters, 2017, 8(4): 389-398.
[14] Hao P Y, Wang L, Zhan Y L. Using moderate-resolution temporal NDVI profiles for high-resolution crop mapping in years of absent ground reference data: A case study of Bole and Manas counties in Xinjiang, China [J]. ISPRS International Journal of Geo-Information, 2016, 5(5): 67.
[15] Wardlow B D, Stephen L E. Large-area crop mapping using time-series MODIS 250 m NDVI data: An assessment for the U.S. Central Great Plains[J]. Remote Sensing of Environment, 2008, 112(3): 1096-1116.
[16] Masialeti I S E, Wardlow B D. A comparative analysis of phenological curves for major crops in kansas[J]. Giscience& Remote Sensing, 2010, 47(2): 241-259.
[17] Shroyer J P, Thompson C, Brown R, et al. Kansas crop planting guide [EB/OL]. [2018-06-20] https://www.bookstore.ksre.ksu.edu/pubs/L818.pdf.
[18] USDA. National Agricultural Statistics Service [EB/OL]. [2018-06-20]http://www.nass.usda.gov/research/Cropland/SARS1a.htm.
[19] 邊金虎,李愛農,宋孟強,等. MODIS植被指數時間序列Savitzky-Golay濾波算法重構[J]. 遙感學報,2010,14(4):725-741.
Bian Jinhu, Li Ai’nong, Song Mengqiang, et al. Reconstruction of NDVI time-series datasets of MODIS based on Savitzky-Golay filter[J]. Journal of Remote Sensing, 2010, 14(4): 725-741. (in Chinese with English abstract)
[20] USGS. Landsat Collections [EB/OL]. [2018-06-20] https://landsat.usgs.gov/ landsat-collections.
[21] Boryan C, Yang Z W, Mueller R, et al. Monitoring US agriculture: the US Department of Agriculture, National Agricultural Statistics Service, Cropland Data Layer Program[J]. Geocarto International, 2011, 26(5): 341-358.
[22] Zhong Y F, Zhang L P. An adaptive artificial immune network for supervised classification of multi-/hyperspectral remote sensing imagery[J]. IEEE Transactions on Geoscience and Remote Sensing, 2012, 50(3): 894-909.
[23] Lhermitte S J, Verbesselt W W, Verstraeten P, et al. A comparison of time series similarity measures for classification and change detection of ecosystem dynamics[J]. Remote Sensing of Environment, 2011, 115(12): 3129-3152.
[24] Breiman L. Random forests [J]. Machine learning, 2001, 45(1): 5-32.
[25] Hao P Y, Zhan Y L, Wang L, et al. Feature selection of time series modis data for early crop classification using random forest: A case study in kansas, USA[J]. Remote Sensing, 2015, 7(5): 5347-5369.
[26] Wu C, Chen J M. Diverse responses of vegetation production to interannual summer drought in North America[J]. International Journal of Applied Earth Observation and Geoinformation, 2013, 21(1): 1-6.
[27] Irons J R, Dwyer J L, Barsi J A. The next landsat satellite: The landsat data continuity mission[J]. Remote Sensing of Environment, 2012, 122(1): 11-21.
[28] Drusch M, Del Bello U, Carlier S, et al. Sentinel-2: ESA’s optical high-resolution mission for GMES operational Services[J]. Remote Sensing of Environment, 2012, 120(1): 25-36.
[29] Hao P, Wang L, Niu Z. Potential of multitemporal Gaofen-1 panchromatic/multispectral images for crop classification: Case study in Xinjiang Uygur autonomous region, China[J]. Journal of Applied Remote Sensing, 2015, 9(6): 096035.
[30] Hao P, Wang L, Zhan Y, et al. Crop classification using crop knowledge of the previous year: Case study in southwest Kansas, USA[J]. European Journal of Remote Sensing, 2016, 49(6): 1061-1077.
[31] 郝鵬宇. 基于多時相遙感數據的農作物分類研究[D]. 北京:中國科學院大學,2017.
Hao Pengyu. Crop Classification Using Time Series Remote Sensing Data [D]. Beijing: University of Chinese Academy of Sciences, 2017. (in Chinese with English abstract)
Early season crop type recognition based on historical EVI time series
Hao Pengyu1, Tang Huajun1※, Chen Zhongxin1, Niu Zheng2
(1./100081,; 2.,,,100101,)
Timely and accurate crop distribution maps derived from satellite observations could assist crop growth monitoring. Although crop mapping methodologies have been widely studied, there are still some drawbacks, such as the limitation of ground reference data and low efficiency of crop type mapping caused by using time series data of the entire year. The objectives of this study are: (1) to develop a new method, which can identify crop types using the crop records of the previous years; (2) to evaluate the performance of the method with different time series length, and try to acquire the crop type map at 30 m spatial resolution. The study area of this paper was the ASD30 of Kansas State, USA. We firstly used the cropland data layer (CDL) data and MODIS EVI(enhanced vegetation index) time series between 2006 and 2013 to generate reference EVI time series with the ABNet algorithm for the major crops in the study area, i.e. alfalfa, corn, sorghum and winter wheat. Then, we acquired the “possible” training samples in 2014 using the CDL records between 2006 and 2013. If a pixel was labeled as “Crop A” more than 4 times among the 8-year CDL records, the pixel was labeled as “possible Crop A” in 2014. Next, we compared the MODIS EVI of the “possible crop A” pixels and the reference EVI time series of Crop A, if the 2 profiles were matched, the “possible Crop A” was confirmed as a training sample of “Crop A”. Finally, we used these training samples and monthly composited Landsat NDVI (normalized differential vegetation index) to identify crop types at 30 m resolution. To analyze the effect of time series length on crop type identification performance, we tried 7 time series lengths (April, April-May, April-June, April-July, April-August, April-September and April-October), used MODIS EVI time series to acquire training samples for each time series length, and then identified crop types using the corresponding training samples and Landsat NDVI time series. Several metrics derived from the confusion matrix, such as overall accuracy, Kappa coefficient, were used to evaluate the classification performance. Results showed that when only time series data in April were used, we acquired 5 088 samples, and 91.86% among these samples had the same crop label with the CDL data. When longer time series data were used, more training samples in 2014 were acquired with higher accuracy. When entire EVI time series data were applied, 10 803 samples were acquired and 10 317 samples had same crop label with CDL data. When using these training samples and monthly composted Landsat NDVI to identify crop types at 30 m resolution, classification accuracies were low if April or April-May time series data were used, and overall accuracies were 66.12% and 52.51%, respectively. When time series length was April-October, overall classification was 94.89%. April-August time series achieved good classification performance, as 10 183 training samples were acquired, 96.32% samples had same label to CDL data, overall classification accuracy was 94.02%, and acreage of major crops was similar to CDL data. Finally, we could conclude: (1) The method proposed in this study can acquire train samples in the classification year when the ground reference data are absent. Using these training samples, we can obtain crop type distribution maps with high accuracy (better than 90%). (2) We can acquire the crop type map of the study area in August with the high classification accuracy which is similar to the result derived from the entire EVI time series, and has the similar crop acreage with CDL data for each crop. In the future, we can enhance this method by improving the previous-year training samples with CDL crop confidence layer.
crops; remote sensing; recognition; reference EVI time series; crop type classification; training samples; artificial annual network; cropland data layer (CDL)
郝鵬宇,唐華俊,陳仲新,牛 錚. 基于歷史增強型植被指數時序的農作物類型早期識別[J]. 農業工程學報,2018,34(13):179-186.doi:10.11975/j.issn.1002-6819.2018.13.021 http://www.tcsae.org
Hao Pengyu, Tang Huajun, Chen Zhongxin, Niu Zheng. Early season crop type recognition based on historical EVI time series[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2018, 34(13): 179-186. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2018.13.021 http://www.tcsae.org
2018-03-05
2018-06-04
博士后創新人才支持計劃(編號:2017BX00286);農業部“948”計劃項目(2016-X38)
郝鵬宇,男,博士,現從事農作物遙感制圖研究。Email:haopy8296@163.com
唐華俊,男,研究員,中國工程院院士,研究方向為農業遙感、全球變化與土地科學的理論和應用。Email:tanghuajun@caas.cn
10.11975/j.issn.1002-6819.2018.13.021
TP701
A
1002-6819(2018)-13-0179-08

