梅花鹿茸生長(zhǎng)中心軟骨組織差異基因表達(dá)分析
劉美欣 趙 雨 張 梅 劉宇昕 胡耀中 幺寶金*
(1.長(zhǎng)春中醫(yī)藥大學(xué)中醫(yī)藥與生物工程研究開(kāi)發(fā)中心,長(zhǎng)春,130117;2.長(zhǎng)春中醫(yī)藥大學(xué)創(chuàng)新實(shí)踐中心,長(zhǎng)春,130117)
鹿茸為鹿科動(dòng)物未骨化密生茸毛的幼角,鹿茸驚人的生長(zhǎng)速度一直是本領(lǐng)域研究的熱點(diǎn),在快速生長(zhǎng)期(生茸后 60 d),鹿茸的生長(zhǎng)速度可達(dá) 2 cm/d[1-4]。鹿茸的生長(zhǎng)主要由鹿茸頂端生長(zhǎng)中心驅(qū)動(dòng)和控制,通過(guò)軟骨內(nèi)骨化過(guò)程實(shí)現(xiàn),生長(zhǎng)中心主要包括茸皮、間充質(zhì)和軟骨組織[5-7]。因此,鹿茸可以作為研究軟骨生長(zhǎng)和發(fā)育機(jī)制的良好模型。鹿茸生長(zhǎng)中心軟骨組織中富含大量血管組織,與正常的軟骨組織存在巨大差異,這與鹿茸具有驚人的生長(zhǎng)速度并且可以修復(fù)再生必然存在聯(lián)系[8-10]。因此,研究不同生長(zhǎng)時(shí)期鹿茸軟骨組織基因表達(dá)變化,對(duì)于揭示鹿茸快速生長(zhǎng)機(jī)制具有重要意義。同時(shí),對(duì)于軟骨組織損傷修復(fù)和骨關(guān)節(jié)退行性疾病的相關(guān)研究具有重要的指導(dǎo)作用。
近年來(lái),隨著高通量測(cè)序技術(shù)的不斷發(fā)展,以轉(zhuǎn)錄組學(xué)為代表的組學(xué)研究在基因表達(dá)分析研究中發(fā)揮重要作用。轉(zhuǎn)錄組指特定組織或細(xì)胞在某個(gè)發(fā)育階段或某種特定生理病理?xiàng)l件下,轉(zhuǎn)錄出來(lái)的全部RNA的總和。轉(zhuǎn)錄組測(cè)序(RNA-seq)是指利用高通量測(cè)序技術(shù)對(duì)RNA進(jìn)行測(cè)序,通過(guò)生物信息學(xué)分析方法,研究特定組織或細(xì)胞的全部轉(zhuǎn)錄本情況,揭示其基因表達(dá)水平與變化,進(jìn)而揭示其具體分子作用機(jī)制。與傳統(tǒng)測(cè)序方法如Sanger法測(cè)序、基因芯片等相比較,具有樣品用量少、分辨率和準(zhǔn)確度高、價(jià)格低和應(yīng)用范圍廣等優(yōu)勢(shì)[11-15]。同時(shí),可以實(shí)現(xiàn)對(duì)無(wú)參考基因組物種進(jìn)行從頭測(cè)序[16-19]。本研究采用 RNA-seq技術(shù)對(duì)快速生長(zhǎng)期和骨化期鹿茸生長(zhǎng)中心軟骨組織進(jìn)行轉(zhuǎn)錄組測(cè)序,通過(guò)Trinity軟件[20]對(duì)測(cè)序結(jié)果進(jìn)行從頭組裝和拼接,通過(guò)生物信息學(xué)分析方法篩選不同生長(zhǎng)時(shí)期鹿茸軟骨組織差異表達(dá)基因,這些研究結(jié)果對(duì)于揭示鹿茸快速生長(zhǎng)機(jī)制具有重要意義。
1 材料與方法
1.1 材料
隨機(jī)選取4歲齡健康的東北梅花鹿(Cervus nippon hortulorum)6只,分別于生茸后60 d(快速生長(zhǎng)期)和90 d(骨化期)時(shí)采集兩側(cè)的鹿茸頂端生長(zhǎng)組織(距離頂端長(zhǎng)度5 cm)。按照Li等[7]的實(shí)驗(yàn)方法分離鹿茸頂端軟骨組織,置于純化水中反復(fù)漂洗后切成小塊,迅速置于液氮罐中保存。
1.2 方法
1.2.1 鹿茸軟骨組織總RNA提取制備
采用TRIzol(Invitrogen,USA)法從鹿茸生長(zhǎng)中心軟骨組織中提取總RNA,通過(guò)Bioanalyzer 2100系統(tǒng)(Agilent Technologies,USA)來(lái)評(píng)估RNA完整性。
1.2.2 測(cè)序文庫(kù)構(gòu)建
采用 TruSeq Stranded mRNA試劑盒(Illumina,USA)進(jìn)行測(cè)序文庫(kù)構(gòu)建。具體方法為:采用oligo(dT)偶聯(lián)磁珠(Life technologies,USA)從總RNA中純化富集mRNA,并進(jìn)行mRNA片段化;經(jīng)逆轉(zhuǎn)錄酶和隨機(jī)引物合成雙鏈cDNA,經(jīng)核糖核酸酶處理除去剩余mRNA;經(jīng)末端修復(fù)和接頭連接后,通過(guò)PCR擴(kuò)增獲得最終的測(cè)序文庫(kù)。
1.2.3 文庫(kù)測(cè)序和生物信息學(xué)分析
在Illumina Hiseq 2000平臺(tái)(Illumina,USA)上進(jìn)行文庫(kù)測(cè)序。采用Illumina HCS 1.1軟件進(jìn)行數(shù)據(jù)轉(zhuǎn)換,去除低質(zhì)量序列和載體序列。采用Trinity軟件進(jìn)行從頭組裝。通過(guò)BLASTX比對(duì)程序,以E值<10-5為評(píng)價(jià)標(biāo)準(zhǔn),將測(cè)序組裝數(shù)據(jù)與nr、Swiss-Prot蛋白數(shù)據(jù)庫(kù)進(jìn)行比對(duì)。采用RPKM法[21]進(jìn)行基因表達(dá)量分析,采用DEGseq軟件包[22]對(duì)快速生長(zhǎng)期和骨化期軟骨組織進(jìn)行差異基因表達(dá)分析,評(píng)價(jià)標(biāo)準(zhǔn)為:差異表達(dá)倍數(shù)在2倍以上(即log2[Fold change]≥1或≤-1,F(xiàn)old change=骨化期 RPKM/快速生長(zhǎng)期RPKM),且假發(fā)現(xiàn)率(FDR)≤0.001。
2 結(jié)果
2.1 測(cè)序和組裝
通過(guò)轉(zhuǎn)錄組測(cè)序,從快速生長(zhǎng)期和骨化期鹿茸軟骨組織中分別獲得4.47和4.45 Gb的測(cè)序數(shù)據(jù)。其中,raw reads分別為45423156和45422448條,去除載體序列和低質(zhì)量序列后,分別獲得44715694和44535200條clean reads,測(cè)序質(zhì)量值Q20值分別為98.44和98.05。通過(guò) Trinity軟件組裝,分別獲得39377和37331條組裝序列(Unigenes),平均長(zhǎng)度分別為1138和1143 nt。Unigenes長(zhǎng)度分布見(jiàn)圖1。
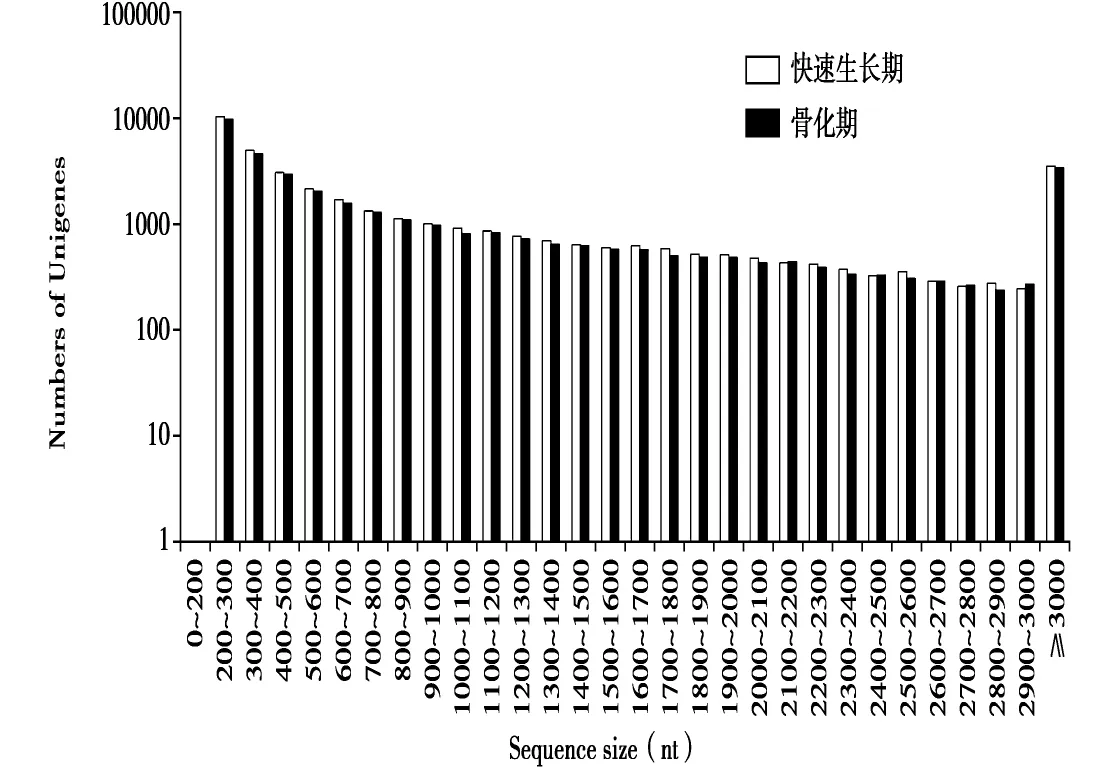
圖1 快速生長(zhǎng)期和骨化期鹿茸軟骨組織Unigenes長(zhǎng)度分布Fig.1 Unigenes length distribution of antler cartilage tissue at rapid growth and ossification stages
2.2 蛋白數(shù)據(jù)庫(kù)比對(duì)及差異基因表達(dá)分析
通過(guò)BLASTX比對(duì)程序,將Unigenes與nr、Swiss-Prot蛋白數(shù)據(jù)庫(kù)進(jìn)行比對(duì),以E值<10-5為評(píng)價(jià)標(biāo)準(zhǔn),同時(shí)與2個(gè)數(shù)據(jù)庫(kù)比對(duì)上的Unigenes共有20551條。針對(duì)這些Unigenes,采用RPKM法進(jìn)行基因表達(dá)量分析,采用DEG-seq軟件包進(jìn)行差異基因表達(dá)分析,共篩選出4422條差異表達(dá)基因(log2[Fold change]≥1或≤-1;FDR≤0.001)。與快速生長(zhǎng)期比較,骨化期顯著上調(diào)的差異表達(dá)基因有2493條,顯著下調(diào)的差異表達(dá)基因有1929條。我們針對(duì)這些差異表達(dá)基因進(jìn)行進(jìn)一步的篩選,找出與軟骨生長(zhǎng)和骨化相關(guān)的差異表達(dá)基因,這些基因主要包括生長(zhǎng)因子、轉(zhuǎn)錄因子和膠原類成分。
2.2.1 生長(zhǎng)因子(growth factor)類差異表達(dá)基因
在快速生長(zhǎng)期和骨化期鹿茸軟骨組織差異表達(dá)基因中,共篩選出7種生長(zhǎng)因子(表1)。其中,與快速生長(zhǎng)期比較,骨化期鹿茸軟骨組織中表達(dá)量顯著下調(diào)的生長(zhǎng)因子有6種,分別為血管內(nèi)皮生長(zhǎng)因子VEGFD、表皮生長(zhǎng)因子樣蛋白EGFL6、胰島素樣生長(zhǎng)因子IGF1、肝細(xì)胞生長(zhǎng)因子HGF、成纖維細(xì)胞生長(zhǎng)因子FGF7和FGF11;血管內(nèi)皮生長(zhǎng)因子VEGF-C表達(dá)量顯著上調(diào)。
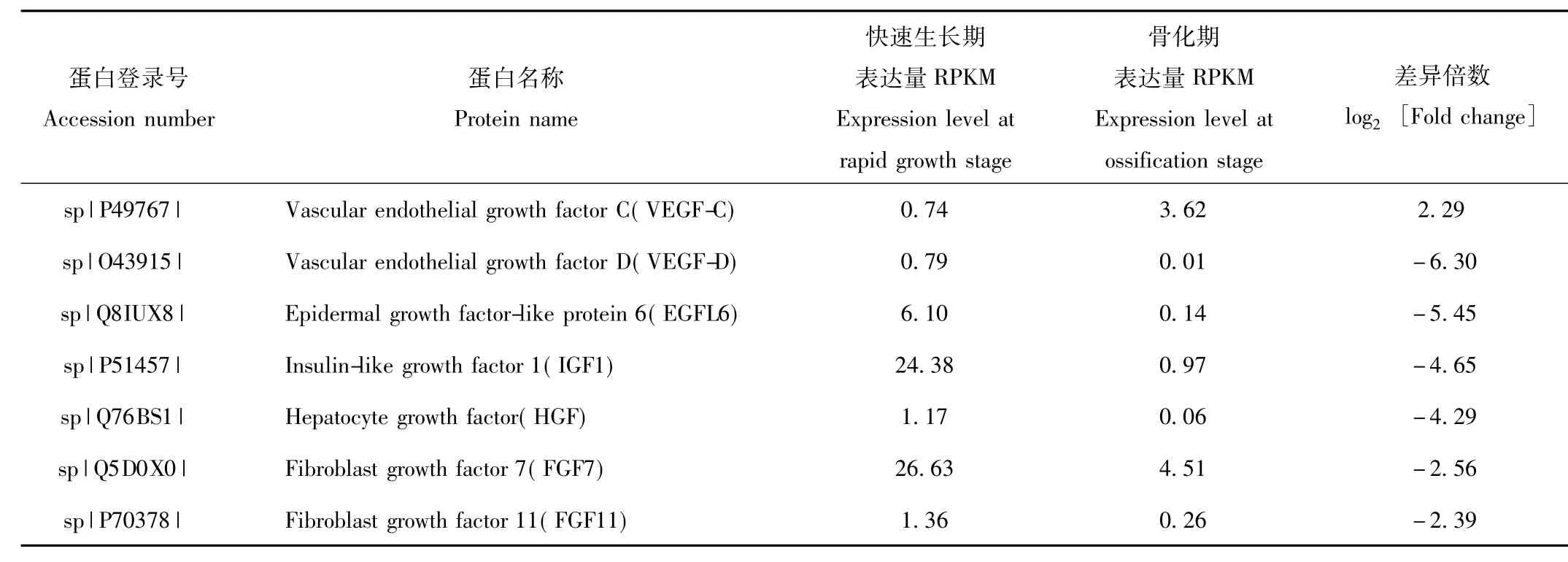
表1 不同生長(zhǎng)時(shí)期鹿茸生長(zhǎng)中心軟骨組織生長(zhǎng)因子類差異表達(dá)基因Tab.1 Differential growth factors of antler growth center cartilage at different growth stages
2.2.2 轉(zhuǎn)錄因子(transcription factor)類差異表達(dá)基因
在快速生長(zhǎng)期和骨化期鹿茸軟骨組織差異表達(dá)基因中,共篩選出12種轉(zhuǎn)錄因子(表2)。其中,與快速生長(zhǎng)期比較,骨化期鹿茸軟骨組織中表達(dá)量顯著下調(diào)的轉(zhuǎn)錄因子有 7種,分別為轉(zhuǎn)錄因子 ATF6β、SOX12、ELF2、ELF4、RUNX1、MAFK和 TCF4;表達(dá)量顯著上調(diào)的轉(zhuǎn)錄因子有5種,分別為轉(zhuǎn)錄因子SOX9、GTF3A、SOX8、ATF3和 MAFF。

表2 不同生長(zhǎng)時(shí)期鹿茸生長(zhǎng)中心軟骨組織轉(zhuǎn)錄因子類差異表達(dá)基因Tab.2 Differential transcription factors of antler growth center cartilage at different growth stages
2.2.3 膠原(collagen)類差異表達(dá)基因
在快速生長(zhǎng)期和骨化期鹿茸軟骨組織差異表達(dá)基因中,共篩選出8種類型膠原(表3)。其中,與快速生長(zhǎng)期比較,骨化期鹿茸軟骨組織中表達(dá)量顯著下調(diào)的膠原有3種,分別為膠原COL16A1、COL8A1和COL14A1;表達(dá)量顯著上調(diào)的膠原有5種,分別為膠原 COL10A1、COL27A1、COL9A1、COL2A1 和COL25A1。
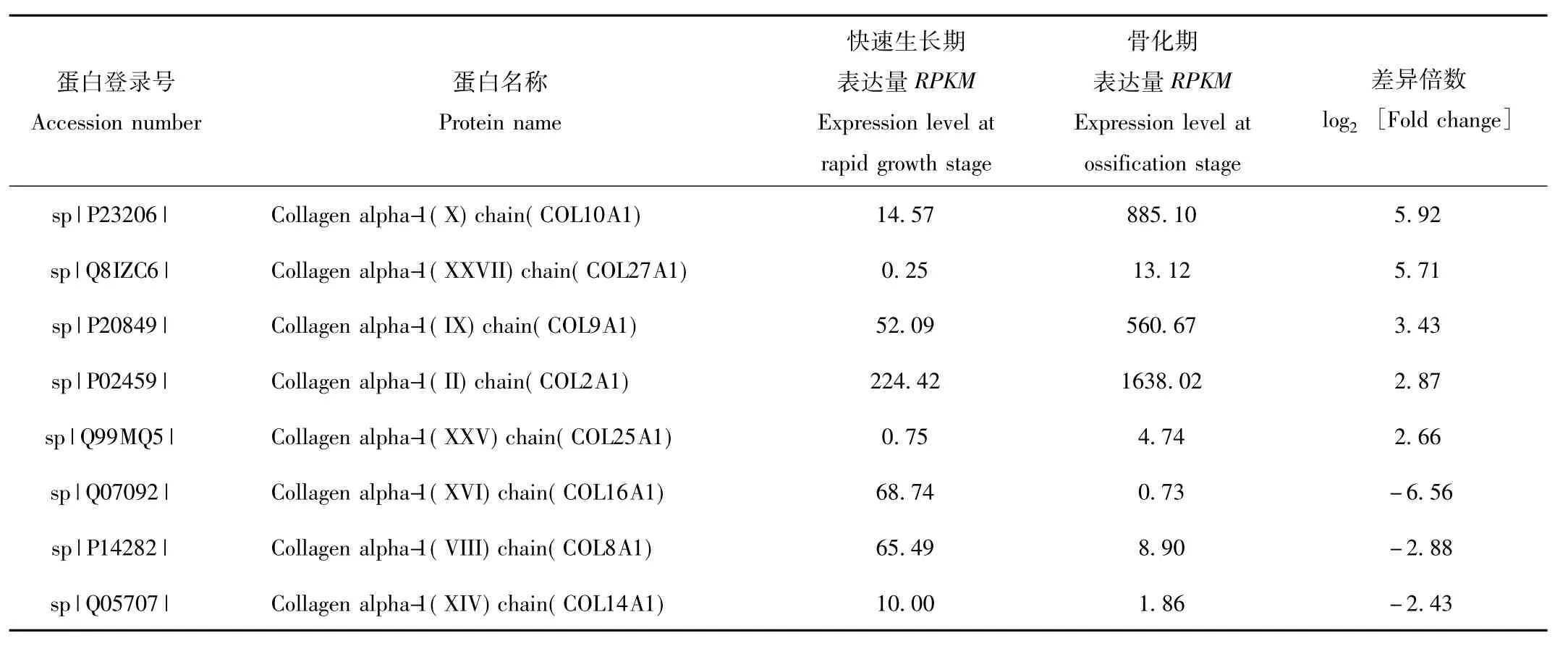
表3 不同生長(zhǎng)時(shí)期鹿茸生長(zhǎng)中心軟骨組織膠原類差異表達(dá)基因Tab.3 Differential collagens of antler growth center cartilage at different growth stages
3 討論
鹿茸的快速生長(zhǎng)機(jī)制一直是本領(lǐng)域的研究熱點(diǎn)。要系統(tǒng)和全面地揭示這一獨(dú)特的生長(zhǎng)機(jī)制,就必須對(duì)鹿茸生長(zhǎng)過(guò)程中的基因表達(dá)變化進(jìn)行深入分析。基于Illumina HiSeqTM2000高通量測(cè)序平臺(tái)的轉(zhuǎn)錄組測(cè)序技術(shù)具有快速、高效、低成本等特點(diǎn),可以對(duì)任意物種的組織或細(xì)胞的全轉(zhuǎn)錄本進(jìn)行檢測(cè),提供全面的轉(zhuǎn)錄組信息,尤其適合于研究缺少基因組信息的非模式生物的轉(zhuǎn)錄組情況。本課題組在前期研究工作中針對(duì)快速生長(zhǎng)期和骨化期鹿茸頂端生長(zhǎng)中心的轉(zhuǎn)錄組進(jìn)行較為深入的分析,篩選出多種調(diào)控鹿茸快速生長(zhǎng)和骨化的差異表達(dá)基因[23-25]。然而,這些研究結(jié)果無(wú)法反映鹿茸生長(zhǎng)過(guò)程中生長(zhǎng)中心的軟骨組織的基因表達(dá)變化。本研究針對(duì)快速生長(zhǎng)期和骨化期鹿茸生長(zhǎng)中心軟骨組織轉(zhuǎn)錄組進(jìn)行深度測(cè)序,采用生物信息學(xué)方法對(duì)測(cè)序結(jié)果進(jìn)行組裝、拼接、比對(duì)和差異基因表達(dá)分析,篩選出7種差異表達(dá)生長(zhǎng)因子,12種差異表達(dá)轉(zhuǎn)錄因子和8種差異表達(dá)膠原。
在快速生長(zhǎng)期和骨化期鹿茸轉(zhuǎn)錄組差異表達(dá)生長(zhǎng)因子中,血管內(nèi)皮生長(zhǎng)因子VEGF-D、表皮生長(zhǎng)因子樣蛋白EGFL6、胰島素樣生長(zhǎng)因子IGF1、肝細(xì)胞生長(zhǎng)因子HGF、成纖維細(xì)胞生長(zhǎng)因子FGF7和FGF11在快速生長(zhǎng)期表達(dá)量較高,在骨化期表達(dá)量顯著下調(diào),而血管內(nèi)皮生長(zhǎng)因子VEGF-C在快速生長(zhǎng)期表達(dá)量較低,在骨化期表達(dá)量顯著上調(diào)。這些研究結(jié)果表明鹿茸的快速生長(zhǎng)受到這些生長(zhǎng)因子的協(xié)同調(diào)控作用。VEGF-D和VEGF-C是2種血管內(nèi)皮細(xì)胞因子亞型,具有促進(jìn)血管和淋巴管形成等功能[26]。表皮生長(zhǎng)因子樣蛋白EGFL6的功能與血管內(nèi)皮生長(zhǎng)因子相似,具有促血管生成作用,尤其在腫瘤組織中表達(dá)量較高[27]。這些研究結(jié)果提示我們這3種生長(zhǎng)因子對(duì)鹿茸生長(zhǎng)過(guò)程中軟骨組織中血管的生成具有重要的調(diào)控作用。胰島素樣生長(zhǎng)因子IGF1是一種多功能生長(zhǎng)因子,對(duì)于細(xì)胞的存活、增殖和分化具有重要調(diào)節(jié)作用。IGF1軟骨特異性基因敲除小鼠出現(xiàn)軟骨細(xì)胞增殖減少,分化和肥大延遲,細(xì)胞凋亡增加,軟骨內(nèi)骨化進(jìn)程遲緩等現(xiàn)象[28]。肝細(xì)胞生長(zhǎng)因子HGF、成纖維細(xì)胞生長(zhǎng)因子FGF7和FGF11三種生長(zhǎng)因子在組織損傷修復(fù)和再生過(guò)程中起著關(guān)鍵的調(diào)節(jié)作用[29-31]。這些研究結(jié)果表明鹿茸快速生長(zhǎng)受到 IGF1、HGF、VEGF和EGF6等多種生長(zhǎng)因子的協(xié)同調(diào)控。
在快速生長(zhǎng)期和骨化期鹿茸轉(zhuǎn)錄組差異表達(dá)轉(zhuǎn)錄因子中,SOX9、GTF3A、SOX8、ATF3和MAFF的表達(dá)量在骨化期表達(dá)量顯著上調(diào),而ATF6β、SOX12、ELF2、ELF4、RUNX1、MAFK和TCF4的表達(dá)量在骨化期表達(dá)量顯著下調(diào)。這些轉(zhuǎn)錄因子主要集中為SOX家族、ATF家族和ELF家族。其中,SOX家族成員包括SOX8、SOX9和SOX12,這3種轉(zhuǎn)錄因子在胚胎發(fā)育、神經(jīng)系統(tǒng)發(fā)育、軟骨及多種組織器官形成和發(fā)育過(guò)程中發(fā)揮重要作用[32]。ATF家族成員包括 ATF3、ATF6β,這2個(gè)轉(zhuǎn)錄因子對(duì)于應(yīng)激條件下軟骨細(xì)胞存活和凋亡具有重要調(diào)控作用[33]。除此之外,RUNX1也是軟骨生長(zhǎng)過(guò)程中重要的調(diào)控因子[34]。這些研究結(jié)果表明鹿茸快速生長(zhǎng)受到SOX8、SOX9、SOX12、ATF3、ATF6β和RUNX1等多種轉(zhuǎn)錄因子的協(xié)同調(diào)控。
在不同生長(zhǎng)時(shí)期鹿茸軟骨組織轉(zhuǎn)錄組數(shù)據(jù)中,我們也發(fā)現(xiàn)了許多表達(dá)量存在顯著差異的膠原類成分,與快速生長(zhǎng)期比較,骨化期鹿茸軟骨組織中表達(dá)量顯著下調(diào)的膠原有 3種,分別為膠原 COL16A1、COL8A1和COL14A1;表達(dá)量顯著上調(diào)的膠原有5種,分別為膠原 COL10A1、COL27A1、COL9A1、COL2A1和 COL25A1。其中,COL10A1、COL9A1和COL2A1廣泛存在于軟骨組織中,是重要的軟骨細(xì)胞外基質(zhì)成分,在軟骨形成和生長(zhǎng)發(fā)育過(guò)程中發(fā)揮重要作用[35]。這些研究結(jié)果表明鹿茸快速生長(zhǎng)和骨化過(guò)程中膠原表達(dá)量發(fā)生巨大改變,有關(guān)這些膠原基因表達(dá)調(diào)控機(jī)制仍然有待于進(jìn)一步研究。
4 結(jié)論
本研究采用Illumina高通量測(cè)序技術(shù),對(duì)快速生長(zhǎng)期和骨化期東北梅花鹿茸生長(zhǎng)中心軟骨組織進(jìn)行轉(zhuǎn)錄組測(cè)序和生物信息學(xué)分析。通過(guò)差異基因表達(dá)分析篩選出7種差異表達(dá)生長(zhǎng)因子,12種差異表達(dá)轉(zhuǎn)錄因子和8種差異表達(dá)膠原。這些差異表達(dá)基因?qū)τ诼谷椎目焖偕L(zhǎng)和骨化具有重要的調(diào)節(jié)作用,本研究結(jié)果為鹿茸快速生長(zhǎng)及骨化機(jī)制研究提供理論基礎(chǔ)。
[1] Chu W,Zhao H,Li J,et al.Custom-built tools for the study of deer antler biology[J].Frontiers in Bioscience(Landmark Ed),2017,22:1622-1633.
[2] Sui Zhigang,Weng Yejing,Zhao Qun,et al.Ionic liquid-based method for direct proteome characterization of velvet antler cartilage[J].Talanta,2016,161:541-546.
[3] Park J,Jeon B,Kang S,et al.Study on the changes in enzyme and insulin-like growth factor-1 concentrations in blood serum and growth characteristics of velvet antler during the antler growth period in sika deer(Cervus nippon) [J].Asian-Australasian Journal of Animal Sciences,2015,28(9):1303 -1308.
[4] Hu Wei,Meng Xingyu,Lu Tiancheng,et al.MicroRNA-1 inhibits the proliferation of Chinese sika deer-derived cartilage cells by binding to the 3'-untranslated region of IGF-1[J].Molecular Medicine Reports,2013,8(2):523 -528.
[5] Price J S,Allen S,F(xiàn)aucheux C,et al.Deer antlers:a zoological curiosity or the key to understanding organ regeneration in mammals[J].Journal of Anatomy,2005,207(5):603 -618.
[6] Price J,Allen S.Exploring the mechanisms regulating regeneration of deer antlers[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2004,359(1445):809 -822.
[7] Li C,Clark D E,Lord E A,et al.Sampling technique to discriminate the different tissue layers of growing antler tips for gene discovery[J].The Anatomical Record,2002,268(2):125-130.
[8] Clark D E,Li C,Wang W,et al.Vascular localization and proliferation in the growing tip of the deer antler[J].The Anatomical Record,2006,288(9):973-981.
[9] Clark D E,Lord E A,Suttie J M.Expression of VEGF and pleiotrophin in deer antler[J].The Anatomical Record,2006,288A(12):1281-1293.
[10] Lai A K W,Hou W L,Verdon D J,et al.The distribution of the growth factors FGF-2 and VEGF,and their receptors,in growing red deer antler[J].Tissue and Cell,2007,39(1):35 -46.
[11] Hrdlickova R,Toloue M,Tian B.RNA-Seq methods for transcriptome analysis[J].Wiley Interdisciplinary Reviews:RNA,2017,8(1):e 1364.
[12] Kukurba K R,Montgomery S B.RNA sequencing and analysis[J].Cold Spring Harbor Protocols,2015(11):951 -969.
[13] Han Y,Gao S,Muegge K,et al.Advanced applications of RNA sequencing and challenges[J].Bioinformatics and Biology Insights,2015,9(S 1):29 -46.
[14] Hoeijmakers W A M,Bártfai R,Stunnenberg H G.Transcriptome analysis using RNA-Seq[M].Malaria Humana Press,2012:221-239.
[15] Mutz K O,Heilkenbrinker A,Lonne M,et al.Transcriptome analysis using next-generation sequencing[J].Current Opinion in Biotechnology,2013,24(1):22 -30.
[16] Lee J H.De Novo gene expression Reconstruction in space[J].Trends in Molecular Medicine,2017,23(7):583-593.
[17] Moreton J,Izquierdo A,Emes R D.Assembly,assessment,and availability of de novo generated eukaryotic aranscriptomes[J].Frontiers in Genetics,2016,6:361.
[18] Unamba C I N,Nag A,Sharma R K.Next generation sequencing technologies:the doorway to the unexplored genomics of non-model plants[J].Frontiers in Plant Science,2015,6:1074.
[19] Góngora-Castillo E,Buell C R.Bioinformatics challenges in de novo transcriptome assembly using short read sequences in the absence of a reference genome sequence [J].Natural Product Reports,2013,30(4):490-500.
[20] Grabherr M G,Haas B J,Yassour M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome [J].Nature Biotechnology,2011,29(7):644-652.
[21] Mortazavi A,Williams B A,Mccue K,et al.Mapping and quantifying mammalian transcriptomes by RNA-Seq [J].Nature Methods,2008,5(7):621-628.
[22] Wang Likun,F(xiàn)eng Zhixing,Wang Xi,et al.DEGseq:an R package for identifying differentially expressed genes from RNA-seq data[J].Bioinformatics,2010,26(1):136-138.
[23] Zhao Yu,Yao Baojin,Zhang Mei,et al.Comparative analysis of differentially expressed genes in sika deer antler at different stages[J].Molecular Biology Reports,2013,40(2):1665 -1676.
[24] Yao Baojin,Zhao Yu,Wang Qun,et al.De novo characterization of the antler tip of Chinese sika deer transcriptome and analysis of gene expression related to rapid growth[J].Molecular and Cellular Biochemistry,2012,364(1/2):93 -100.
[25] Yao Baojin,Zhao Yu,Zhang Haishan,et al.Sequencing and de novo analysis of the Chinese sika deer antler-tip transcriptome during the ossification stage using Illumina RNA-Seq technology[J].Biotechnology Letters,2012,34(5):813 -822.
[26] Hu K,Olsen B R.The roles of vascular endothelial growth factor in bone repair and regeneration[J].Bone,2016,91:30-38.
[27] Wang Xuanchun,Gong Ye,Wang Daijun,et al.Analysis of gene expression profiling in meningioma:deregulated signaling pathways associated with meningioma and EGFL6 overexpression in benign meningioma tissue and serum [J].PLoS One,2012,7(12):e52707.
[28] Wang Yongmei,Bikle D D,Chang Wenhan.Autocrine and paracrine actions of IGF-I signaling in skeletal development[J].Bone Research,2013,1(3):249 -259.
[29] Miyagi H,Thomasy S M,Russell P,et al.The role of hepatocyte growth factor in corneal wound healing[J].Experimental Eye Research,2018,166:49-55.
[30] Seo H S,Lee D J,Chung J H,et al.Hominis placenta facilitates hair re-growth by upregulating cellular proliferation and expression of fibroblast growth factor-7[J].BMC Complementary and Alternative Medicine,2016,16(1):187.
[31] Yang J,Kim W J,Jun H O,et al.Hypoxia-induced fibroblast growth factor 11 stimulates capillary-like endothelial tube formation[J].Oncology Reports,2015,34(5):2745 -2751.
[32] Kamachi Y,Kondoh H.Sox proteins:regulators of cell fate specification and differentiation [J].Development,2013,140(20):4129-4144.
[33] Yuan Xiaoliang,Liu Haiqing,Li Linfu,et al.The roles of endoplasmic reticulum stress in the pathophysiological development of cartilage and chondrocytes [J].CurrentPharmaceuticalDesign,2017,23(11):1693-1704.
[34] LeBlanc K T,Walcott M E,Gaur T A,et al.Runx1 activities in superficial zone chondrocytes,osteoarthritic chondrocyte clones and response to mechanical loading[J].Journal of Cellular Physiology,2015,230(2):440-448.
[35] Stocco E,Barbon S,Radossi P,et al.Autologous chondrocytes as a novel source for neo-chondrogenesis in haemophiliacs[J].Cell and Tissue Research,2016,366(1):51-61.

