Bacillus mucilaginosus莢膜多糖脫除與全蛋白雙向電泳體系的建立
閆玉潔, 李 會, 王向棟, 馬 洋, 史勁松, 許正宏
(江南大學 藥學院,江蘇 無錫 214122)
微生物多糖是某些微生物生長代謝過程中,在特殊的營養條件下,合成的對細胞具有保護作用的生物聚合物[1-2]。
膠質芽孢桿菌于20世紀初分離得到,它可分解云母、長石等硅酸鹽礦石而釋放出磷、鉀元素,且在生長過程中能夠產生豐富的胞外多糖。
目前,對于多糖BMPS過量合成的研究工作僅限于常規的發酵條件優化[3-4],導致其生產效率偏低、生產成本偏高,限制了該多糖的大規模工業應用。隨著蛋白質組學技術的迅猛發展,作者希望通過蛋白組學的方式,分析不同條件下的差異蛋白,找出影響多糖合成的關鍵酶,并對其在分子水平和蛋白質水平上進行代謝調控,引導物質代謝流量朝著目標產物多糖的合成方向偏移,從而實現多糖的過量合成。然而由于蛋白質組自身的復雜性和不穩定性,至今尚未得到通用、簡便、高效的技術支持[5],目前雙向電泳技術結合質譜鑒定技術是差異蛋白質組學最常用的方法之一。作者研究了夾膜多糖的脫除,對菌體總蛋白質的提取方法進行改進,并優化雙向電泳的條件,建立一套高分辨率、高靈敏度的雙向電泳技術體系。
1 材料與方法
1.1 實驗材料
1.1.1 菌株與培養條件 菌株:膠質芽孢桿菌SM-01,江南大學藥學院制藥工程實驗室分離并保藏。
培養條件:從固體培養基上刮取適量菌體接入到50 mL的種子培養基中,置于30℃,200 r/min的往復式搖床中培養24 h;再將種子液按照體積分數4%接種量接入到發酵培養基中,置于30℃,200 r/min的往復搖床中發酵培養24 h。
1.1.2 試劑 IPG線性預制干膠條、兩性電解質等:購自美國Bio-Rad公司;尿素、硫脲、二硫蘇糖醇(DTT)、碘乙酰胺(IAA)、甘氨酸、三羥甲基氨基甲烷(Tris)、四甲基乙二胺(TEMED)、過硫酸銨、溴酚藍、低熔點瓊脂糖、十二烷基磺酸鈉(SDS)、pH7.5 Tris飽和酚、考馬斯亮藍G-250、蛋白測定試劑盒等:購自上海生工集團;蔗糖、甲醇、丙酮、冰醋酸、三氯乙酸(TCA)等:均為國產分析純產品。
1.1.3 主要儀器 Ettan IPG-phor 3型等電聚焦電泳儀、Ettan DALTsix垂直電泳系統、Image MasterLabScan凝膠電泳圖像分析系統:GE公司產品;CR22GH型高速冷凍離心機:日本日立公司產品;PDQuest 8.0.1分析軟件:美國Bio-Rad公司產品。
1.2 實驗方法
1.2.1 多糖去除與菌體收集
1)超純水洗滌法 取100 mL發酵液,在4℃、12 000 r/min條件下離心10 min,棄去上清。用50mL超純水懸浮洗滌菌體,在4℃、12 000 r/min條件下離心10 min,重復3次得菌體沉淀。
2)高濃度鹽酸單次處理法 取100 mL發酵液,加入等體積的體積分數50%鹽酸,攪拌1 h,在4℃、12 000 r/min條件下離心10 min,棄去上清。超純水懸浮洗滌菌體3次得菌體沉淀。
3)低濃度鹽酸多次處理法 取100 mL發酵液,加入等體積的體積分數30%鹽酸,攪拌1 h,在4℃、12 000 r/min條件下離心10 min,棄去上清。沉淀中加入50 mL體積分數10%鹽酸,攪拌30 min,離心,重復一次,超純水懸浮洗滌菌體3次得菌體沉淀。
1.2.2 蛋白質提取
1)TCA-丙酮法 將收集到的菌體置于陶瓷研缽中,加入液氮研磨成粉,將粉末轉移到50 mL離心管中。加入TCA-丙酮溶液30 mL,-20℃放置過夜,用于沉淀蛋白質。在4℃、14 000 r/min條件下離心30 min。用預冷的丙酮溶液(含1 g/L的DTT)懸浮洗滌沉淀,-20℃靜置1 h,在4℃、14 000 r/min條件下離心30 min,重復2次,得蛋白質沉淀。
2)酚抽提法 根據Rocco[6]的方法并加以改進,將收集到的菌體用液氮在研缽中研磨成粉,加入5 mL酚提取緩沖液(0.5 mol/L Tris-HCl pH 7.5,0.7 mol/L蔗糖,0.1 mol/L KCl,0.05 mol/L EDTA),4 ℃放置 10min。加入等體積pH 7.5的Tris飽和酚,渦旋震蕩混勻。在4℃、14 000 r/min條件下離心30 min,收集上層酚相,加入與酚相體積相同的提取緩沖液,重復上述提取步驟2次。沉淀分別用預冷的甲醇和丙酮懸浮洗滌2次,得蛋白質沉淀。
3)TCA-丙酮與酚抽提結合法 將收集到的菌體用液氮在研缽中研磨成粉,加入30 mL TCA-丙酮溶液沉淀蛋白質,-20℃放置過夜,在4℃、14 000 r/min條件下離心30 min,加入5 mL酚提取緩沖液,4℃放置10 min。加入等體積pH 7.5 Tris飽和酚,渦旋震蕩混勻。在4℃、14 000 r/min條件下離心30 min,收集上層酚相,加入與酚相體積相同的提取緩沖液,將上述步驟重復2次。沉淀用預冷的甲醇懸浮洗滌2次,沉淀中加入30 mL TCA-丙酮溶液,-20℃放置過夜,在4℃、14 000 r/min條件下離心30 min。用預冷的丙酮溶液 (含1 mg/mL的DTT)懸浮洗滌沉淀,-20℃靜置1 h,在4℃、14 000 r/min條件下離心30 min,重復2次,得蛋白質沉淀。
蛋白質沉淀中加入500 μL蛋白裂解液。至沉淀完全溶解后,并于在4℃、10 000 r/min條件下離心30 min,用Bradford方法定量蛋白質[7]。
1.2.3 雙向電泳
1)第一向等電聚焦 移取300 μg全蛋白于1.5 mL EP管中,加入上樣緩沖液至總體積為300μL,充分混勻后,緩慢均勻加入聚焦槽。將膠條膠面朝下覆蓋于樣品上,趕出氣泡,加入2~3 mL的礦物油封閉聚焦槽,20℃進行等電聚焦(IEF)。
2)膠條平衡 聚焦完成后,取出膠條,膠面朝上放入平衡盤內,分別用平衡液Ⅰ(6 mol/L尿素,75 mmol pH 8.8 Tris-HCl,體積分數20%甘油,體積分數4%SDS,質量分數2%DTT)和平衡液Ⅱ(6 mol/L尿素,75 mmol pH 8.8 Tris-HCl,體積分數20%甘油,體積分數4%SDS,質量分數2%碘乙酰胺)平衡10 min。
3)第二向SDS-PAGE分離 將膠條從平衡槽中取出,快速浸入 TGS Buffer(Tris 3 g,SDS 1 g,甘氨酸 14.4 g,ddH2O定容至1 L)中,去除表面氣泡。在封有質量分數12%SDS-PAGE的玻璃膠板中加入融化好的覆蓋瓊脂糖,將膠條壓入其中,待覆蓋瓊脂糖凝固,將玻璃膠板放入裝有TGS Buffer的電泳槽。
4)染色 將凝膠從玻璃板中剝離,加入約500 mL超純水清洗1 min。加入適量的染色液(2.5 g/L考馬斯亮藍G-250,體積分數10% 冰醋酸,40%甲醇)置于搖床上染色約4 h,更換脫色液(體積分數10%冰醋酸,40%甲醇),脫色至點清晰,背景不明顯。
5)圖像采集與分析 采用 Image MasterLabScan凝膠電泳圖像分析系統對雙向凝膠電泳圖譜進行掃描,并用PDQuest 8.0.1分析軟件進行圖像分析處理。
2 結果與分析
2.1 不同多糖預處理方法對菌體蛋白提取的影響
由于B.mucilaginosus SM-01能夠合成大量的莢膜多糖,菌體被該多糖緊緊包裹,難以去除,對后續菌體蛋白的提取帶來極大不便。使用超純水洗滌多次仍無法有效洗去菌體外莢膜多糖,且處理后蛋白濃度過低,無法達到實驗要求,見圖1(b)與表1。由于該多糖易溶于酸性溶液中,故采用不同濃度的鹽酸單次或多次處理菌體。圖1(c)和圖1(d)顯示,兩種酸法處理后莢膜多糖能夠有效去除,且細胞完整,不影響后續實驗。表1顯示,低濃度鹽酸多次處理法所得的蛋白濃度更高,且考慮到高濃度鹽酸可能對細胞產生一定的傷害,故后續實驗采用低濃度鹽酸多次處理法。
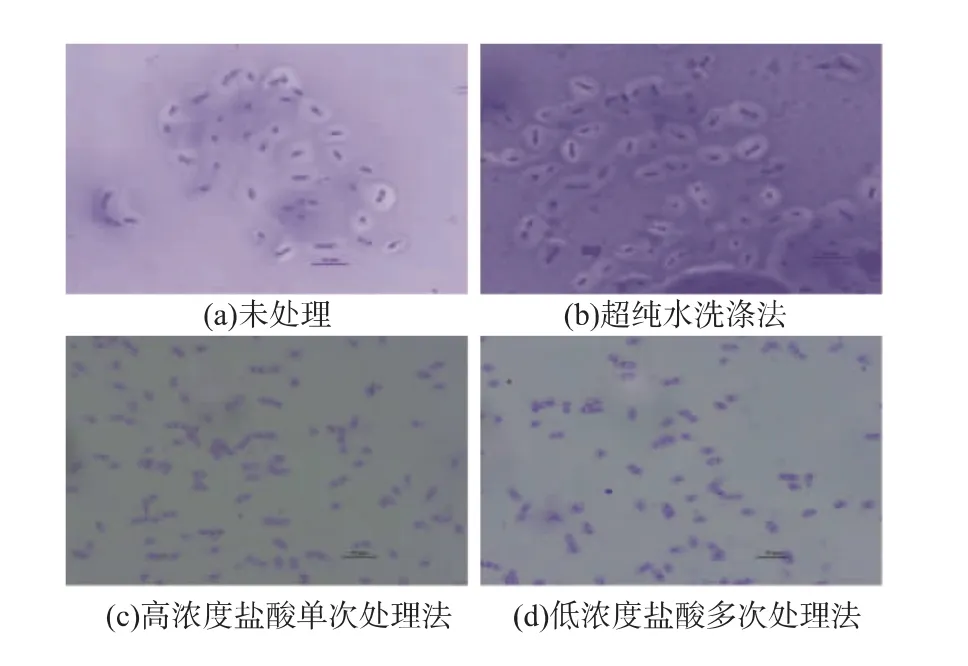
圖1 不同多糖去除方法對細胞的影響Fig.1 Comparison of cells with different BMPS dissolution methods
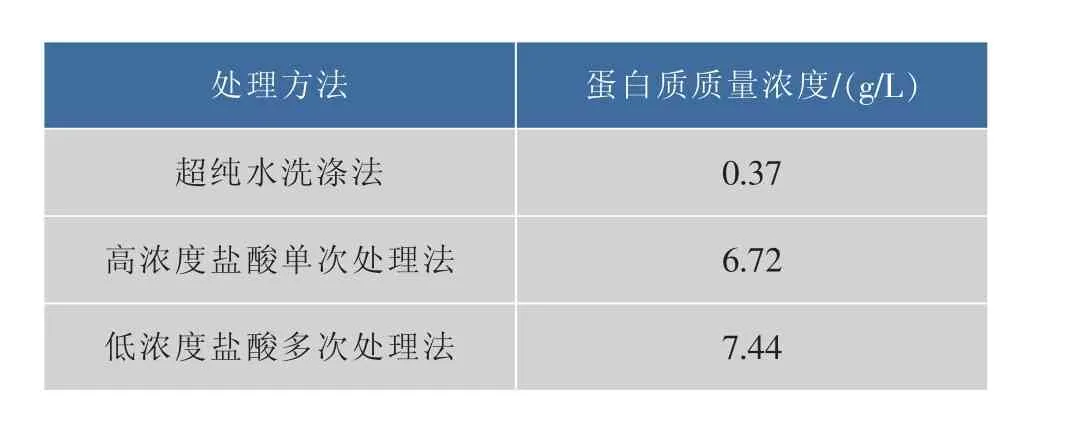
表1 不同多糖去除方法得到的蛋白質質量濃度Table 1 Comparison protein concentration with different BMPS dissolution methods
2.2 不同菌體總蛋白提取方法對雙向電泳圖譜的影響
蛋白質提取方法是雙向電泳成敗的關鍵[8],使用上述方法處理菌體后,仍有部分多糖存在,多糖難以去除且可溶于蛋白裂解液中,使蛋白質提取面臨許多困難。TCA丙酮法是目前微生物全蛋白提取常用的方法[9-10],而酚抽提法則主要應用于植物組織蛋白的提取,有研究表明酚抽提法同樣適用于細菌,甚至效果優于TCA丙酮法。作者比較了TCA丙酮法、酚抽提法和TCA丙酮與酚抽提結合法3種蛋白質提取方法對雙向電泳圖譜的影響,圖2(a)和圖2(b)表明,單獨采用TCA丙酮法和酚抽提法時得到的圖譜不清晰,且蛋白點聚集成團,基本無法分離,分析原因為莢膜多糖沒有完全除去。圖2(c)中,用TCA丙酮與酚抽提結合法得到的蛋白圖譜,蛋白點基本得以分離,圖譜較清晰。以上結果表明,TCA丙酮與酚抽結合法能夠較好的除去多糖等雜質,提取得到的蛋白質純度較高。因此,后續實驗采用TCA丙酮與酚抽結合法作為B.mucilaginosus SM-01菌體蛋白質的提取方法。

圖2 不同蛋白質提取方法的雙向電泳圖譜Fig.2 Comparison of 2-DE maps with different protein extraction methods
2.3 不同pH范圍膠條對雙向電泳圖譜的影響
每個蛋白質都有各自不同的等電點(pI),這決定了蛋白圖譜中蛋白點的橫向分布。因此選擇合適pH范圍的IPG膠條對雙向電泳圖譜具有重要影響,較寬pH范圍的IPG膠條能包含所有的蛋白質,但蛋白點可能聚集,難以有效分離;較窄pH范圍IPG膠條有利于提高圖譜的分辨率,但可能丟失pH值范圍之外的蛋白質。作者首先采用17 cm、pH 3~10的線性IPG預制干膠條,得到的結果如圖3(a)所示,蛋白點較集中,主要分布在pH 4~7附近,膠條沒有得到有效的利用,而且蛋白點出現了堆積現象,分離效果不理想,進一步用PDQuest 8.0.1軟件分析檢測到447個蛋白點。基于上述實驗結果,改用pH范圍為4~7的線性IPG預制干膠條,得到的結果如圖3(b)所示。此時蛋白點的分離效果較圖3(a)顯著提高,很好的解決了蛋白點重疊的問題,用PDQuest 8.0.1軟件可檢測得到616個蛋白點。因此,作者選擇采用pH 4~7的IPG預制干膠條進行雙向電泳的第一向等電聚焦電泳。
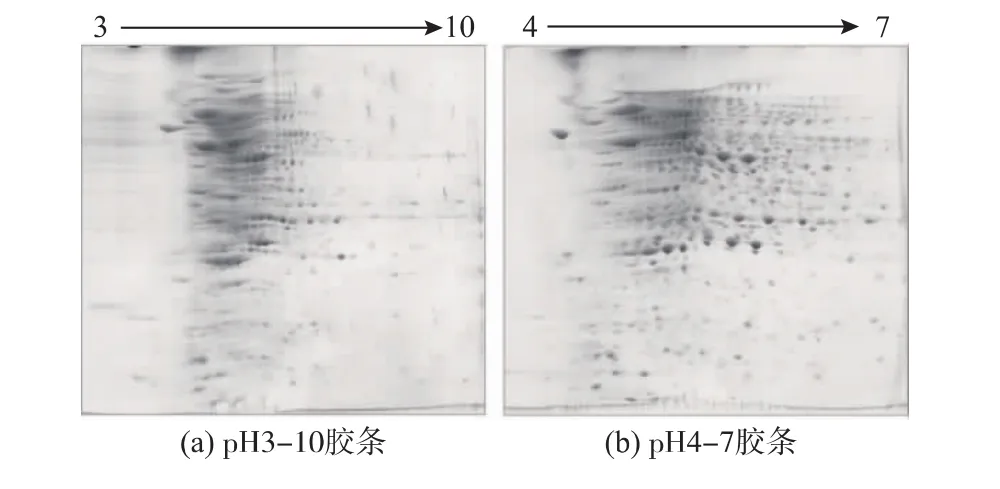
圖3 不同pH范圍膠條的雙向電泳圖譜Fig.3 Comparison of 2-DE maps with different IPG strips
2.4 不同等電聚焦程序對雙向電泳圖譜的影響
等電聚焦是雙向電泳中至關重要的一步,聚焦時間不足容易造成高相對分子質量部分蛋白聚焦不完全以及點不圓等現象,聚焦時間過長則容易導致蛋白在其等電點位置時不穩定而產生橫條紋。作者設置了3個不同的等電聚焦程序:程序I(100 V 30 min,1 000 V 3 h,6 000 V 5 h,6 000 V 50 000 Vh,500 V 20 h)、程序Ⅱ(100 V 30 min,1 000 V 3 h,8 000 V 5 h,8 000 V 60 000 Vh,500 V 20 h)和程序Ⅲ (100 V 30 min,1 000 V 3 h,10 000 V 5 h,10 000 V 70 000 Vh,500 V 20 h)。 3 個程序的區別為最高電壓和聚焦功率不同,結果如圖4所示。程序I條件下的蛋白圖譜可分離的蛋白點極少,高相對分子質量部分出現較多橫條紋;程序Ⅲ得到的結果見圖4(c),程序I相比得到改善,但高相對分子質量部分的橫條紋依然較明顯,考慮可能的原因分別是聚焦不完全和聚焦過度。程序Ⅱ對應的圖譜蛋白點更加清晰,橫條紋的問題大大的得到了改善,用PDQuest 8.0.1分析檢測可得737個蛋白點。因此,本實驗選用程序Ⅱ進行雙向電泳的第一向等電聚焦電泳。

圖4 不同等電聚焦程序的雙向電泳圖譜Fig.4 Comparison of 2-DE maps with different IEF programs
3 結 語
建立了適用于B.mucilaginosus SM-01的全蛋白提取和雙向電泳方法。確定采用低濃度鹽酸多次處理法去除菌體外莢膜多糖,然后用TCA丙酮/酚抽提結合法提取全蛋白,所得到的蛋白質純度較高。采用17 cm pH 4~7的線性IPG預制干膠條,在8 000 V 60 000 Vh的最高聚焦程序下進行第一向等電聚焦。在此條件下,圖譜蛋白質點多達737個,圖譜分離效果好,蛋白點清晰且背景較低,適用于B.mucilaginosus SM-01的蛋白質組學研究。
[1]SEVIOUR R J,MCNEIL B,FAZENDA M L,et al.Operating bioreactors for microbial exopolysaccharide production[J].Critical Reviews in Biotechnology,2011,31:170-185.
[2]YOON S,HONG E,KIM S,et al.Optimization of culture medium for enhanced production of exopolysaccharide from Aureobasidium pullulans[J].Bioprocess and Biosystems Engineering,2012,35:167-172.
[3]LI Donghua,YANG Bo.Application of response surface methodology to optimize the spore production of B.mucilaginosus GM1[J].Science and Technology of Food Industry,2012,33(22):206-209.(in Chinese)
[4]LIU Wuxing,XU Xushi.Study on fermentation of Bacillus mucilaginosus[J].Journal of Nanchang University (Natural Science),2002,26(3):299-302.(in Chinese)
[5]WRIGHTON K H.Technology:proteomics gets more selective[J].Nature Reviews Molecular Cell Biology,2013,14(3):131-131.
[6]ROCCO M,D'AMBROSIO C,ARENA S,et al.Proteomic analysis of tomato fruits from two ecotypes during ripening[J].Proteomics,2006,6(13):3781-3791.
[7]BRADFORD M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of Protein-Dye binding[J].Analytical Biochemistry,1976,72:248-254.
[8]WISNIEWSKI J R,ZOUGMAN A,NAGARAJ N,et al.Universal sample preparation method for proteome analysis[J].Nature Methods,2009,6(5):359.
[9]SHENG L,ZHU G,TONG Q.Comparative proteomic analysis of Aureobasidium pullulans in the presence of high and low levels of nitrogen source[J].Journal of Agricultural and Food Chemistry,2014,62(43):10529-10534.
[10]ZHAO Shaohui,ZHOU Jingwen,DU Guocheng,et al.A preliminary study on differential proteomics of Corynebacterium crenatum in two oxygen supply models[J].Journal of Food Science and Biotechnology,2014,33(3):235-240.(in Chinese)

