一株食竇魏斯氏菌的分離鑒定及其質粒的序列分析
張廣峰,張楠笛,趙婷婷,許 琴,譚 霄,向文良
(西華大學食品與生物工程學院,四川省食品生物技術重點實驗室, 古法發酵(釀造)生物技術研究所,四川成都 610039)
魏斯氏菌屬于乳酸菌,廣泛分布于各類發酵食品、土壤等環境中。不僅具有β-葡糖苷酶活性[1]、產細菌素能力[2-3]和較好的抗真菌性[4-5],而且產生的乳酸和胞外多糖對食品的風味有一定影響[6]。故近年來,魏斯氏菌在微生物學、醫藥學、發酵工業、食品加工等方面的應用受到研究者的廣泛關注[7-8]。在以往得到的研究成果中,乳酸菌的諸多生理功能都與其攜帶的質粒相關,如產胞外多糖、產細菌素、抗生素抗性等[9]。因此對質粒生物學和遺傳學的研究受到了極大關注。
許多魏斯氏菌擁有一個或多個天然質粒,但是相對于乳桿菌屬、乳球菌屬、腸球菌屬等質粒,對魏斯氏菌質粒研究較少[9-10]。Park[11]、金紅星[12]、Kim[13]等分別從食竇魏斯氏菌(Weissellacibaria)分別分離出3、2、6個質粒,王海娟等[14]從融合魏斯氏菌(Weissellaconfusa)中分離出了2個質粒,并對部分質粒完成了序列分析及載體構建。數據庫中魏斯氏菌質粒的相關數據較少,以致于不能推斷質粒閱讀框中的具體功能[13]。所以豐富魏斯氏菌質粒數據庫對魏斯氏菌屬質粒復制、遺傳、進化、功能等方面具有重要意義。
中國泡菜中蘊含豐富的乳桿菌質粒資源[15],因此本研究以中國四川泡菜為分離源,篩選其中含質粒乳酸菌并對質粒進行全序列測定及功能分析,以期為構建良好的載體提供理論基礎。
1 材料與方法
1.1 材料與儀器
泡菜液 四川盈宇食品有限公司;大腸桿菌(EscherchiacoliDH5α) 天根生化科技(北京)有限公司;pGEM-T Simple Vector 普洛麥格(北京)生物技術有限公司;溶菌酶、限制性核酸內切酶 美國Thermo scientific;Taq DNA聚合酶、T4 DNA連接酶 Promega有限公司;質粒小量提取試劑盒 OMEGA公司;膠回收試劑盒 天根生化科技有限公司;土壤DNA Extraction試劑盒 福際生物技術有限公司;LB培養基、LB液體培養基、MRS液體培養基和改良MRS培養基(含0.75 g/100 mL的CaCO3) 均按文獻[16]配制。
Biometra PCR儀 德國耶拿分析儀器有限公司;Allegra X-15R冷凍離心機 貝克曼庫爾特公司;720BR電泳凝膠成像分析儀 Bip-rad(美國);SGSP-02恒溫隔水式培養箱 黃石恒豐器械有限公司;DYY-8C型電泳儀 北京市六一儀器廠。
1.2 實驗方法
1.2.1 乳酸菌的分離純化 取25 mL泡菜液于225 mL滅菌生理鹽水中混合均勻,經10倍梯度稀釋后涂布于改良MRS 培養基中,37 ℃倒置培養48 h。挑取周圍形成透明圈的菌落劃線純化,同時進行革蘭氏染色和過氧化氫酶實驗。將革蘭氏染色結果為陽性、過氧化氫酶陰性的菌株初步判定為乳酸菌[17]。
1.2.2 質粒DNA的提取 將純化的乳酸菌接種至MRS液體培養基,37 ℃過夜培養。采用質粒小量提取試劑盒提取質粒DNA,經過以下修改:菌體以轉速12000 r/min離心2 min、ddH2O洗滌后重懸于含有30 mg/mL溶菌酶的SolutionI中,37 ℃溫育1 h,其余操作按照說明書進行。1%瓊脂糖凝膠電泳檢測提取質粒DNA。
1.2.3 含質粒乳酸菌的鑒定 乳酸菌的生化特征分析按照《乳酸菌細菌分類鑒定及實驗方法》[18]進行。參照文獻[19]方法提取含質粒乳酸菌基因組DNA。16S rDNA的PCR擴增引物為Eu27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′)。擴增條件:95 ℃預變性5 min,95 ℃變性1 min,50 ℃退火1 min,72 ℃延伸2 min,30個循環,72 ℃保持10 min。擴增產物連接到pGEM-T后轉化到E.coliDH5α中,篩選陽性克隆子對16S rDNA測序。所得序列提交至NCBI進行同源性比對,利用MEGA 5.0軟件構建系統發育樹。
1.2.4 質粒序列測定 根據質粒條帶,回收其中的大質粒命名為pA010。分別利用不同的限制性內切酶EcoR I、BamH I和PstI對分離純化的質粒pA010進行酶切分析,回收酶切反應液。參照都立輝等[17]方法補平酶切后的黏性末端并在3′端引入堿基A,回收其中DNA片段同pGEM-T載體連接并轉化到E.coliDH5α中,篩選陽性克隆測序。以測序結果中最長序列片段為基礎設計引物 W1CF(5′-TTTTCCACTTCGTTATTCTTG-3′)和 W1CR(5′-TTGACAACGACAAGTATTAGAGC-3′)擴增質粒pA010剩余片段。PCR擴增條件:95 ℃ 5 min,95 ℃ 1 min、50 ℃ 1 min、72 ℃ 2 min、30 個循環,72 ℃ 10 min。將PCR擴增片段與載體連接、轉化到E.coliDH5α,挑選陽性克隆測序,拼接各個測定序列獲得質粒pA010的全序列。
1.2.5 質粒的生物信息學分析 利用NCBI網站上的ORF Finder(Open reading frame finder)尋找該質粒可能存在的開放閱讀框(ORF),然后對質粒的核苷酸及氨基酸序列進行分析。DNAman6.0繪制質粒圖譜,CDD網站用于假定蛋白保守結構域的尋找,CLUSTAL X實現序列的多重比對。通過分析質粒上的序列信息,推導質粒復制方式,并將該質粒全序列提交GenBank數據庫。
2 結果與分析
2.1 含質粒菌株的分離鑒定
從四川泡菜中分離到11株含溶鈣圈的菌株,編號為W1~W11,均呈乳白色、邊緣整齊的圓形菌落,油鏡下觀察顯示菌體為桿狀、紫色。對這11株菌進行質粒普查發現只有菌株W5攜帶2個天然質粒(圖1)。將其中大質粒命名為pA010,分離純化該質粒,通過酶切和PCR擴增對其進行序列測定。

圖1 四川泡菜中乳酸菌的質粒提取電泳圖Fig.1 Plasmid extracts electrophoresis of Lactic acid bacteria in Sichuan Pickle注:M為DNA Marker;1~11為乳酸菌W1~11的質粒提取液。
對含質粒菌株W5進行常規生理生化實驗,結果如表1。根據形態學和生化特征,檢索乳酸菌分類系統,初步確定該菌株屬于魏斯氏屬(Weissella)。提取含質粒菌株W5的基因組DNA并進行16S rDNA擴增,其擴增產物序列大小為1463 bp。將菌株序列與GenBank數據庫中標準菌株的序列進行BLAST比對發現,該菌株與食竇魏斯氏菌(W.cibaria)的16S rDNA相似性達99%。該菌株與標準菌株的16S rDNA的系統發育表明,該菌株與食竇魏斯氏菌具有較大的親緣關系(圖2)。一般來講,在種分類等級上,如果2個分類單位間的16S rDNA序列同源性大于97.5%,則認為屬于同一個種[20]。結合菌株的形態學和生理生化特征,這株含內源質粒的乳酸菌被鑒定為食竇魏斯氏菌。

圖2 含質粒菌株W5基于16 S rDNA序列的系統發育樹Fig.2 Phylogenetic tree of strain W5 containing plasmid based on 16S rDNA sequence

表1 含質粒菌株W5部分生理生化特征Table 1 Part of physiological and biochemical results of strain W5 containing plasmid
2.2 質粒全序列測定
將回收純化的質粒pA010分別用限制性內切酶EcoR I、BamH I和PstI進行酶切分析,發現該質粒僅有4個EcoR I酶切位點(圖3)。回收酶切反應液,末端補平后連入pGEM-T,經測序分析EcoR I酶切的4個片段長度分別為5762、2820、2677、371 bp。以最長序列片段為基礎設計引物,對pA010進行PCR擴增,以確定各個質粒片段的連接順序,最終得到pA010的完整序列(GenBank登錄號為MF951102)。

圖3 質粒pA010酶切分析Fig.3 Restriction enzyme analysis of pA010注:M為DNA Marker;E、B、P分別代表EcoR I、BamH I和Pst I酶切pA010。
2.3 質粒pA010的一般結構分析
食竇魏斯式菌W5較大質粒pA010序列大小為11630 bp,為一個閉合環狀雙鏈DNA分子,其中GC含量34.6%。通過NCBI網站上的ORF Finder軟件查找ORF,搜索到12個序列長度大于300 bp的ORFs。由于一個具有起始密碼子和終止密碼子的ORF不一定都是蛋白質的編碼序列,因此搜索到的ORFs中還有非編碼基因和重復序列,所以再將序列進行BLASTp蛋白質同源片段分析[21]。12個ORFs中有4個ORFs編碼已知功能蛋白,其他8個ORFs編碼假定蛋白(表2)。通過DANman6.0查找質粒的EcoR I、BamH I和PstI位點發現,質粒中無BamH I和PstI的酶切位點、4個EcoR I酶切位點,此結果與圖4一致。為標識已知功能蛋白、假定蛋白和EcoR I酶切位點在質粒中的位置,通過DANman6.0構建質粒圖譜,如圖4所示。

表2 質粒pA010推斷的ORFs編碼的蛋白質Table 2 The putative ORFs and the encoded proteins of plasmid pA010

圖4 食竇魏斯式菌W5中pA010的質粒圖譜Fig.4 Plasmid map of pA010 from W. cibaria W5
2.4 質粒pA010的功能分析
ORF2編碼了一個INT_C_like_3,與植物乳桿菌中酪氨酸重組酶顯示了99%的相似性,與乳酸片球菌質粒pSMB74中的酪氨酸重組酶顯示了95%的相似性。這個蛋白屬于DNA breaking/re-joining enzymes超家族,可能與質粒多聚體的解離有關[22]。Motlagh A等[23]表明,這個蛋白經常被發現在乳酸菌的θ型復制質粒。
ORF3編碼復制蛋白Rep,與植物乳桿菌G63內源質粒pG6302復制蛋白的相似性達100%,與植物乳桿菌內源質粒pLME300復制蛋白的相似性達93%。這些同源Rep有一個保守的涉及質粒復制起始的Rep_3結構域(pfam01051),這個結構域屬于RepA_N質粒家族,一般為θ型復制質粒所有[15]。而且pLME300[24]和pG6302[25]被認為是一種θ型復制的質粒,所以pA010被認為是一種θ型復制的質粒。復制蛋白的上游515 bp有類似起始質粒復制的重復序列,預測可能是復制起始區(圖5)。復制子區域包含3個同向重復序列(Direct repeats,DR)和3個反向重復序列(Invert repeats,IR),還有一段富含A、T序列。Xi等[26]認為正向重復序列是復制蛋白的目標且在質粒的不相容性上有重要作用,而且A/T富含區被認為是復制的起始。
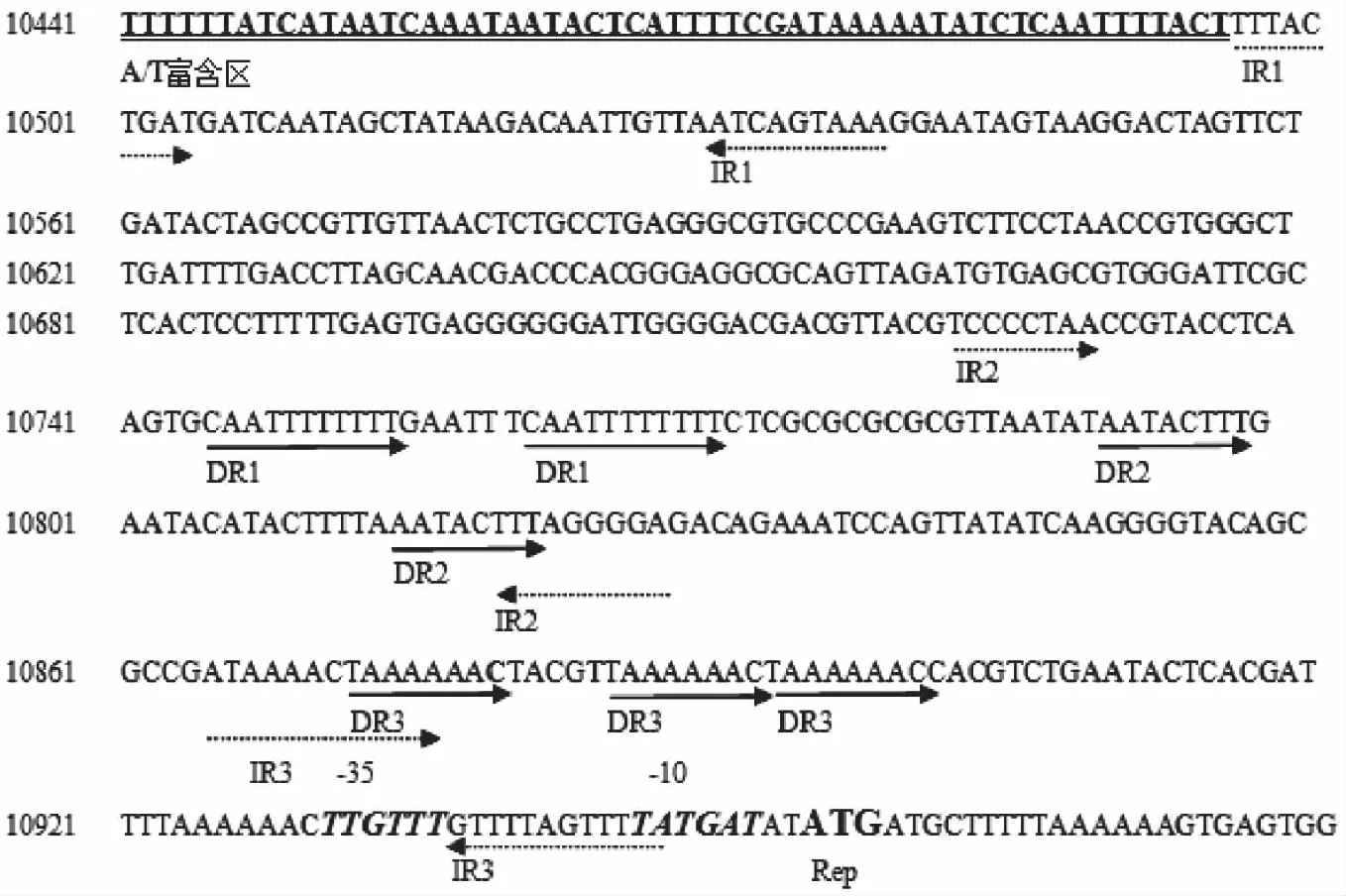
圖5 質粒pA010假定的復制起始區Fig.5 The predicted origin region of replication of pA010注:雙下劃線為A/T富含區;實線箭頭為正向重復(DR);虛線箭頭為反向重復(IR);加粗斜體字母為-10和-35區;加粗ATG為復制蛋白(Rep)的起始點。
ORF11和ORF12編碼的蛋白分別與乳酸片球菌H中質粒pSMB74的PemK和PemI蛋白顯示了100%和94%的相似性。Fang等[25]報道,PemK和PemI是一個II型毒素和抗毒素系統(type II Toxin and Antitoxin system,II-TA),其中PemK編碼穩定的毒素,PemI編碼不穩定的抗毒素。ORF11和ORF12之間重疊7個堿基,提示兩個基因可能共同構成一個操縱子(圖6)。細菌質粒編碼的TA系統作為沉溺系統(plasmid addiction systems,PAS),通過細胞分裂后死亡的機制維持質粒的穩定性[27]。而且TA系統利于細菌持留感染的形成(細菌進入一種低生長或休眠狀態),在這種狀態下細胞對周圍環境的適應力可以得到很大提升,如對抗生素的耐藥增加、在有害環境下保持一部分生存個體等[28]。在植物乳桿菌NC7中的質粒p256[29]、植物乳桿菌CD033中質粒pCD033[26]和唾液乳桿菌UCC118中的質粒pSF118[27]中都編碼了一個TA系統。近年來,乳酸菌中TA研究受到了相當的重視,不僅是因為質粒攜帶的TA基因維持細胞質粒的遺傳穩定性,更是由于乳酸菌中TA的研究對乳酸菌中菌株的多樣化的元基因組分析以及乳酸菌開發有重要意義[30]。

圖6 pemI、pemK在pA010上的遺傳結構及其蛋白序列同源性比對 Fig.6 Genetic structure and sequence alignment of the pemI/pemK locus in pA010注:A為pemI/pemK遺傳結構示意圖,箭頭所示pemI和pemK的轉錄方向;B為pemI編碼產物與PemI蛋白序列比對; C為pemK編碼產物與PemK蛋白序列比對,pSMB74、p256、pSF118、pCD033分別表示P. acidilactici H、L. plantarum NC7、L. salivarius UC118、L.plantarum CD033中的質粒。
3 結論
本實驗以四川泡菜為分離源,從中篩選到一株含質粒的食竇魏斯氏菌。其中大質粒pA010全序列結果顯示該質粒全長11630 bp,GC含量為34.6%,同時編碼了四個可識別功能蛋白:酪氨酸重組酶、復制蛋白、抗毒素PemI和毒素PemK。通過分析這些蛋白的功能發現,該質粒可能是θ型復制質粒,而且毒素-抗毒素系統對質粒穩定以及魏斯氏菌耐受周圍環境提供了可能。
[1]Lee K W,Han N S,Kim J H. Purification and characterization of beta-glucosidase fromWeissellacibaria37[J]. Journal of Microbiology & Biotechnology,2012,22(12):1705-1713.
[2]Li S W,Chen Y S,Lee Y S,et al. Comparative genomic analysis of bacteriocin-producingWeissellacibaria110[J]. Applied Microbiology and Biotechnology,2017,101(3):1-11.
[3]王筱夢,江蕓,孫芝蘭,等. 1株產細菌素乳酸菌的鑒定及所產細菌素的誘導合成現象[J]. 食品科學,2016,37(21):170-175.
[4]Baek E,Kim H,Choi H,et al. Antifungal activity ofLeuconostoccitreumandWeissellaconfusain rice cakes[J]. J Microbiol,2012,50(5):842.
[5]常偉,饒瑜,胡懷容,等. 一株抗真菌食竇魏斯氏菌AT6的特性分析及培養條件優化[J]. 食品工業科技,2014,35(6):193-196.
[6]Galle S,Schwab C,Arendt E,et al. Exopolysaccharide-formingWeissellastrains as starter cultures for sorghum and wheat sourdoughs[J]. Journal of Agricultural & Food Chemistry,2010,58(9):5834.
[7]王海娟,戴雨珂,潘渠. 魏斯氏菌的研究現狀[J]. 成都醫學院學報,2014,9(6):747-750.
[8]Lee K W,Park J Y,Jeong H R,et al. Probiotic properties ofWeissellastrains isolated from human faeces[J]. Anaerobe,2012,18:96-102.
[9]孫大慶,李洪飛,宋大巍,等.乳桿菌屬天然質粒研究進展[J]. 食品科學,2015,36(11):251-255.
[10]孫大慶,李洪飛,楊健. 植物乳桿菌編碼復制起始蛋白天然質粒的系統進化分析[J]. 微生物學通報,2017,44(5):1047-1055.
[11]Park M S,Kim S H,Kim J D,et al. Molecular characterization of plasmid DNA fromWeissellacibariaisolated from Kimchi[J]. Korean Journal of Genetics,2007,29(1):29-35.
[12]金紅星,楊希寅,成文玉. 辣白菜中分離含內源性質粒的魏斯氏菌及其鑒定[J]. 中國釀造,2012,31(1):77-79.
[13]Kim S Y,Oh C G,Lee Y J,et al. Sequence analysis of a cryptic plasmid pKW2124 fromWeissellacibariaKLC140 and construction of a surface display vector[J]. Journal of Microbiology & Biotechnology,2013,23(4):545.
[14]王海娟,戴雨珂,蘇森森,等. 一個融合魏斯氏菌隱蔽質粒的序列分析[J]. 生物技術通報,2016,32(4):217-221.
[15]楊四佳,王穎,李宇婷,等. 泡菜中乳桿菌的快速檢出和乳桿菌質粒資源的初步調查[J]. 食品與生物技術學報,2013,32(6):639-644.
[16]杜連祥,路福平. 微生物學實驗技術[M]. 北京:中國輕工業出版社,2014:349-354.
[17]都立輝,霍貴成,鞠興榮,等. 一株植物乳桿菌的分離鑒定及其隱蔽質粒的序列分析[J]. 食品科學,2010,31(17):236-239.
[18]凌代文. 乳酸細菌分類鑒定及實驗方法[M]. 北京:中國輕工業出版社,1999:117-127.
[19]Xiang W L,Liang H Z,Liu S,et al. Isolation and performance evaluation of halo tolerant phosphate solubilizing bacteria from the rhizospheric soils of historic Dagong Brine Well in China[J]. World Journal of Microbiology and Biotechnology,2011,27(11):2629-2637.
[20]StackebrandT E,Gebel B M. Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic Bacteriology,1994,44(4):846-849.
[21]Silvia H,Reingard G,Stefan H. Characterization of theLactobacillusplantarumplasmid pCD033 and generation of the plasmid free strainL.plantarum3NSH[J]. Plasmid,2015,81(11):9-20.
[22]Rob V H,Raphael L,Gipsi L M,et al. Towards a more accurate annotation of tyrosine-based site-specific recombinases in bacterial genomes[J]. Mobile DNA,2012,3(1):6.
[23]Motlagh A,Bukhtiyarova M,Ray B. Complete nucleotide sequence of pSMB 74,a plasmid encoding the production of pediocin AcH inPediococcusacidilactici[J]. Letters in Applied Microbiology,1994,18(6):305.
[24]Gfeller K Y R,Melle L,Teuber M,et al. Sequence and genetic organization of the 19.3-kb erythromycin-and dalfopristin-resistance plasmid pLME300 fromLactobacillusfermentumROT1[J]. Plasmid,2003,50(3):190-201.
[25]Fang F,Flynn S,Li Y,et al. Characterization of endogenous plasmids fromLactobacillussalivariusUCC118[J]. Applied & Environmental Microbiology,2008,74(10):3216-3228.
[26]Xi X,Fan J,Hou Y,et al. Characterization of three cryptic plasmids fromLactobacillusplantarumG63 that was isolated from Chinese pickle[J]. Plasmid,2013,70(3):321-328.
[27]孫瑞,寧德剛. 細菌Ⅱ型毒素-抗毒素系統活性的調控[J]. 微生物學通報,2016,43(12):2714-2719.
[28]楚海榮,付玉榮,伊正君. 細菌毒素-抗毒素系統研究進展[J]. 細胞與分子免疫學雜志,2015,31(2):271-274.
[29]S?rvig E,Skaugen M,Naterstad K,et al. Plasmid p256 fromLactobacillusplantarumrepresents a new type of replicon in lactic acid bacteria,and contains a toxin-antitoxin-like plasmid maintenance system[J]. Microbiology,2005,151(2):421-431.
[30]徐謂,李洪軍,賀稚非. 乳酸菌中毒素-抗毒素系統研究進展[J]. 食品科學,2015,36(19):260-264.

