玉米ZmLTP3基因啟動子的克隆及功能分析
寧慧宇, 江 敏, 張丙林, 鄒華文
(長江大學農學院,湖北荊州 434025)
植物轉脂蛋白(lipid transfer proteins,簡稱LTPs)是植物體內具有結合和轉運脂類功能的一類小分子蛋白。又因其對各種脂類(如輔酶A、磷脂和脂肪酸等)具有較高的親和力,又被稱為非專一性轉脂蛋白(non-specific lipid transfer proteins,簡稱nsLTPs)[1-3]。植物轉脂蛋白是一類堿性蛋白,分子內部都含有1個由8個半胱氨酸組成的保守基序(C-Xn-C-Xn-CC-Xn-CXC-Xn-C-Xn-C)。這8個半胱氨酸形成4對二硫鍵,所以轉脂蛋白分子具有較高的耐高溫、耐變性等特性[4]。最初根據分子量大小,植物LTPs成員被分為2種類型:Ⅰ型、Ⅱ型[5]。此后,Edstam等根據半胱氨酸之間的距離、保守內含子的位置以及翻譯后糖基磷脂酰肌醇錨(GPI-anchor)的添加與否,把LTPs重新劃分為A、B、C、D、E、F、G、H、J和K型[1]。因為具有膜間轉移脂分子的功能,植物LTPs最初被認為參與生物膜系統的生物合成[6]。然而隨著N-端信號肽的發現和胞外定位,使人們開始重新認識LTPs的功能。現有的研究表明,LTPs涉及植物多種生理過程,包括參與蠟質的合成和運輸、生殖器官的發育、提高植物抗性、促進細胞壁的伸長,此外還有調節果膠降解活性等[7-9]。其中研究較多的是其在蠟質的合成和運輸、生殖器官的發育以及在提高植物抗性中的作用。
在之前的研究中,筆者從玉米中克隆到1個轉脂蛋白家族成員,并命名為ZmLTP3(GenBank登錄號 JX435819.1),過量表達ZmLTP3基因的擬南芥表現出明顯的抗鹽性[10]。在美國國立生物技術信息中心(National Center for Biotechnology Information,簡稱NCBI)數據庫中,ZmLTP3基因上游啟動子序列并未測通。為了闡述ZmLTP3基因的作用機制,本研究克隆并測序ZmLTP3基因上游完整的啟動子序列,并對此序列進行生物信息學分析,擬構建融合GUS報告基因的植物表達載體,將其轉化擬南芥,并對轉基因擬南芥純合體株系進行GUS組織化學染色,驗證啟動子功能。本研究為深入分析ZmLTP3基因的作用機制奠定了基礎。
1 材料與方法
1.1 材料
植物材料擬南芥(Columbia 生態型)、玉米B73、大腸桿菌菌株 DH5α、農桿菌GV3101、植物表達載體pGreen0029-GUS等,均由筆者所在實驗室保存并提供。
1.2 方法
1.2.1 啟動子克隆及序列分析 利用十六烷基三甲基溴化銨(簡稱CTAB)法[11]提取3葉1心期的玉米葉片基因組DNA。在NCBI網站(https://www.ncbi.nlm.nih.gov/)上下載得到ZmLTP3基因ATG上游 2 881 bp(不包括gap的堿基數)的啟動子序列,并在此序列內部缺口的兩端設計上下游引物,擴增完整的啟動子序列。上游引物序列為5′-GGCGACTGTGACACTATCC-3′,下游引物序列為5′-AAGTAGGGCTATCGAAACAGG-3′。得到完整序列后,在NCBI網站上比對測序結果與數據庫中序列的異同。在Plantcare網站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)上預測測序啟動子序列中所含有的順式作用元件。
1.2.2 擬南芥的遺傳轉化 以測序的啟動子序列為模板,在-1 301 bp處設計上游引物,擴增啟動子片段,并將此啟動子片段插入到植物表達載體pGreen0029-GUS中GUS的上游。同時,也把筆者所在實驗室保存的35S啟動子片段插入到上述載體中作為陽性對照。擬南芥的培養、基因轉化及轉基因株系的分子鑒定參考文獻[12]。在GUS基因序列內部設計1對引物,用于對轉基因擬南芥的PCR檢測,上游引物序列為5′-TTCTGATTAACCACAAAC-3′,下游引物序列為5′-CGGTTCGTTGGCAATACTCC-3′。
1.2.3 啟動子功能分析 分別取轉空載體、35S、ZmLTP3基因啟動子載體的陽性植株和野生型擬南芥葉片,參照Gross等的方法[13]進行GUS染色。
2 結果與分析
2.1 ZmLTP3基因啟動子克隆
得到準確、完整的啟動子序列,是啟動子功能分析的第1步。但是查詢NCBI數據庫發現,ZmLTP3基因起始密碼子ATG上游-1 270 bp處有未測通的缺口,并且序列內部含有重復序列及反向重復序列(圖1)。為了得到準確的序列,筆者在距缺口較遠的位置設計上下游引物, 擴增此片段。測序結果表明,從上游引物到ATG只有1 302 bp的距離,而不是NCBI數據庫中的2 492 bp (不包括缺口的堿基數)。BLAST結果表明,測序的啟動子序列與網上序列一致性為99%,序列內部沒有重復及反向重復序列(圖2)。


2.2 ZmLTP3基因啟動子序列分析
利用Plantcare網站分析發現,測序序列含有眾多順式作用元件,包括14個TATA-box,這14個TATA-box中離ATG最近的位于-149 bp處,離ATG最遠的位于-1 196 bp處,此外,測序序列中還含有7個CAAT-box(表1)。除了TATA-box、CAAT-box等啟動子必需的元件外,該啟動子片段包含許多重要的結合位點及功能元件,例如參與植物防御和逆境響應的調控元件(TC-rich repeats)、參與脫落酸響應的作用元件(ABRE)、參與干旱誘導響應的元件(MBS)、參與水楊酸響應的元件(TCA-element)、眾多光響應元件等。這些功能元件的存在,說明ZmLTP3基因可以響應多種逆境脅迫(包括生物逆境以及非生物逆境)的誘導,并且在逆境脅迫中發揮作用。

表1 ZmLTP3基因啟動子區順式作用元件
2.3 ZmLTP3基因啟動子的擬南芥轉化及鑒定
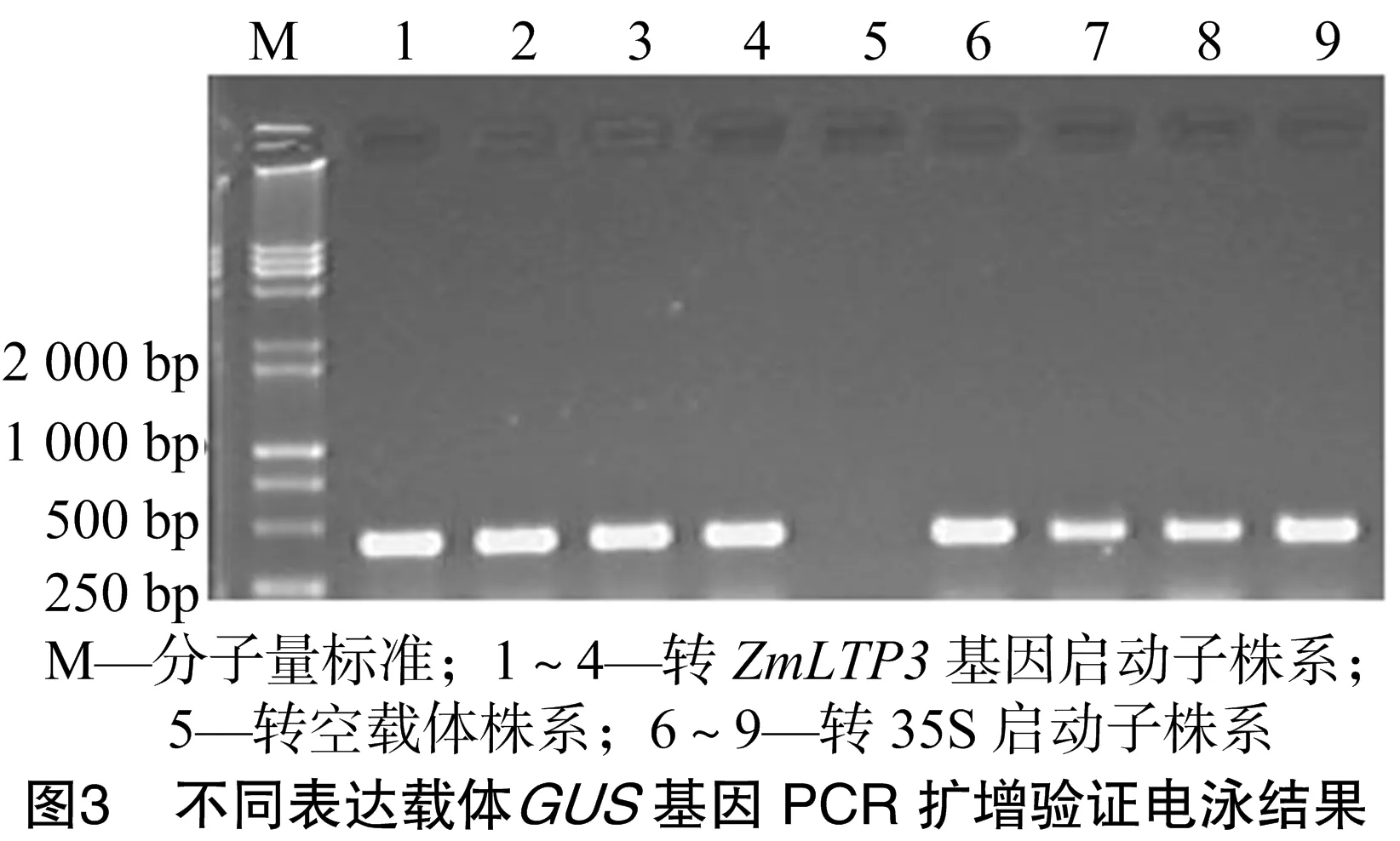
花苞用浸蘸法轉化擬南芥后,收取T0代種子,消毒后播種在含有50 μg/L卡那霉素的MS培養基上培養,種子發芽后,選取正常生長狀態的幼苗進行PCR鑒定。如圖3所示,所選擇的轉基因株系在400 bp左右都有1條陽性條帶,而野生型株系的對照擴增結果為陰性,表明所采樣的擬南芥植株均為成功轉化了的陽性株系,可以用來進行下一步試驗。
2.4 ZmLTP3基因啟動子的功能鑒定
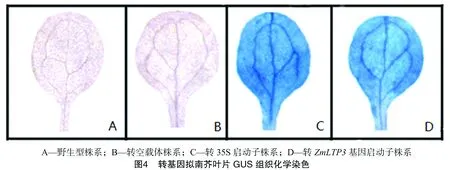
在本試驗中,分別采取成熟階段的不同擬南芥植株的葉片作為試驗材料進行GUS染色。如圖4所示,野生型及空白載體轉化的擬南芥植株葉片未顯示藍色,而轉35S啟動子及ZmLTP3基因啟動子的轉基因擬南芥株系都呈現不均勻的藍色,且轉35S啟動子的擬南芥株系染色較轉ZmLTP3基因啟動子的深,暗示ZmLTP3基因啟動子的表達強度不及35S啟動子。
3 討論與結論
從現有的資料來看,研究者大多集中在對LTPs生物學功能的探究,而對其作用機制則很少有報道,尤其是對其上游的分子調控機制也鮮有系統的研究。另外,盡管第1條編碼植物LTP的cDNA是從玉米中獲得的,但是之后關于玉米中LTP基因家族及其功能的研究很少有深入的報道。本研究通過對ZmLTP3基因啟動子的分析,為進一步闡明ZmLTP3基因作用機制,尤其是其上游調控機制打下了基礎。
盡管玉米基因組測序工作的完成給玉米分子生物學的研究帶來了極大的方便,但是由于玉米基因組結構的復雜性,尚有一些未測通的片段,這也給相關工作的開展帶來了諸多不便。例如,本研究中ZmLTP3基因上游的啟動子序列就沒有完全測通。本試驗以NCBI中公布的ZmLTP3基因上游序列為基礎,在序列的缺口上下游合適位置設計引物,成功地克隆了啟動子序列,填補了NCBI中此序列的缺口,為其他研究者進行相關研究提供了便利。BLAST結果表明,克隆序列與NCBI中公布的序列一致性為99%。
Plantcare在線分析表明,測序序列除具有TATA-box及CAAT-box等典型的啟動子基本順式作用元件外,還含有多種響應生物脅迫及非生物脅迫的元件,暗示ZmLTP3基因可能參與多種逆境脅迫的信號傳導過程,這也與筆者之前的試驗結果相吻合[10]。為了驗證此啟動子序列的功能,構建植物表達載體,轉化擬南芥,GUS染色結果初步證明所克隆的序列具有啟動子功能。進一步的研究,如啟動子核心序列的鑒定、反式作用因子的篩選及驗證等則需要進行后續試驗。
[1]Edstam M M,Viitanen L,Salminen T A. Evolutionary history of the Non-Specific lipid transfer proteins[J]. Molecular Plant,2011,4(6): 947-964.
[2]Kader J C. Lipid-transfer proteins in plants[J]. Annual Review of Plant Physiology and Plant Molecular Biology,1996,47(1): 627-654.
[3]Smith L J,Roby Y,Allison J R,et al. Molecular dynamics simulations of barley and maize lipid transfer proteins show different ligand binding preferences in agreement with experimental data[J]. Biochemistry,2013,52(30): 5029-5038.
[4]Edstam M M,Laurila M,H?glund A,et al. Characterization of the GPI-anchored lipid transfer proteins in the mossPhyscomitrellapatens[J]. Plant Physiology and Biochemistry,2014,75: 55-69.
[5]Yeats T H,Rose J K. The biochemistry and biology of extracellular plant lipid-transfer proteins (LTPs)[J]. Protein Science,2008,17(2): 191-198.
[6]Kader J C,Julienne M,Vergnolle C. Purification and characterization of a spinach-leaf protein capable of transferring phospholipids from liposomes to mitochondria or chloroplasts[J]. European Journal of Biochemistry,1984,139(2): 411-416.
[7]Fan Y,Du K,Gao Y,et al. Transformation ofLTPgene intoBrassicanapusto enhance its resistance toSclerotiniasclerotiorum[J]. Russian Journal of Genetics,2013,49(4): 380-387.
[8]Orellana S,Yanez M,Espinoza A,et al. The transcription factor SlAREB1 confers drought,salt stress tolerance and regulates biotic and abiotic stress-related genes in tomato[J]. Plant Cell and Environment,2010,33(12): 2191-2208.
[9]Yang Z B,Eticha D,Rotter B,et al. Physiological and molecular analysis of polyethylene glycol-induced reduction of aluminium accumulation in the root tips of common bean (Phaseolusvulgaris)[J]. New Phytologist,2011,192(1): 99-113.
[10]Zou H W,Tian X H,Ma G H,et al. Isolation and functional analysis ofZmLTP3,a homologue to arabidopsisLTP3[J]. International Journal of Molecular Sciences,2013,14(3): 5025-5035.
[11]Zou H W,Wu Z Y,Yang Q,et al. Gene expression analyses of ZmPti1,encoding a maize Pti-like kinase,suggest a role in stress signaling[J]. Plant Science,2006,171(1): 99-105.
[12]Zou H W,Wu Z Y,Zhang X H,et al. Over-expression ofZmPti1,a homologue toPti1,increases salt tolerance ofArabidopsisthaliana[J]. African Journal of Biology,2010,9(5): 656-662.
[13]Gross J,Stein R J,Fett-Neto A G,et al. Iron homeostasis related genes in rice[J]. Genetics and Molecular Biology,2003,26(4): 477-497.

