夏黑葡萄附生酵母菌分離及多相分類鑒定
,,,,,
(南京農業大學食品科技學院,江蘇南京 210095)
夏黑葡萄附生酵母菌分離及多相分類鑒定
楊雨蒙,徐敬國,胡伊旻,黃思琪,史雅凝,辛志宏*
(南京農業大學食品科技學院,江蘇南京 210095)
葡萄野生酵母是釀造優質葡萄酒的重要菌種來源,為發現有價值的野生葡萄酵母,本研究以夏黑葡萄為研究對象,采用平板劃線分離的方法得到3株葡萄野生酵母YYMPT-1、YYMPT-2和YYMPT-3,利用掃描電子顯微鏡(SEM)進行形態觀察,PCR擴增26S核糖體DNA的D1/D2區(26S rDNA D1/D2)、核糖體內轉錄間隔區(ITS1-5.8S-ITS2)和肌動蛋白基因(Actin gene)區域,并構建系統發育樹,將3株酵母菌分別鑒定為有孢漢遜酵母(Hanseniasporauvarum)、近玫色鎖擲酵母(Sporidioboluspararoseus)和假絲酵母(Candidazemplinina)。本研究結果為優質葡萄酒釀造和改善葡萄酒風味提供了3株具有潛在工業化應用價值的資源菌。
酵母菌,形態學特征,系統發育樹,多相鑒定
葡萄酒發酵是一個復雜的過程,在釀造期間需多種微生物的參與,其中天然酵母是最重要的發酵菌群。優良的酵母菌不僅可以提高葡萄酒的風味和品質,而且對葡萄酒特色的形成具有至關重要的作用[1-4]。夏黑葡萄是釀造紅葡萄酒的優良品種,其果肉飽滿、含糖量高、口感好、果汁呈紫紅色、無核,是葡萄酒釀造過程中較為普遍使用的品種[5-6]。研究發現,葡萄表皮附生有豐富的野生酵母,對其進行分離鑒定,在釀造優質葡萄酒和改善酒的品質方面具有重要的實際應用價值。
酵母菌的鑒定包括表型鑒定和分子生物學鑒定兩類方法[7]。表型鑒定是一種傳統的菌種鑒定方法,一般通過單菌落的表型及顯微形態觀察判斷菌株的種屬,但該方法耗時、耗力、工作量大,需要專業的人員,且只能將酵母鑒定到屬水平,不能反映與其它物種之間的親緣關系[8]。
近年來,隨著分子生物學的發展,通過PCR擴增酵母菌的保守基因序列并構建系統發育樹從而確定酵母菌的種屬地位,這為酵母菌的分類鑒定提供了準確有效的鑒定方法。研究表明,酵母菌的26S核糖體DNA的D1/D2區域具有較高的變異性,不同種菌株之間堿基差異較大,而同種的不同菌株之間堿基差異一般不超過1%,可作為將酵母鑒定至種級水平的靶點[9];核糖體內轉錄間隔區(ITS1-5.8S-ITS2)也具有較高的變異率,根據序列間的差異,可作為親緣關系較近的酵母菌鑒定標準[10];肌動蛋白基因(Actin gene)區是普遍存在于真核細胞中的微絲構造蛋白,其核苷酸序列在種內具有高度保守性,廣泛應用于酵母菌的鑒定[11]。然而單一分子靶點鑒定有時難以將相似度較高的種區分開來,大大限制了鑒定結果的準確性。如Scorzetti[12]等研究發現,C.magnus、F.magnus和F.floriforme的26S區基因序列完全相同,因此只通過26S序列分析無法區分這3個種,這時就需要其他的輔助鑒定方法。
多相分類鑒定是綜合微生物表型特征和多個分子生物學特征對微生物進行分類的方法,可更加科學、準確地確定微生物的種屬地位[13]。Janos等人[14]通過擴增ITS、β-tubulin和鈣調蛋白基因序列并分別構建系統發育樹,鑒定出了一株黑色曲霉新種——巴西曲霉(Aspergillusbrasiliensissp. nov.)。白飛榮等[15]通過形態學和ITS/β-tubulin分子序列系統發育分析,從香山黃櫨枯萎病葉片中分離得到4種真菌:細極鏈格孢(Alternariatenuissima)、草酸青霉(Penicilliumoxalicum)、臘梅擬莖點霉(Phomopsischimonanthi)和葡萄莖枯病菌(Phomaglomerata),為香山紅葉的枯萎病提供了有效的防治方法。劉洋等從諾尼果實中分離得到2株酵母菌,采用態學觀察、生理生化特征以及ITS和26S序列分析,將這兩株菌分別鑒定為Eremotheciumcoryli和Kodamaeaohmeri,為開發利用諾尼果實提供了有價值的資源菌[16]。
夏黑作為紅葡萄酒釀造的優良品種,其附生酵母菌對釀造葡萄酒具有重要作用。本研究以夏黑為對象,采用平板劃線法分離篩選葡萄附生酵母,利用掃描電子顯微鏡(SEM)進行形態觀察,分別PCR擴增26S、ITS和Actin區域,經TA克隆測序后構建系統發育樹,確定酵母菌的種屬地位,以期為葡萄酒釀造和改善葡萄酒風味提供具有工業化應用價值的菌種資源。
1 材料與方法
1.1材料與儀器
夏黑葡萄 采自江蘇省南京市葡萄采摘園、土豆采自衛崗蘇果社區超市;葡萄糖、酵母粉、瓊脂粉、NaCl、蛋白胨、KH2PO4、MgSO4·7H2O、孟加拉紅、戊二醛、氨芐青霉素(Amp) 南京壽德試劑器材有限公司。
D3390-01E.Z.N.A真菌DNA微量提取試劑盒 美國Omega公司;2×Taq Master Mix PCR試劑 諾唯贊生物;DP1502多功能DNA純化回收試劑盒 北京百泰克生物技術科技有限公司;pMDTM19-T Vector Cloning Kit 日本TaKaRa公司;DYCP-31DN電泳儀 北京市六一儀器廠;Microfuge 22R臺式微量冷凍離心機 美國Beckman公司;TP600型梯度PCR儀 日本TaKaRa公司;JS-380C全自動數碼凝膠成像分析儀 上海培清科技有限公司;S4800型掃描電鏡 日本日立高新技術公司。
1.2酵母菌分離純化培養基
PDA分離培養基(1 L):將土豆去皮并稱取200 g切碎,自來水1 L煮沸約30 min,8層紗布過濾,濾汁定容至1 L,加入葡萄糖20.0 g、瓊脂粉20.0 g、NaCl 30.0.0 g混勻,121 ℃、30 min高壓滅菌備用。
孟加拉紅培養基(1 L):加入蛋白胨5.0 g、葡萄糖10.0 g、KH2PO41.0 g、MgSO4·7H2O 0.5 g、瓊脂粉20.0 g、1/3000孟加拉紅水溶液100 mL、自來水900 mL混勻,121 ℃、30 min高壓滅菌備用。
YPD培養基(1 L):加入葡萄糖20.0 g、酵母粉10.0 g、蛋白胨20.0 g、瓊脂粉20.0 g,自來水定容至1 L溶解混勻,121 ℃、30 min高壓滅菌備用。
1.3菌株分離
將剪刀、研缽、鑷子紫外殺菌,在無菌條件下,用剪刀將葡萄剪碎置于研缽中研磨,然后將研磨物全部歸入100 mL ddH2O,37 ℃、180 r/min搖床培養1 h,將葡萄混合物梯度稀釋,涂板至以上3種培養基(添加氯霉素終濃度100 μg/mL),30 ℃培養2~3 d,待平板上長出單菌落,挑取單菌落劃線純化,并在相應培養基斜面試管中劃線接種,置4 ℃保藏。
1.4形態學觀察
挑取劃線純化的單菌落,經戊二醛固定、乙醇脫水、真空干燥進行電鏡觀察,觀測參數為加速電壓:15 kV,樣品臺:三軸馬達臺。
1.5基因組DNA的提取
從斜面保種中挑取單菌落至相應培養基劃線活化,于30 ℃恒溫培養箱中培養2~3 d。基因組DNA的提取嚴格按照真菌基因組試劑盒(Fungal DNA Kit 50)說明書操作。提取的基因組DNA在1%瓊脂糖凝膠中電泳檢測(110 V,40 min),溴化乙錠(EB)染色。4 ℃保存備用,或于-20 ℃中長期保存。
1.6基因片段PCR擴增
1.6.1 26S區域PCR擴增 26S區域擴增選擇酵母菌通用擴增引物NL-1(5′-GCATATCAATAA GCGGAGGAAAAG-3′)/NL-4(5′-GGTCCGTGTTTC AAGACGG-3′),PCR反應條件為94 ℃預變性5 min,94 ℃變性1 min,52 ℃退火1 min,72 ℃延伸90 s,共36個循環,最后72 ℃延伸10 min[17]。
1.6.2 ITS區域PCR擴增 ITS區域的擴增選擇真核生物ITS通用擴增引物ITS1(5′-TCCGTA GGTGAACCTGCGG-3′)/ITS4(5′-TCCTCCGCTTAT TGATATGC-3′)[18],PCR反應條件為94 ℃預變性2 min,94 ℃變性30 s,59 ℃退火30 s,72 ℃延伸90 s,共35個循環,最后72 ℃延伸7 min[19]。
1.6.3 Actin區域PCR擴增 Actin區域的擴增引物Act-HV-F(5′-TGGTGGAGCAATGATTTTGA-3′)/Act-HV-R(5′-GTTGTTGATGAAGCCCAATC-3′),PCR反應條件為94 ℃預變性5 min,94 ℃變性30 s,63 ℃退火30 s,72 ℃延伸2 min,共35個循環,最后72 ℃延伸7 min[20]。
PCR擴增反應均采用25 μL反應體系:ddH2O 9.5 μL、2×taq Mix 12.5 μL、10 μmol/L Primer-F 1 μL、10 μmol/L Primer-R 1 μL、DNA 1 μL。1%瓊脂糖凝膠電泳檢測PCR擴增產物,置于4 ℃保存備用。
1.7基因序列TA克隆
采用DP1502多功能DNA純化回收試劑盒回收PCR產物,純化的PCR產物與pMD19-T載體連接12 h,然后轉化至-70 ℃保存的100 μL感受態細胞大腸桿菌DH5α中,冰中放置30 min,取出后42 ℃熱激90 s,迅速加入預熱的890 μL LB液體培養基,37 ℃、200 r/min振蕩培養1 h。取適量菌液均勻涂布于含氨芐青霉素Amp(100 ng/mL)的LB固體培養基中,平板倒置過夜培養12~16 h,用滅菌牙簽挑取白色單菌落于1 mL LB液體中37 ℃、200 r/min過夜培養。取2 μL菌液直接做PCR,引物為M13-47和M13-RV,1%瓊脂糖凝膠電泳檢測是否含有目的片段。PCR采用25 μL的反應體系:ddH2O 8.5 μL、2×taq Mix 12.5 μL、10 μmol/L Primer-F 1 μL、10 μmol/L Primer-R 1 μL、菌液2 μL[21]。
1.8測序及系統發育學分析
將含有目的條帶的菌液送上海杰李生物科技有限公司測序。NCBI(https://blast.ncbi.nlm.nih.gov/Blast.cgi)BLAST進行同源性檢索,下載與該序列相似性較高的核酸序列。使用Mega 6.0軟件,采用鄰接法(neighbor joining method)構建系統發育樹。
2 結果與分析
2.1菌株表型特征鑒定
從以上三種培養基共分離得到10株酵母菌,其中孟加拉紅培養基分離得到6株、PDA培養基分離得到2株、YPD培養基分離得到2株。但分子鑒定去除重復后,僅有3株菌株。掃描電鏡結果(圖1)顯示:YYMPT-1酵母細胞呈圓形、臘腸形、皺縮扁長、多邊芽殖,與鑒定手冊[22-23]中Hanseniaspora屬的描述特征基本一致,初步鑒定屬于Hanseniaspora屬。YYMPT-2呈豆形或腎形,與鑒定手冊中Sporidiobolus屬的描述特征一致,初步鑒定屬于Sporidiobolus屬。YYMPT-3呈橢圓型、卵型或長型,各細胞間僅以狹小的面積相連,為藕節狀的假菌絲,與鑒定手冊中Candida屬的描述特征一致,初步鑒定為Candida屬。

圖1 3株分離酵母菌的掃描電子顯微鏡圖片
2.2菌株分子生物學特征鑒定
2.2.1 菌株26S、ITS、Actin序列擴增 菌株經PCR擴增獲得的26S序列全長572 bp,擴增ITS序列全長為500 bp左右,擴增Actin序列全長為800 bp左右(圖2)。
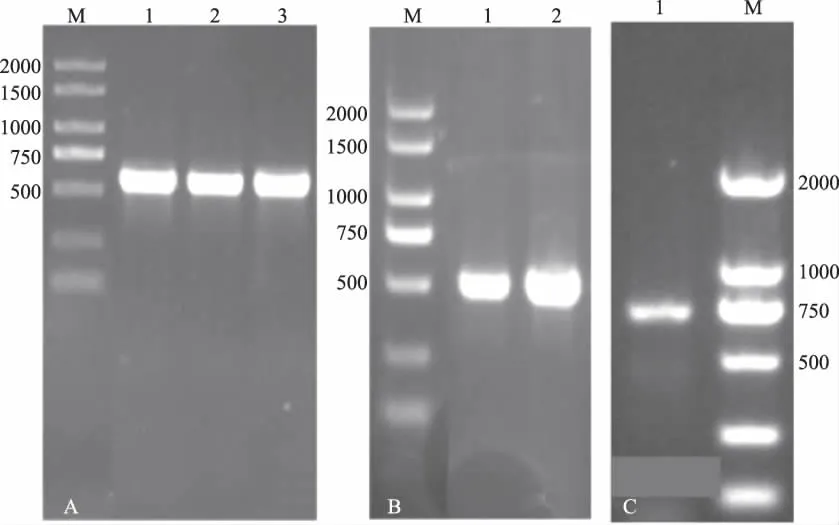
圖2 26S rDNA、ITS、Actin基因片段擴增電泳圖譜
2.2.2 菌株YYMPT系統發育學分析 將菌株YYMPT-1序列在NCBI中進行BLAST比對。結果顯示其26S序列與H.uvarum的26S同源性最高,相似度達100%,選取其中同源性較高菌株的26S基因序列,使用MEGA 6.0軟件構建系統發育樹,從26S系統發育樹(圖3)中可以看出,菌株YYMPT-1(KY952908)與H.uvarum(U84229)聚為一枝,自展值為93,表現出相當近的親緣關系;ITS序列與H.uvarum的ITS同源性最高,相似度達100%,選取其中同源性較高菌株的ITS基因序列,使用MEGA 6.0軟件構建系統發育樹,從ITS系統發育樹(圖4)中可以看出,菌株YYMPT-1(KY952911)與H.uvarum(AJ512432)聚為一枝,自展值為99,表現出相當近的親緣關系;Actin序列與H.uvarum的Actin同源性最高,相似度達100%,選取其中同源性較高菌株的ITS基因序列,使用MEGA 6.0軟件構建系統發育樹,從Actin系統發育樹(圖5)中可以看出,菌株YYMPT-1(KY952913)與H.uvarum(AM039456)聚為一枝,自展值為100,表現出相當近的親緣關系。
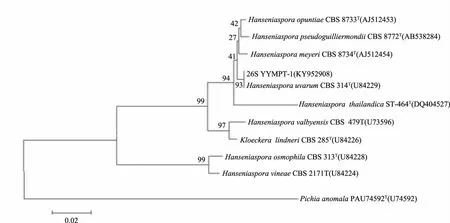
圖3 YYMPT-1基于26S rDNA基因序列構建的系統發育樹
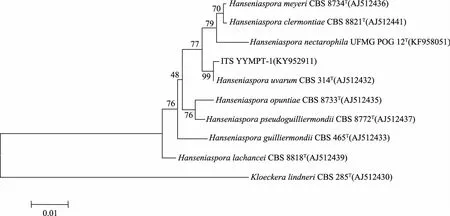
圖4 YYMPT-1基于ITS rDNA基因序列構建的系統發育樹
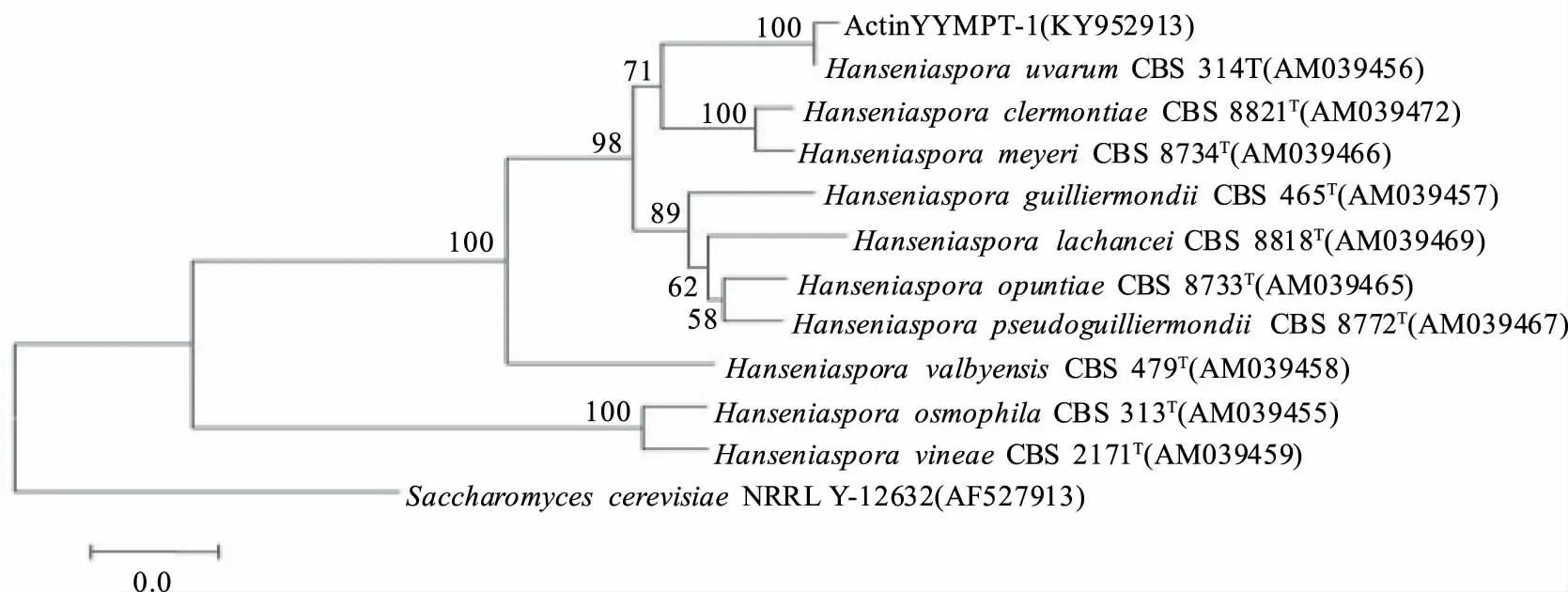
圖5 YYMPT-1基于Actin基因rDNA基因序列構建的系統發育樹
綜合26S、ITS和Actin基因序列分析結果及菌落表型特征,且與酵母菌鑒定手冊標準菌株對照后,將菌株YYMPT-1鑒定為葡萄有孢漢遜酵母(H.uvarum)。按照同樣的方法,通過表型特征及分子生物學鑒定,將菌株YYMPT-2和YYMPT-3分別鑒定為近玫色鎖擲酵母(S.pararoseus)和假絲酵母(C.zemplinina)。
3 討論
傳統的酵母菌鑒定方法主要依據形態學或生理生化分析,但由于培養時長及培養基和培養條件的改變,容易造成鑒定結果的不穩定或不確定性。相較于菌落表型觀察,分子生物學研究酵母的基因型,以酵母菌26S、ITS、Actin基因保守序列為研究靶點,更能反映菌株的遺傳本質,提供基因水平的鑒定信息,使鑒定結果更具準確性和遺傳特異性。隨著酵母數據庫信息量的不斷擴大與公開,分子生物學分析手段越來越成為酵母鑒定的常規工具。酵母菌26S rDNA位于核糖體大亞基5′端,其中D1/D2兩個區域主要用于系統發育研究和菌種鑒定[24-25]。Kurtzman和Rohbnett 等[26]對子囊菌綱的假絲酵母26S區域進行測序,認為堿基差異在1%以上就為不同的種;核糖體內轉錄間隔區(ITS),是一段非編碼區域,由位于18S rDNA和5.8S rDNA之間的ITS1區及5.8S rDNA和28S rDNA之間的ITS2區組成,該序列區種內高度保守,種間存在較大差異,為酵母菌鑒定提供豐富的遺傳信息;Actin是普遍存在于真核細胞中的微絲構造蛋白,其核苷酸序列在種內也高度保守,廣泛應用于酵母菌的鑒定。目前,26S、ITS及Actin區域序列分析已經成為最常用的DNA序列分析方法,其鑒定酵母菌快速準確,使酵母菌的分類鑒定從一般表型特征鑒定深化為分子生物學特征鑒定[27-28]。
本研究采用微生物表型觀察和PCR擴增26S、ITS、Actin基因片段,將分離自夏黑葡萄表皮中的3株酵母菌分別鑒定為有孢漢遜酵母(H.uvarum)、近玫色鎖擲酵母菌(S.pararoseus)和假絲酵母(C.zemplinina)。有孢漢遜酵母和假絲酵母在葡萄酒制備過程中,對葡萄酒的色澤、香味、口感等有重要影響,是最重要的微生物菌群。有孢漢遜酵母可發酵產生更高的乙酸酯類化合物,如乙酸苯乙酯和乙酸異戊酯,提高葡萄酒的香氣[29]。假絲酵母可以產生大量的甘油、琥珀酸和醋酸,不僅賦予葡萄酒柔和性、圓潤感和粘稠度,而且使葡萄酒具有強烈的蜂蜜、杏、和乙酸乙酯香[30]。
4 結論
本研究從夏黑葡萄中分離到3株葡萄野生酵母YYMPT-1、YYMPT-2和YYMPT-3,PCR擴增3株菌DNA的26S、ITS和Actin基因序列,構建系統發育樹,結合形態學觀察,將3株酵母菌分別鑒定為有孢漢遜酵母(H.uvarum)、近玫色鎖擲酵母菌(S.pararoseus)和假絲酵母(C.zemplinina)。本研究結果為葡萄酒釀造和改善葡萄酒風味提供了3株具有潛在工業化應用價值的資源菌,今后將對3株菌的理化性質及其在葡萄酒釀造過程中的作用進行深入研究,為改善葡萄酒品質提供科學依據。
[1]謝俊云,姚笛,郭瑜,等. 貝達葡萄酒中酵母菌的分離鑒定[J]. 農產品加工,2016(1):19-21.
[2]李鳳梅,譚婷婷,徐麗,等. 葡萄自然發酵過程中酵母菌和細菌的篩選、鑒定及系統發育分析[J]. 釀酒科技,2015(3):53-57.
[3]張春芝,莫寅斌. 寧夏產區釀酒葡萄酵母菌初步分類鑒定及多樣性研究[J]. 中國釀造,2014(10):49-54.
[4]蘇龍,劉樹文,何玲,等. 東北山葡萄酒自然發酵酵母菌群的研究[J]. 食品與生物技術學報,2007,26(3):110-115.
[5]公維庶. 釀酒品種“夏黑”寒地生產技術[J]. 黑龍江生態工程職業學院學報,2013(3):23-24.
[6]魯海東,倪天飛,楊義忠. 夏黑葡萄特征特性及栽培技術[J]. 現代農業科技,2011(9):120.
[7]楊靜靜,孟鎮,鐘其頂,等. 黃酒生產中酵母菌多相鑒定技術研究[J]. 中國釀造,2011(8):53-56.
[8]李丙超,胡衛東,唐文才,等. 傳統發酵香腸中菌種的分子生物學鑒定[J]. 食品與機械,2015(3):20-22.
[9]盧君,李艷. 分子生物學技術在葡萄酒相關酵母菌分類鑒定中的應用及研究進展[J]. 釀酒科技,2009(6):92-94.
[10]王會會.煙臺干紅葡萄酒發酵過程酵母菌群研究[D]. 濟南:山東輕工業學院,2011.
[11]范丙友,李芳,張文婷,等. 芍藥肌動蛋白基因的克隆及表達分析[J]. 中草藥,2013(15):2136-2142.
[12]Scorzetti G,Fell JW,Fonseca A,et al. Systematics of basidiomycetous yeasts:a comparison of large subunit D1/D2 and internal transcribed spacer rDNA regions[J]. FEMS Yeast Res,2002,4(2):495-517.
[13]張欣,劉勇,李金霞,等. 中性蛋白酶生產菌種AS1.398的多相復核鑒定[J]. 食品與發酵工業,2015(5):54-58.
[15]白飛榮,劉洋,李輝,等. 香山黃櫨枯萎病葉片真菌的分離與鑒定[J]. 生物學通報,2014(5):51-53.
[16]劉洋,白飛榮,姚粟,等. 兩株西沙群島野生諾尼種子內生酵母CICC32991與CICC32993的多相分類鑒定[J]. 中國真菌學雜志,2014(4):199-202.
[17]Guo J,Wang H,Liu D,et al. Isolation ofCunninghamellabigeloviisp nov CGMCC 8094 as a new endophytic oleaginous fungus fromSalicorniabigelovii[J]. Mycological Progress,2015,14(113).
[18]McCullough MJ,Clemons KV,McCusker JH,et al. Intergenic transcribed spacer PCR ribotyping for differentiation ofSaccharomycesspeciesand interspecific hybrids[J]. Journal of Clinical Microbiology,1998,36(4):1035-1038.
[19]燕勇,李衛平,高雯潔,等. rDNA-ITS序列分析在真菌鑒定中的應用[J]. 中國衛生檢驗雜志,2008(10):1958-1961.
[20]Neza C,Peter R,Maudy T S. Phylogenetic placement of Hanseniaspora-Kloeckera species using multigene sequence analysis with taxonomic implications:descriptions ofHanseniasporapseudoguilliermondiisp. nov. andHanseniasporaoccidentalis var. citrica var. nov.[J]. International Journal of Systematic and Evolutionary Microbiology,2006,56:1157-1165.
[21]曹娜,湛東銳,李連強,等. 分子生物學與形態學相結合鑒定鹽生海蘆筍內生真菌Salicorn 15[J]. 食品科學,2012(23):180-183.
[22]KREGER-VAN R N J W. The yeasts:a taxonomic study[M]. Amsterdam:Elsevier Science Publishers BV,1984:130-137,585,766-768.
[23]孫萬儒.酵母菌[J]. 生物學通報,2007(11):5-10.
[24]劉寧,劉延琳.核糖體RNA基因在酵母分類鑒定中的應用[J].中國農業科學,2010(22):4701-4708.
[25]杭姣,陳亞利,陳可泉,等. 酵母核酸常用的鑒定方法及其應用[J]. 食品科技,2014(12):2-7.
[26]Kurtzman CP,Robnett CJ. Identification and ascomycetous yeasts from analysis of nuclear large subunit(26S)ribosomal DNA partial sequences[J]. Antonie van Leeuwenhoek,1998,73:331-371.
[27]王鳳梅,馬利兵,潘建剛.分子生物學技術在酵母菌鑒定中的應用[J].綜述與述評,2009(10):1-4.
[28]楊靜靜,孟鎮,鐘其頂,等.分子生物學技術在酵母菌多相分類鑒定中的應用[J].中國釀造,2011(4):16-20.
[29]郝瑞穎,王肇悅,張博潤,等.葡萄酒中釀酒酵母產生的重要香氣化合物及其代謝調控[J].中國食品學報,2012(11):121-127.
[30]李婧,羅蘇蘇,張曉旭.葡萄相關酵母多樣性及其菌種鑒定的研究進展[J]. 食品工業科技,2016(24):391-394.
IsolationofepiphyticyeastsfromtheXiaheigrapeanditsidentificationbypolyphasicprocedure
YANGYu-meng,XUJing-guo,HUYi-min,HUANGSi-qi,SHIYa-ning,XINZhi-hong*
(College of Food Science and Technology,Nanjing Agricultural University,Nanjing 210095,China)
Wild yeast strains are important resources for brewing wine. In this study,in order to find valuable wild yeast strains,streak plate separation procedures were used and three strains,YYMPT-1,YYMPT-2 and YYMPT-3,were obtained using Xiahei grape as object. They were identified asHanseniasporauvarum,SporidioboluspararoseusandCandidazemplininaby amplifying D1/D2 domain of 26S ribosomal gene(26S rDNA),internal transcribed spacer sequence(ITS1-5.8S-ITS2)and actin gene by polymerase chain reaction(PCR),and established their phylogenetic tree,as well as observed their morphological features by scanning electron microscopy(SEM). The results from the current research provided 3 potential strains used for industrial applications for the production and improvement of wine quality with desirable flavor.
yeast;morphological features;phylogenetic tree;polyphasic identification
2017-05-18
楊雨蒙(1991-),女,碩士研究生,研究方向:食品營養與化學,E-mail:2015108059@njau.edu.cn。
*
辛志宏(1974-),男,博士,教授,研究方向:食品營養與化學,E-mail:xzhfood@njau.edu.cn。
2017年農產品質量安全風險評估項目(GJFP201701101);江蘇省“青藍工程”。
TS255.1
A
1002-0306(2017)22-0100-06
10.13386/j.issn1002-0306.2017.22.020

