鴨坦布蘇病毒甲基轉(zhuǎn)移酶的序列分析和結(jié)構(gòu)預(yù)測(cè)
武學(xué)東,張悅天,賈仁勇,3
鴨坦布蘇病毒甲基轉(zhuǎn)移酶的序列分析和結(jié)構(gòu)預(yù)測(cè)
武學(xué)東1,2,張悅天1,2,賈仁勇1,2,3
為了確定鴨坦布蘇病毒(duck Tembusu virus, DTMUV)發(fā)揮甲基轉(zhuǎn)移酶(methyltransferase, MTase)功能的氨基酸序列。本研究運(yùn)用生物信息學(xué)方法,以已知的黃病毒MTase的序列和結(jié)構(gòu)為模板,對(duì)GenBank上注入的DTMUV MTase序列進(jìn)行同源比對(duì)分析和結(jié)構(gòu)預(yù)測(cè)。分析結(jié)果表明,DTMUV MTase與WNV,DENV-2,JEV三種病毒MTase的核酸序列同源性分別為64.76%,64.55%,67.33%,氨基酸序列同源性分別為74.71%,68.45%,75.15%;其可能結(jié)構(gòu)含有SAM結(jié)合位點(diǎn)且具有典型的4個(gè)α螺旋和7個(gè)β折疊的黃病毒MTase的結(jié)構(gòu)特點(diǎn);同時(shí),DTMUV MTase序列上存在黃病毒MTase經(jīng)典保守位點(diǎn)K-D-K-E;從而為確定DTMUV MTase屬于黃病毒MTase家族成員提供一些理論依據(jù)。
鴨坦布蘇病毒;甲基轉(zhuǎn)移酶;序列分析;結(jié)構(gòu)預(yù)測(cè)
黃病毒RNA具有5′帽子結(jié)構(gòu),由于病毒在細(xì)胞質(zhì)中復(fù)制而無(wú)法利用宿主內(nèi)核加帽機(jī)制,這些病毒已經(jīng)進(jìn)化出了自己的甲基轉(zhuǎn)移酶來(lái)進(jìn)行基因組RNA的N7和2′-O甲基化。黃病毒NS5蛋白的N端約300個(gè)氨基酸是編碼MTase的區(qū)域,催化病毒RNA帽子結(jié)構(gòu)中腺苷酸核糖的N7和2′-O甲基化。N7和2′-O甲基化是基因組5′帽子結(jié)構(gòu)形成過(guò)程中所必需的[1]。兩步甲基化按照GpppA-RNA →m7Gppp A-RNA→m7Gppp Am-RNA先進(jìn)行N7甲基化后進(jìn)行2′-O甲基化,從而形成Ⅰ型帽子結(jié)構(gòu)[2]。
鴨坦布蘇病毒(DTMUV)屬于黃病毒科,黃病毒屬,基因組5′端具有Ⅰ型帽子結(jié)構(gòu),3′端無(wú)polyA尾巴結(jié)構(gòu)[3-5]。在形成帽子結(jié)構(gòu)過(guò)程中,必然有甲基轉(zhuǎn)移酶(MTase)的參與。而DTMUV發(fā)揮MTase功能的蛋白序列還未確定。目前,DENV,WNV,YFV等黃病毒的MTase晶體結(jié)構(gòu)均已被解析[6-8],從而可以為DTMUV MTase的序列分析和結(jié)構(gòu)預(yù)測(cè)提供參考。晶體結(jié)構(gòu)顯示,黃病毒的MTase包括核心區(qū),N端和C端的亞結(jié)構(gòu)域。本研究參照其他黃病毒,運(yùn)用生物信息學(xué)方法對(duì)GenBank上標(biāo)注的DTMUV MTase序列進(jìn)行分析和結(jié)構(gòu)預(yù)測(cè),對(duì)其發(fā)揮MTase功能的真實(shí)性和可靠性做出理論判斷。進(jìn)而,為DTMUV MTase的研究提供理論依據(jù)。
1 材料與方法
1.1DTMUV基因序列 DTMUV基因序列為本實(shí)驗(yàn)室分離的CQW1株DTMUV基因序列,序列號(hào)為KM233707.1。
1.2DTMUV MTase核酸序列同源比對(duì)分析 利用DNAMAN軟件對(duì)CQW1株DTMUVMTase對(duì)應(yīng)的核酸序列和在GenBank中隨機(jī)抽取的DENV,JEV,WNV 3種病毒MTase對(duì)應(yīng)的核酸序列進(jìn)行同源比對(duì)分析。
1.3DTMUV MTase氨基酸序列的同源比對(duì)分析 利用DNAMAN軟件對(duì)CQW1株DTMUVMTase對(duì)應(yīng)的氨基酸序列和在GenBank中隨機(jī)抽取的DENV,JEV,WNV 3種病毒MTase對(duì)應(yīng)的氨基酸序列進(jìn)行同源比對(duì)分析。
1.4DTMUV MTase結(jié)構(gòu)預(yù)測(cè) 利用SWISS-MODEL在線軟件和PyMOL軟件對(duì)DTMUV MTase的三級(jí)結(jié)構(gòu)進(jìn)行預(yù)測(cè)。
2 結(jié) 果
2.1DTMUV基因組結(jié)構(gòu) DTMUV為黃病毒科,黃病毒屬,恩塔雅病毒群。DTMUV病毒基因組為單股正鏈RNA,有一個(gè)ORF框編碼3種結(jié)構(gòu)蛋白和7種非結(jié)構(gòu)蛋白以及一個(gè)含有23aa的2K蛋白。基因組5′端具有Ⅰ型帽子(m7GpppAmG)結(jié)構(gòu),其非編碼區(qū)長(zhǎng)為94 nt;3′端不具有polyA尾巴結(jié)構(gòu),其非編碼區(qū)長(zhǎng)為618 nt[3-4,9]。如圖1所示。
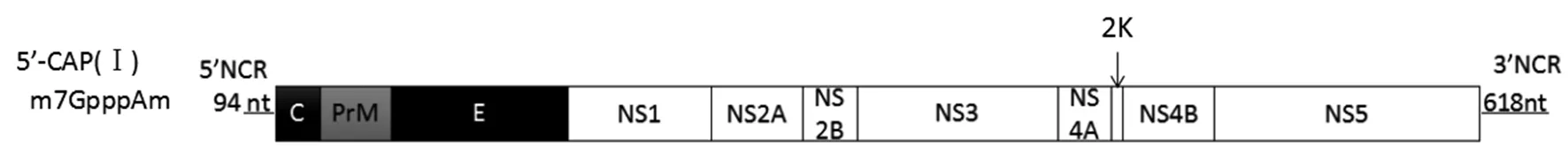
圖1 DTMUV基因組結(jié)構(gòu)Fig.1 Structure of the genome of DTMUV
2.2DTMUV MTase核酸序列同源比對(duì)分析 由本實(shí)驗(yàn)室分離的CQW1株DTMUVMTase對(duì)應(yīng)的核酸序列和在GenBank中隨機(jī)抽取的DENV,JEV,WNV 3種病毒MTase對(duì)應(yīng)的核酸序列利用DNAMAN軟件進(jìn)行同源比對(duì)分析。根據(jù)比對(duì)結(jié)果發(fā)現(xiàn)四種病毒的MTase核酸同源性為79.04%,并且MTase的K61-D146-K182-E218經(jīng)典保守位點(diǎn)[1,10-11]對(duì)應(yīng)的密碼子均相同(圖2)(注:aaa和aag均能編碼K賴氨酸)。同時(shí),利用DNAMAN對(duì)4種病毒MTase核酸序列兩兩比對(duì)后,同源性分別為63.58%,70.87%,64.76%,65.54%,64.55%,67.33%(圖4)。

注:***標(biāo)注為K61-D146-K182-E218對(duì)應(yīng)的密碼子*** label to mark codons of K61-D146-K182-E218圖2 MTase核酸序列同源比對(duì)分析Fig.2 Homologous analysis of MTase nucleic acid sequences
2.3DTMUV MTase氨基酸序列的同源比對(duì)分析
同樣,由本實(shí)驗(yàn)室分離的CQW1株DTMUVMTase對(duì)應(yīng)的氨基酸序列和從GenBank中隨機(jī)抽取的DENV,JEV,WNV 3種病毒MTase對(duì)應(yīng)的氨基酸序列利用DNAMAN進(jìn)行比對(duì)分析。根據(jù)序列比對(duì)分析結(jié)果發(fā)現(xiàn)4種病毒的MTase氨基酸序列同源性較高為82.65%,經(jīng)典保守位點(diǎn)K61-D146-K182-E218在4種病毒MTase氨基酸序列中均一致,非常保守(圖3)。同時(shí),利用DNAMAN對(duì)四種病毒MTase氨基酸序列兩兩比對(duì)后,同源性分別為68.82%,81.18%,70.83%,74.71%,68.45%,75.15%(圖4)。

圖3 MTase氨基酸序列同源比對(duì)分析Fig.3 Homologous analysis of MTase amino acid sequences
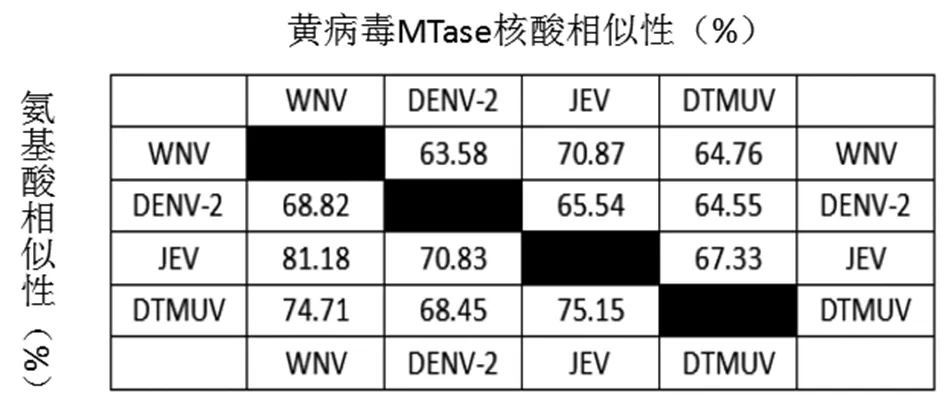
圖4 黃病毒MTase核酸和氨基酸相似性Fig.4 Similarity of flaviviruses MTase in nucleotide and protein level
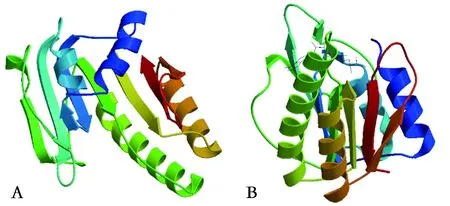
注:A: DTMUV; B: WNV(下同)A: DTMUV; B: WNV (The same below)圖5 MTase核心區(qū)的三級(jí)結(jié)構(gòu)Fig.5 Tertiary structure of MTase core region
2.4DTMUV MTase結(jié)構(gòu)預(yù)測(cè) 利用SWISS-MODEL和PyMOL軟件預(yù)測(cè)了DTMUV MTase序列的三級(jí)結(jié)構(gòu)。結(jié)果顯示,它展示的是DTMUV MTase的核心區(qū)結(jié)構(gòu),該區(qū)域是其功能區(qū),與其他黃病毒MTase核心區(qū)的三級(jí)結(jié)構(gòu)十分相似。DTMUV MTase的可能結(jié)構(gòu)為4個(gè)α螺旋和7個(gè)β折疊,屬于黃病毒MTase的特有結(jié)構(gòu),與DENV,WNV,JEV等黃病毒MTase核心區(qū)的結(jié)構(gòu)基本一致(圖5)。黃病毒MTase含有SAM結(jié)合位點(diǎn),在甲基化過(guò)程中作為甲基供體的結(jié)合區(qū)域[8,12-13]。同時(shí),SWISS-MODEL軟件預(yù)測(cè)結(jié)果顯示了在DTMUV MTase結(jié)構(gòu)上同樣含有SAM結(jié)合位點(diǎn)相似區(qū),可推測(cè)該區(qū)域?yàn)榧谆^(guò)程中作為甲基供體的結(jié)合區(qū)域。在大多數(shù)細(xì)胞和病毒中,2′-O甲基轉(zhuǎn)移酶的活性部位存在K-D-K-E氨基酸四分體且高度保守,它們?cè)谌?jí)結(jié)構(gòu)上聚集在一起形成活性口袋。同樣的,黃病毒MTase在結(jié)構(gòu)上具有由經(jīng)典保守氨基酸四分體(K61-D146-K182-E218)位點(diǎn)形成的活性口袋且高度保守[11,14-15]。而利用PyMOL軟件預(yù)測(cè)結(jié)果顯示,在DTMUV MTase可能結(jié)構(gòu)上也能發(fā)現(xiàn)氨基酸四分體K61-D146-K182-E218的存在,并在三級(jí)結(jié)構(gòu)上聚集在一起形成活性口袋(圖6)。
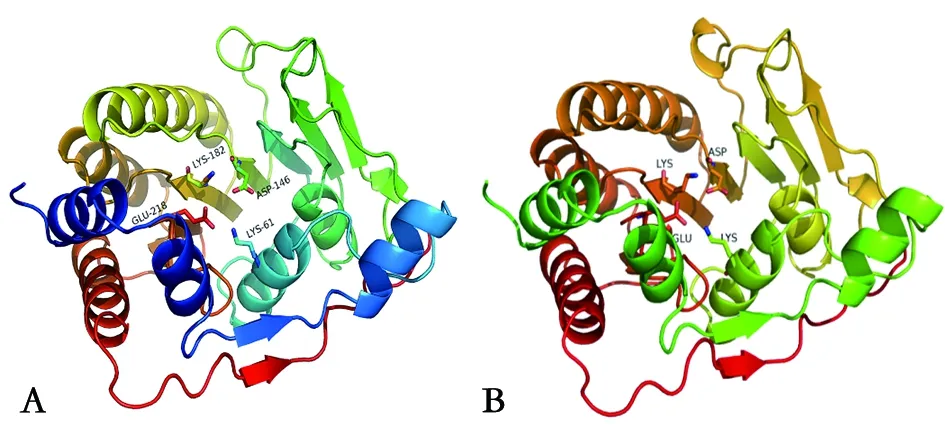
圖6 MTase的三級(jí)結(jié)構(gòu)Fig.6 Tertiary structure of MTase
3 討 論
黃病毒含有I型帽子結(jié)構(gòu)。因?yàn)辄S病毒RNA是在細(xì)胞質(zhì)中進(jìn)行復(fù)制而不能利用宿主的酶系統(tǒng),所以進(jìn)化出了自身的酶系統(tǒng),黃病毒的NS5蛋白N端具有甲基轉(zhuǎn)移酶功能區(qū)域,其結(jié)構(gòu)形成了黃病毒MTase的特有結(jié)構(gòu)特征。本研究將DTMUV與DENV-2,JEV,WNV的MTase核心區(qū)的核酸序列和氨基酸序列同源比對(duì)及兩兩比對(duì)分析,能夠得到較高的同源性。其構(gòu)成酶活口袋的關(guān)鍵位點(diǎn)K-D-K-E在DTMUV MTase的序列上也很保守,從核酸序列和氨基酸序列的同源比對(duì)分析上能夠初步提供DTMUV MTase屬于黃病毒MTase家族成員的一些理論依據(jù)。利用SWISS-MODEL和PyMOL軟件預(yù)測(cè)的DTMUV MTase核心區(qū)可能結(jié)構(gòu)具有典型的黃病毒MTase 4個(gè)α螺旋和7個(gè)β折疊的結(jié)構(gòu)特征,且含有一個(gè)SAM結(jié)構(gòu),進(jìn)一步為DTMUV MTase能夠發(fā)揮甲基轉(zhuǎn)移酶功能提供了一些理論依據(jù)。然而,DTMUV MTase是否具有MTase功能需要實(shí)質(zhì)性地去檢測(cè)其酶活性。目前DENV,JEV,WNV等[2,15-16]黃病毒的體外酶活性已經(jīng)被檢測(cè),為DTMUV MTase的酶活測(cè)定提供了參考。接下來(lái)的工作,我們將進(jìn)行體外測(cè)定DTMUV MTase的酶活活性以及功能相關(guān)研究。
近年來(lái),隨著黃病毒MTase的研究逐漸深入,黃病毒MTase發(fā)揮的功能,在病毒中充當(dāng)?shù)慕巧皩?duì)宿主的影響已經(jīng)有大量的研究報(bào)道。黃病毒MTase發(fā)揮功能的核心區(qū)是由SAM結(jié)合位點(diǎn)、RNA結(jié)合位點(diǎn)和GTP結(jié)合位點(diǎn)來(lái)執(zhí)行其功能,SAM結(jié)合位點(diǎn)作為甲基供體的結(jié)合區(qū)域,GTP結(jié)合區(qū)結(jié)合m7G,RNA結(jié)合位點(diǎn)結(jié)合未加帽的病毒RNA。當(dāng)SAM結(jié)構(gòu)類似物存在時(shí),會(huì)和SAM競(jìng)爭(zhēng)該區(qū)域,從而影響甲基化活性;同時(shí),該區(qū)域的保守位點(diǎn)突變會(huì)影響MTase活性[11,17]。GTP結(jié)合區(qū)既可以結(jié)合GTP的鳥(niǎo)苷部分進(jìn)行鳥(niǎo)苷酸轉(zhuǎn)移,也可以結(jié)合m7Gppp A-RNA的m7G進(jìn)行甲基化反應(yīng);該區(qū)域位點(diǎn)突變會(huì)影響2′-O甲基化,但不影響N7甲基化[12,14];同時(shí),在甲基化過(guò)程中,加入外源GTP會(huì)與m7G會(huì)發(fā)生競(jìng)爭(zhēng),相互拮抗對(duì)方結(jié)合GTP結(jié)合區(qū)[18]。RNA結(jié)合位點(diǎn)研究尚少,有報(bào)道稱在GTase反應(yīng)到MTase反應(yīng)時(shí),RNA需要重新定位[8]。在黃病毒MTase酶活口袋上存在經(jīng)典保守位點(diǎn)K-D-K-E氨基酸四分體,該四分體的突變會(huì)影響MTase的N7和2′-O甲基化功能,當(dāng)N7甲基化功能缺失后,突變病毒將不能復(fù)制[2];當(dāng)2′-O甲基化功能缺失后,突變病毒復(fù)制能力有所下降但不影響病毒增殖,會(huì)降低病毒毒力,接種小鼠后能產(chǎn)生很好的免疫原性,作為疫苗免疫小鼠能夠起到很好的保護(hù)效果[15-16]。最近又有重大發(fā)現(xiàn),MTase使病毒RNA形成與宿主細(xì)胞mRNA非常相似的帽子結(jié)構(gòu),從而導(dǎo)致宿主天然免疫系統(tǒng)無(wú)法識(shí)別和清除,形成天然免疫逃逸[19-20]。并且有研究表明,黃病毒2′-O甲基轉(zhuǎn)移酶甲基化形成5′帽子結(jié)構(gòu)的病毒能夠逃逸IFIT1的抗病毒作用[21]。從另一個(gè)角度發(fā)現(xiàn),IFIT1優(yōu)先結(jié)合2′-O未甲基化的病毒RNA,使真核翻譯起始因子無(wú)法結(jié)合病毒RNA,最終抑制病毒的復(fù)制和翻譯[22-23]。目前為止,黃病毒MTase的大量研究報(bào)道有效的闡明了MTase在病毒侵染與宿主抗病毒之間所起的重要作用;而鴨坦布蘇病毒MTase的研究將會(huì)豐富黃病毒MTase的研究?jī)?nèi)容。使用生物信息學(xué)的方法只能將前人大量的試驗(yàn)結(jié)果進(jìn)行總結(jié),從而便于對(duì)未知結(jié)構(gòu)和功能的蛋白質(zhì)進(jìn)行預(yù)測(cè)。但是,要驗(yàn)證鴨坦布蘇病毒MTase的具體功能還需具體試驗(yàn)數(shù)據(jù)來(lái)提供更為可靠的依據(jù)。
[1] Zhou Y, Ray D, Zhao Y, et al. Structure and function of flavivirus NS5 methyltransferase[J]. J Virol, 2007, 81(8):3891-3903.
[2] Ray D, Shah A, Tilgner M, et al. West Nile virus 5′-cap structure is formed by sequential guanine N-7 and ribose 2′-O methylations by nonstructural protein 5[J]. J Virol, 2006, 80(17): 8362-8370.
[3] Liu P, Lu H, Li S, et al. Duck egg drop syndrome virus: an emerging Tembusu-related flavivirus in China[J]. Sci China Life Sci, 2013, 56(8):701-710.
[4] Liu P, Lu H, Li S, et al. Genomic and antigenic characterization of the newly emerging Chinese duck egg-drop syndrome flavivirus: genomic comparison with Tembusu and Sitiawan viruses[J]. J General Virol, 2012, 93(10):2158-2170.
[5] Wang J, Liu W, Meng G, et al. Isolation and genome characterization of a novel duck Tembusu virus with a 74 nucleotide insertion in the 3′ non-translated region[J]. Avian Pathol, 2015, 44(2): 92-102.
[6] Geiss BJ, Thompson AA, Andrews AJ, et al. Analysis of flavivirus NS5 methyltransferase cap binding[J]. J Mol Biol, 2009, 385(5): 1643-1654.
[7] EgloffMP,BenarrochD,SeliskoB, et al. An RNA cap (nucleoside‐2′‐O‐)‐methyltransferase in the flavivirus RNA polymerase NS5: crystal structure and functional characterization[J]. Embo J, 2002, 21(11): 2757-2768.
[8] Yap LJ, Luo D, Chung KY, et al. Crystal structure of the dengue virus methyltransferase bound to a 5′-capped octameric RNA[J]. PLoS One, 2010, 5(9): e12836.
[9] Tang Y, Diao Y, Gao X, et al. Analysis of the complete genome of Tembusu virus, a flavivirus isolated from ducks in China[J]. Transbound Emerg Dis, 2012, 59(4):336-343.
[10] Dong H, Fink K, Züst R, et al. Flavivirus RNA methylation[J]. J General Virol, 2014, 95(4):763-778.
[11] Kroschewski H, Lim SP, Butcher RE, et al. Mutagenesis of the dengue virus type 2 NS5 methyltransferase domain[J]. J Biologic Chem, 2008, 283(283):19410-19421.
[12] Dong H, Ren S, Zhang B, et al. West Nile virus methyltransferase catalyzes two methylations of the viral RNA cap through a substrate-repositioning mechanism[J]. J Virol, 2008, 82(9): 4295-4307.
[13] Egloff MP, Decroly E, Malet H, et al. Structural and functional analysis of methylation and 5′-RNA sequence requirements of short capped RNAs by the methyltransferase domain of dengue virus NS5[J]. J Mol Biol, 2007, 372(3): 723-736.
[14] Dong HP, Chang DC, Xie XP, et al. Biochemical and genetic characterization of dengue virus methyltransferase[J]. Virology, 2010, 405(2):568-578.
[15] Züst R, Dong H, Li XF, et al. Rational design of a live attenuated dengue vaccine: 2′-O-methyltransferase mutants are highly attenuated and immunogenic in mice and macaques[J]. PLoS Pathog, 2013, 9(8): e1003521.
[16] Li SH, Dong H, Li XF, et al. Rational design of a flavivirus vaccine by abolishing viral RNA 2′-O methylation[J]. J Virol, 2013, 87(10): 5812-5819.
[17] Chung KY, Dong H, Chao AT, et al. Higher catalytic efficiency of N-7-methylation is responsible for processive N-7 and 2′-O methyltransferase activity in dengue virus[J]. Virology, 2010, 402(1):52-60.
[18] Benarroch D, Egloff MP, Mulard L, et al. A structural basis for the inhibition of the NS5 dengue virus mRNA 2′-O-methyltransferase domain by ribavirin 5′-triphosphate[J]. J Biologic Chem, 2004, 279(34):35638-35643.
[19] Daffis S, Szretter KJ, Schriewer J, et al. 2′-O methylation of the viral mRNA cap evades host restriction by IFIT family members[J]. Nature, 2010, 468(7322):452-456.
[20] Züst R, Cervantesbarragan L, Habjan M, et al. Ribose 2′-O-methylation provides a molecular signature for the distinction of self and non-self mRNA dependent on the RNA sensor Mda5[J]. Nat Immunol, 2011, 12(2):137-143.
[21] Szretter KJ, Daniels BP, Cho H, et al. 2′-O methylation of the viral mRNA cap by West Nile virus evades Ifit1-dependent and -independent mechanisms of host restriction in vivo[J]. PLoS Pathog, 2012, 8(5):e1002698.
[22] Habjan M, Hubel P, Lacerda L, et al. Sequestration by IFIT1 impairs translation of 2′O-unmethylated capped RNA[J]. PLoS Pathog, 2013, 9(10):e1003663.
[23] Kimura T, Katoh H, Kayama H, et al. Ifit1 inhibits Japanese encephalitis virus replication through binding to 5′ capped 2′-O unmethylated RNA[J]. J Virol, 2013, 87(18):9997-10003.
SequenceanalysisandstructurepredictionofthemethyltransferaseofduckTembusuvirus
WU Xue-dong1,2, ZHANG Yue-tian1,2, JIA Ren-yong1,2,3
(1.ResearchCenterforAvianDiseases,CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Chengdu611130,China;2.InstituteofPreventiveVeterinaryMedicine,CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Chengdu611130,China;3.KeyLaboratoryofAnimalDiseasesandHumanHealthofSichuanProvince,SichuanAgriculturalUniversity,Chengdu611130,China)
In order to determine the amino acid sequence of functionthat play a role of methyltransferase (MTase) activity in duck Tembusu virus (DTMUV),we have made sequence analysis and structure prediction of the MTase in DTMUV by bioinformatics methods, as well as using the sequence and structure of MTase in other flaviviruses which have been reported. The bioinformatical analysis results showed that the MTase and the MTase of other three kinds of flaviviruses had the higher homology in nucleotide sequence and amino acid sequence, for 64.76%, 64.55%, 67.33% and 74.71%, 68.45%, 75.15% respectively, and the same basic structural characteristics which contained SAM binding site and had a typical of 4 alpha screw and 7 beta folding. At the same time, there were classical conservative sites K-D-K-E of flavivirus MTase in DTMUV MTase sequence. In conclusion, they imply that the MTase of DTMUV might belong to MTase family of flaviviruses .
duck Tembusu virus; methyltransferase; sequence analysis; structure prediction
Jia Ren-yong, Email: jiary@sicau.edu.cn
10.3969/j.issn.1002-2694.2017.10.005
四川省科技廳應(yīng)用基礎(chǔ)研究項(xiàng)目資助 (No.2017JY0014)
賈仁勇,Email:jiary@sicau.edu.cn
1.四川農(nóng)業(yè)大學(xué)禽病防治研究中心, 成都 611130;
2.四川農(nóng)業(yè)大學(xué)預(yù)防獸醫(yī)研究所, 成都 611130;
3.動(dòng)物疫病與人類健康四川省重點(diǎn)實(shí)驗(yàn)室, 成都 611130
373
A
1002-2694(2017)10-0877-05
Funded by the Application and Base Research Project of Sichuan Science and Technology Department (No.2017JY0014)
2017-06-30編輯李友松

