基于機器學習的COX抑制劑預測模型研究
聶長森,白 勇,柳賢德
(1.海南大學 信息科學技術學院,海南 海口 570228;2.海南大學 農學院,海南 海口 570228)
基于機器學習的COX抑制劑預測模型研究
聶長森1,白 勇1,柳賢德2
(1.海南大學 信息科學技術學院,海南 海口 570228;2.海南大學 農學院,海南 海口 570228)
針對目前COX(環氧合酶)抑制劑較少且抑制效果差的問題,以及傳統的化學實驗篩選COX抑制劑分子的方法中成本高且效率低的問題,基于機器學習算法,提出并建立了一種COX抑制劑的預測模型。該模型可高效且準確地找到COX抑制劑,通過大量搜集文獻中的數據建立數據集,使用Mold2軟件計算化合物分子描述符,利用自組織特征映射神經網絡(SOM)劃分訓練集和測試集,應用隨機森林(RF)和支持向量機(SVM)等機器學習算法分別建立了COX抑制劑預測模型。實驗對比發現,SOM結合RF算法較傳統化學實驗方法具有更好的預測精度,且預測效率也有大幅提升。實驗研究表明,基于自組織神經網絡和隨機森立的機器學習方法建立的COX抑制劑預測模型,具有很好的分類預測效果,可以為COX抑制劑的分析與預測提供有力的研究工具。
COX抑制劑;機器學習方法;自組織特征神經網絡;隨機森林;支持向量機
0 引 言
如何高效地篩選具有高活性的COX抑制劑對于探索人體炎癥的治療具有非常重要的意義。環氧合酶是體內花生四烯酸代謝過程中最主要的限速酶,存在兩種亞型:原生型(COX-1)和誘導型(COX-2)[1-3]。COX-1存在于血管、腎臟和胃,具有生理保護作用,如維持胃腸道黏膜的完整性,調節腎血流量和血小板功能;COX-2是一種誘導酶,在組織損傷、炎癥時,細胞因子和其他炎性介質誘導激活炎癥部位COX-2,由此產生PGG2/PGH2,從而出現炎癥反應。人類對環氧合酶抑制劑的研究一直是藥物研究的熱點,自1999年第一代特異性COX-2抑制劑,即昔布類藥物—塞來昔布和羅非昔布先后在國外和國內上市以來,專家學者們對COX抑制劑的研究從未停止。現已證明,COX-1不僅參與炎癥并且有加重炎癥的作用,而COX-2似乎主要參與早期炎癥,而在慢性炎癥階段反而有抗炎作用。那么如何尋找一種COX抑制劑,對COX-1和COX-2都具有抑制作用,科學家們對此進行了大量研究。基本思想是基于分子描述符和機器學習算法,對前人實驗的數據進行分析,利用計算機高效提取COX抑制劑的特征,建立COX抑制劑的預測模型,并利用現有的COX抑制劑進行驗證。
設計一個高效的COX抑制劑預測模型的任務非常艱巨,雖然近年來科學家們對COX抑制劑的研究較多,但是已知的抑制劑化合物非常有限,所采用的化合物來源于文獻搜集,一共54個,對COX-1和COX-2具有有效的抑制作用。由于COX抑制劑的數據庫非常有限,給預測模型的建立帶來了很大的挑戰,但是機器學習算法憑借其優良的數據篩選特性,一直以來在化和物結構預測、藥代動力學、藥效動力學等方面均有非常好的效果。為此,在實驗中分別建立了隨機森林模型和支持向量機的COX抑制劑預測模型,并將自組織神經網絡(SOM)[4-8]分別與這兩種算法相結合,建立了四種預測模型,并進行了對比驗證。
1 材料與方法
分別采用SOM結合隨機森林(RF)及支持向量機(SVM)等機器學習算法建立了COX抑制劑的分析和預測模型。采用的實驗數據來源于文獻檢索的COX抑制劑,利用ChemBioDraw軟件繪制得到其二維(2D)結構,然后使用化合物分子格式轉換軟件openbabel將分子結構轉換為sdf格式進行保存,使用著名的Mold2軟件進行分子描述符的計算[9-13]。化合物量化處理后,利用SOM進行訓練集和測試集的劃分,然后使用隨機森林算法對訓練集進行學習,對測試集進行預測分析,通過與SVM算法的預測結果對比后發現,SOM結合RF算法的預測正確率較高。
1.1分子描述符
采用國家毒理學研究中心(NCTR)設計的Mold2軟件進行分子描述符的計算。每個化合物的分子描述符有777個數據,分別代表化合物的不同結構和屬性,由于許多文獻和書籍中都有詳細的描述[14],故只做簡單介紹。化合物的分子描述符可以分為經驗描述符和理論描述符。
經驗描述符來源于物質的實驗數據,如溶點、沸點等,因此有其自身的缺點,例如當化合物缺少相應的實驗值時,則不能進行QSAR研究。為確保實驗的有效性和預測的正確率,不采用經驗描述符。
1.2數據集
采用的數據集為54個COX抑制劑,全部從文獻中搜集,并且來源于同一個實驗室。這些抑制劑的IC50值范圍從小于0.05 μM到大于50 μM。因為這是對化合物進行分類預測,所以根據IC50值將所有抑制劑劃分為兩類:8個高活性類(IC50值低于1 μM)和46個低活性類(IC50值高于1 μM)。圖1列出了兩個代表性COX抑制劑的母本結構。
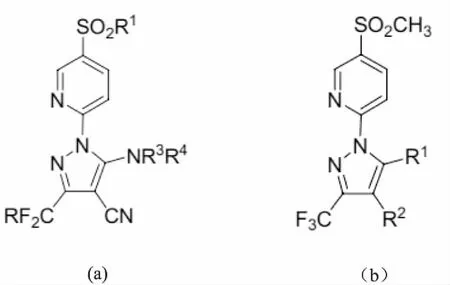
圖1 代表性COX抑制劑母本結構
1.3訓練集和測試集的劃分
利用計算機進行化合物的特征提取,并進行藥性預測,一個重要任務是要進行訓練集和測試集的劃分。訓練集作為輸入,由隨機森林等智能算法進行特征提取[15],并建立訓練模型。測試集利用建立好的模型進行預測,也就是進行特征匹配,以期得到好的預測效果。測試集是檢驗訓練模型預測結果與實驗結果是否一致的重要一步。所以訓練集和測試集的劃分是否合理,直接決定實驗結果的有效性。
訓練集和測試集的劃分方法主要有以下幾種:
(1)隨機劃分方法。
該方法是最簡單、最易實現也是使用最多的一種訓練集和測試集的劃分方法。在實現該算法時,只需利用編程語言中的隨機函數進行分類即可,但值得注意的是,由于訓練集和測試集的劃分,對實驗結果至關重要。使用該方法一般需要對訓練集和測試集進行多次劃分,然后取平均結果作為劃分模型。
基于現實調查的數據分析,我們對中小學師生的創新現狀進行調查,準確把握實驗起點。1998年9月,選取重慶42中、53中、沙坪壩區實驗一小起始年級班進行實驗前測。2001年,對重慶、新疆、廣東及我國香港地區的實驗學校進行大樣本調查,收到有效問卷47 548份,分析數據近1 000萬條,寫出了報告,得到了專家的認可。
(2)主成分分析(PCA)劃分方法。
主成分分析是用于數值分類研究的一種重要方法,目前的應用也較為廣泛。其基本思想是實現多維問題低維化,用二維或者三維歐氏空間的直觀散點圖來刻劃類群或作其他分析。但是實驗最重要的是根據化合物的分子描述符提取特征,并根據數據分布,使訓練集和測試集具有最相似的分布,以達到預期效果,所以對目前的實驗并不十分合適。
(3)SOM劃分方法[16]。
該網絡是一個由全連接的神經元陣列組成的無教師、自組織、自學習網絡。該網絡空間中不同區域的神經元具有各自不同的分工,可以根據輸入空間中的輸入向量進行學習和分類。SOM神經網絡是一種發展較為成熟,經過實驗驗證的人工智能算法,對訓練集和測試集的劃分取得了非常好的效果。
1.4機器學習方法
采用了兩種最新的機器學習算法—RF[17-22]和SVM[23-24],通過RF和SVM方法將COX抑制劑的預測轉化為一個二元的分類問題,即通過訓練集訓練后預測一種化合物是COX抑制劑或者不是COX抑制劑。對于RF和SVM算法的具體實現原理,這里不做詳述,只對其思想做簡單介紹。
RF是一種決策樹自然生長且很多個決策樹預測器組合在一起的分類方法[25]。每棵決策樹依賴于對輸入向量進行隨機獨立抽樣所獲得的數值,且森林中的所有決策樹都具有相同的分布。每棵樹都不受干涉地自然生長到最大規模,然后對一個新的數據點給出自己的預測。也就是說,這顆樹投票決定這一新數據點的類別。當大量的決策樹生成以后,整個森林就選擇最多數的投票結果作為對這個數據點類別的判定。
SVM是一種基于統計學習理論中結構風險最小化(SRM)原則的方法[26],而統計學習理論是一種著名的與核函數相關的機器學習方法。SVM方法通過使用核函數,把輸入變量投射到高維特征空間,然后從輸入向量中選擇一個所謂支持向量的小的子集。在變換后的空間中,通過最大間隔的原則構建一個最優化的分類超平面,從而把這些輸入向量分成了兩種不同的類別。
1.5特征選擇方法與模型建立
采用SOM、RF、SVM三種算法建立模型。其中自組織特征映射神經網絡是一種發展比較成熟的特征分類算法,主要作為訓練集和測試集的劃分模型。因為訓練集和測試集劃分的主要目的就是確保訓練集的點占據整個數據集空間,測試集的點接近訓練集的點。使用SOM神經網絡的方法對于獨立預測集進行選擇,這種方法是基于化合物的化學空間來選擇測試集分子。使用3×3的SOM神經網絡,把所有化合物映射到9個位置。相似的對象映射到相似的位置。在這個網絡中按照訓練集和測試集3∶2的比例進行選擇。訓練集用來建立分類模型,而測試集用來評估模型的預測能力。其中,訓練集包含33個分子(29個低活性,4個高活性),測試集包含21個分子(17個低活性,4個高活性)。然后分別建立隨機森林和支持向量機模型進行訓練和測試,并對兩個模型的預測結果進行對比。
2 實驗結果與分析
2.1模型的預測性能
對COX抑制劑進行預測,根據劃分的訓練集進行訓練,測試集的數據根據預測結果與實際化合物的活性是否相同,來對預測的正確率進行判斷。將預測正確率記為P,測試集高活性化合物數量為M,低活性化合物數量為N,預測正確的高活性化合物數量為m,預測正確的低活性化合物數量為n。則總預測正確率為:
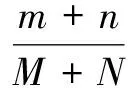
(1)
對高活性化合物的預測正確率為:
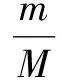
(2)
對低活性化合物的預測正確率為:
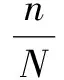
(3)
分別采用四種方法進行預測,正確率如表1所示。
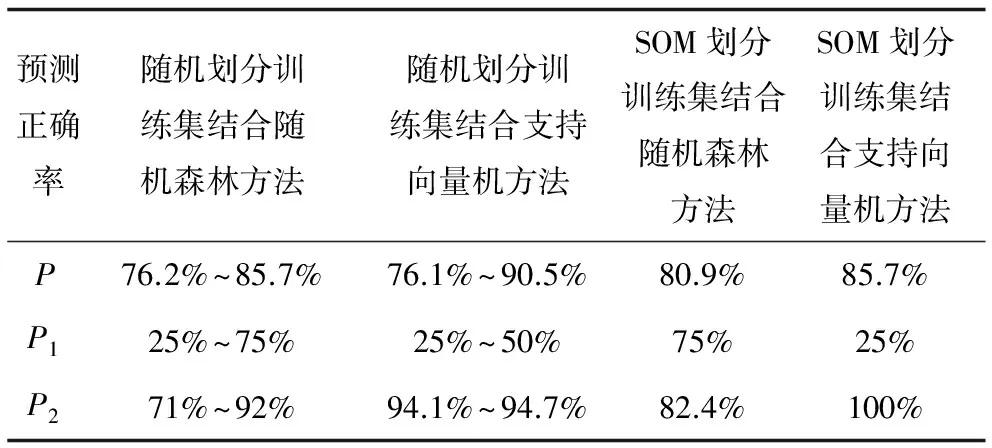
表1 四種預測模型正確率
2.2模型的分析
通過表1發現,訓練集和測試集的劃分方法,對實驗結果有著至關重要的作用。運用隨機劃分的方法,明顯出現了預測結果不穩定的現象,這主要是由于每次隨機劃分的訓練集和測試集不能各自完整地代表整個數據集的特征,并且每次實驗受劃分的高活性化合物和低活性化合物的數量影響較大,導致結果非常不穩定,預測正確率波動較大。而采用自組織特征映射神經網絡方法劃分訓練集和測試集,實驗結果比較穩定,而且SOM結合RF算法[27-29]的整體預測正確率在80.9%左右,SOM結合SVM算法的正確率穩定在85.7%左右。
可以發現,雖然SOM結合SVM算法的整體預測正確率較高[30-34],但是對于高活性化合物的預測正確率卻相當低,所以對于今后的研究幫助不是很大。SOM結合RF算法的預測正確率相對比較理想。
2.3模型的驗證
通過文獻調研查詢了15個COX抑制劑藥物,對研究建立的COX抑制劑預測模型進行驗證。首先使用ChemBioDraw軟件繪制這15個藥物的二維結構,然后使用openbabel軟件進行格式轉換,并使用Mold2軟件計算分子描述符。將這15個化合物的分子描述符輸入研究建立的基于SOM和隨機森林算法的預測模型進行預測,預測結果如表2所示。其中,預測結果‘1’表示該藥物為高活性,預測結果‘2’表示該藥物為低活性。
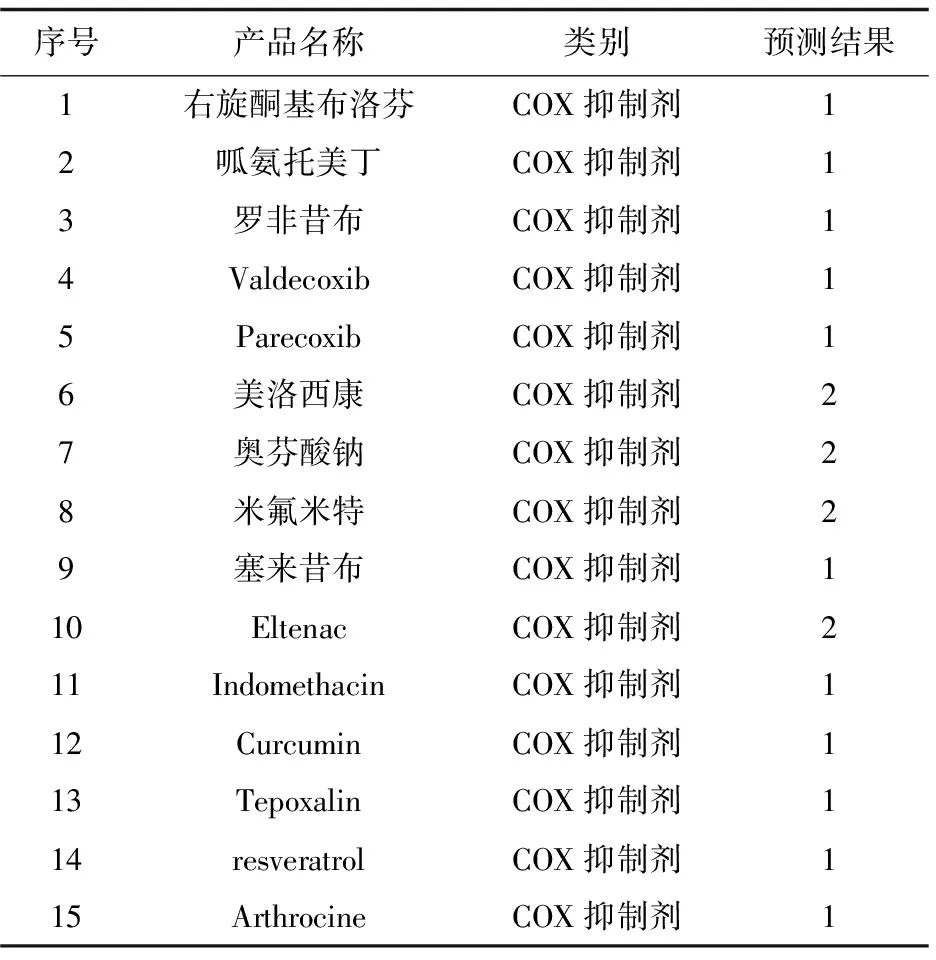
表2 COX抑制劑藥物預測結果
由預測結果可知,該模型將這15個化合物預測為高活性COX抑制劑,由于這些藥物已經上市并投入使用,多數是COX的高活性抑制劑。預測結果表明有73.3%的藥物為高活性,符合實際情況,表明該研究在COX抑制劑的預測方面具有重要的現實意義。
3 結束語
將分子描述符和機器學習方法應用于COX抑制劑,提出并建立了COX抑制劑的分類和預測模型。研究結果表明,相對于SOM結合SVM算法、隨機劃分訓練集結合RF算法、隨機劃分訓練集結合SVM算法,SOM結合RF的機器學習算法預測準確率高且效果好,同時還可節省大量時間和資源成本。
[1] Sakya S M,DeMello K M L,Minich M L,et al.5-heteroatom substituted pyrazoles as canine COX-2 inhibitors.Part 1:structure-activity relationship studies of 5-alkylamino py-razoles and discovery of a potent,selective,and orally active analog[J].Bioorganic & Medicinal Chemistry Letters,2006,16(2):288-292.
[2] Sakya S M,Cheng H,DeMello K M L,et al.5-heteroatom-substituted pyrazoles as canine COX-2 inhibitors. Part 2:structure-activity relationship studies of 5-alkylethers and 5-thioethers[J].Bioorganic & medicinal Chemistry Letters,2006,16(5):1202-1206.
[3] Sakya S M,Hou X,Minich M L,et al.5-heteroatom substituted pyrazoles as canine COX-2 inhibitors. Part III:molecular modeling studies on binding contribution of 1-(5-methylsulfonyl) pyrid-2-yl and 4-nitrile[J].Bioorganic & Medicinal Chemistry Letters,2007,17(4):1067-1072.
[4] Kobuchi Y, Tanoue M. Learning and forgetting-how they should be balanced in SOM algorithm[C]//IEEE international conference on neural networks.San Francisco,CA,USA:IEEE,2004:745-749.
[5] 姚登舉,楊 靜,詹曉娟.基于隨機森林的特征選擇算法[J].吉林大學學報:工學版,2014,44(1):137-141.
[6] 黃亞捷,葉回春,張世文,等.基于自組織特征映射神經網絡的中國耕地生產力分區[J].中國農業科學,2015,48(6):1136-1150.
[7] 謝倩倩,李訂芳,章 文.基于集成學習的離子通道藥物靶點預測[J].計算機科學,2015,42(4):177-180.
[8] 聶 斌,郝竹林,桂 寶,等.基于隨機森林的中藥寒、熱藥性代謝組學判別方法研究[J].江西中醫藥大學學報,2015(2):82-86.
[9] 閆樹英,陳志宏,惠 娜,等.基于RF和KNN的三種肝炎分類模型的建立[J].寧夏醫學雜志,2015,37(6):496-498.
[10] 劉建偉,劉 媛,羅雄麟.半監督學習方法[J].計算機學報,2015,38(8):1592-1617.
[11] 何 冰,羅 勇,李秉軻,等.基于分子描述符和機器學習方法預測和虛擬篩選乳腺癌靶向蛋白HEC1抑制劑[J].物理化學學報,2015,31(9):1795-1802.
[12] 任 偉,孔德信.定量構效關系研究中分子描述符的相關性[J].計算機與應用化學,2009,26(11):1455-1458.
[13] 郝 明.基于化學信息學方法的藥物分子計算研究[D].大連:大連理工大學,2012.
[14] 俞書浩.功能基因組學和化學信息學協同的藥物研發數據挖掘方法[D].上海:上海交通大學,2013.
[15] 曹正鳳.隨機森林算法優化研究[D].北京:首都經濟貿易大學,2014.
[16] 白耀輝,陳 明.利用自組織特征映射神經網絡進行可視化聚類[J].計算機仿真,2006,23(1):180-183.
[17] 張華偉,王明文,甘麗新.基于隨機森林的文本分類模型研究[J].山東大學學報:理學版,2006,41(3):5-9.
[18] 袁芳娟.基于隨機森林的年齡估計[D].天津:河北工業大學,2012.
[19] 雍 凱.隨機森林的特征選擇和模型優化算法研究[D].哈爾濱:哈爾濱工業大學,2008.
[20] 李欣海.隨機森林模型在分類與回歸分析中的應用[J].應用昆蟲學報,2013,50(4):1190-1197.
[21] 游 偉,李樹濤,譚明奎.基于SVM-RFE-SFS的基因選擇方法[J].中國生物醫學工程學報,2010,29(1):93-99.
[22] 馬景義,謝邦昌.用于分類的隨機森林和Bagging分類樹比較[J].統計與信息論壇,2010,25(10):18-22.
[23] 董 婷.支持向量機分類算法在MATLAB環境下的實現[J].榆林學院學報,2008,18(4):94-96.
[24] 曹東升.化學生物信息學新方法及其在醫藥研究中的應用[D].長沙:中南大學,2013.
[25] 劉孝良.基于半監督學習的隨機森林算法研究與應用[D].青島:中國海洋大學,2013.
[26] 廖明橋.基于支持向量機的半監督式分類學習方法[D].哈爾濱:哈爾濱工程大學,2013.
[27] 劉曉東.基于組合策略的隨機森林方法研究[D].大連:大連理工大學,2013.
[28] 方匡南,吳見彬,朱建平,等.隨機森林方法研究綜述[J].統計與信息論壇,2011,26(3):32-38.
[29] 王愛平,萬國偉,程志全,等.支持在線學習的增量式極端隨機森林分類器[J].軟件學報,2011,22(9):2059-2074.
[30] 陳永健.半監督支持向量機分類方法研究[D].西安:陜西師范大學,2014.
[31] 張燦淋.基于支持向量機的半監督式增量學習研究[D].杭州:浙江工業大學,2014.
[32] 趙 瑩.半監督支持向量機學習算法研究[D].哈爾濱:哈爾濱工程大學,2010.
[33] 周志華.基于分歧的半監督學習[J].自動化學報,2013,39(11):1871-1878.
[34] 楊南海,黃明明,赫 然,等.基于最大相關熵準則的魯棒半監督學習算法[J].軟件學報,2012,23(2):279-288.
StudyonCOXInhibitorPredictionModelBasedonMachineLearning
NIE Chang-sen1,BAI Yong1,LIU Xian-de2
(1.College of Information Science & Technology,Hainan University,Haikou 570228,China;2.College of Agriculture,Hainan University,Haikou 570228,China)
In allusion of the lack in COX (Cyclooxygenase) inhibitor and its poor inhibition effect,moreover for the reason that the traditional COX inhibitor screening must be performed through chemical experiment in high cost and low efficiency,a forecast model of COX inhibitors based on machine learning algorithm is proposed and established.It can find COX inhibitor efficiently and accurately.In the establishing process the data set with huge collection of data in the literature has been built up and then the molecular descriptors with the software of Mold2 has been calculated and divided into training set and testing set with the method of SOM.However,two ML methods,Support Vector Machine (SVM) and Random Forest (RF),are employed to develop a prediction method for searching inhibitors and non-inhibitors of COX from the literature.The verification experiments show that the algorithm of SOM and RF has a better prediction accuracy,which also has a higher efficiency compared with the traditional chemical methods.The results of investigation demonstrate that the COX inhibitor prediction models based on SOM and RF has a good classification prediction effect and provides powerful instrument for analysis and prediction of COX inhibitor.
COX inhibitors;machine learning;SOM;random forests;support vector machines
TP301
A
1673-629X(2017)10-0074-04
2016-11-22
2017-03-13 < class="emphasis_bold">網絡出版時間
時間:2017-07-19
國家自然科學基金資助項目(31660733);海南省應用技術研發與示范推廣專項(ZDXM2015065);海南省社會發展科技專項(SF201421)
聶長森(1994-),男,碩士研究生,研究方向為移動通信與智能信息處理;白 勇,教授,博士,研究方向為移動通信與智能信息處理;柳賢德,副教授,博士,通訊作者,研究方向為獸醫公共衛生學。
http://kns.cnki.net/kcms/detail/61.1450.TP.20170719.1113.082.html
10.3969/j.issn.1673-629X.2017.10.016

