基于PCR—DGGE分析云南牛干巴的細菌群落結構
劉姝韻++尚思奇++孫燦++王桂瑛++程志斌++肖蓉++蔣進仙++汪善榮++廖國周
摘 要:以黃牛后腿肉為原料,采用傳統工藝加工云南牛干巴,分別在腌制前、腌制中期、腌制后期、成熟1 個月、成熟2 個月與成熟3 個月6 個加工階段取樣,應用聚合酶鏈式反應-變性梯度凝膠電泳(polymerase chain reaction-denaturing gradient gel electrophorsis,PCR-DGGE)技術分析不同加工階段牛干巴的細菌群落結構及多樣性差異。結果表明:牛干巴具有較為豐富的細菌多樣性,同時存在明顯的優勢種群結構變化。腌制階段的細菌種類最為豐富;腌制前與腌制后的樣品中細菌群落結構差異明顯,同一階段細菌群落結構組成類似;存在于牛干巴加工過程中的細菌主要為葡萄球菌屬(Staphylococcus)、嗜冷桿菌(Psychrobacter)及明串珠菌屬(Leuconostoc)等,其中腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp. dextranicum)是優勢菌種;此外,牛干巴加工過程中還存在著不可培養(unculturable bacterium)的菌群。
關鍵詞:云南牛干巴;聚合酶鏈式反應-變性梯度凝膠電泳;細菌群落結構;多樣性
Analysis of Bacterial Community Structure of Yunnan Dry-Cured Beef by Polymerase Chain
Reaction-Denaturing Gradient Gel Electrophorsis
LIU Shuyun1,2, SHNAG Siqi1,2, SUN Can1, WANG Guiying1,2, CHENG Zhibin2, XIAO Rong1, JIANG Jinxian1,
WANG Shanrong3,*, LIAO Guozhou2,*
(1.College of Food Science and Technology, Yunnan Agricultural University, Kunming 650201, China;
2.Livestock Product Processing Engineering and Technology Research Center of Yunnan Province, Kunming 650201, China;
3.Yunnan Rural Leader College, Yunnan Agricultural University, Kunming 650201, China)
Abstract: Yunnan dry-cured beef was processed from beef hindquarter using the traditional method. Six samples were collected before curing, during the mid- and late-curing stages, and after one, two and three months of fermentation and ripening, respectively and then the bacterial community structure and diversity in dry-cured beef at different processing stages were analyzed using polymerase chain reaction-denaturing gradient gel electrophorsis (PCR-DGGE). The results showed that dry-cured beef was relatively rich in bacterial diversity, and the dominant bacterial populations in it were highly diverse. The highest bacterial diversity was found during the curing process of meat; there was a significant difference in the bacterial community structure between cured and uncured meat. The bacterial community structure remained similar at the same stage of processing. The bacterial community mainly consisted of Staphylococcus, Psychrobacter and Leuconostoc, with the predominance of L.mesenteroides subsp. dextranicum, along with unculturable bacteria, during the processing of dry-cured beef.
Key words: Yunnan dry-cured beef; polymerase chain reaction-denaturing gradient gel electrophorsis (PCR-DGGE); bacterial community structure; diversity
DOI:10.7506/rlyj1001-8123-201709008
中圖分類號:TS251.5 文獻標志碼:A 文章編號:1001-8123(2017)09-0044-07endprint
引文格式:
劉姝韻, 尚思奇, 孫燦, 等. 基于PCR-DGGE分析云南牛干巴的細菌群落結構[J]. 肉類研究, 2017, 31(9): 44-50. DOI:10.7506/rlyj1001-8123-201709008. http://www.rlyj.pub
LIU Shuyun, SHNAG Siqi, SUN Can, et al. Analysis of bacterial community structure of yunnan dry-cured beef by polymerase chain reaction-denaturing gradient gel electrophorsis[J]. Meat Research, 2017, 31(9): 44-50. DOI:10.7506/rlyj1001-8123-201709008. http://www.rlyj.pub
云南牛干巴是云南地區一種傳統的民族肉制品,它是以黃牛后腿肉為原料,經腌制、晾曬、風干發酵而成的塊狀自然發酵肉制品[1]。牛干巴制作工藝考究、肉質酥脆、風味獨特且營養豐富,但作為一種傳統的腌臘肉制品,它在加工過程中主要依靠環境中的微生物生長來進行發酵,產品質量不穩定,其腌制發酵過程會形成復雜的微生物菌群,并產生一些對人體有害的有機化合物,降低了牛干巴的食用安全性[2]。
聚合酶鏈式反應-變性梯度凝膠電泳(polymerase chain reaction-denaturing gradient gel electrophorsis,PCR-DGGE)是研究微生物群落組成及親緣關系的分子指紋技術,可以高效、直接、快速、全面地分析微生物組成及群落結構,是細菌分類和鑒定的有力工具[3-6]。如今,依賴于培養的微生物分析方法已經無法準確描述完整的菌群結構,具有很大的局限性,而PCR-DGGE技術作為一種免培養方法可以有效避免傳統培養方法的不足。雖然該技術在肉品微生物學領域中的應用起步較晚,但近年來其在原料肉和多種肉制品的微生物群落結構分析中得到了廣泛的應用,能夠對發酵肉制品發酵和成熟過程中的微生物菌群結構多樣性及變化進行分析和研究[7-12]。目前,運用不依賴于傳統培養的(分子生物學)技術對云南牛干巴微生物方面的研究鮮有報道,已有報道多是采用傳統分離培養技術對牛干巴的菌群結構進行研究[1,13-16]。
基于此,本研究采用PCR-DGGE技術對云南牛干巴加工過程中6 個工藝點(腌制前、腌制中期、腌制后期、成熟1 個月、成熟2 個月與成熟3 個月)的微生物變化規律進行系統分析,為進一步了解和認識云南少數民族傳統發酵肉制品中微生物的變化規律,改善牛干巴品質,提高其商品價值提供理論基礎,促進云南牛干巴產業的持續發展。
1 材料與方法
1.1 材料與試劑
檢驗合格的新鮮黃牛后腿肉(剔除筋膜) 云南省昆明市威遠街農貿市場。
食鹽 云南鹽化股份有限公司;硝酸鈉(食品級)、硝酸銀、甲醛、冰醋酸、氫氧化鈉(均為分析純或分析純配制試劑) 國藥集團化學試劑有限公司;VC(食品級) 昆明振華制藥廠;CTAB環境微生物DNA提取試劑盒、50×TAE Buffer 北京美億美生物技術有限公司;Poly-Gel DNA Extraction Kit 廣州飛揚生物科技有限公司;dNTPs、rTaq、pMD18-T Cloning Kit 寶生物工程(大連)有限公司;DNA純化試劑盒、DH5α感受態細胞 北京天根生物科技有限公司;引物 南京
金斯瑞生物科技有限公司;40%甲叉雙丙烯酰胺(分
析純) 伯樂生命醫學產品(上海)有限公司;尿素、過硫酸銨(分析純) 美國Amresco公司;四甲基乙二胺(N,N,N,N-tetramethylethylenediamine,TEMED)(分析純) 美國Sigma公司。
1.2 儀器與設備
BS11OS精密分析天平 北京賽多利斯天平有限公司;Centrifuge 5415D離心機 德國Eppendorf股份
公司;T-gradient PCR儀 德國Biometra公司;Gel-Doc 2000凝膠成像儀 美國Bio-Rad公司;JY-SPFT電泳儀 北京君意東方電泳設備有限公司;DCode變性梯度凝膠電泳儀 伯樂生命醫學產品(上海)有限公司。
1.3 方法
1.3.1 牛干巴傳統加工工藝
取新鮮黃牛后腿肌肉5 塊,于11月份開始腌制,溫度在0~l0 ℃之間,腌制15 d左右。具體加工工藝如下:黃牛后腿肉→冷涼→稱質量→擦腌制料→入壇壓實,密封腌制→晾曬→堆碼擠壓→風干→成品→切片→包裝
腌制料:市售食鹽(40 g/kg)、硝酸鈉(0.4 g/kg)、VC(0.01 g/kg)。
分別在腌制前(腌制第0天)、腌制中期(腌制第7天)、腌制后期(腌制第15天)、成熟1 個月、成熟2 個月及成熟3 個月6 個工藝點進行取樣,每個工藝點取5 個樣品。將樣品分別編號:腌制前(A)為A1、A2、A3、A4、A5,腌制中期(B)為B1、B2、B3、B4、B5,腌制后期(C)為C1、C2、C3、C4、C5,成熟1 個月(D)為D1、D2、D3、D4、D5,成熟2 個月(E)為E1、E2、E3、E4、E5,成熟3 個月(F)為F1、F2、F3、F4、F5。
用于PCR-DGGE分析的樣品:從每個工藝點的5 個牛干巴樣品中隨機抽取3 個,分別為A1、A3、A4,B1、B3、B4,C1、C3、C4,D1、D3、D4,E1、E3、E4,F1、F3、F4。
1.3.2 樣品中總DNA的提取endprint
采用FastDNATM SPIN Kit For Soil試劑盒方法。稱取0.5 g樣品,按照試劑盒步驟進行樣品微生物總DNA的提取,保存于-20 ℃待測。
1.3.3 細菌16S rDNA片段的PCR擴增
參考文獻[17]中的方法,以樣品基因組DNA為模板,采用細菌通用引物GC-338F和518R擴增樣品的16S rDNA高變區序列。所用引物序列如表1所示。
PCR擴增體系(50 μL):10×PCR Buffer 5 μL、dNTP(2.5 mmol/L)3.2 μL、rTaq(5 U/μL)0.4 μL、GC-338F(20 μmol/L)1 μL、518R(20 μmol/L)1 μL、模板DNA 50 ng,用dd H2O補至50 μL。
PCR擴增條件:94 ℃預變性5 min,94 ℃變性1 min,55 ℃復性45 s,72 ℃延伸1 min,30 個循環;最終72 ℃延伸10 min。
PCR產物采用DNA Gel Extraction Kit純化回收,用1.2%瓊脂糖凝膠電泳檢測,Gel-Doc 2000凝膠成像系統拍照。
1.3.4 PCR產物的DGGE分析
取10 μL PCR產物進行DGGE分析。采用變性梯度為35%~55%、濃度為7%的聚丙烯酰胺凝膠在1×TAE緩沖液中電泳5 h(150 V,60 ℃)。電泳結束后,采用銀染法染色,條帶顯現后立即拍照。
1.3.5 DGGE圖譜中優勢條帶的回收與測序
參考文獻[18]中的方法。用滅菌手術刀切下選取的DGGE條帶,按試劑盒步驟回收目的條帶。將純化后的DNA片段按1.3.2、1.3.3節中的方法重新進行PCR擴增,隨后將DNA片段切膠回收、純化后,連接到Pmd18-T載體上,并轉化至DH5α感受態細胞中,篩選陽性克隆,與GenBank中的序列進行比對,得出條帶所代表的細菌類型。每個回收條帶選取3 個克隆進行序列測定。
測序結果采用DNAstar和Cluster軟件進行序列分析,下載最相似的菌株序列作為系統發育樹的參考序列,然后采用MEGA軟件,Neighbor-Joining法構建系統發育樹,自展數為1 000。
1.3.6 細菌多樣性指數計算
細菌多樣性指數是研究群落物種數和個體數以及均勻度的綜合指標。根據電泳圖譜中樣品的條帶數目及每個條帶的強度(灰度),對各樣品的香農指數(H)、均勻度(E)和豐富度(S)等指標進行分析,它們可以用來比較不同樣品的微生物多樣性[19]。采用Quantity one軟件對DGGE圖譜中每個樣品的電泳條帶數目和條帶密度進行數字化分析,參考文獻[18]中的方法,按照下式計算H和E。
式中:pi為樣品中單一條帶的強度在該樣品所有條帶總強度中所占的比率;N為單一泳道上所有條帶的豐度,Ni為第i條帶的豐度;S為某樣品中所有條帶數目的總和。
2 結果與分析
2.1 牛干巴細菌組DNA的提取
由圖1可知,本研究中的DNA提取方法可以順利地從牛干巴樣品中提取出細菌DNA片段,且所提取的DNA完整性較好,有利于后續的PCR擴增。
2.2 細菌16S rDNA的PCR擴增
由圖2可知,以GC-338F和518R為引物擴增細菌DNA的16S rDNA序列,得到的擴增產物大小符合目的片段(200 bp)要求,所有樣品的電泳圖中均有較亮的擴增條帶出現且條帶清晰,因此本研究的PCR擴增條件適用于接下來的DGGE電泳分析。
2.3 PCR產物的DGGE分析及主要條帶測序
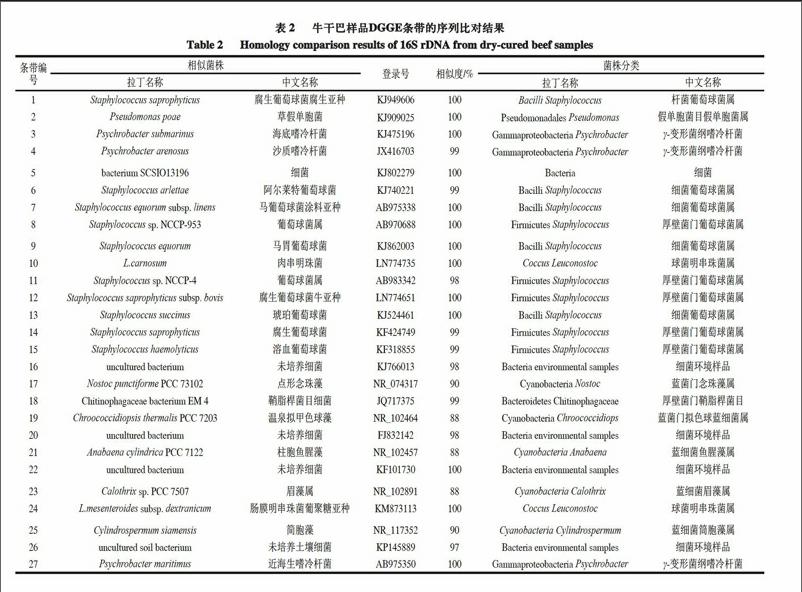
DGGE電泳圖譜中條帶越多說明樣品的生物多樣性越豐富,不同位置的條帶代表不同種屬的細菌;條帶的亮度反映了對應細菌的豐度,條帶越亮,表明該種細菌的相對含量可能越大,從而反映出微生物的數量和種類[20-21]。
由圖3~4可知,不同階段樣品的電泳條帶分離效果較好,條帶的亮度各有不同,共檢測到28 條不同條帶,顯示出牛干巴中豐富的細菌種類,其中樣品A有19 個主要條帶,樣品B 21個,樣品C 18個,樣品D 21個,樣品E 19個,樣品F 20個。
由表2可知,切膠測序的27 個條帶中有24 條可以在GenBank中找到與其序列同源性較高(>90%)的種群,根據同源性分析結果,這24 條DGGE條帶序列中有10 條屬于葡萄球菌屬(Staphylococcus),4 條屬于未培養細菌(uncultured soil bacterium),3 條屬于嗜冷桿菌(Psychrobacter),2 條屬于明串珠菌屬(Leuconostoc),1 條屬于假單胞菌屬(Pseudomonas),1 條屬于細菌(Bacteria),1 條屬于念珠藻屬(Nostoc),1 條屬于鞘脂桿菌目(Chitinophagaceae),1 條屬于筒胞藻屬(Cylindrospermum)。其中條帶10與肉明串珠菌(L.carnosum)、條帶24與腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp. dextranicum)的序列同源性都達到了100%;牛干巴腌制前樣品(A)的最亮條帶為21:柱胞魚腥藻(Anabaena cylindrica PCC 7122),腌制中期樣品(B)的最亮條帶為16和21,其中條帶16為未培養細菌(uncultured bacterium),腌制后期樣品(C)的最亮條帶為10、13、16和24,其中條帶10為肉明串珠菌(L.carnosum),條帶13為琥珀葡萄球菌(Staphylococcus succinus),條帶24為腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp. dextranicum),成熟1 個月樣品(D)的最亮條帶為5、9、13和24,其中條帶5為細菌(bacterium SCSIO13196),條帶9為馬胃葡萄球菌(Staphylococcus equorum);成熟2 個月樣品(E)的最亮條帶為4、5、16、20、24、27,其中條帶4為沙質嗜冷桿菌(Psychrobacter arenosus),條帶20為未培養細菌(uncultured bacterium),條帶27為近海生嗜冷桿菌(Psychrobacter maritimus);成熟3 個月樣品(F)的最亮條帶為2、5、9、10、14、24,其中條帶2為草假單胞菌(Pseudomonas poae),條帶14為腐生葡萄球菌(Staphylococcus saprophyticus)。endprint
由PCR-DGGE圖譜可知,牛干巴樣品的整個加工過程均檢測出了腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp. dextranicum),由此可以推斷,腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp. dextranicum)是牛干巴加工過程中的優勢菌種。另外,條帶16代表的未培養細菌(uncultured bacterium)也是牛干巴加工過程中的優勢菌群,其在牛干巴加工過程中的作用有待進一步研究和驗證。
2.4 樣品間的微生物相似性分析
由表3可知,整體上看各樣品間的DGGE圖譜相似性都不高,體現出牛干巴中細菌組成的多樣性。依據樣品間的相似系數構建聚類分析圖,對不同階段牛干巴樣品中細菌群落結構的相似性進行比較。由圖5可知,18 個牛干巴樣品中的細菌被分成2 類,腌制前與腌制成熟期樣品的細菌群落結構差異明顯,表明牛干巴樣品的細菌組成與腌制、成熟的不同階段之間存在明顯的相關性。
牛干巴腌制前(A)與腌制中期(B)的相似度為75.3%,腌制中期(B)與腌制后期(C)的相似度為64.8%,腌制后期(C)與成熟1 個月(D)的相似度為68.1%,成熟1 個月(D)與成熟2 個月(E)的相似度為67.0%,成熟2 個月(E)與成熟3 個月(F)的相似度為74.2%。從腌制前到成熟3 個月,相鄰加工階段樣品間的相似度先下降后上升,其中腌制中期與腌制后期樣品間的相似度最低。同一階段的不同樣品間相似度也較低,如腌制前的樣品1和樣品3之間的相似度僅為55.1%,這說明牛干巴在腌制過程中細菌種類較為豐富。
2.5 牛干巴樣品的PCA
由圖6可知,主成分因子1(PC1)的貢獻率為41.5%,主成分因子2(PC2)的貢獻率為16.5%。根據主成分1,B4、C1、D1、D4、E3、F3共6 個樣品明顯地歸于一類,A4、B1、B3、C3、E4歸于一類,A1、A3、D3、E1、F1歸于一類,而樣品C4、F4則離散在圈外,沒有明顯的類聚,與其他樣品差異明顯。
2.6 牛干巴樣品的微生物多樣性
H用來評價微生物群落的多樣性,H值越大,說明微生物的種類和數量越多;E表示微生物分布的均勻程度;S表示微生物種類的多寡[22]。由表4可知,隨著牛干巴加工過程的進行,成熟2 個月樣品的H、E及S值最大,說明此時各樣品的微生物種類和數量最多,且分布較均勻。
3 討 論
通過DGGE圖譜可以看出牛干巴樣品的細菌組成在腌制前與腌制成熟期有著明顯差異,尤其是進入腌制期后,樣品間的相似度下降,表明在腌制前期就產生了不同種類的細菌;腌制中期與腌制后期之間的相似度則更低;同一階段的不同樣品間相似度也較低,如腌制前的樣品1和樣品3之間的相似度僅為55.1%,這說明牛干巴在腌制過程中產生細菌的種類較為豐富,同時也表明牛干巴樣品的細菌組成與不同腌制階段之間存在明顯的相關性。
目前關于PCR-DGGE技術應用于肉制品微生物研究的報導較多集中在發酵香腸的微生物區系研究、香腸成熟過程中微生物種群的動態變化研究、主要發酵微生物種類的分離鑒定[23]以及不同地區發酵香腸微生物多樣性的差異研究等[24]。應用PCR-DGGE技術對各階段牛干巴樣品中細菌菌落結構進行研究的結果表明,整體來說,牛干巴中細菌多樣性較為豐富,優勢菌群為葡萄球菌屬和明串珠菌屬菌,這與陳韻等[25]對貴州侗族酸肉中細菌多樣性的研究結果相似。不同時期的牛干巴樣品中均檢測出了腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp.dextranicum),說明其為牛干巴加工過程中的優勢菌種,與孫燦等[1]的研究結果一致。
Cocolin等[26]對意大利發酵香腸菌群分布的研究發現,香腸發酵前葡萄球菌屬為優勢菌,并且在其成熟過程中有3 種葡萄球菌起主要作用;Silvestri等[27]的研究表明,22 種市售色拉米香腸樣品中最常見的為清酒乳桿菌和彎曲乳桿菌2 種乳酸菌。Fontana等[28-29]采用PCR-DGGE技術研究阿根廷香腸,發現在香腸發酵初期,優勢菌群為植物乳桿菌和腐生葡萄球菌。Paleari等[30]發現在用于Bresaola火腿加工的牛肉原料中存在乳酸菌、葡萄球菌和腸細菌,而產品中則主要存在乳酸菌、葡萄球菌屬細菌;肖蓉等[16]使用傳統分離篩選方法在牛干巴加工過程中篩選到的優勢菌為微球菌、乳桿菌、假單胞菌、埃希氏菌和片球菌;劉洋等[31]發現低鹽臘肉加工過程中的優勢菌是葡萄球菌和乳酸菌,它們的數量較其他菌株高出一個數量級;馬曉燕[32]從發酵牛肉加工過程中共篩選出8 株優勢細菌,包括5 株葡萄球菌、2 株乳酸菌和1 株芽孢桿菌。以上研究結果均與本研究相似,乳酸菌和葡萄球菌在肉制品中的生長可以使肉中的蛋白質分解為肽和氨基酸,同時產生大量風味物質,提高產品的營養價值。
本研究檢測到Uncultured 16、Uncultured 20、Uncultured 22、Uncultured 26非培養細菌,表明利用PCR-DGGE技術得出的結果更加全面、精確。但是由于環境的復雜性、PCR擴增中引物的偏好性、并非DGGE圖譜中的所有條帶均能成功測序等因素均會影響該技術的準確性,因此針對云南牛干巴加工過程中細菌多樣性的變化規律研究還需結合環境因素進行更全面、更深入的分析。
4 結 論
云南牛干巴具有較為豐富的細菌多樣性,同時存在明顯的優勢種群結構變化。牛干巴在腌制階段的細菌種類最為豐富;18 個牛干巴樣品的細菌組成被分成2 類,腌制前與腌制后的樣品中細菌群落結構差異明顯,同一階段細菌群落結構組成類似;存在于牛干巴加工過程中的細菌主要為葡萄球菌屬(Staphylococcus)、嗜冷桿菌(Psychrobacter)及明串珠菌屬(Leuconostoc)等,其中腸膜明串珠菌葡聚糖亞種(L.mesenteroides subsp.dextranicum)是優勢菌種;此外,牛干巴加工過程中還存在著不可培養(uncultured bacterium)的菌群。相較于傳統的分離培養法,采用PCR-DGGE技術分析牛干巴中的微生物菌群結構能夠得出更加全面和直觀的結果,可以為今后進一步改進云南牛干巴的加工工藝、提高其食用安全性提供理論依據。endprint
參考文獻:
[1] 孫燦, 肖蓉, 尹豐, 等. 云南牛干巴加工過程產生物胺的微生物消長規律[J]. 肉類研究, 2015, 29(4): 6-9.
[2] MARCOBAL A, DE LAS RIVAS B, LANDETE J M, et al. Tyramine and phenylethylamine biosynthesis by food bacteria[J]. Critical Reviews in Food Science and Nutrition, 2012, 52(5): 448-467.
DOI:10.1080/10408398.2010.500545.
[3] PENNACCHIA C, ERCOLINI D, VILLANI F. Spoilage-related microbiota associated with chilled beef stored in air or vacuum pack[J]. Food Microbiology, 2011, 28(1): 84-93. DOI:10.1016/j.fm.2010.08.010.
[4] DING Xiaofei, WU Chongde, HUANG Jun, et al. Eubacterial and archaeal community characteristics in the man-made pit mud revealed by combined PCR-DGGE and FISH analyses[J]. Food Research International, 2014, 62(8): 1047-1053. DOI:10.1016/j.foofres.2014.05.045.
[5] CHEN Huibin, WANG Meiying, LIN Xiangzhi, et al. Bacterial microbiota profile in gills of modified atmosphere-packaged oysters stored at 4 ℃[J]. Food Microbiology, 2017, 61(1): 58-65. DOI:10.1016/j.fm.2016.08.006.
[6] EL SHEIKHA A F, MONTET D. How to determine the geographical origin of seafood?[J]. Critical Reviews in Food Science and Nutrition, 2016, 56(2): 306-317. DOI:10.1080/10408398.2012.745478.
[7] COCOLIN L, DOLCI P, RANTSIOU K. Biodiversity and dynamics of meat fermentations: the contribution of molecular methods for a better comprehension of a complex ecosystem[J]. Meat Science, 2011, 89(3): 296-302. DOI:10.1016/j.meatsci.2011.04.011.
[8] 高永超, 王加寧, 遲建國, 等. 污染土壤微生物群落結構分子鑒定技術研究進展[J]. 生物技術, 2012, 22(2): 91-96. DOI:10.3969/j.issn.1004-311X.2012.02.052.
[9] Lü Xucong, WENG Xiaolan, ZHANG Wen, et al. Microbial diversity of traditional fermentation starters for Hong Qu glutinous rice wine as determined by PCR-mediated DGGE[J]. Food Control, 2012, 28(2): 426-434. DOI:10.1016/j.foodcont.2012.05.025.
[10] 牛會敏, 魏法山, 袁乾乾. PCR-DGGE技術在肉制品中微生物分析中的應用[J]. 食品安全導刊, 2015(9): 119-120. DOI:10.16043/j.cnki.cfs.2015.27.072.
[11] TREMONTE P, SORRENTINO E, PANNELLA G, et al. Detection of different microenvironments and Lactobacillus sakei, biotypes in Ventricina, a traditional fermented sausage from central Italy[J]. International Journal of Food Microbiology, 2017, 242(2): 132-140. DOI:10.1016/j.ijfoodmicro.2016.11.009.
[12] 全拓. 肉制品中主要微生物的檢測與研究[D]. 重慶: 西南大學, 2012: 10-12.
[13] 肖蓉, 徐昆龍, 施忠芬, 等. 輻照牛干巴在貯藏過程中微生物變化的研究[J]. 食品工業科技, 2009, 30(1): 286-288. DOI:10.13386/j.issn1002-0306.2009.01.071.
[14] 胡凱弟, 甘芳瑗, 劉書亮, 等. 不同處理方式對牛干巴貯藏期品質的影響[J]. 食品與發酵工業, 2015, 41(7): 220-225. DOI:10.13995/j.cnki.11-1802/ts.201507041.endprint
[15] 徐昆龍, 肖蓉. 臘牛肉加工中微生物消長規律性研究[J]. 中國畜牧獸醫文摘, 2005(5): 73-74. DOI:10.3969/j.issn.1004-7034.2005.04.045.
[16] 肖蓉, 徐昆龍, 劉旭川. 云南黃牛干巴(臘牛肉)中細菌的分離與鑒定[J]. 中國牛業科學, 1998, 24(3): 21-22.
[17] 趙乾宇. 飼養密度對籠養蛋雞小腸組織結構和盲腸細菌種群的影響[D]. 大慶: 黑龍江八一農墾大學, 2015: 24.
[18] 鄭艷, 姚婷. PCR-DGGE分析甘薯酸漿自然發酵過程中細菌多樣性[J]. 食品科學, 2016, 37(7): 99-103. DOI:10.7506/spkx1002-6630-201607019.
[19] KUANG Y, TANI K, SYNNOTT A J, et al. Characterization of bacterial population of raw milk from bovine mastitis by
culture-independent PCR-DGGE method[J]. Biochemical Engineering Journal, 2009, 45(1): 76-81. DOI:10.1029/1999JC900266.
[20] 欒同青, 李志軍, 鐘其頂, 等. 黃酒釀造過程細菌群落結構變化初步研究[J]. 食品工業科技, 2013, 34(12): 177-180. DOI:10.13386/j.issn1002-0306.2013.12.055.
[21] ERCOLINI D. PCR-DGGE fingerprinting: novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods, 2004, 56(3): 297-314. DOI:10.1016/j.mimet.2003.11.006.
[22] 王建輝, 楊晶, 劉永樂, 等. 基于PCR-DGGE技術對冷藏過程中草魚肌肉的細菌群落結構分析[J]. 中國食品學報, 2014, 14(9): 216-223. DOI:10.16429/j.1009-7848.2014.09.035.
[23] RENOUF V, CLAISSE O, MIOT-SERTIER C, et al. Lactic acid bacteria evolution during winemaking: use of rpoB gene as a target for PCR-DGGE analysis[J]. Food Microbiology, 2006, 23(2): 136-145. DOI:10.1016/j.fm.2005.01.019.
[24] MOROT-BIZOT S C, LEROY S, TALON R. Staphylococcal community of a small unit manufacturing traditional dry fermented sausages[J]. International Journal of Food Microbiology, 2006, 108(2): 210-217. DOI:10.1016/j.ijfoodmicro.2005.12.006.
[25] 陳韻, 胡萍, 湛劍龍, 等. 運用PCR-DGGE技術分析貴州侗族酸肉中細菌多樣性[J]. 食品與機械, 2014, 30(2): 188-191. DOI:10.3969/j.issn.1003-5788.2014.02.047.
[26] COCOLIN L, MANZANO M, AGGIO D, et al. A novel polymerase chain reaction (PCR)-denaturing gradient gel electrophoresis (DGGE) for the identification of Micrococcaceae strains involved in meat fermentations. Its application to naturally fermented Italian sausages[J]. Meat Science, 2001, 58(1): 59-64. DOI:10.1016/S0309-1740(00)00131-5.
[27] SILVESTRI G, SANTARELLI S, AQUILANTI L, et al. Investigation of the microbial ecology of Ciauscolo, a traditional Italian salami, by culture-dependent techniques and PCR-DGGE[J]. Meat Science, 2007, 77(3): 413-423. DOI:10.1016/j.meatsci.2007.04.015.
[28] FONTANA C, VIGNOLO G, COCCONCELLI P S. PCR-DGGE analysis for the identification of microbial populations from Argentinean dry fermented sausages[J]. Journal of Microbiological Methods, 2005, 63(3): 254-263. DOI:10.1016/j.mimet.2005.03.010.
[29] FONTANA C, SANDRO C P, VIGNOLO G. Monitoring the bacterial population dynamics during fermentation of artisanal Argentinean sausages[J]. International Journal of Food Microbiology, 2005, 103(2): 131-142. DOI:10.1016/j.ijfoodmicro.2004.11.046.
[30] PALEARI M A, MORETTI V M, BERETTA G, et al. Cured products from different animal species[J]. Meat Science, 2003, 63(4): 485-489. DOI:10.1016/S0309-1740(02)00108-0.
[31] 劉洋, 崔建云, 任發政, 等. 低鹽臘肉在加工過程中的菌相變化初探[J]. 食品工業科技, 2005, 26(8): 49-50. DOI:10.3969/j.issn.1002-0306.2005.08.011.
[32] 馬曉燕. 牛肉發酵過程中優勢微生物的分離鑒定及應用研究[D]. 保定: 河北農業大學, 2004: 49.endprint

