鹽角草PsaH基因的克隆及生物信息學分析
郝曉燕,李建平,足木熱木·吐爾遜,高升旗,黃全生
(新疆農業科學院核技術生物技術研究所,烏魯木齊 830091)
鹽角草PsaH基因的克隆及生物信息學分析
郝曉燕,李建平,足木熱木·吐爾遜,高升旗,黃全生
(新疆農業科學院核技術生物技術研究所,烏魯木齊 830091)
目的從鹽生植物鹽角草(Salicorniaeuropaea)中克隆得到一個參與光合反應光系統I(PSI)復合蛋白H亞基的PsaH基因,分析該基因進行生物信息學,為研究該基因的作用奠定基礎并為改良作物耐鹽性分子育種提供候選基因。方法以鹽生植物鹽角草為實驗材料,采用RT-PCR方法克隆PsaH基因,利用NCBI、MEGA、Expasy等生物信息學工具對其核酸序列、編碼的氨基酸序列及蛋白質的結構和功能進行分析。結果克隆得到一個與耐鹽有關的基因,命名為SePsaH,屬于光系統I(PSI)家族H亞基成員,其開放閱讀框(ORF)為438 bp,基因編碼145個氨基酸,預測蛋白分子量為15.3 kD,等電點9.84,是親水性蛋白;系統進化樹分析表明其與菠菜親緣關系較近,通過對該蛋白保守性分析發現其含有4個保守性結構域。結論獲得鹽角草SePsaH基因,為進一步研究該基因耐鹽功能及其在鹽生植物耐鹽機制中的作用提供了基礎。
鹽角草;SePsaH基因;生物信息
0 引 言
【研究意義】干旱、鹽堿、極端溫度等非生物脅迫是嚴重影響植物生長和發育造成農作物減產的主要因素。所有這些脅迫都會引發植物細胞內一系列的生理生化反應來響應對逆境的應答機制。據世界糧農組織的估計,世界上大約有20%的耕地和接近50%的灌溉用地受到土壤鹽漬化的嚴重危害[1-3]。我國大約有0.33×108hm2(5億畝)鹽堿地,并且其面積有不斷增加的趨勢。鹽角草(Salicorniaeuropaea)是藜科(Chenopodiaceae)鹽角草屬(SaliconiaL)的一年生低矮草本,生于平原荒漠區湖邊潮濕鹽土上,是典型的鹽生植物,是我國最重要的鹽土荒漠建群種之一。由于其蛋白質組分良好,且種子脂類含量高,有獨特的生物生態學特征和廣泛的經濟、生態價值[4]。因此,利用現代生物學技術,通過對鹽生植物耐鹽基因的發掘和功能研究能夠為改良作物耐鹽性提供理論基礎,還將對有效地改良和利用鹽漬土提供思路[5]。【前人研究進展】植物耐鹽的機制是包含相互關聯、相互協調、協同作用的一系列生理生化功能的復雜機制。深入研究鹽角草耐鹽相關基因及其功能對于了解鹽生植物耐鹽機理具有重要意義。馬金彪[6]等通過RACE技術從鹽角草中克隆SeHKT1基因,通過生物信息學方法和qRT-PCR方法研究發現,該基因響應鹽脅迫誘導。孫偉民[7]通過轉基因煙草證實了鹽角草SePEAMT和SeCMO能夠提高煙草甜菜堿表達量從而提高煙草的耐鹽性。臧潔[8]等利用RACE技術克隆了鹽角草中的Cu/Zn-SOD基因,通過對過表達菌株證實其的耐鹽功能。【本研究切入點】目前,有關鹽生植物的研究工作雖然早已起步[9,10],主要集中在生態學、形態學、生理學及藥物化學等幾個方面,缺乏系統性研究。利用RT-PCR技術從鹽生植物鹽角草中獲得耐鹽相關基因SePsaH全長,并對其進行生物信息學分析[11-13],以便進一步的了解其分子生物學功能。【擬解決的關鍵問題】研究利用RT-PCR克隆了鹽角草SePsaH基因,并對其推導的氨基酸序列進行生物信息學分析,有助于進一步研究SePsaH基因在提高植物耐鹽性功能方面奠定基礎。
1 材料與方法
1.1 材 料
1.1.1 植物材料
鹽角草(Salicorniaeuropaea)種子收獲與阜康水庫鹽堿地,生長4周大小的鹽角草,以400 mM NaCl脅迫處理48 h取樣,迅速置于液氮中冷凍,-70℃冰箱保存備用。
1.1.2 質粒、菌株、試劑
大腸桿菌(Escherichiacoli)Trans-T1感受態細胞、DNA Marker、TaqDNA聚合酶、pEASY-Blunt Zero Cloning Kit購自TransGen Biotech;;PowerScriptTMII 反轉錄試劑盒、限制性內切酶均購自NEB公司,DNase 酶Ⅰ、LATaq酶、ExTaq酶、dNTP均購自TaKaRa 公司,T4DNA 連接酶購自Promega 公司,DNA 回收試劑盒購自于OMEGA,引物由北京華大生物科技有限公司合成。
1.2 方 法
1.2.1 RNA的提取與純化
材料的總RNA提取按照Invitrogen公司Triozol 試劑說明書進行。以DNaseⅠ純化處理后,取適量進行甲醛變性膠電泳檢測,判斷RNA 完整性和純度。另外,取適量RNA 用紫外分光光度計測定RNA 濃度。
1.2.2 cDNA的合成
第一鏈cDNA的合成按照Fermentas公司的First Strand cDNA Synthesis Kit操作說明進行,采用的反轉錄引物為Oligo(dT)15。取1 μg 總RNA 作為模板進行RT-PCR,20 μL體系中總RNA 1 μg,DEPC水6 μL,Oligo(dT)151 μL,65℃ 5 min,冰上放置5 min,RNase inhibitor 1.0 μL,5×Reaction buffer 4 μL,dNTP(10 mmol/L)2 μL 和M-MLV(200 U/μL)1 μL,42℃溫育60 min,70℃變性5 min,冰上5 min,-70℃保存備用。
1.2.3 SePsaH基因的克隆
使用Primer Primer 5.0軟件在開放閱讀框兩側設計一對能有效擴增SePsaH的特異引物(SePsaH-ORF-F:5’-ATGGCTTCTCTAGCAACCTTTGCC-3’;SePsaH-ORF-R:5’-TTAGATCTTGCCACGAGGTCCG-3’,擴增片段長度為438 bp)。以合成的第一鏈cDNA為模板,進行PCR擴增。反應體系為50 μL:cDNA 1 μL,引物各1 μL,ExTaq酶0.5 μL,10×PCR Buffer (Mg2+plus)5 μL,dNTP Mixture 4 μL,加ddH2O至50 μL。PCR程序:94℃預變性 3 min;94℃ 變性30 s,60℃ 復性30 s,72℃ 延伸30 s,30個循環;72℃延伸10 min; 4℃保存。瓊脂糖凝膠電泳檢測后用DNA回收試劑盒回收目的片段連接到pEASY-Blunt Zero載體進行克隆,陽性克隆經酶切鑒定后送北京華大基因科技有限公司完成DNA測序。
1.2.4 SePsaH基因的生物信息學
SePsaH基因的核苷酸和氨基酸序列分別用NCBI的BLASTn和BLASTp(http://bladt.ncbi.nlm.nih.gov/Blast.cgi)進行相似性分析;用ClustalX1.8對SePsaH與GenBank數據庫中登錄的相似基因進行多序列比對,用MEGA5.0通過鄰接算(Neighbor-Joining)法完成進化樹繪制。利用在線軟件Protparam (htpp://www.expasy.org/protparam/)分析蛋白的理化性質;用TMHMM軟件(http://www.cbs.dtu.dk/services/TMHMM/預測蛋白的跨膜區。
2 結果與分析
2.1 SePsaH基因的克隆
以400 mM NaCl脅迫處理48 h的鹽角草cDNA為模板,SePsaH-ORF全長引物用RT-PCR 法擴增得到預期長度為438 bp的產物。經連接克隆載體、酶切鑒定、測序分析后,得到完整的開放閱讀框438 bp,編碼145個氨基酸。BLASTP分析發現,該序列與菠菜有高度同源的序列,且長度接近。因此,將其命名為SePsaH。用在線軟件ExPasy預測SePsaH基因所編碼的蛋白質理化性質。預測結果表明,SePsaH蛋白分子量為15.4kD,等電點為9.84,分子式為C699H1109N183O204S1,蛋白質的半衰期為30 h,不穩定指數為47.81,為不穩定蛋白。SePsaH蛋白有8個負電荷氨基酸殘基 (Asp+Glu),16個正電荷氨基酸殘基 (Arg+Lys)。圖1
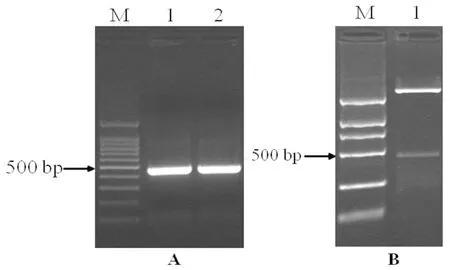
A, M:100 bp Marker;1和2:以cDNA為模板的擴增產物; B, M:DL2000 Marker;1:pEASY-Blunt Zero-SePsaH雙酶切鑒定
A, M:100 bp Marker;1 and 2:the products of cDNA by RT-PCR; B, M:DL2000 Marker;1:pEASY-Blunt Zero-SePsaHdigested byEcoRI and KpnI
圖1 SePsaH基因的擴增和雙酶切鑒定
Fig.1 RT-PCR products of SePsaH gene full-length cDNA sequence from Salicornia europaea and identification by double-enzyme digestion
2.2SePsaH基因的結構域及同源性
通過NCBI在線預測SePsaH蛋白功能結構域,發現該蛋白屬于光系統I H亞基。利用NCBI的Blast在線分析SePsaH蛋白序列。分析結果表明,SePsaH蛋白與菠菜(Spinaciaoleracea) 同源性最高,相似性為92%,與木薯(Manihotesculenta)、蓖麻(Ricinuscommunis) 相似性為85%,與毛果楊(Populustrichocarpa)的相似性為84%,與等其它植物的氨基酸有著高度保守性。該結果表明,研究所克隆的SePsaH基因是光系統I(PSI) H亞基成員。圖2
對包含SePsaH基因在內的6種植物的同源蛋白進行保守區分析發現,他們包含4個保守的結構功能域:1個N端的高酸性富集結構域,1個轉移肽,1個中心疏水結構域和1個C端的保守結構域。這些分析表明該基因編碼的氨基酸具有跨膜的功能。圖3

圖2 蛋白保守區預測
Fig.2 Prediction of SePsaH conserved domains

AhPsaH (花生Arachishypogaea,AB184258);NsPsaH (煙草樟子松Nicotianasylvestris, BAA04634); RcPsaH (蓖麻Ricinuscommunis, XP002516025);PtPsaH (毛果楊Populustrichocarpa, XP002304206); AtPsaH (Arabidopsisthaliana擬南芥,NP175633);SePsaH(鹽角草Salicorniaeuropaea)
圖3 SePsaH基因與其它植物PsaH基因的氨基酸序列比對分析
Fig.3 Comparison of amino acid sequences homology of SePsaH with different plant PsaH
2.3 SePsaH基因系統進化樹
將鹽角草SePsaH氨基酸序列與NCBI數據庫中其他植物中的同源基因的氨基酸序列利用MEGA5.0軟件,用相鄰連接法構建系統進化樹。進化樹分析表明,SePsaH在植物中普遍存在,具有較強的保守性,整體可分為三組:大豆、花生、紅三葉、毛果楊、蓖麻、煙草樟子松聚為一組,其中楊毛果和蓖麻聚為一亞組,其余聚為一亞組。葡萄、玉米聚為一組,鹽角草、菠菜和擬南芥聚為一組。由此可以看出鹽角草與菠菜和擬南芥同類基因的親緣關系較近,推測其在結構和功能上也可能有一定的相似性。圖4
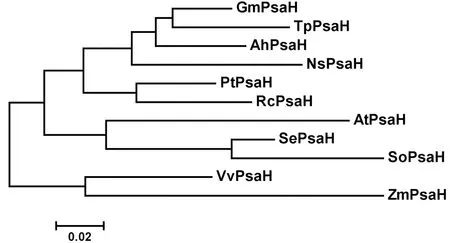
NsPsaH (煙草樟子松Nicotianasylvestris, BAA04634); AhPsaH (花生Arachishypogaea,AB184258); GmPsaH (大豆Glycinemax, ACU13848);TpPsaH (紅三葉Trifoliumpretense, AAQ21121); PtPsaH (毛果楊Populustrichocarpa, XP002304206);RcPsaH (蓖麻Ricinuscommunis, XP002516025);AtPsaH (Arabidopsisthaliana擬南芥,NP175633); SoPsaH (菠菜Spinaciaoleracea, CAA34749);VvPsaH (葡萄Vitisvinifera,XP002284496);ZmPsaH (玉米Zeamays,NP010104905)
圖4 SePsaH系統進化樹
Fig.4 Phylogenetic tree analysis of SePsaH
2.4 SePsaH基因疏水性及跨膜性預測
用ExPasy軟件在線預測SePsaH蛋白的疏水性,研究表明,SePsaH蛋白的最高值為2.44,在第105和106個氨基酸處,疏水性強,總平均疏水性指數(GRAVY)-0.148;在第134個氨基酸處的低值為-2.3,親水性強,可知該蛋白是親水性蛋白;通過TMHMM分析SePsaH蛋白的跨膜性預測,該蛋白具有1個跨膜結構域,位于101~211位氨基酸。圖5,圖6
2.5 SePsaH基因編碼蛋白的高級結構預測
通過PredictProtein預測SePsaH蛋白質的二級結構發現Helix(螺旋)占18.6%,Strand(延伸鏈)占21.4%,Loop(環)占60%。用SWISS-MODEL對SePsaH和SoPsaH蛋白質的三級結構進行了建模預測,從結果可以看出兩個同源性最高的蛋白自三級結構預測結果是十分相似的,與二級結構預測結果也相符。同時,亞細胞定位預測結果顯示SePsaH是定位在真核細胞中葉綠體膜上,與蛋白質功能預測結果相符。圖7
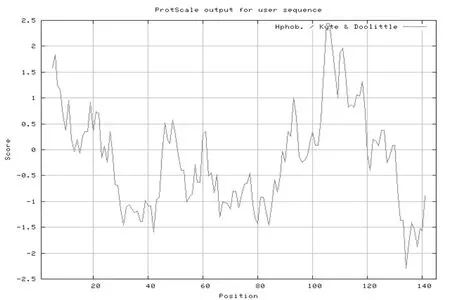
圖5 SePsaH蛋白的疏水性預測
Fig.5 Prediction of the SePsaH protein hydrophobicity
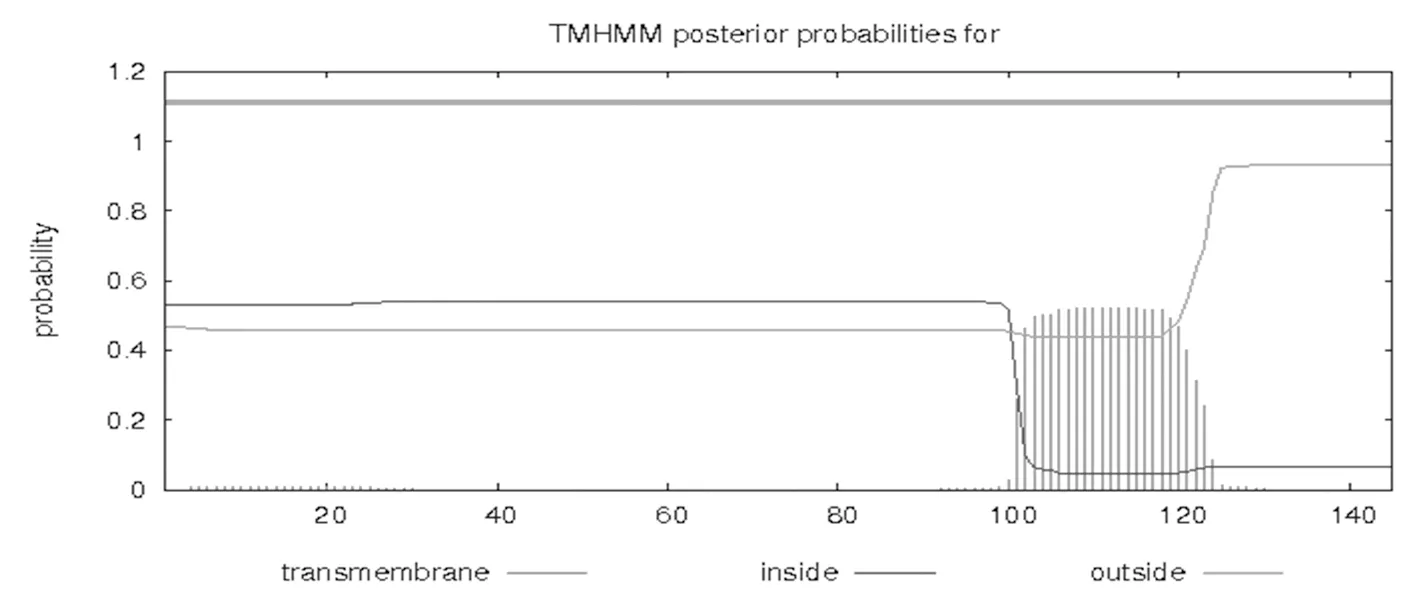
圖6 SePsaH蛋白的跨膜性預測
Fig.6 The transmembrane prediction of the SePsaH protein

圖7 A:SePsaH 編碼蛋白質的三級結構預測;B:SoPsaH編碼蛋白質的三級結構預測
Fig.7 A:Predicted tertiary structure of SePsaH protein; B: Predicted tertiary structure of SoPsaH protein
3 討 論
植物的生長發育會受到各種非生物和生物脅迫的影響,如鹽堿、高溫、干旱等極端環境和病原菌的侵染等[13,14]。相對于生物脅迫、鹽堿、干旱、極端溫度等非生物脅迫對植物生長發育的影響極大,是引起農作物減產的首要原因。在這些脅迫下,植物的光合作用會受到嚴重的抑制,使蛋白質代謝異常[15,16],從而造成植物體內生理干旱[17],進一步抑制植物的生長和發育。但鹽生植物鹽角草適應鹽漬環境的一個特殊方式就是葉片的肉質化,從而在逆境條件下儲存水分,保證葉片進行光合作用,完成正常的生理功能[18]。光合作用(Photosynthesis)是綠色植物、藻類和某些細菌在可見光照射下,經過光反應和碳反應,利用葉綠素,將二氧化碳和水轉化為有機物(主要是淀粉),并釋放出氧氣的生化過程。光合作用是一系列復雜的代謝反應的總和,是生物界賴以生存的基礎。在鹽脅迫下,植物要生存和適應逆境的生態條件,往往要合成相容性的物質,以提高原生質和葉綠體等重要細胞器中有機質的濃度[19],來維持其存在大量鹽分的液泡之間的滲透平衡[20,21],防止原生質和葉綠體等細胞器脫水使植物受到破壞。
在鹽脅迫下,隨著鹽濃度的提高光合電子傳遞速度明顯下降。鹽角草葉片和角質膜厚,氣孔器下陷,具表皮毛,柵欄組織發達,葉片極度肉質化,利用同化枝執行光合功能,且同化枝內部具有粘液和含晶細胞,貯水組織發達。鹽角草是通過這些生理結構的改變來適應旱生和鹽生環境的[22,23]。光合系統蛋白復合體的生理功能是吸收光能,進行光化學反應,產生強的氧化劑,使水裂解釋放氧氣,并把水中的電子傳至質體醌[24]。PsaH是編碼參與PSI蛋白復合體H亞基的基因,在光合系統組成與反應中心的電子傳遞中起著重要的作用。研究獲得了鹽角草的SePsaH基因,對于候選研究PsaH在鹽角草中的功能以及探索鹽生植物耐鹽機制中的作用奠定了基礎。
4 結 論
研究利用RT-PCR方法從鹽角草中成功分離得到一個參與光合反應光系統的PsaH基因。生物信息學分析得出SePsaH基因具有光合反應PSI_H亞基家族典型的結構特征,屬于PsaH基因家族;該基因的開放閱讀框為438 bp,編碼145個氨基酸,對其蛋白質的亞細胞定位預測結果表明其為定位在葉綠體膜上的穩定蛋白;通過跨膜結構分析,其含有1個跨膜結構域,4個保守結構功能域;系統進化分析表明鹽角草的SePsaH基因與菠菜和擬南芥聚為一類,親緣關系最近,這些都與其蛋白質功能預測的結果相符。蛋白質氨基酸序列同源比對分析結果表明,PsaH蛋白與菠菜同源性達到92%,與木薯和蓖麻相似性為85%,與毛果楊的相似性為84%,不同物種之間的PsaH蛋白還是比較保守的。
References)
[1] 趙可夫,李法曾. 中國鹽生植物[M]. 北京:中國科學技術出版社, 2000.
ZHAO Ke-fu, LI Fa-zeng. (2000).HalophytesinChina[M]. Beijing: Science and Technology of China Press. (in Chinese)
[2] 郗金標, 張福鎖, 田長彥,等. 新疆鹽生植物[M]. 北京: 科學出版社, 2006.
XI Jin-biao, ZHANG Fu-suo, TIAN Chang-yan, et al. (2006).HalophytesinXinjiang[M]. Beijing: Science Press. (in Chinese)
[3] Flower T J, Yeo A R.et al. (1997). Breeding for salinity resistance in crop plants-the role of molecular biology.Acta.PhysiologiaePlantarum, 19(4): 427-433.
[4] 李梅梅, 吳國華, 趙振勇, 等.新疆5種藜科鹽生植物的飼用價值[J]. 草業科學, 2017, 34(2):361-368.
LI Mei-mei, WU Guo-hua, ZHAO Zheng-yong, et al. (2017). Feeding value evaluation of Xinjiang five chenopod halophytes [J].PrataculturalScience, 34(2): 361-368. (in Chinese)
[5] Sunkar, R,, Bartels, D., Kirch, HH. (2003). Overexpression of a stress inducible aldehyde dehydrogenase gene from Arabidopsis thaliana in transgenic Plants improves stress tolerance.PlantJournal, (35):452-464.
[6] 馬金彪, 張大勇, 張梅茹, 等. 鹽角草高親和鉀離子轉運蛋白SeHKT1基因的克隆及表達分析[J].生物技術通報, 2015, 31(11):159-165.
MA Jin-biao, ZHANG Da-yong, ZHANG Mei-ru, et al. Clone and Expression Analysis of The High-affinity K+Transporter Gene SeHKT1 from the Halophyte Salicornia europaea [J].BiotechnologyBulletin, 31(11):159-165. (in Chinese)
[7] 孫偉民. 鹽角草SeCMO、SePEAMT基因在大腸桿菌中的表達及轉雙基因煙草耐旱性研究[D]. 大連:大連理工大學碩士學位論文, 2009.
SUN Wei-ming. (2009).ExpressionofSeCMOandSePEAMTgenesinE.coliandDroughtToleranceofTransgenicTobaccowithSePEAMY+SeCMOGene[D]. Mater Thesis. Dalian University of Techonology, Dalian. (in Chinese)
[8] 臧潔, 余梅, 王先磊, 等. 鹽角草Cu/Zn-SOD基因的克隆及耐鹽性分析[J].農業生物技術學報, 2013, 21(7): 847-854.
ZANG Jie, YU Mei, WANG Xian-lei, et al. Cloning and Salt-tolerance Analysis of the Salicornia europaea Gene Cu/Zn-SOD [J].JournalofAgriculturalBiotechnology, 21(7): 847-854. (in Chinese)
[9] 蔡倫,張富春,馬紀,等. 新疆3種藜科鹽生植物NHx基因的克隆與序列分析比較[J].植物生理學通訊,2005,41(3):383-387.
CAI Lun, ZHANG Fu-chun, MA Ji, et al. (2005). Cloning and Sequence Analysis of NHX Genes from Three Species of Halophytes from Xinjiang [J].PlantPhysiologyCommunications, 41(3):383-387. (in Chinese)
[10] Shao, Q., Zhao, C., Han, N., & Wang, B. S. (2008). Cloning and expression pattern of sshkt1 encoding a putative cation transporter from halophyte suaeda salsa.DNASequencetheJournalofDNASequencing&Mapping, 19(2):106.
[11] Liu, S., Wang, H., Kebin, W. U., & Xiong, X. (2017). Cloning and bioinformatics analysis of gpdh in rapeseed.ActaAgriculturaeBoreali-Sinica, 32(2):109-116.
[12] 周興文, 周銀慧, 趙英, 等. 金花茶CnBCH基因cDNA全長克隆及生物信息學分析[J].分子植物育種, 2017, 15(1):77-83.
ZHOU Xing-wen, Zhou Yin-hui, Zhao Ying, et al. (2017). Cloning and Bioinformatics Analysis of the Full-length of cDNA Sequence ofCnBCHGene from Golden Camellia (Camellianitidisima) [J].MolecularPlantBreeding, 15(1):77-83. (in Chinese)
[13]路東曄, 賀玉嬌, 金娜, 等. 沙柳SpsLAS基因克隆及分子生物信息學分析[J].分子植物育種, 2017, 15(2):483-491.
LU Dong-ye, He Yu-jiao, Jin Na, et al. (2017). Cloning and Bioinformatics Analysis ofSpsLASGene inSalixpsammophila[J].MolecularPlantBreeding, 15 (2):483-491. (in Chinese)
[14] 劉鋼, 肖前谷, 段瑞君, 等. 北美海蓬子SbNHX1基因耐鹽性及功能結構域分析[J].西北植物學報, 2009, 29(5):867-873.
LIU Gang, XIAO Qian-gu, DUAN Rui-jun, et al. (2009). Functional Domain and Salt-tolerance Analysis ofSbNHX1 Gene fromSalicorniabigeloviiTorr.[J]ActaBotBoreal, 29(5):867-873. (in Chinese)
[15] 劉倩, 高婭妮, 柳旭, 等. 植物對鹽堿脅迫的響應機制研究進展[J].生態學報, 2017, 37(16):25-33.
LIU Qian, GAO Ya-ni, LIU Xu, et al. (2017). Review on the mechanisms of the response to salinity-alkalinity stress in plants [J].ActaEcologicaSinica, 37(16): 25-33.
[16] Chen, A. P., Wang, G. L., Qu, Z. L., Lu, C. X., Liu, N., & Wang, F., et al. (2007). Ectopic expression of thcyp1, a stress-responsive cyclophilin gene from thellungiella halophila, confers salt tolerance in fission yeast and tobacco cells.PlantCellReports, 26(2):237-245.
[17] 李煥勇, 楊秀艷, 唐曉倩, 等. 植物響應鹽脅迫組學研究進展[J].西北植物學報, 2016, 36(12):2 548-2 557.
LI Huan-yong, YANG Xiu-yan, TANG Xiao-yan, et al. (2016). Omics Research Progress of Plant under Salt Stress [J].ActaBotBoreal, 36 (12): 2,548-2,557. (in Chinese)
[18] 林植芳, 劉楠. 活性氧調控植物生長發育的研究進展[J].植物學報, 2012, (47): 74-86.
LIN Zhi-fang, LIU Nan. (2012). Research Progress in the Control and Regulation of Plant Growth and Development by Reactive Oxygen Species [J].BulletinofBotany, 47(1): 74-86. (in Chinese)
[19] QIU Li-li, Zhao Qi, Zhang Yu-hong, et al. (2017). Research Advances in Stress-responsive Plant Plasma Membrane Proteomic [J].ChineseBulletinofBotany, 52(2): 128-147.
[20] 雍艷華, 張霞, 王紹明, 等. 新疆典型鹽生植物營養器官鹽分積累與生態化學計量特征[J].植物生態學報, 2016, 40(12): 1 267-1 275.
YONG Yan-hua, ZHANG Xia, WANG Shao-ming, et al. (2016). Salt accumulation in vegetative organs and ecological stoichiometry characteristics in typical halophytes in Xinjiang, China [J].ChineseJournalofPlantEcology, 40 (12): 1,267-1,275. (in Chinese)
[21] Nakashima, K., & Yamaguchi-Shinozaki, K. (2006). Regulons involved in osmotic stress-responsive and cold stress-responsive gene expression in plants.PhysiologiaPlantarum, 126(1): 62-71.
[22] Yoshimura, K., Miyao, K., Gaber, A., Takeda, T., Kanaboshi, H., & Miyasaka, H., et al. (2004). Enhancement of stress tolerance in transgenic tobacco plants overexpressing chlamydomonas glutathione peroxidase in chloroplasts or cytosol.PlantJournal, 37(1):21-33.
[23] Yoshimura K, Miyao K, Gaer A, Takeda T, Kanaboshi H, Miyasaka H,Shigeoka S. (2004) Enhancement of stress tolerance in transgenic tobacco Plants overexpressing Chlamydomonas glutathione peroxidase in chloroplasts or cytosol.PlantJournal, (37):21-33.
[24]Sunkar R, Bartels D, Kirch HH. (2003) Overexpression of a stress inducible aldehyde dehydrogenase gene from Arabidopsis thaliana in transgenic Plants improves stress tolerance.PlantJournal, (35):452-464.
Abstract:【Objective】 To clone a novelPsaHgene fromSalicorniaeuropaea, and analyze its biological information for better understanding its role in function of salt-tolerance.【Method】Salicorniaeuropaeawas used as the plant material to clone the full-length cDNA sequence ofSePsaHby RT-PCR. The encoding region and amino acid sequence ofPsaHgene, and the structure and function of protein encoded by protein were analyzed by NCBI, MEGA and Expasy and other online Bioinformatics bioinformatics software forSePsaHgene.【Result】 Full-length cDNA sequence encoding photosystem I reaction center subunit H was cloned fromSalicorniaeuropaeaand designated by the name ofSePsaH, which was an opening reading frame of 438bp encoding 145 amino acids. The putative protein molecular weight was 15.3kD and its theoretical isoelectric points was 9.84,SePsaHwas a hydrophilic protein; Phylogenetic analysis showed thatSePsaHgene andSpinaciaoleraceawere closely related; Through the conservative analysis of the protein, we found that there were 4 conserved domain structures.【Conclusion】SePsaHgene was cloned, which has laid the foundation for further study on the gene function and the role in salt-tolerance ofSalicorniaeuropaea.
Keywords:Salicorniaeuropaea; photosystem I reaction center subunit H; bioinformatics
CloningandBioinformaticsAnalysisofSalt-toleranceofPsaHGenefromSalicorniaeuropaea
HAO Xiao-yan, LI Jian-ping, Zumuremu Turxun, GAO Sheng-qi, HUANG Quan-sheng
(InstituteofNuclearandBiologicalTechnologies,XinjiangAcademyofAgriculturalSciences,Urumqi830091,China)
10.6048/j.issn.1001-4330.2017.09.006
S188
A
1001-4330(2017)09-1613-08
2017-07-18
新疆維吾爾自治區自然科學基金項目“堿蓬SsPSI基因提高轉基因煙草耐旱性的研究”(2015211A028);新疆農業科學院優秀青年科技人才基金“鹽生植物鹽角草耐鹽基因的發掘與功能研究”(xjnky-2012-003)
郝曉燕(1980-),女,新疆人,副研究員,碩士,研究方向為植物抗逆分子生物學,(E-mail)hxy29@126.com
黃全生(1964-),男,新疆烏魯木齊人,研究員,博士,博士生導師,研究方向為作物耐逆分子生物學,(E-mail)hquansheng@126.com
Supported by: Natural Science Foundation of Xinjiang "Study on Improving Drought Resistance of Transgenic Tobacco by Suaeda Salsa SsPSI Gene" (2015211A028) and Outstanding Young Scientific Talent Foundation of Xinjaing Academy of Agricultural Sciences "Discovery and Function of Salt Tolerant Genes in Halophytes" (xjnky-2012-003)
Corresponding author:HUANG Quan-sheng(1964-),male, Urumqi, Xinjiang, Professor, supervisor, the main research directions for crop adversity molecular biology,(E-mail) hquansheng@126.com

