基于SNP標記的桃矮化基因精細定位
魯振華,牛良,張南南,姚家龍,崔國朝,曾文芳,潘磊,王志強
(1中國農業科學院鄭州果樹研究所/國家桃葡萄品種改良中心/農業部果樹育種技術重點實驗室,中國鄭州 450009;2新西蘭植物與食品研究所,新西蘭 Auckland 1142)
園藝
基于SNP標記的桃矮化基因精細定位
魯振華1,牛良1,張南南1,姚家龍2,崔國朝1,曾文芳1,潘磊1,王志強1
(1中國農業科學院鄭州果樹研究所/國家桃葡萄品種改良中心/農業部果樹育種技術重點實驗室,中國鄭州 450009;2新西蘭植物與食品研究所,新西蘭 Auckland 1142)
【目的】矮化型桃樹體矮小、節間短,是盆栽觀賞和砧木育種的重要遺傳資源。明確矮化性狀形成的遺傳機制并對桃矮化基因進行精細定位,是建立目標性狀分子輔助選種體系和遺傳改良的前提,可為有目標的選育矮化觀賞桃和砧木品種奠定基礎。【方法】以‘05-2-144’(‘97矮’ב鴛鴦垂枝’桃)套袋自交獲得的395個后代單株構建的分離群體為材料。參考桃基因組信息并基于Sanger技術開發的SNP標記對親本和后代單株進行分析,在擴大群體單株中進行連鎖關系分析,確定連鎖的SNP標記,初步定位目標基因。在定位區域內基于二代測序技術開發更多的基因型和表型一致的SNP標記,對后代單株進行基因分型,完成目標性狀的精細定位。然后在精細定位區域內開發SNP標記,即雜交群體雙親均為Aa雜合基因型,對‘10-7’ב96-5-1’雜交后代89個單株進行分子鑒定,以驗證基因定位結果的準確性。【結果】通過對桃單株‘05-2-144’自交后代實生苗表型鑒定表明,普通型和矮化型單株數分別為300株和95株,性狀分離比例接近3∶1(P值為0.67;χ2為0.19),符合孟德爾遺傳規律,桃矮化性狀受隱性單基因控制。用于分子鑒定的單株來源于‘10-7’(普通型)ב96-5-1’(普通型)雜交組合,共獲得89個后代單株,其中普通型66株;矮化型23株(P值為0.854;χ2為0.034)。基于Sanger測序技術開發了SNP標記,在桃基因組數據庫Pp06上25 230 425 bp和27 191 090 bp處獲得了連鎖的SNP標記,且目標基因位于這兩個標記的右側,初步獲得了連鎖的SNP分子標記。在此基礎上,對親本進行66.89X深度測序,繼續開發符合Aa雜合基因型的SNP標記位點。根據參考基因組和物理距離區間共設計了15對SNP引物,其中12對引物與重測序結果中SNP的類型一致,3對引物與重測序結果中SNP的類型不一致,連鎖標記的基因分型成功率為80.0%。通過基于SNP基因分型分析,最終完成了目標性狀的精細定位,位點位于Pp06的28 712 165 bp(引物為JXSNP-5)和28 899 661 bp(引物為JXHRM-SNP-3)之間,遺傳距離分別為0.38 cM和0.13 cM,物理距離約為277 kb,精細定位區域內有54個已知轉錄本。在定位區域內桃基因組Pp06的28 108 436 bp處和29 247 763 bp處開發 SNP標記用于雜交后代表型的鑒定,結果表明所有后代單株基因型和表型鑒定結果完全一致,鑒定準確率為100%。【結論】本研究精細定位了桃矮化基因,物理距離約為277 kb,為基因克隆、親本早期篩選以選育矮化觀賞桃和砧木品種等奠定了基礎。
桃;矮化基因;SNP;精細定位
Abstract:【Objective】 Dwarfing peach, due to the small trees, shorter internodes, is an important genetic resources in ornamental and rootstock breeding program. The fine mapping of peach dwarfing genes and explication of the genetic mechanism will provide a basis for the establishment of a molecular-assisted selection system and desired variety improvement, which could be used in breeding dwarf ornamental peach and rootstock varieties. 【Method】 Based on the results of genetic analysis in this study, 395 individuals generated from a self-pollinated population of 05-2-144 were selected for fine mapping the dwarfing gene in peach (Prunus persica (L.) Batsch). By referencing the peach genome sequence, SNP markers were developed in the parents and progenies to generate markers linked to the locus and used to map the gene based on Sanger sequencing. Subsequently, the parent was resequenced to generate SNP markers in the mapping region and acquire heterozyous SNPs for fine mapping. Within the fine mapping region, SNP primers were designed to verify the phenotype of 89 individuals generated from the F1segregation population of 10-7×96-5-1.【Result】The segregating population of 05-2-144 was generated to assess the genetic characteristics,resulting in observed 3:1 (300 standard type and 95 dwarfing type individuals) Mendelian ratio fitting with the expected ratio for a monogenic recessive genetic control (P=0.67, χ2=0.19). Moreover, the ratio of standard type to dwarf type corresponded to the expected 3:1 (66 standard type and 23 dwarfing type individuals) segregation (P=0.854, χ2=0.034) for molecular detection. Based on Sanger sequencing results, the linked SNP markers were identified in the position 25 230 425 bp and 27 191 090 bp on Pp06 of peach genome and the locus were on the right side of these two markers. For fine mapping, the parent of this segregation population was resequenced with 66.89X depth and identified the heterozygous SNP to develop SNP markers. Totally, fifteen SNP primers were designed and 12 SNPs (80.0%) were consistent with resequencing data. The dwarfing locus of this type was narrowed on Pp06 between the position 28 712 165 bp and 28 899 661 bp with genetic distances of 0.38 cM and 0.13 cM. The physical region of fine mapping was 277 kb containing 54 known transcripts. Within 28 108 436bp and 29 247 763 bp on Pp06,SNP markers were developed and detected the progeny phenotype of a segregation population of 10-7×96-5-1 with 100%accuracy, constituted of 89 individuals. 【Conclusion】 The dwarfing gene was fine mapped on Pp06 within a physical distance 277 kb. The results of this study will be helpful to clone dwarfing gene and select parents for breeding dwarfing ornamental peach and rootstock varieties.
Key words:peach; dwarfing gene; SNP; fine mapping
0 引言
【研究意義】桃[Prunus persica (L.)Batsch]是中國栽培面積較大的落葉果樹之一。2014年中國桃栽培面積約799 500 hm2,居世界桃栽培第一位(FAO)。其中,矮化型桃因其節間短、成花好是觀賞桃和砧木育種的發展方向之一。通過對桃矮化基因進行精細定位是建立目標性狀分子輔助選種體系和品種改良的基礎。【前人研究進展】中國是桃起源中心,具有豐富的遺傳資源。作為重要的農藝性狀之一,樹型種類多樣,包括普通生長型(Standard type)、柱型(Pillar type)、半矮生型(Semi-dwarf type)、垂枝型(Weeping type)、直立型(Upright type)、短枝型(Spur type)和矮化型(Dwarf type)等幾類[1]。其中,矮化型桃的明顯特征是節間長度極短、樹冠密。LAMMERTS[2]最早報道了來源中國的矮化桃遺傳資源,之后多位科研人員開始了一系列的研究[3],并育成了不少矮化桃品種,如‘Bonfire’‘Leprechaun’等[4]。國內育種家同步選育了矮化觀賞型桃品種,宗學普等[5]開展了桃矮化品種的選育,選出的矮化油桃新品系‘矮麗紅’。YAMAMOTO等[6]是最早開展桃矮化基因的定位工作,將控制桃矮化的 dw定位在連鎖群 6的前端。HOLLENDER等[7]采用重測序的方法將來源日本觀賞桃的 dw進行定位,獲得了編碼赤霉素受體的候選基因(GID1c)。【本研究切入點】矮化型桃有多種類型,由于樹體矮小,是盆栽觀賞和砧木的重要育種資源。雖然,我國擁有重要的矮化性狀遺傳資源,但是桃矮化基因定位研究工作開展較少,相關的分子輔助選種體系還沒有建立。【擬解決的關鍵問題】本研究以來源中國的矮化桃品種為材料,通過構建雜交群體對控制桃矮化性狀基因進行精細定位,獲得緊密連鎖的SNP標記,為建立目標性狀的分子鑒定體系,有目的選育矮化觀賞桃和砧木品種奠定基礎。
1 材料與方法
田間試驗于 2014—2016年在中國農業科學院鄭州果樹研究所新鄉試驗基地(河南新鄉)進行,室內試驗在中國農業科學院鄭州果樹研究所農業部果樹育種技術重點實驗室(河南鄭州)進行。
1.1 研究材料
根據該矮化性狀的遺傳特征,以單株‘05-2-144’為親本自交構建分離群體,其中‘05-2-144’來源于2005年‘97矮’(矮化型)ב鴛鴦垂枝’桃(非矮化型)雜交單株。于 2014年和 2015年分別以‘05-2-144’為親本自交,共獲得 395個后代單株。用于分子鑒定的親本為‘10-7’(普通型)ב96-5-1’(普通型),雙親均來源于97矮毛桃,共獲得89個后代單株;2個組合的后代桃核經自然干燥后用錘子將外殼敲開,取種子進行包衣(包衣劑,先正達),晾干后,放于濕潤的粗砂中進行層積處理,次年1月取出萌發的種子,種植于穴盤中用于表型觀察和后續研究。
1.2 基因組DNA的提取
基因定位親本和自交后代單株葉片基因組 DNA的提取采用 CTAB法[8],略作修改。獲得沉淀 DNA后,加入200 μL體積的0.1×TE溶解沉淀DNA,同時加入0.5 μL的RNase,去除RNA污染(長期保存在-20℃冰箱,常用則存于4℃冰箱)。采用NanoDrop 1000 spectrophotometer(Themo)和1%的瓊脂糖膠檢測DNA的純度濃度和完整度,稀釋成工作液濃度(25 ng·μL-1)備用。自交親本提取DNA純化后送樣建庫。
1.3 基于Sanger測序SNP標記的開發
參考桃基因組序列信息(Genome Database for Rosaceae數據庫Version 2.0)中桃基因組Pp 01—08,采用 primer3Web Version 4.0(http://primer3.ut.ee/)設計引物,約每1—2 Mb設計1對引物,引物退火溫度在 59—63℃,引物長度20—23 bp,引物擴增片段長度為1 600 bp或750 bp左右。基于Sanger測序開發基因型和表型一致的SNP標記,用于目標基因的定位。
1.4 基于二代測序SNP標記的開發
構建好文庫后對親本‘05-2-144’進行深度測序,在儀器HiSeq 4000(Illumina,CA)上進行,測序深度約為70X。在定位區域內采用Integrative Genomics Viewer 2.3(IGV)對Bam格式數據進行分析[9],確定目標區域內基因型為Dwdw的SNP,以設計候選標記,用于連鎖SNP標記的開發。
1.5 基于HRM技術的SNP基因分型
根據深度測序的結果設計引物。采用HRM master mix(Roche)試劑進行 PCR 擴增,反應總體積為 15 μL。其中含15 ng模板DNA、0.17 μmol·L-1正/反引物和1μL HRM master熒光染料(Roche)。在羅氏LightCycler 480II定量PCR儀(Roche)進行PCR擴增和HRM分析。高分辨率熔解曲線分析采用Gene Scanning軟件(version1.50),具體參考魯振華等[10]。
1.6 矮化性狀的精細定位與驗證
根據表型鑒定結果,在矮化型和普通型各兩個子代和親本中尋找連鎖的 SNP標記。具體基因型表現為,自交親本單株‘05-2-144’基因型為Dwdw,矮化型子代基因型為 dwdw,普通型子代基因型為 DwDw和Dwdw,在4個子代中獲得連鎖的SNP標記后,擴大至分離后代各20個單株中,確定連鎖后進一步擴大至全部樣品,完成初步定位。同時結合親本二代測序結果開發的 SNP標記,在群體所有后代單株中進行SNP分析,確定交換單株并獲得緊密連鎖的 SNP標記。遺傳距離的計算采用 JoinMap(3.0)[11],以 F2群體方法進行遺傳距離計算,參數設置為默認。
2 結果
2.1 雜交群體表型鑒定和遺傳特征分析
通過對‘05-2-144’自交后代實生苗觀察,2014年得到102株雜種苗,其中普通型和矮化型分別為78株和24株,二者比例接近3∶1(P值為0.732;χ2為0.118)。為進一步對目標基因進行精細定位,于2015年進行了自交,共得到293株自交后代單株,對其進行目測觀察,區分普通型和矮化型幼苗分開定植。其中普通型222株,矮化型71株,二者比例接近3∶1(P值為0.761;χ2為0.092)。用于分子鑒定的單株來源于‘10-7’(普通型)ב96-5-1’(普通型)雜交組合,共獲得89個后代單株,其中普通型 66株;矮化型 23株(P值為 0.854;χ2為0.034)。目標性狀分離比例符合孟德爾遺傳規律,矮化性狀受隱性單基因控制。同時對定植2年后植株表型進行了再鑒定,幼苗與定植后2年植侏表型觀察結果一致(圖1)。
2.2 矮化基因的初步定位
參考桃基因組(https://www.rosaceae.org/gb/gbrowse/prunus_persica_v2.0.a1/ Version 2.0)序列信息,設計了桃基因組Pp 01—08的引物,約每1—2 Mb設計1對引物,引物擴增片段長度為1 600 bp或750 bp左右(表1)。基于Sanger測序法獲得SNP標記,并根據子代基因型和表型確定候選SNP標記,并在雜交群體后代單株中(普通型和矮化型各 4株)進行序列測定獲得多態性的SNP標記進行初步分析。在Pp06的25 230 425 bp和27 191 090 bp處找到了可能連鎖的SNP標記。在獲得可能的 SNP標記后擴大了雜交群體,在普通型和矮化型各20個后代單株中進一步驗證連鎖關系,進而在全部樣品中驗證。通過擴大群體分析,在 Pp06 上 25 230 425 bp(LG6-25.2)和 27 191 090 bp(LG6-27)處獲得了與dw連鎖的SNP標記,根據重組單株以及重組單株數目的變化表明,目標基因位于獲得的兩個標記右側。

圖1 普通型桃(左)和矮化型桃(右)植株表型Fig. 1 Phenotype of standard type (left) and dwarf type (right)peach
2.3 矮化基因的精細定位
在初步獲得連鎖的SNP分子標記后,對親本進行深度測序。根據親本‘05-2-144’基因型和表型開發更多Aa基因型的SNP標記。后擴大分離群體進行連鎖關系驗證,以獲得緊密連鎖的 SNP標記。親本‘05-2-144’測序共產生Raw data 17.956 Gb,過濾后的Clean data 17.098 Gb,測序質量較高,其中Q20≥94.9%,Q30≥88.57%,全基因組GC含量為39.04%。參考基因組大小為227 411 381 bp,所有樣本的比對率為 96.71%,對參考基因組(排除 N區)的平均覆蓋深度為66.89X,1X覆蓋度(至少有一個堿基的覆蓋)在98.77%以上,可用于后續的變異檢測及相關分析。根據開發的SNP標記,在參考基因組(Version 2.0)30.003191 Mb處獲得了另一側連鎖的SNP標記。基于重測序數據和定位區間,共設計了15對SNP引物,其中12對引物與重測序SNP基因型一致,3對引物與重測序SNP基因型不一致成功率為80.0%(表1)。利用其中SNP標記進行了目標性狀的精細定位,在桃基因組Pp06的28 712 165 bp(JXSNP-5)和28 989 661 bp(JXHRM-SNP-3)之間,物理距離區間約為277 kb(圖2)。

圖2 桃矮化基因的遺傳距離分析Fig. 2 Linkage map of dwarfing gene for peach
2.4 精細定位區域的候選基因
參考桃基因組數據庫(Version 2.0)在精細定位區域277 kb內,包含已知轉錄本54個,具體如表2所示。精細定位區域內基因包括螺旋-環-螺旋(bHLH)、DHHC類鋅指蛋白、GAG反轉座子、信號肽酶、受體類激酶、赤霉素受體(GA不敏感矮化型)、未知功能蛋白等(表2)。其中,GIDC基因可能是候選基因,這與HOLLENDER等[7]報道一致。
2.5 植株表型的分子鑒定
根據已經獲得的緊密連鎖的 SNP標記的物理位置,并基于參考桃基因組數據在新的親本(‘10-7’X‘96-5-1’)中開發基因型和表型一致的 SNP標記用于分子鑒定,在精細定位目標基因兩翼分別設計1對SNP引物,驗證分子標記對雜交后代單株表型鑒定的準確性。其中引物YZ-SNP260k1位于Pp06的28 108 436 bp處,SNP類型為C/T;引物YZ-SNP260k2位于Pp06的29 247 763 bp處,SNP類型為G/A。通過標記基因型和表型比較,89株后代單株的驗證符合率為100%(圖3)。
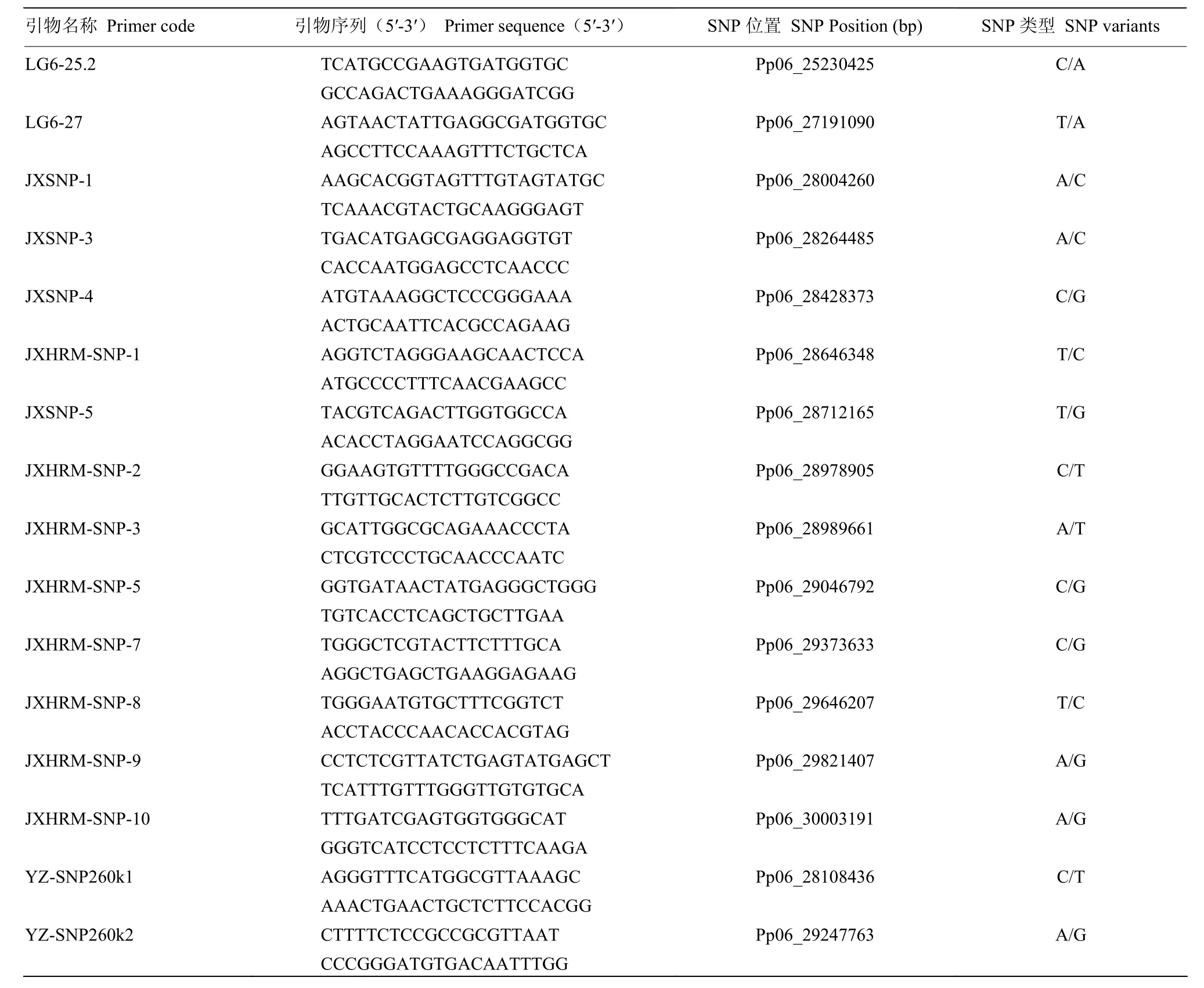
表1 本研究所用引物信息Table 1 Primers information used in this study
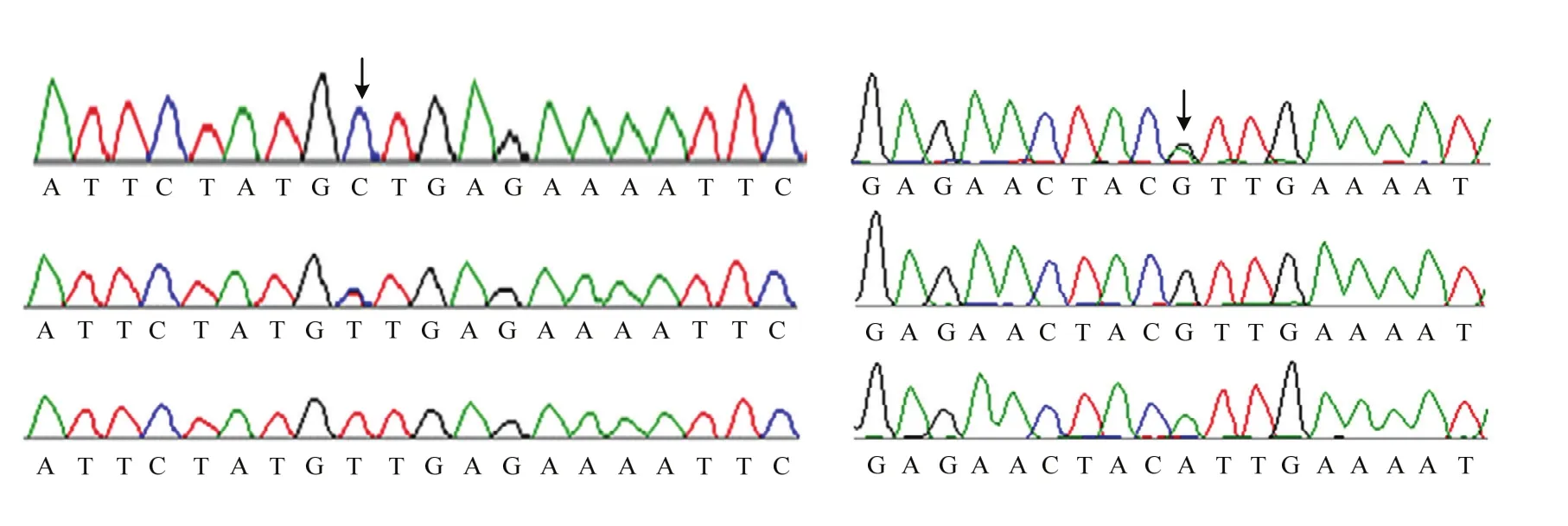
圖3 基于SNP標記桃矮化性狀的分子鑒定Fig. 3 Dwarfing trait detection for peach based on SNP Marker

表2 精細定位區域內候選基因Table 2 Candidate genes in fine mapping region

續表2 Continued table 2
3 討論
在遺傳圖譜構建方面,SNP標記優勢突出[12-13]。近年來隨著生物技術的進步,特別是基于二代測序的標記技術加速了作物基因的精細定位和分子輔助選種體系的建立[14-17]。建立桃樹分子輔助選種體系是實現目標性狀早期鑒定的關鍵環節,可加速育種進程,實現多個優良性狀的聚合,提高果樹的經濟價值。如何準確鑒定目標性狀是育種者關心的科學問題。精細定位和克隆目標性狀基因是關鍵,在精細定位區域內開發與目標性狀緊密連鎖的分子標記,包括 SSR、SNP和 Indel等,而克隆到目標基因是實現對目標性狀直接鑒定的重要途徑。在桃中,已完成紅肉性狀[18]、黃/白肉[19-20]、果皮顏色亮度[21]、肉質類型[22]、分枝角度[23]和矮化性狀[7]等基因的直接分子鑒定。以上研究為加快桃遺傳改良進程奠定了基礎。
果樹樹型性狀與果實性狀不同,在苗期即可實現部分性狀的表型鑒定,但對于隱性單基因和多基因控制的樹體性狀,很難實現早期的表型鑒定,仍需通過基因定位建立輔助選種體系[22]。果樹作物中,蘋果最先在生產中利用了柱型性狀,實現了高產、高密栽培,兼顧省時和省力[23-27]。對桃樹型研究主要集中在株高和節間長度方面[28-30]。YAMAMOTO等[6]將控制桃株高矮化基因定位在 LG6上;張妤艷等[31]采用 Fl雜交分離群體共 123個單株,獲得了與矮化性狀連鎖的SRAP標記,遺傳距離3.2 cM。最近,HOLLENDER等[7]采用BSA-seq重測序的方法對來源于日本矮化品種‘Juseito’的BD(Brachytic dwarfism)性狀的dw位點進行了定位,獲得了候選基因赤霉素受體(GID1c),并驗證了基因的功能。盡管功能驗證表明GID1c與株高表型有關,但該矮化基因是從900 Kb的定位區間內篩選的候選基因,本研究則提供了進一步的數據支持。本研究定位的 dw基因與日本的矮化品種‘Juseito’的dw位于同一區域,但二者是否存在不同的變異形式以及來源是否相同,仍需進一步研究。
4 結論
本研究對來源‘97矮’桃單株的矮化性狀基因進行了精細定位,目標性狀位點位于Pp06的28 712 165—28 899 661 bp,物理距離約為277 kb,包含54個已知轉錄本。在精細定位目標基因兩翼分別設計 1對SNP引物,89株后代單株的分子鑒定符合率為100%,研究結果為揭示桃矮化性狀的遺傳機制并建立目標性狀的分子輔助選種體系奠定了基礎。
[1]BASSI D, DIMA A, SCORZA R. Tree structure and pruning response of six peach growth forms. Journal of the American Society for Horticultural Science, 1994, 119(3): 378-382.
[2]LAMMERTS W E. The breeding of ornamental edible peaches for mild climates.I. inheritance of tree and flower characters. AmericanJournal of Botany, 1945, 32: 53-61.
[3]SCORZA R. Characterization of four distinct peach tree growth types.Journal of the American Society for Horticultural Science, 1984,109(4): 455-457.
[4]MOORE J N, ROM R C, BROWN S A, KLINGAMAN G L.‘Bonfire’ dwarf peach, ‘Leprechaun’ dwarf nectarine, and ‘Crimson Cascade’ and ‘Pink Cascade’ weeping peaches. Hortscience, 1993,28(8): 854.
[5]宗學普, 張貴榮, 王志強, 劉淑娥. 矮化型油桃新品種-矮麗紅. 落葉果樹, 1997(2): 26.ZONG X P, ZHANG G R, WANG Z Q, LIU S E. Dwarf nectarine variety - ‘Ailihong’. Deciduous Fruits, 1997(2): 26. (in Chinese)
[6]YAMAMOTO T, SHIMADA T, IMAI T, YAEGAKI H, HAJI T,MATSUTA N, YAMAGUCHI M, HAYASHI T. Characterization of morphological traits based on a genetic linkage map in peach.Breeding Science, 2001, 51: 271-278.
[7]HOLLENDER C A, HADIARTOT, SRINIVASAN C, SCORZA R,DARDICK C. A brachytic dwar fi sm trait (dw) in peach trees is caused by anonsense mutation within the gibberellic acid receptor PpeGID1c.New Phytologist, 2015, 210(1): 227-239.
[8]DOYLE J J, DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin, 1987, 19:11-15.
[9]ROBINSON J T, THORVALDSDóTTIR H, WINCKLER W,GUTTMAN M, LANDER E S, GETZ G, MESIROV J P. Integrative genomics viewer. Nature Biotechnology, 2011, 29: 24-26.
[10]魯振華, 牛良, 張南南, 崔國朝, 潘磊, 曾文芳, 王志強. 基于HRM獲得與桃Tssd緊密連鎖的SNP標記. 中國農業科學, 2017,50(8): 1505-1513.LU Z H, NIU L, ZHANG N N, CUI G C, PAN L, ZENG W F, WANG Z Q. SNP marker tightly linked to Tssd for peach using high resolution melting analysis. Scientia Agricultura Sinica, 2017, 50(8):1505-1513. (in Chinese)
[11]VAN OOIJEN J W, VOORRIPS R E. JionMap version 3.0: Software for the calculation of genetic linkage maps. Plant Research International,Wageningen, The Netherlands, 2001.
[12]BALL A, STAPLEY J, DAWSON D, BIRKHEAD T, BURKE T,SLATE J. A comparison of SNPs and microsatellites as linkage mapping markers: Lessons from the zebra finch (Taeniopygia guttata).BMC Genomics, 2010, 11(1): 218.
[13]MARTíNEZ-GARCíA P J, PARFITT D E, OGUNDIWIN E A,FASS J, CHAN H M, AHMAD R, LURIE S, DANDEKAR A,GRADZIEL T M, CRISOSTO C H. High density SNP mapping and QTL analysis for fruit quality characteristics in peach (Prunus persica L.). Tree Genetics and Genomes, 2013, 9: 19-36.
[14]TAKAGI H, ABE A, YOSHIDA K, KOSUGI S, NATSUME S,MITSUOKA C, UEMURA A, UTSUSHI H, TAMIRU M, TAKUNO S, INNAN H, CANO L M, KAMOUN S, TERAUCHI R. QTL-seq:rapid mapping of quantitative trait loci in rice by whole genome resequencing of DNA from two bulked populations. The Plant Journal, 2013, 74: 174-183.
[15]ABE A, KOSUGI S, YOSHIDA K, NATSUME S, TAKAGI H,KANZAKI H, MATSUMURA H, YOSHIDA K, MITSUOKA C,TAMIRU M, INNAN H, CANO L, KAMOUN S, TERAUCHI R.Genome sequencing reveals agronomically important loci in rice using MutMap. Nature Biotechnology, 2012, 30(2): 174-179.
[16]李娜, 王吉明, 尚建立, 李楠楠, 徐永陽, 馬雙武. 西瓜枯萎病生理小種 1抗性 QTL精細定位與 InDel標記開發. 中國農業科學,2017, 50(1): 131-141.LI N, WANG J M, SHANG J L, LI N N, XU Y Y, MA S W.Fine-mapping of QTL and development of InDel markers for Fusarium oxysporum race 1 resistance in watermelon. Scientia Agricultura Sinica, 2017, 50(1): 131-141. (in Chinese)
[17]LU Z H, NIU L, CHAGNé D, CUI GG C, PAN L, FOSTER T,ZHANG R P, ZENG W F, WANG Z Q. Fine mapping of the temperature-sensitive semi-dwarf (Tssd) locus regulating the internode length in peach (Prunus persica). Molecular Breeding, 2016, 36: 20.DOI: 10.1007/s11032-016-0442-6.
[18]SHEN Z J, CONFOLENT C, LAMBERT P, PO?SSEL J-L,QUILOT-TURION B, YU M L, MA R J, PASCAL T. Characterization and genetic mapping of a new blood-flesh trait controlled by the single dominant locus DBF in peach. Tree Genetics and Genomes,2013, 9: 1435-1446.
[19]ADAMI M, FRANCESCHI P D, BRANDI F, LIVERANI A,GIOVANNINI D, ROSATI C, DONDINI L, TARTARINI S.Identifying a carotenoid cleavage dioxygenase (ccd4) gene controlling yellow/white fruit flesh color of peach. Plant Molecular Biology Reporter, 2013, 31(5): 1166-1175.
[20]MA J J, LI J, ZHAO J B, ZHOU H, REN F, WANG L, GU C, LIAO L,HAN Y P. Inactivation of a gene encoding carotenoid cleavage dioxygenase (CCD4) leads to carotenoid-based yellow coloration of fruit flesh and leaf midvein in Peach. Plant Molecular Biology Reporter, 2014, 32(4): 246-257.
[21]BRETó M P, CANTíN C M, IGLESIAS I, ARúS P, EDUARDO I.Mapping a major gene for red skin color suppression (highlighter) in peach. Euphytica, 2017, 213: 14. DOI: 10.1007/s10681-016-1812-1.
[22]PAN L, ZENG W F, NIU L, LU Z H, WANG X B, LIU H, CUI G C,ZHU Y Q, CHU J F , LI W P, FANGWC, CAI Z G, LI G H, WANG Z Q. PpYUC11, a strong candidate gene for the stony hard phenotype in peach (Prunus persica L. Batsch), participates in IAA biosynthesis during fruit ripening. Journal of Experimental Botany, 2015. Doi:10.1093/jxb/erv400.
[23]DARDICK C, CALLAHAN A, HORN R, RUIZ KB, ZHEBENTYAYEVA T, HOLLENDER C, WHITAKER M, ABBOTT A, SCORZA R.PpeTAC1 promotes the horizontal growth of branches in peach trees and is a member of a functionally conserved gene family found in diverse plants species. The Plant Journal, 2013, 75: 618-630.
[24]OTTO D, PETERSEN R, BRAUKSIEPE B, BRAUN P, SCHMIDT E R. The columnar mutation (‘Co gene’) of apple (Malus × domestica)is associated with an integration of a Gypsy-like retrotransposon.Molecular Breeding, 2013, 33(4): 863-880.
[25]MORIYA S, OKADA K, HAJI T, YAMAMOTO T, ABE K. Fine mapping of Co, a gene controlling columnar growth habit located on apple (Malus × domestica Borkh.) linkage group 10. Plant Breeding,2012, 131(5): 641-647.
[26]OGNJANOV V, VUJANIC-VARGA D, GASIC K. Breeding columnar apples in Novi Sad. Acta Horticulturae, 1999, 484: 207-209.
[27]MORIYA S, IWANAMI H, KOTODA N, TAKAHASHI S,YAMAMOTO T, ABE K. Development of a marker-assisted selection system for columnar growth habit in apple breeding. Journal of the Japanese Society for Horticultural Science, 2009, 78: 279-287.
[28]VERDE I, BASSIL N, SCALABRIN S, GILMORE B, LAWLEY C T.Correction: Development and evaluation of a 9K SNP array for peach by internationally coordinated SNP detection and validation in breeding germplasm. PLoS ONE, 2012, 7(6): e35668. DOI: 10.1371/journal.pone.0035668.
[29]CANTíNA C M, CRISOSTOB C H, OGUNDIWINB E A,GRADZIELB T, TORRENTSC J, MORENOA M A, GOGORCENA Y. Chilling injury susceptibility in an intra-speci fi c peach [Prunus persica (L.) Batsch]progeny. Postharvest Biology and Technology,2010, 58: 79-87.
[30]VERDE I, QUARTA R, CEDROLA C, DETTORI M T. QTL analysis of agronomic traits in a BC1 peach population. Acta Horticulturae, 2002,592: 291-297.
[31]張妤艷, 俞明亮, 馬瑞娟, 蔡志翔, 沈志軍, 許建蘭. 與桃樹性高/矮性狀相關的SRAP標記. 果樹學報, 2011, 28(4): 586-590.ZHANG Y Y, YU M L, MA R J, CAI Z X, SHEN Z J, XU J L.Identification of SRAP markers linked to peach gene with the character of tall normal/brachytic dwarf. Journal of Fruit Science,2011, 28 (4): 586-590. (in Chinese)
(責任編輯 趙伶俐)
Fine Mapping of Dwarfing Gene for Peach Based on SNP Markers
LU ZhenHua1, NIU Liang1, ZHANG NanNan1, YAO JiaLong2, CUI GuoChao1, ZENG WenFang1,PAN Lei1, WANG ZhiQiang1
(1Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences/National Peach and Grape Improvement Center/Key Laboratory of Fruit Breeding Technology of Ministry of Agriculture, Zhengzhou 450009, China;2The New Zealand Institute for Plant & Food Research Limited, Auckland 1142, New Zealand)
2017-05-02;接受日期:2017-07-25
國家自然科學基金(31500558)、中國農業科學院創新工程(CAAS-ASTIP-2017-ZFRI)、中央級科研院所基本科研業務費專項(1610192017702)
聯系方式:魯振華,Tel:0371-55001909;E-mail:luzhenhua@caas.cn。通信作者王志強,Tel:0371-65330988;E-mail:wangzhiqiang@caas.cn

