羊口瘡病毒重慶株 F1L基因克隆與生物信息學分析
李鵬飛,馮 將,劉 嬡,包細明,張 益,李 婷,鮮思美,2(.貴州大學 動物科學學院,貴陽 550025;2.貴州省動物疫病研究所,貴陽 550025)
羊口瘡病毒重慶株 F1L基因克隆與生物信息學分析
李鵬飛1,馮 將1,劉 嬡1,包細明1,張 益1,李 婷1,鮮思美1,2
(1.貴州大學 動物科學學院,貴陽 550025;2.貴州省動物疫病研究所,貴陽 550025)
旨在分析OrfV-CQ株 F1L基因的分子特點,預測其編碼蛋白的生物學功能。對OrfV-CQ株 F1L基因進行PCR擴增、克隆及序列測定,利用生物信息學相關軟件進行序列分析并對其編碼蛋白的二級結構、B細胞表位、T細胞表位、保守結構域、跨膜結構域和信號肽進行預測。結果表明,OrfV-CQ株 F1L基因長1 023 bp,編碼340 個氨基酸,與Shanxi株 F1L基因的核苷酸和氨基酸同源性分別為98.4%和95.0%。OrfV-CQ 株 F1L基因編碼蛋白的α-螺旋、β-片層、β-轉角、無規則卷曲分別為11.21%、10.3%、2.73%、75.76%;可能存在5 個B細胞優勢抗原表位,3 個CTL表位,4 個Th細胞表位,無跨膜結構域和信號肽。
羊口瘡病毒; F1L基因;克隆;生物信息學分析
羊口瘡(Orf)是由羊傳染性膿皰病毒(Orf virus,OrfV)引起的一種接觸性、傳染性、嗜上皮性人畜共患傳染病[1],其主要特征病變為皮膚、口唇、鼻部和口腔黏膜等處形成增生性皮膚損傷,多發于6 月齡內的羔羊[2]。OrfV粒子長200~350 nm,寬140~220 nm,基因組長度為130~150 kb,不同毒株基因組長度有10~25 kb的差異[3]。 F1L基因位于OrfV的第59 個開放閱讀框,編碼的39 ku蛋白是表面微管的重要組成,該蛋白與病毒吸附、侵入宿主細胞及病毒成熟有關,還可以誘導機體產生中和抗體并介導細胞免疫[4-5]。本研究采用PCR技術擴增羊口瘡病毒重慶分離株(OrfV-CQ) F1L基因,進行序列測定后,采用在線程序和生物信息學分析軟件對其核苷酸序列和推導的氨基酸序列進行生物信息學分析和預測,為重慶地區羊口瘡的防治、OrfV的遺傳變異研究奠定基礎。
1 材料與方法
1.1 主要材料
OrfV-CQ株、大腸桿菌DH5α感受態細胞、pMD18-T載體均由貴州省動物疫病研究所保存;T4DNA連接酶、PCR反應試劑、限制性內切酶NotⅠ和EcoRⅠ、DL 2000 DNA Marker 購自TaKaRa公司;2×TaqPCR Master Mix、質粒小量提取試劑盒、DNA凝膠回收試劑盒、DNA病毒基因組提取試劑盒購自北京康為世紀生物科技有限公司。
1.2 引物的設計與合成
根據GenBank公布的OrfV標準序列(No.AY040083),設計合成1 對引物用于擴增 F1L基因,F:5′-CCGGAATTCATGGATCCACCCGAAAT-3′(EcoRⅠ);R:5′-ATAAGAATGCGGCCGCTCACACGATGGCCGTGAC-3′(NotⅠ),劃線部分為酶切位點,預期擴增片段長度為1 023 bp。
1.3 F1L基因的克隆與鑒定
使用DNA病毒基因組提取試劑盒提取痂皮組織 DNA。以提取的 DNA為模板,進行 PCR擴增。使用膠回收試劑盒回收并純化目的片段,連接至 pMD18-T載體,轉化至大腸桿菌 DH5α感受態細胞。培養后提取重組質粒,經 PCR和雙酶切鑒定后,陽性重組質粒送生工生物工程(上海)股份有限公司測序。
1.4 序列分析
采用NCBI的BLAST對比驗證所測序列,并使用在線程序和生物信息學分析軟件對所測核苷酸序列與選取的參考序列進行比對和生物信息學分析。
2 結果與分析
2.1 FlL基因的PCR擴增與克隆鑒定
以提取的病毒DNA為模板進行PCR擴增,得到長約1 023 bp的片段(圖1)。用設計的特異性引物對重組質粒進行PCR擴增,得到約1 023 bp的特異性片段(圖2)。經EcoRⅠ和NotⅠ雙酶切,得到2 條條帶,即載體帶和目的條帶(圖3)。經測序證實目的基因正確插入到pMD18-T載體中。

M.DNA Marker;1.羊口瘡病毒 OrfV
2.2 序列及遺傳進化分析
經測序拼接后得到OrfV-CQ株 F1L基因長度為1 023 bp,使用DNAStar推導其編碼340個氨基酸(圖4)。應用DNAStar將OrfV-CQ株 F1L序列與GenBank中收錄的17株OrfV F1L基因序列進行比對分析,核苷酸序列同源性為93.8%~98.4%(圖5),氨基酸同源性為11.5%~95.0%(圖6),其與Shanxi株(No:HQ221964)的核苷酸和推導氨基酸的同源性分別為95.0%和98.4% 。依據OrfV F1L基因核苷酸序列繪制遺傳進化樹(圖7),OrfV-CQ株與國內毒株中Shanxi株的親緣關系最近,與Jinlin株的親緣關系最遠;與國外毒株中SA00株的親緣關系最近,與OV-Torino的親緣關系最遠。

M.DNA Marker;1.pMD18-T- F1L重組質粒 Recombinant plasmid of pMD18-T- F1L
圖2重組質粒PCR鑒定
Fig.2IdentificationofrecombinantplasmidbyPCR

M. DNA Marke;1.pMD18-T- F1L重組質粒 Recombinant plasmid of pMD18-T- F1L
圖3重組質粒雙酶切鑒定
Fig.3Doubleenzymedigestionofrecombinantplasmid
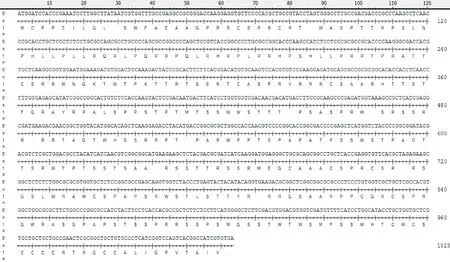
圖4 OrfV-CQ株 F1L基因的核苷酸序列及推導的氨基酸序列Fig.4 Nucleotide sequence and derived amino acid sequences of F1L gene of OrfV-CQ strain
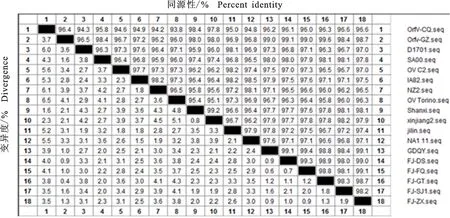
圖5 OrfV-CQ株 F1L基因的核苷酸同源性分析Fig.5 Nuccleotide homology analysis of F1L gene of OrfV-CQ
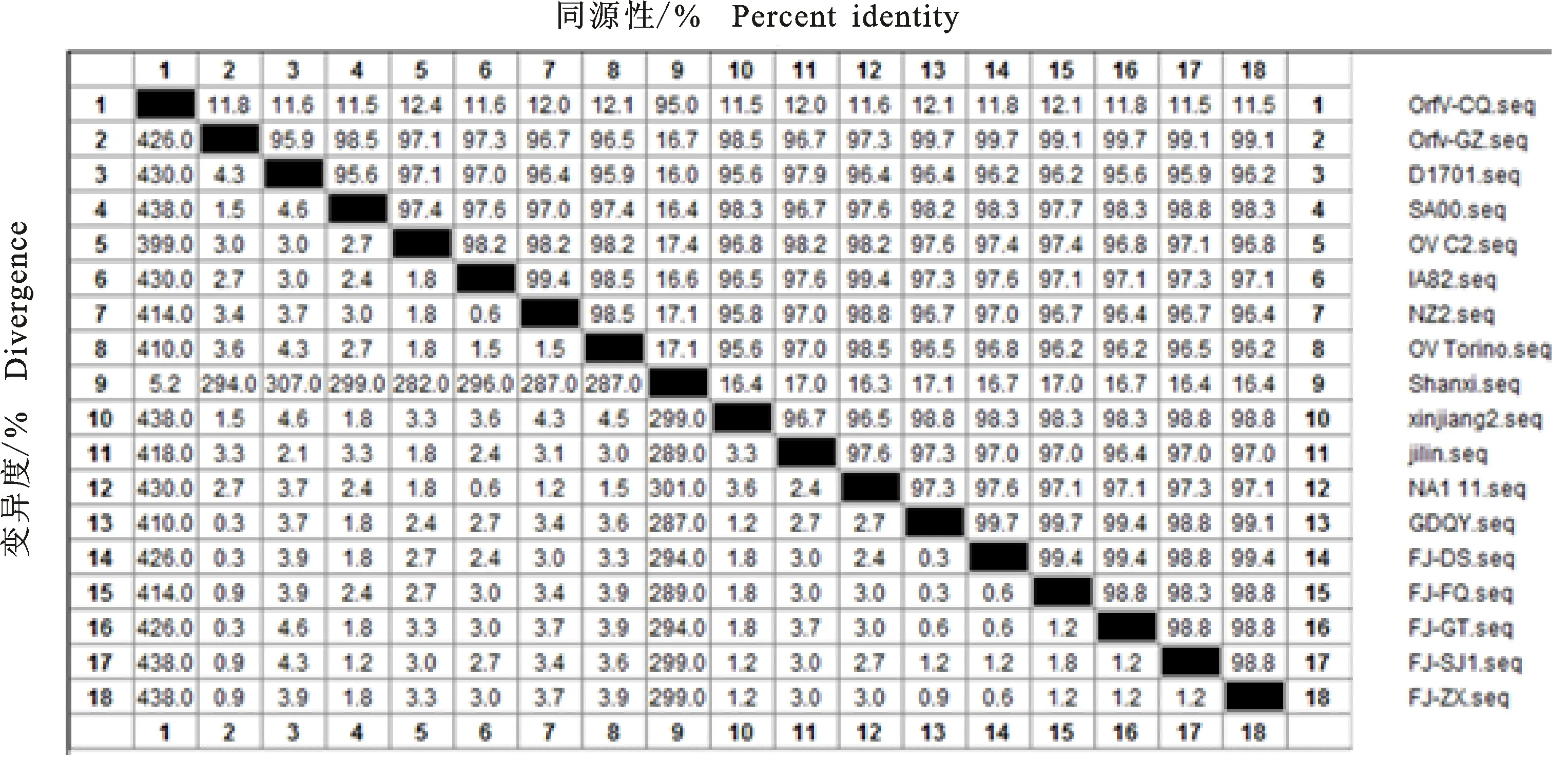
圖6 OrfV-CQ株 F1L基因編碼蛋白質的氨基酸同源性分析Fig.6 Amino acaid homology analysis of F1L gene of OrfV-CQ
2.3OrfV-CQ株F1L基因編碼蛋白的B細胞表位預測
2.3.1 二級結構預測 使用SOPMA在線服務器進行OrfV-CQ株 F1L基因編碼蛋白的二級結構預測(圖8),其二級結構包括α-螺旋(h,11.21%)、β-轉角(t,2.73%)、β-片層(e,10.3%)和無規則卷曲(c,75.76%) 4種類型。
2.3.2 B細胞表位多參數分析 使用DNAStar程序設置高于閾值的氨基酸殘基作為參考區域,對OrfV-CQ株 F1L基因編碼蛋白B細胞表位各參數進行預測(圖9),該基因編碼蛋白在15~320位氨基酸區域具有較好的親水性;在20~305位氨基酸區域具有較好的柔韌性,其柔韌性分布較平均;抗原性及表面可及性較高;310位氨基酸以后,各類B細胞表位參數為負值,故C端的肽段各參數優勢較低。
2.3.3 B細胞抗原表位綜合分析 根據B細胞表位多參數綜合分析,OrfV-CQ株 F1L蛋白可能存在優勢抗原表位的各肽段區域如表1所示。
2.4OrfV-CQ株F1L基因編碼蛋白的T細胞表位預測
2.4.1 CTL表位預測 使用nHLAPred 在線程序,將閾值設置為0.5,分別對HLA-A2、HLA-A*0201、HLA-A*0201、HLA-A*0202 、HLA-A*0203和HLA-A*0205 5種不同分子結合肽進行預測(表2),36~40位的SLNPH、41~44位的LLPL和332~340位的ALIGPVTAI為OrfV-CQ F1L蛋白的CTL表位。

圖7 OrfV-CQ株 F1L基因核苷酸序列的系統進化樹Fig.7 Phylogenetic tree of F1L gene of OrfV-CQ on nucleotide sequence

圖8 OrfV-CQ株F1L蛋白的二級結構預測Fig.8 Secondary structure prediction of OrfV-CQ F1L protein

圖9 OrfV-CQ F1L蛋白B細胞表位參數預測Fig.9 Bcell epitopes prediction for F1L protein of OrfV-GZ by the DNAStar software
2.4.2 Th表位預測 使用ProPred在線程序分別對DRB1-0101、DRB1-0102、DRB1-0301 3種不同Th表位類型進行預測(表3),44~49位的LLRQRL、61~69位的LRRHPSHLL、122~130位的YRPALSPPS和273~281位的WRASGPAPS為OrfV-CQ F1L蛋白的Th表位。

表1 應用不同參數預測OrfV-CQ F1L蛋白抗原表位的肽段位置Table 1 Prediction results of epitopes of OrfV-CQ F1L protein with different parameter
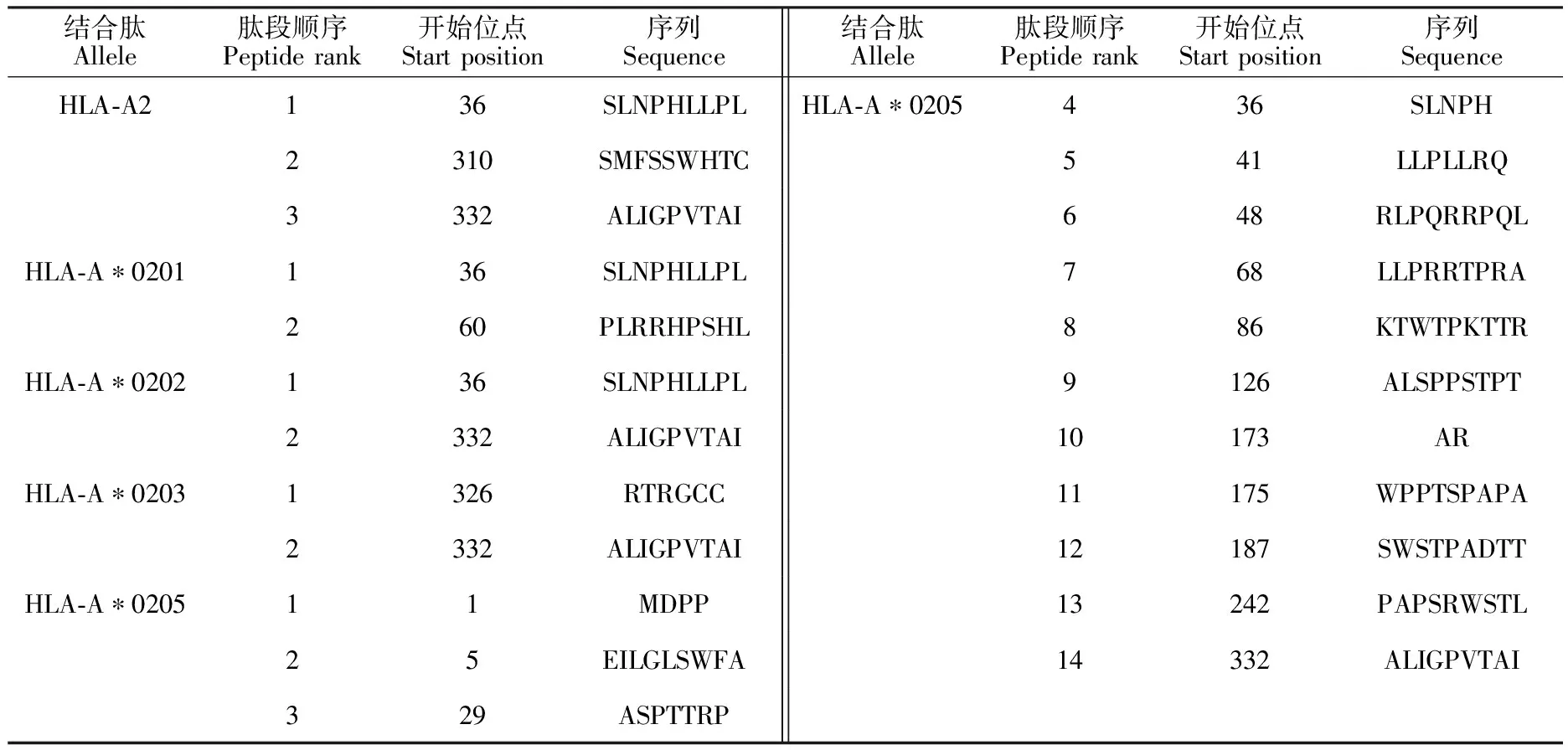
表2 OrfV-CQ F1L CTL表位預測Table 2 Prediction for CTL epitopes of OrfV-CQ F1L
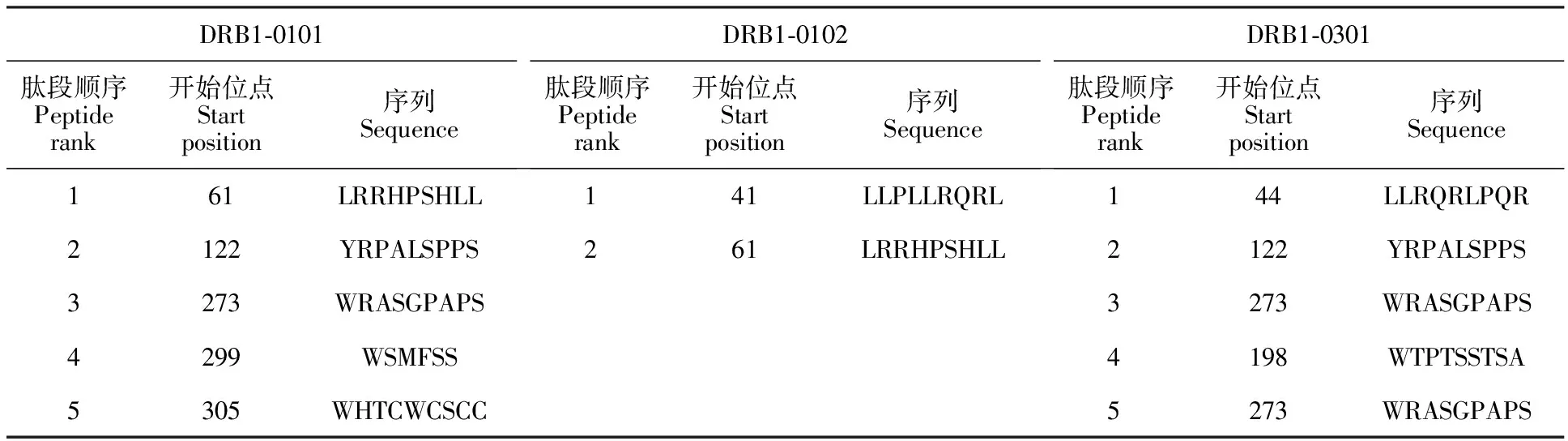
表3 OrfV-CQ F1L Th表位預測Table 3 Prediction for cell epitopes of OrfV-CQ F1L
2.5 OrfV-CQ株 F1L蛋白的保守結構域預測
采用NCBI上的CDART工具分析OrfV-CQ株 F1L蛋白的保守結構域(圖10)。結果顯示該蛋白無保守結構域。
2.6 OrfV-CQ株 F1L蛋白的跨膜結構預測
采用TMHMM 2.0 Server在線軟件預測OrfV-CQ株 F1L蛋白的跨膜結構域(圖11)。結果顯示該蛋白無跨膜結構域。
2.7 OrfV-CQ株 F1L蛋白的信號肽預測
采用SignalP 4.1在線軟件預測OrfV-CQ株 F1L蛋白的信號肽(圖12),結果發現該蛋白無信號肽,即為非分泌蛋白。
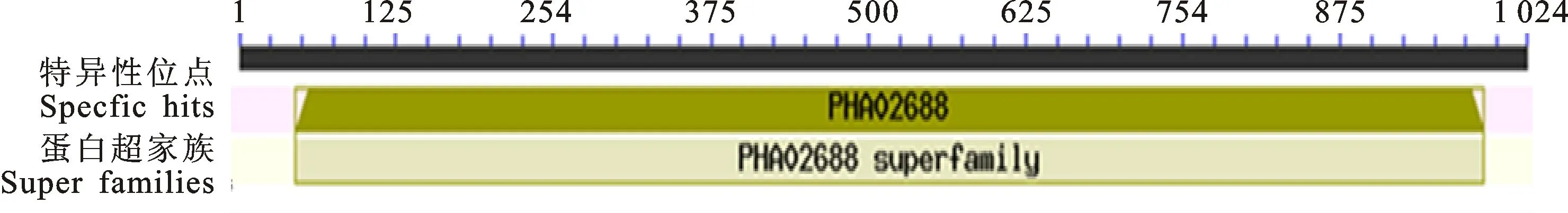
圖10 OrfV-CQ F1L蛋白的保守結構域分析Fig.10 Conserved domains analysis of F1L protein of OrfV-CQ

圖11 OrfV-CQ株 F1L 蛋白的跨膜結構域預測Fig.11 Prediction of transmembrance domains of F1L protein of OrfV-CQ
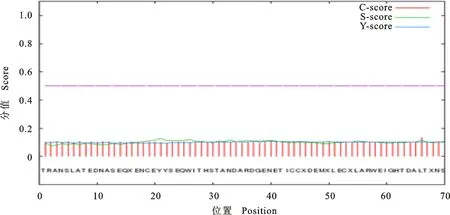
圖12 OrfV-CQ株 F1L蛋白的信號肽預測Fig.12 Signal peptide analysis of F1L protein of OrfV-CQ
3 討 論
3.1 OrfV F1L基因的長度分析
本研究結果表明,OrfV-CQ株 F1L基因序列長1 023 bp,DNAStar推導其編碼340個氨基酸。與GenBank登錄的17株 F1L基因序列比對發現,不同地區、不同宿主的OrfV F1L基因長度存在變異性[6-7]。其中,與重慶毗鄰的OrfV貴州分離株(OrfV-GZ株) F1L基因為1 029 bp[8],OrfV F1L基因存在的長度變異性可能與病毒的適應性相關,但目前未見相關報道。
3.2 OrfV-CQ株 F1L基因同源性分析
本研究發現,不同毒株 F1L基因的同源性較高,但存在不同的堿基改變或缺失。OrfV-CQ株 F1L基因與參考序列的核苷酸同源性為93.8%~98.4%,其他研究人員分析各地的 F1L基因核苷酸序列也高度同源[9-10]。OrfV-CQ株 F1L基因編碼的氨基酸與Shanxi株的同源性為95.0%,但與其他16株OrfV F1L的氨基酸同源性較低,為11.5%~12.4%。編碼氨基酸同源性較低的原因,可能是在進化過程中堿基的缺失或變異,從而導致編碼的蛋白質出現較大變異。
3.3 OrfV-CQ 株 F1L蛋白的生物信息學分析
本研究通過在線程序和生物學軟件對OrfV-CQ 株 F1L基因編碼蛋白的二級結構、信號肽及可能存在的T、B淋巴細胞抗原表位進行預測和分析。結果表明,α-螺旋和β-片層占21.51%,二者化學鍵能較高,存在于蛋白質內部且不易變形,有利于維持病毒囊膜蛋白的穩定性,但難與抗體結合,故不利于抗原表位的形成;β-轉角和無規則卷曲占78.49%,二者的結構常盤旋、扭曲并在蛋白表面展示,對抗體結合和B細胞抗原表位形成有利,因此這些區域常含有B細胞優勢抗原表位[11]。本研究綜合二級結構、親水性、表面可及性、柔韌性和抗原性等參數預測OrfV-CQ株 F1L基因編碼蛋白可能存在的5個B細胞優勢表位,分別可能存在于49~50、90~92、170~190、195~197、288~289。
CTL表位能夠被T細胞抗原受體(TCR)特異性識別,它是可以與MHC-I類分子結合,進而誘導相應的CTL發生免疫應答的一類短肽,一般由8~10個連續的氨基酸殘基構成,活化并決定CTL的特異性殺傷效應[12]。本研究使用nHLAPred在線程序對CTL表位進行預測。結果表明,OrfV-CQ F1L蛋白上有3個CTL表位,分別是36~40位的SLNPH、41~44位的LLPL和332~340位的ALIGPVTAI。這些CTL表位與MHC-I類分子結合形成復合物呈遞至CTL細胞以激活其細胞毒作用。
Th細胞表位是通過識別、加工外源蛋白,從而使外源蛋白與MHC-II類分子結合的一類抗原表位(外源性抗原T細胞表位)。本研究利用ProPred在線程序對Th表位進行預測。結果表明,OrfV-CQ F1L蛋白上有4個Th表位,分別是44~49位的LLRQRL、61~69位的LRRHPSHLL、122~130位的YRPALSPPS和273~281位的WRASGPAPS。這些Th表位與MHC-II類分子結合后,被抗原遞呈細胞轉運至細胞表面與Th細胞受體結合,從而對體液免疫和細胞免疫應答進行協調[13]。
綜合保守結構域、信號肽和跨膜結構域分析發現,OrfV-CQ株 F1L基因編碼蛋白沒有保守結構域、信號肽和跨膜結構域。而吉艷紅等[14]報道預測 F1L基因序列無跨膜區和信號肽;李瑞芳等[15]研究發現OrfV-shz株 F1L基因編碼的氨基酸序列第1~70位存在信號肽,第285~313位存在跨膜區。這些差異的存在可能是由于不同毒株中堿基改變或缺失引起,有待進一步研究。
Reference:
[1] ZHANG K SH,LU ZH X,SHANG Y J,etal.Diagnosis and phylogenetic analysis of Orf virus from goats in China:a case report[J].VirologyJournal,2010,7:78.
[2] NANDI S,DE UK,CHOWDHURY S.Current status of contagious ecthyma or Orf disease in goat and sheep-a global perspective[J].SmallRuminantResearch,2011,96(2/3):73-82.
[3] 趙 魁,賀文琦,高 豐.羊傳染性膿皰皮炎病毒研究進展.中國畜牧醫,2008,35(11):133-137.
ZHAO K,HE W Q,GAO F.The research progress of Orf virus[J].ChinaAnimalHusbandryandVeterinaryMedicine,2008,35(11):133-137(in Chinese).
[4] SCAGLIARINI A,GALLINA L,POZZO F D,etal.Heparin binding activity of Orf virus F1L protein[J].VirusResearch,2004,105(2):107-112.
[5] GALLINA L,SCAGLIARINI A,CIULLI S,etal.Prosperi cloning and expression of the ORF virus F1L gene,possible use as a subunit vaccine[J] .VeterinaryResearchCommunications,2004,28(1):291-293.
[6] 趙 魁.羊傳染性膿皰病毒重組 DNA疫苗的構建與實驗免疫研究[D].長春:吉林大學,2010.
ZHAO K.Study on the construction and experimental immunity of recombinant DNA vaccine against Orf virus[D].Changchun:Jilin University,2010(in Chinese with English abstract).
[7] 尚 辰,黃 勇,張秀娟,等.陜西關中某羊場羊口瘡病毒的分離鑒定與基因序列分析[J].西北農業學報,2014,23(12):25-32.
SHANG CH,HUANG Y,ZHANG X J,etal.Isolation and identification of Orf virus Shaanxi Guanzhong and sequence analysis of B2L, F1L and VIR genes[J].2014,23(12):25-32(in Chinese with English abstract).
[8] 劉 嬡,楊 鈺,鮮思美,等.羊口瘡病毒貴州株 F1L基因克隆及生物信息學分析[J].中國畜牧獸醫,2015,42(1):53-60.
LIU A,YANG Y,XIAN S M,etal.Cloning and bioinformatic analysis of F1L gene of Orf Virus from Guizhou province[J].ChinaAnimalHusbandry&Veterinary,2015,42(1):53-60(in Chinese with English abstract).
[9] ZHANG K,XIAO Y,MENG Y.etal.Phylogenetic analysis of three orf virus strains isolated from different districts in Shandong province,East China[J].JournalofVeterinaryMedicalScience,2015,77(12):1639-1645.
[10] LI W,NING ZH Y,HAO W B,etal.Isolation and phylogenetic analysis of orf virus from the sheep herd outbreak in northeast China[J].BMCVeterinaryResearch,2012,8:229.
[11] SAHA S,RAGHAVE G P.Prediction of ontinuous B-cell epitopes in an antigen using recurrent neural network[J].Proteins,2006,65(1):40-48.
[12] BHASIN M,RAGHAVA G P.Prediction of CTL epitopes using QM,SVM and ANN techniques[J].Vaccine,2004,22(23-24):3195-3204.
[13] AL-ATTIYAH R,MTTSTAFA A S.Computer assisted prediction of HLA-DR binding and experimental analysis for human promiscuous Th1-cell pepfides in the 24kDa secreted lipoprotein(LppX) ofMycobacteriumtuberculosis[J].ScandinavianJournalofImmunology,2004,59(1):16-24.
[14] 吉艷紅,尚佑軍,王光祥,等.羊口瘡病毒 F1L基因的克隆及其B細胞表位預測[J] 中國獸醫科學,2010,40(11):1101-1105.
JI Y H,SHANG Y J,WANG G X,etal.Cloning and expression of F1L gene of Orf virus in Xinjiang[J].ChineseVeterinaryScience,2010,40(11):1101-1105(in Chinese with English abstract).
[15] 李瑞芳,喬 軍,張 輝,等.羊傳染性膿包病毒新疆流行株 F1L基因克隆及表達[J].生物技術,2012,22(3):13-16.
LI R F,QIAO J,ZHANG H,etal.Cloning and expressin of F1L gene of Orf virus in Xinjiang[J].Biotechnology,2012,22(3):13-16(in Chinese with English abstract).
CloningandBioinformaticAnalysisofF1LGeneinChongqingOrfVirus
LI Pengfei1,FENG Jiang1,LIU Ai1,BAO Ximing1,ZHANG Yi1,LI Ting1and XIAN Simei1,2
(1.College of Animal Science,Guizhou University,Guiyang 550025,China;2.Institute of Animal Disease,Guizhou Province,Guiyang 550025,China)
In order to analyze the F1L gene of Orf virus in Chongqing (OrfV-CQ) and predict the function of its derived protein. The F1L gene was amplified,cloned and sequenced by PCR,and the secondary structure,B-cell preponderant epitope,T-cell preponderant epitope,conserved domains analysis,transmembrance domain and signal peptide of its derived protein were predicted by the bioinformatics softwares. The results indicated that the F1L gene of OrfV-CQ was at the length of 1 023 bp,which encoded 340 amino acids and shared the 98.4% and 95.0% at the consistency of nucleotide sequence and amino acid sequence with Shanxi strain,respectively. The proportion of the alpha helix in the protein encoded by F1L gene of OrfV-CQ was 11.21%,β-turn was 2.73%,andom coils was 75.76% andβ-sheet was 10.3%.The protein may exist 5 B-cell epitopes,3 CTL epitopes and 4 Th epitopes,no transmembrance domains and signal pepide were found.
Orf virus(OrfV); F1L gene;Clone;Bioinformatics analysis
2016-09-07
2016-11-02
Science and Technology Department of Guizhou Privince [No.LH(2014)7667];Science and Technology Innovation Talents Team of Animal Disease Prevention and Control and Veterinary Public Health Security of Guizhou Privince[No. (2015)4016].
LI Pengfei,male,master student. Research area:preventive veterinary medicine.E-mail:lpf0405@yeah.net
S855.3
A
1004-1389(2017)09-1281-08
(責任編輯:顧玉蘭Responsibleeditor:GUYulan)
日期:2017-09-12
網絡出版地址:http://kns.cnki.net/kcms/detail/61.1220.S.20170912.1740.006.html
2016-09-07
2016-11-02
貴州省科學技術基金[LH(2014)7667];貴州省動物疫病防控與獸醫公共衛生保障科技創新人才團隊[(2015) 4016]。
李鵬飛,男,碩士研究生,研究方向為預防獸醫學。E-mail:lpf0405@yeah.net
鮮思美,女,副教授,博士,研究方向為預防獸醫學。E-mail:xiansimei@163.com
CorrespondingauthorXIAN Simei,female,associate professor,Ph.D.Research area:preventive veterinary medicine.E-mail:xiansimei@163.com

