基于高通量測(cè)序技術(shù)對(duì)三種太歲樣品細(xì)菌組成的分析
王朝江++王世清


摘要:利用Illumina公司MiSeq技術(shù),檢測(cè)3種太歲樣品細(xì)菌群落的差異性,分析太歲樣品的細(xì)菌多樣性和群落結(jié)構(gòu),揭示細(xì)菌在太歲構(gòu)成中的作用和在不同太歲間的內(nèi)在聯(lián)系。結(jié)果表明,3種太歲樣品的高通量測(cè)序分析共得到有效序列64 114條,歸為4 033個(gè)OTU,屬于35個(gè)門、61個(gè)綱、89個(gè)目、201個(gè)科及625個(gè)屬,還有一些未有明確的分類學(xué)信息。變形菌門和厚壁菌門為優(yōu)勢(shì)門,在2、8、9號(hào)太歲樣品中所占比例分別是63.95%、79.65%、76.82%。2、8、9號(hào)太歲中的最優(yōu)勢(shì)屬及所占比例分別為Edaphobacter (23.84%)、梭菌屬(12.94%)、芽孢桿菌屬(17.60%)。3種太歲樣品各自的優(yōu)勢(shì)屬不盡相同,沒有共有且占比均大的屬。3種太歲樣品的細(xì)菌群落的多樣性豐富,且三者的細(xì)菌組成差異較大,不能建立起明顯的內(nèi)在聯(lián)系。
關(guān)鍵詞:太歲;細(xì)菌多樣性;高通量測(cè)序
中圖分類號(hào):Q93 文獻(xiàn)標(biāo)識(shí)碼:A 文章編號(hào):0439-8114(2017)13-2543-05
DOI:10.14088/j.cnki.issn0439-8114.2017.13.037
Analysis of Bacterial Diversity and Community Structure from Three Taisui Based on High-throughput Sequencing
WANG Chao-jiang1,WANG Shi-qing2
(1. Institute of Genetics and Physiology of Hebei Academy of Agriculture and Forestry Science, Shijiazhuang 050051, China;
2. Shijiazhuang No.44 Middle School, Shijiazhuang 050051, China)
Abstract: By Miseq technology of Illumina company, the bacterial community structure from three samples were studied. The bacterial diversity and community structure were analysised in order to identify the role of bacteria in composition of Taisui and internal relations between different Taisui. 64 114 non-redundant sequences were obtained which were classified into 4 033 OTUs. These OTUs were belonged to 35 phyla, 61 classes, 89 orders, 201 families, 625 genera, and some unclassification groups. In the sample numbered 2, 8, 9, the dominant phyla were Proteobacteria and Firmicutes, respectively the proportions of two phyla were 63.95%, 79.65%, 76.82%. The most dominant genus in the sample numbered 2, 8, 9 was Edaphobacter (23.84%), Clostridium (12.94%) and Bacillus (17.60%) respectively. The dominant genera were different from each other and there was no common preponderant genus. High bacterial diversities were found in all samples, also great differences existed between them, and there were no clear internal relations to be established between different samples.
Key words: Taisui;bacterial diversity;high-throughput sequencing
“肉芝狀如肉,乃生物也,白者如截肪,黃者如紫金,皆光明洞徹如堅(jiān)冰也”[1]。根據(jù)《本草綱目》中對(duì)太歲的描述,民間把1992年在中國陜西周至渭河灘發(fā)現(xiàn)的第一個(gè)肉團(tuán)狀不明物體及后來在河北、天津、山西、北京、吉林、新疆、內(nèi)蒙古、甘肅等地發(fā)現(xiàn)的多個(gè)不明物體稱之為太歲。目前,太歲在生物界的地位尚沒有科學(xué)定論,僅少數(shù)學(xué)者在不同方面進(jìn)行了初探。鄭科研等[2]和朱春玉等[3]在其化學(xué)構(gòu)成及生物學(xué)組分方面進(jìn)行了研究。在其微生物構(gòu)成方面,戴璐[4]利用培養(yǎng)方法得到了2種黏菌,并用平板培養(yǎng)結(jié)合克隆文庫及T-RFLP得知渭河太歲衍生體是由18種真菌組成;王欣[5]分離培養(yǎng)了同一樣本中的細(xì)菌獲得35株細(xì)菌和1株黏細(xì)菌,并用T-RFLP發(fā)現(xiàn)其中還有藍(lán)細(xì)菌。林澗等[6]用平板培養(yǎng)了2種太歲得到了假絲酵母菌、黏質(zhì)紅酵母和根癌農(nóng)桿菌,2種太歲克隆文庫各得到3個(gè)屬的細(xì)菌。本研究筆者曾針對(duì)1種太歲構(gòu)建了完整的16S rDNA克隆文庫,發(fā)現(xiàn)太歲樣品中細(xì)菌多樣性較豐富,且蘊(yùn)藏著許多未知的微生物資源。
高通量測(cè)序技術(shù)(High-throughput sequencing)具有通量高、速度快、準(zhǔn)確度高以及運(yùn)行成本低等特點(diǎn),能夠彌補(bǔ)克隆文庫方法的工作量量大、信息量少等不足,已被廣泛應(yīng)用于生命科學(xué)的各個(gè)領(lǐng)域,尤其是可以系統(tǒng)地研究不同個(gè)體或環(huán)境中微生物菌群結(jié)構(gòu)和多樣性,對(duì)于全面、深入地認(rèn)識(shí)了解個(gè)體、環(huán)境及其與微生物的關(guān)系有明顯的推動(dòng)作用。近年來,利用高通量測(cè)序技術(shù)對(duì)包括人體[7]、動(dòng)物[8]、植物、昆蟲[9]、海洋生物、土壤[10]、活性污泥[11]等相關(guān)的微生物群落多樣性研究都已開展,而對(duì)太歲的研究結(jié)果鮮見報(bào)道。本研究利用Illumina公司MiSeq技術(shù),對(duì)3種不同產(chǎn)地和形態(tài)的太歲深層組織內(nèi)細(xì)菌群落多樣性異同進(jìn)行分析,為揭示太歲的微生物構(gòu)成及不同太歲的內(nèi)在聯(lián)系提供數(shù)據(jù)支持。
1 材料與方法
1.1 材料
3株太歲分別產(chǎn)自山東青島、新疆伊犁、河南三門峽,編號(hào)為2、8、9。2號(hào)太歲采集于河道淤泥上,重350 kg,長(zhǎng)橢圓體,極富彈性,內(nèi)部組織如熔融玻璃,溶于水;8號(hào)太歲采集于牧場(chǎng)表土層,重15 kg,扁枕頭狀,質(zhì)如豬肥肉膘;9號(hào)太歲采集于黃河岸邊,重20 kg,具尖圓柱體,質(zhì)如硬橡皮,表皮和內(nèi)部組織褐色。為避免保藏階段外源菌感染干擾,提取DNA的樣品取樣位置在表層下2~3 cm處,重0.2 g。
1.2 DNA提取和擴(kuò)增
使用OMEGA公司E.Z.N.A Soil DNA試劑盒提取樣品的總基因組DNA。利用瓊脂糖凝膠檢測(cè)DNA完整性。采用引物341F(5′-CCTACGGGNGG- CWGCAG -3′)和805R(5′-GACTACHVGGGTATCTAATCC-3′)擴(kuò)增細(xì)菌16S rDNA的V3-4區(qū)片段[12]。PCR反應(yīng)體系50 μL:5 μL 10×PCR buffer,0.5 μL dNTP(10 mmol/L),10 ng基因組DNA,0.5 μL引物F(50 μmol/L),0.5 μL引物R(50 μmol/L),0.5 μL Plantium Taq(5 U/μL)。PCR反應(yīng)程序如下:94 ℃ 3 min,94 ℃ 30 s,45 ℃ 20 s,65 ℃ 30 s,5個(gè)循環(huán),94 ℃ 20 s,55 ℃ 20 s,72 ℃ 30 s,20個(gè)循環(huán),72 ℃延伸5 min。第二輪擴(kuò)增,引入Illumina橋式PCR兼容引物。每個(gè)樣品做3個(gè)重復(fù),PCR產(chǎn)物切膠回收純化并用Qubit 2.0檢測(cè)DNA濃度,同一樣品PCR產(chǎn)物等量混合后測(cè)序。
1.3 測(cè)序及數(shù)據(jù)處理
使用Miseq平臺(tái)測(cè)序,測(cè)序序列中含有barcode序列,以及測(cè)序時(shí)加入的引物和接頭序列。首先需要去除引物接頭序列,再根據(jù)PE reads之間的overlap關(guān)系,將成對(duì)的reads拼接成一條序列,然后按照barcode標(biāo)簽序列識(shí)別并區(qū)分樣品得到各樣本數(shù)據(jù),最后對(duì)各樣本數(shù)據(jù)的質(zhì)量進(jìn)行質(zhì)控過濾,得到各樣本有效數(shù)據(jù)。
1.4 數(shù)據(jù)分析
1.4.1 測(cè)序數(shù)據(jù)優(yōu)化 使用Usearch去除預(yù)處理后序列中非擴(kuò)增區(qū)域序列,而后對(duì)序列進(jìn)行測(cè)序錯(cuò)誤校正,并調(diào)用Uchime進(jìn)行鑒定嵌合體。隨后,將去除嵌合體的序列與數(shù)據(jù)庫代表性序列進(jìn)行Blastn比對(duì),低于閾值的比對(duì)結(jié)果被認(rèn)為是靶區(qū)域外序列,并進(jìn)行剔除。
1.4.2 OTU聚類與分類學(xué)分析 將所有樣本序列按照序列間的距離進(jìn)行聚類,根據(jù)序列之間的相似性將序列分成不同的操作分類單元(OTU,Operational Taxonomic Units)。采用RDP classifier貝葉斯算法對(duì)97%相似度水平的OTU代表序列進(jìn)行分類學(xué)分析,得到每個(gè)OTU對(duì)應(yīng)的物種分類信息,并在界、門、綱、目、科、屬水平統(tǒng)計(jì)各個(gè)樣品的菌落組成。
1.4.3 細(xì)菌的多樣性分析 通過對(duì)單個(gè)樣品中物種多樣性的分析(α多樣性)可以反映細(xì)菌群落的豐度和多樣性,包括計(jì)算群落分布豐度(Community richness)的指數(shù)Chao值(http://www.mothur.org/wiki/Chao)和ACE值(http://www.mothur.org/wiki/Ace),計(jì)算群落分布多樣性(Community diversity)的Shannon指數(shù)(http://www.mothur.org/wiki/Shannon)及Simpson指數(shù)(http://www.mothur.org/wiki/Simpson)。
2 結(jié)果與分析
2.1 樣品測(cè)序深度驗(yàn)證
稀釋曲線(Rarefaction curve)反映了樣品的取樣深度,可以用來評(píng)價(jià)測(cè)序量是否足以覆蓋所有類群。用樣品豐富度和Shannon指數(shù)做稀釋曲線(圖1、圖2)。由圖1、圖2可知,3個(gè)樣品豐富度稀釋曲線均基本趨于平緩,但仍未達(dá)到飽和,而Shannon指數(shù)的稀釋曲線已經(jīng)進(jìn)入平臺(tái)期,這說明本次高通量測(cè)序達(dá)到了測(cè)序要求,能夠較真實(shí)地反映樣本的細(xì)菌群落,不過仍有少量微生物種類尚未被發(fā)現(xiàn)。
Coverage(http://www.mothur.org/wiki/Coverage)表示各樣品文庫的覆蓋率,其數(shù)值越高,則樣本中序列沒有被測(cè)出的概率越低。該指數(shù)實(shí)際反映了本次測(cè)序結(jié)果是否代表樣本的真實(shí)情況。計(jì)算公式如下:
C=1-■
式中,n1為只含有一條序列的OTU數(shù)目,N為所有個(gè)體數(shù)目,此處為序列總數(shù)。2號(hào)、8號(hào)、9號(hào)太歲樣品的覆蓋度分別為0.940 5、0.969 0、0.947 3,太歲樣品的覆蓋度均高于0.940 0,說明本次測(cè)序結(jié)果均能較好地反映樣本的真實(shí)情況。
2.2 細(xì)菌的多樣性
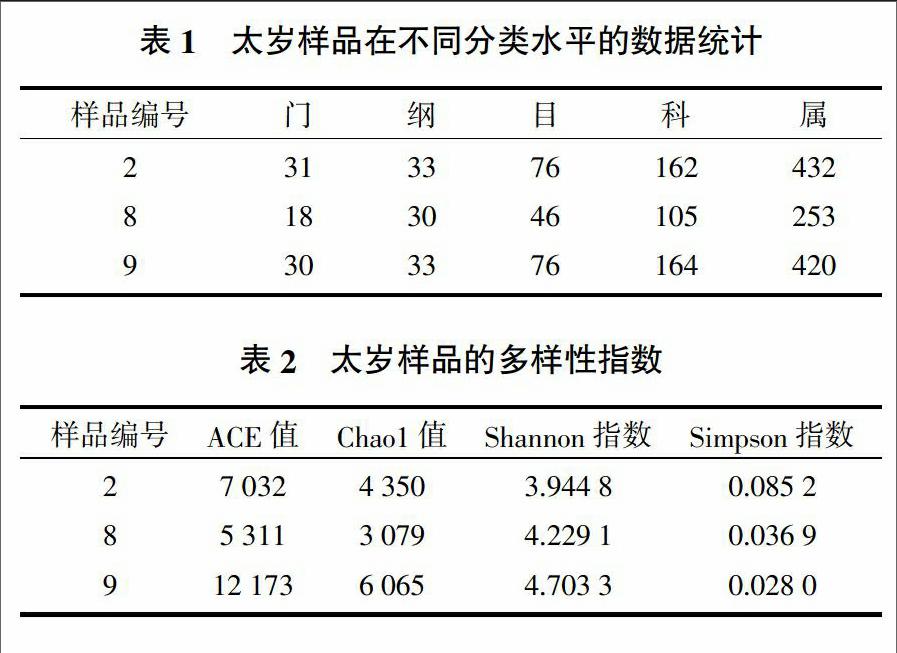
對(duì)3個(gè)樣品的高通量測(cè)序,共得到有效序列 64 114條,共歸類為4 033個(gè)OTU,它們歸屬于35個(gè)門、61個(gè)綱、89個(gè)目、201個(gè)科及625個(gè)屬,還有一些未有明確的分類學(xué)信息。其中2號(hào)樣品得到有效序列20 883條,歸類為1 856個(gè)OTU;8號(hào)樣品得到有效序列16 672條,歸類為788個(gè)OTU;8號(hào)樣品得到有效序列26 606條,歸類為2 040個(gè)OTU。如表1所示,每個(gè)樣品在不同分類水平上的種類數(shù)量都很豐富,說明3個(gè)樣品細(xì)菌都有豐富的多樣性。表2顯示每個(gè)樣品的群落豐度都很高,并且根據(jù)Shannon指數(shù)和Simpson指數(shù)可以看出它們有豐富的細(xì)菌群落多樣性。
2.3 細(xì)菌群落的組成分析
如圖3顯示,2號(hào)樣品細(xì)菌群落的優(yōu)勢(shì)門是厚壁菌門(Firmicutes,34.00%)、變形菌門(Proteobacteria,29.95%)、酸桿菌門(Acidobacteria,27.84%)、擬桿菌門(Bacteroidetes,3.24%)。8號(hào)樣品細(xì)菌群落的優(yōu)勢(shì)門是變形菌門(44.47%)、厚壁菌門(35.18%)、擬桿菌門(12.72%)、浮霉菌門(Planctomycetes,3.39%)。9號(hào)樣品細(xì)菌群落的優(yōu)勢(shì)門是變形菌門(46.48%)、厚壁菌門(30.34%)、擬桿菌門(18.93%)、放線菌門(Actinobacteria,1.78%)。其中變形菌門和厚壁菌門在3個(gè)樣品中都占主要優(yōu)勢(shì),這2個(gè)門在2號(hào)、8號(hào)和9號(hào)3個(gè)樣品中所占比例分別是63.95%、79.65%和76.82%。
表3是各個(gè)太歲樣品中豐度大于1%的目及其所占比例,2號(hào)樣品豐度大于1%的目有15個(gè),8號(hào)樣品豐度大于1%的目有14個(gè),9號(hào)樣品豐度大于1%的目有13個(gè)。3個(gè)樣品共有的豐度大于1%的目有梭菌目(Clostridiales)、擬桿菌目(Bacteroidales)、芽孢桿菌目(Bacillales)、假單胞菌目(Pseudomonadales)、伯克霍爾德氏菌目(Burkholderiales)、鞘鞍醇單胞菌目(Sphingomonadales)、根瘤菌目(Rhizobiales)、腸桿菌目(Enterobacteriales)、乳桿菌目(Lactobacillales)。2號(hào)樣品的優(yōu)勢(shì)目是芽孢桿菌目(25.86%)和假單胞菌目(11.66%),8號(hào)樣品的優(yōu)勢(shì)目是梭菌目(26.38%)和紅螺菌目(Rhodospirillales,15.39%),9號(hào)樣品的優(yōu)勢(shì)目是芽孢桿菌目(20.49%)和擬桿菌目(14.48%)。
圖4是屬水平上的每個(gè)太歲樣品的細(xì)菌群落組成。由圖4可知,3個(gè)樣品的細(xì)菌群落構(gòu)成有很大差異,其在屬的水平上未有明確分類信息的類群所占比例也不同。8號(hào)樣品中未有明確分類信息的占13.81%,2號(hào)和9號(hào)樣品中未有明確分類信息的分別占2.59%、2.88%。
3個(gè)太歲樣品優(yōu)勢(shì)屬也不相同,2號(hào)樣品的優(yōu)勢(shì)屬是Edaphobacter(23.84%),芽孢桿菌屬(Bacillus,22.04%),假單胞菌屬(Pseudomonas,8.07%);8號(hào)樣品優(yōu)勢(shì)屬為梭菌屬(Clostridium,12.94%),固氮螺菌屬(Azospirillum,9.44%),假單胞菌屬(5.73%);9號(hào)樣品的優(yōu)勢(shì)屬是芽孢桿菌屬(17.60%),假單胞菌屬(7.64%),生絲單胞菌屬(Hyphomonas,7.38%)。而且2號(hào)、8號(hào)和9號(hào)樣品中豐度各在前3位的屬所占比例分別是53.95%、28.11%和32.62%。在3個(gè)太歲樣品中共有的屬(豐度>1%)只有3個(gè):芽孢桿菌屬、假單胞菌屬和鞘鞍醇單胞菌屬(Sphingomonas)。芽孢桿菌屬在2號(hào)、8號(hào)和9號(hào)樣品中所占比列分別是22.04%、4.31%、17.60%,其豐度排位是2、4、1;假單胞菌屬在2號(hào)、8號(hào)和9號(hào)樣品中所占比列分別是8.07%、5.73%、7.64%,其豐度排位是3、5、2;鞘鞍醇單胞菌屬在2號(hào)、8號(hào)和9號(hào)樣品中所占比列分別是1.34%、2.11%、5.60%,其豐度排位是12、12、5。這表明沒有一種細(xì)菌在各自太歲樣品中占絕對(duì)優(yōu)勢(shì)(豐度>60%),且3個(gè)太歲樣品在細(xì)菌屬的組成上有很大差異,缺乏占比例高的共有類群。
從3個(gè)樣品的韋恩圖(圖5)可以看出,它們的并集是143個(gè)OTU,2號(hào)和8號(hào)樣品交集是173個(gè)OTU,2號(hào)和9號(hào)樣品的交集是393個(gè)OTU,8號(hào)和9號(hào)樣品的交集是239個(gè)OTU。各個(gè)樣品的交集較少,說明這3個(gè)太歲樣品相同OTU較少,但是相對(duì)來說2號(hào)和9號(hào)交集較多。
3 討論
本研究采用高通量測(cè)序技術(shù)分析了3種太歲樣品的細(xì)菌群落組成及多樣性。3個(gè)太歲樣品的4 033個(gè)OTU歸屬于35個(gè)門、61個(gè)綱、89個(gè)目、201個(gè)科及625個(gè)屬,還有一些未有明確的分類學(xué)信息。Shannon指數(shù)和Simpson指數(shù)表明3個(gè)太歲樣品都有豐富的細(xì)菌多樣性。本研究3個(gè)太歲樣品的Shannon指數(shù)分別是3.944 8、4.229 1、4.703 3,較長(zhǎng)春太歲的Shannon指數(shù)2.22大[7],說明3個(gè)樣品的細(xì)菌多樣性更豐富。
在細(xì)菌的群落結(jié)構(gòu)上,3個(gè)太歲樣品的優(yōu)勢(shì)門是變形菌門和厚壁菌門,所占比例大于60%,與長(zhǎng)春太歲結(jié)果相一致。而3種太歲的優(yōu)勢(shì)屬有很大差異,2號(hào)樣品的優(yōu)勢(shì)屬是Edaphobacter、芽孢桿菌屬、假單胞菌屬;8號(hào)樣品優(yōu)勢(shì)屬為梭菌屬、固氮螺菌屬、假單胞菌屬;9號(hào)樣品的優(yōu)勢(shì)屬是芽孢桿菌屬、假單胞菌屬、生絲單胞菌屬。用16S rDNA克隆文庫方法得到的長(zhǎng)春太歲的優(yōu)勢(shì)屬為短波單胞菌屬、葡萄球菌屬和食酸菌屬,與本研究3種太歲優(yōu)勢(shì)屬也不相同,這說明不同太歲樣品的細(xì)菌的優(yōu)勢(shì)屬并不相同,這可能與太歲生長(zhǎng)的環(huán)境有關(guān)。另外,3個(gè)太歲各自的優(yōu)勢(shì)屬中沒有1種占絕對(duì)優(yōu)勢(shì)(豐度大于60%),排名前三位屬的豐度相加,2號(hào)接近60%,8號(hào)9號(hào)僅30%;共有的3個(gè)屬(豐度大于1%),豐度沒有同時(shí)位列前三,共有且豐度最靠前的芽孢桿菌屬在8號(hào)豐度僅排第4位,不存在共有且占比均大的屬;OTU并集少,說明3種太歲樣品的細(xì)菌群落結(jié)構(gòu)差異較大。所以,可以大膽推測(cè),細(xì)菌尤其是本研究所用引物測(cè)得的細(xì)菌,不是太歲本體的根本組成,而是其同生境共存微生物。不同產(chǎn)地形態(tài)太歲之間,不大可能依靠細(xì)菌建立起內(nèi)在聯(lián)系。因此,為揭示太歲生命本質(zhì),需要更改引物或探討除細(xì)菌之外的其他微生物組成。
參考文獻(xiàn):
[1] 李時(shí)珍.本草綱目[M].上海:商務(wù)印書館,1954.
[2] 鄭科研,李文英.不明物體“太歲”的初步研究[J].西北大學(xué)學(xué)報(bào)(自然科學(xué)版),2010,40(6):1012-1016.
[3] 朱春玉,白婷婷,姜秋實(shí),等.“太歲”生物學(xué)組分的研究[J].微生物學(xué)雜志,2011,31(1):1-5.
[4] 戴 璐.“大型黏菌復(fù)合體”黏菌和真菌多樣性的初步研究[D]. 西安:西北大學(xué),2007.
[5] 王 欣.“大型粘菌復(fù)合體”細(xì)菌多樣性及抑菌功能初步研究[D].西安:西北大學(xué),2007.
[6] 林 澗,熊向華,葛 欣,等.2種太歲樣品中微生物的分離和鑒定[J].生物技術(shù)通訊,2013(6):825-827.
[7] COSTELLO E K,LAUBER C L,HAMADY M,et al. Bacterial community variation in human body habitats across space and time[J]. Science,2009,326(5960):1694-1697.
[8] CAIDOS L,RUSCH A,ILARDO M. Ribosomal tag pyrosequeuciug of DNA and RNA from benthic coral reef microbiota: Community spatial structure,rare members and nitrogen-cycling guilds[J]. Enviroumental Microbiology,2011,13(5):1138-1152.
[9] YI C,TIAN B,JI X,et al. Associated bacteria of different life stages of Meloidogyne incognita, using pyrosequencing-based analysis[J]. Journal of Basic Microbiology,2015,55(8):950-960.
[10] ACOSTA-MARTINEZ V,DOWD S,SUN Y,et al. Tag encoded pyrosequencing analysis of bacterial in a single soil type as affected by management and land use[J]. Soil Biology and Biochemistry,2008,40(11):2762-2770.
[11] SANAPAREDDY N,HAMP T J,GONZALEZ L C,et al. Molecular diversity of a North Carolina waste water treatment plant as revealed by pyrosequencing[J]. Applied and Environmental Microbiology,2009,75(6):1688-1696.
[12] ZAKRZEWSKI M,GOESMANN A,JAENICKE S,et al. Profiling of the metabolically active community from a production-scale biogas plant by means of high-throughput metatranscriptome sequencing[J]. Journal of Biotechnology,2012,158(4): 248-258.

