脹果甘草查爾酮異構酶基因的克隆及序列分析
尹彥超,張曉冬,馬永生,周姍,高智強,胡婷,劉穎
北京中醫藥大學 中藥學院,北京 100102
脹果甘草查爾酮異構酶基因的克隆及序列分析
尹彥超,張曉冬,馬永生,周姍,高智強,胡婷,劉穎
北京中醫藥大學 中藥學院,北京 100102
目的:對脹果甘草甘草苷生物合成途徑的關鍵酶查爾酮異構酶(CHI)進行基因克隆及序列分析。方法:根據烏拉爾甘草CHI基因cDNA序列設計引物,以脹果甘草主根總RNA為模板RT-PCR擴增CHI基因cDNA序列,并對其進行分析。結果:克隆得到20條脹果甘草CHI基因cDNA序列,開放讀框全長690 bp,編碼229個氨基酸殘基。20條脹果甘草CHI基因的cDNA序列存在22個變異位點,一致性為99.54%,可分為6種單倍型;其編碼的氨基酸序列存在14個變異位點,一致性為98.98%。脹果甘草CHI基因編碼蛋白為穩定性親水蛋白,相對分子質量為24.5×103,等電點為5.3~6.2,不含信號肽,無跨膜區,二級結構以α螺旋和無規則卷曲為主,保守結構域包含一個查耳酮超家族結構域。同源性分析顯示,不同物種間的CHI基因同源性較低。結論:克隆了脹果甘草CHI基因cDNA序列,為進一步研究不同基原甘草中甘草苷生物合成的分子調控機制奠定了基礎,并為優質甘草的分子選育提供了理論依據。
脹果甘草;查爾酮異構酶;生物信息學分析
[Key words]Glycyrrhiza inflataBat.;chalcone isomerase;bioinformatic analysis
甘草是我國最常用的大宗藥材之一,具有補脾益氣、抗炎解毒、鎮咳祛痰、調和諸藥等作用[1]。《中國藥典》規定的甘草基原是豆科植物烏拉爾甘草(Glycyrrhiza uralensisFisch.)、脹果甘草(G.inflataBat.)或光果甘草(G.glabraL.)的干燥根和根莖[1]。烏拉爾甘草、光果甘草、脹果甘草雖然同為藥典采用的甘草基原植物,但其化學成分和藥理活性卻相差較大[2-3]。目前關于甘草的研究多集中于烏拉爾甘草,涉及功能基因克隆[4]、基因多態性研究[5]、化學成分分析[6]等方面,而關于脹果甘草的研究報道則相對較少。
甘草的活性成分主要為甘草酸和甘草苷,《中國藥典》規定本品按干燥品計算,含甘草苷(C21H22O9)不得少于0.50%[1]。甘草苷屬于黃酮類化合物,其生物合成途徑受眾多酶的調控,其中查耳酮異構酶(chalcone isomerase,CHI)是關鍵酶之一,催化分子內的環化反應[7],對甘草苷的合成具有至關重要的作用。CHI分為2種類型,Ⅰ型CHI只能以查耳酮為底物,催化柚皮素查爾酮合成柚皮素;Ⅱ型CHI既可以查耳酮為底物,也可以6'-脫氧查耳酮為底物[8],催化查爾酮類的異甘草素合成甘草素,甘草素與α-D-吡喃葡萄糖合成甘草苷(圖1)。因此,對CHI開展相關研究,可以在一定程度上揭示甘草苷生物合成的分子機制,從而有望提高栽培甘草中甘草苷的積累水平。
鑒于CHI在黃酮代謝途徑中的關鍵地位,目前已從金銀花[9]、水母雪蓮[10]、芍藥[11]等植物中克隆得到其cDNA序列。就甘草而言,3種基原甘草中烏拉爾甘草CHI基因cDNA序列已有報道[12],而脹果甘草中該基因尚未完成克隆。因此,我們采用RT-PCR技術,以脹果甘草主根總RNA為模板克隆CHI基因,并進行序列比對及生物信息學分析,為進一步研究功能基因多態性對甘草苷積累的影響及優質甘草的分子育種奠定基礎。
1 材料和方法
1.1 材料
植物材料取自栽培兩年生的新鮮脹果甘草主根,液氮速凍,-80℃保存備用。大腸桿菌DH5α感受態細胞、pMD-19T載體、ESPY spin植物RNA快速提取試劑盒、瓊脂糖凝膠純化回收試劑盒、M-MLV(Rnase H-)購于北京博邁德生物技術有限公司;LA-TaqDNA聚合酶、Oligo(dT)購于TaKaRa公司;引物合成由上海生工生物工程公司(Sangon)完成。微量移液器(Eppendorf);1-13000型離心機(Sigma);DYY-6C型電泳儀(北京六一儀器廠);BG-gdsAUTO510型凝膠成像系統(上海培清科技有限公司);酶標儀(Biotek Epoch)。
1.2 脹果甘草主根總RNA的提取及RT-PCR
取脹果甘草根于液氮中研磨至粉末,用RNA提取試劑盒提取總RNA,1.0%瓊脂糖凝膠電泳檢查RNA的完整性,用酶標儀測定所得RNA的純度和濃度。以脹果甘草總RNA為模板,Oligo(dT)為引物,用M-MLV反轉錄試劑盒反轉錄成cDNA。
1.3 脹果甘草CHI基因擴增
根據文獻[12]報道的烏拉爾甘草CHI序列,用Primer Premier 5.0設計上游引物GF(5'-ATGGC GGGAGCAGCACCAGTA-3')和下游引物GR(5'-T CAGTTTCCGTTTCCAAT-3'),以上述反轉錄合成的cDNA為模板進行PCR擴增。50μL反應體系包括模板cDNA 2.0μL、10×緩沖液5.0μL、dNTP(2.5 mmol/L)5.0μL、引物GF和GR(10μmol/L)各2.0μL、LA-TaqDNA聚合酶1.0μL、無RNase的水33.0μL。反應程序:95℃ 2 min;95℃ 20 s,58℃ 30 s,72℃ 45 s,35個循環;72℃ 5 min。于4℃保存。
1.4 PCR產物膠回收、克隆及序列測定
經1.0%瓊脂糖凝膠電泳檢測后,切下目的片段,膠回收純化后與pMD-19T于16℃連接12 h,將連接產物轉入大腸桿菌DH5α感受態細胞,涂于含50 mg/L氨芐西林(Amp)的LB固體培養基上,37℃倒置培養12~16 h,隨機挑選50個單菌落接種于含50 mg/L Amp的LB液體培養基中,于37℃、200 r/min條件下培養 12~16 h,行菌液PCR,1.0%瓊脂糖凝膠電泳檢測PCR產物,驗證為陽性克隆的用于測序。測序由上海生工生物工程有限公司完成。
1.5 生物信息學分析
將得到的序列在NCBI網站進行BLAST比對。用Editseq將所得序列翻譯成氨基酸序列,通過 在 線 工 具 SignaIP、Prot Scale、Prot Param、SW ISS-Model和SOPMA等分析其二級結構、三級結構、信號肽、跨膜結構域、保守結構域及基本理化性質,并通過NCBI在線數據庫查找已注冊的CHI序列,用軟件MEGA5.1構建系統進化樹。
2 結果
2.1 脹果甘草CHI基因序列分析
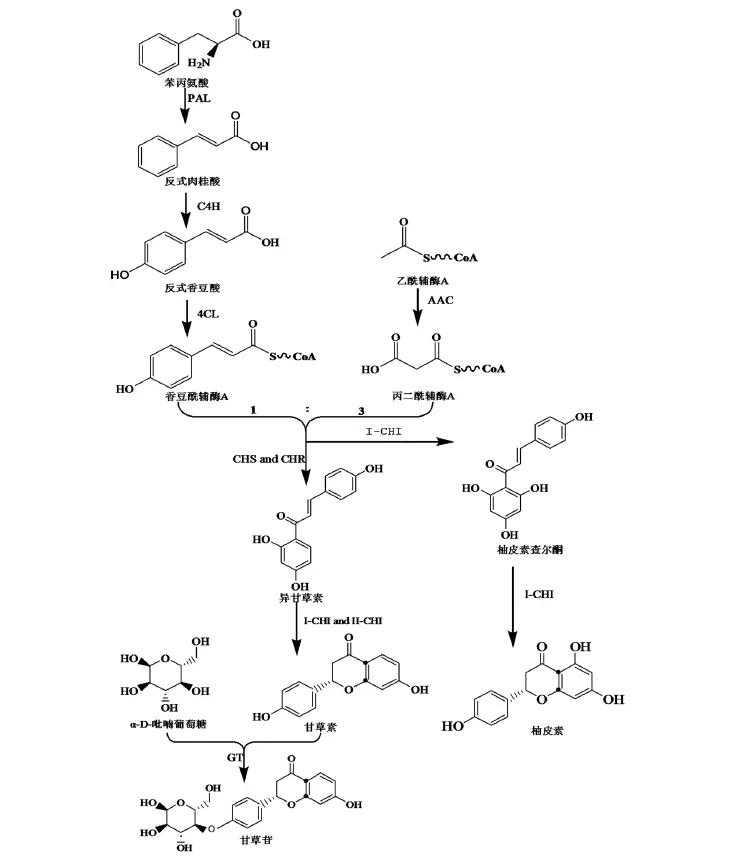
圖1 甘草苷合成途徑
共獲得20條長度約為690 bp的脹果甘草CHI基因cDNA序列(圖2),與目標條帶長度一致。測序結果顯示,所得20條cDNA序列長度均為690 bp。DNAMAN比對結果顯示20條cDNA序列中存在22個變異位點,可分為6種單倍型J1~J6,一致性為99.54%(表1)。將這6種單倍型與文獻[12]報道的烏拉爾甘草CHI基因cDNA序列進行比對,發現其一致性為98.55%~100%,因此可確定獲得的序列為脹果甘草CHI基因cDNA序列。氨基酸序列比對結果顯示存在14個變異位點,一致性為98.98%(表2)。在GenBank完成所有序列的注冊,注冊登錄號見表3。在兩兩比對時發現J3與J1、J2、J4、J5、J6之間的一致性分別為99.86%、98.55%、99.13%、99.42%和99.86%,較其他高;J2與J1、J3、J4、J5、J6之間的一致性分別為98.41%、98.55%、97.68%、97.97%和98.41%,較其他低。因此,挑選這2條序列進行后續生物信息學分析。
2.2 理化性質分析
用ProtParam軟件預測6條脹果甘草CHI編碼蛋白的理化性質,結果見表3。脹果甘草CHI編碼的蛋白由229個氨基酸殘基構成,相對分子質量約24.5×103,等電點為5.3~6.2,不穩定指數均小于40,表明其為穩定蛋白,半衰期為30 h。總平均親水性為-0.125~-0.040,均為負值且介于-2.0~2.0,說明脹果甘草CHI為親水性蛋白。
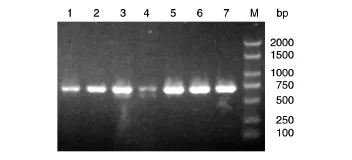
圖2 CHI擴增結果
2.3 脹果甘草CHI的同源性分析
利用DNAMAN將所得6條脹果甘草CHI氨基酸序列與其他植物的該氨基酸序列進行同源性比較,結果表明脹果甘草J3與文獻[12]報道的烏拉爾甘草一致性最高,為 100%;其次與綠豆(AJZ72654.1)的一致性較高,為76.42%;而與擬南芥(OAP11712.1)的一致性最低,僅為7.48%。J2與文獻[12]報道的烏拉爾甘草一致性最高,為97.38%;其次為綠豆(AJZ72654.1),74.67%;與擬南芥(OAP11712.1)的一致性最低,僅為7.14%。可見CHI在不同物種間的同源性較低。
根據親緣關系的遠近,在NCBI數據庫中挑選有代表性的20個其他物種的CHI序列,利用MEGA5.1軟件,將其與本研究所得脹果甘草CHI氨基酸序列構建Neighbor-Joining系統進化樹,如圖3所示。27條氨基酸序列共分為2大類群,其中6條脹果甘草CHI序列與文獻報道的烏拉爾甘草聚到一支,且與豆科植物綠豆(AJZ72654.1)在進化關系上最近,與枸杞(AIC33515.1)、郁金香(AGJ50586.1)、紫花苜蓿(AGZ04578.1)等較遠,與擬南芥(OAP11712.1)、黃芩(AMW 91737.1)等相距更遠,該結果與序列分析結果相符。
2.4 脹果甘草CHI二級結構預測
用DNAMAN和SOPMA對J3和J2的二級結構進行預測,結果如圖4。J3由34.06% α螺旋、21.40%延伸鏈、11.79% β折疊、32.75%不規則卷曲組成;J2由31.44% α螺旋、20.09%延伸鏈、11.79% β折疊、36.68%不規則卷曲組成。J3與J2在29~140、175~218位均存在α螺旋,30~34、163~167、222~224位均存在β折疊,123~127、142~145、159~160位為延伸鏈,35~38、88~91、105~107、172~174位為不規則卷曲,由此可推斷兩者在氨基酸二級結構上相近。
2.5 脹果甘草CHI結構域分析及三級結構預測
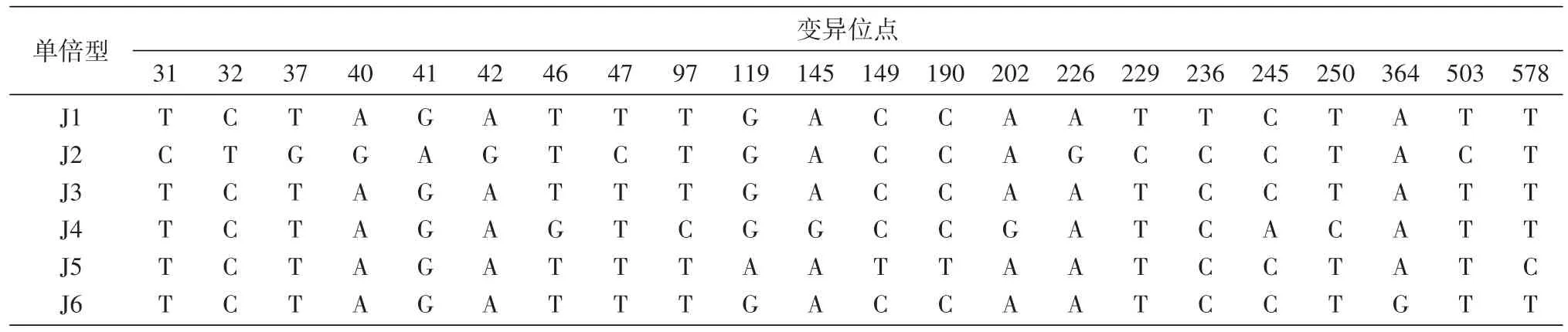
表1 脹果甘草CHI基因cDNA序列變異位點統計表
對J3、J2進行結構域分析,結果顯示均包含一個查爾酮3超家族結構域。采用SWISS-Model分別對J3和J2進行蛋白質三級結構同源性建模,結果如圖5所示,以X-ray為方法,1jx1.1.A為模板建立的J3模型和J2模型立體結構相似。2.6 脹果甘草CHI信號肽的預測及分析
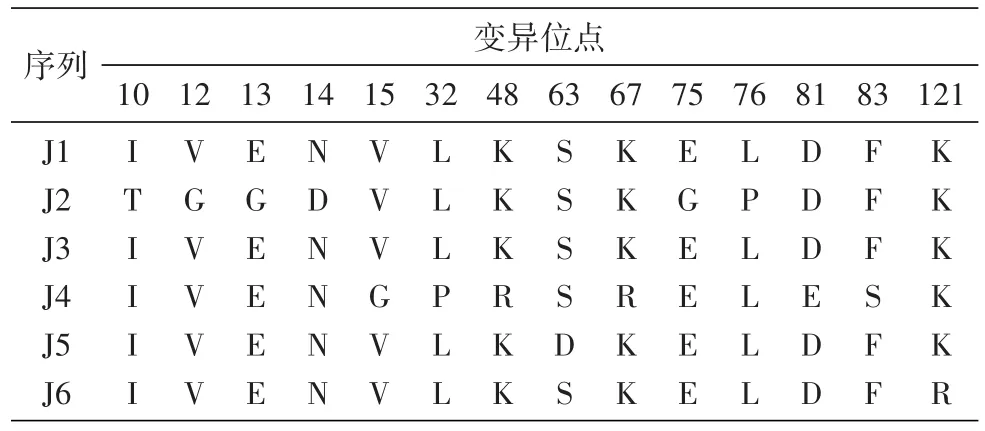
表2 脹果甘草CHI氨基酸序列變異位點統計表
用SignalP 4.1 Server對J3、J2進行信號肽預測,結果如圖6。J3序列中第24位氨基酸殘基具有最高的原始剪切位點分值0.116,第2號氨基酸殘基具有最高的信號肽分值0.154,第11號氨基酸殘基具有最高的綜合剪切位點分值0.119。J2序列中第9位氨基酸殘基具有最高的原始剪切位點分值0.109,第2號氨基酸殘基具有最高的信號肽分值0.131,第12號氨基酸殘基具有最高的綜合剪切位點分值0.106。由此可見,脹果甘草CHI不存在信號肽酶切位點,不具有信號肽。
2.7 脹果甘草CHI跨膜結構域的預測

表3 脹果甘草CHI氨基酸序列理化性質
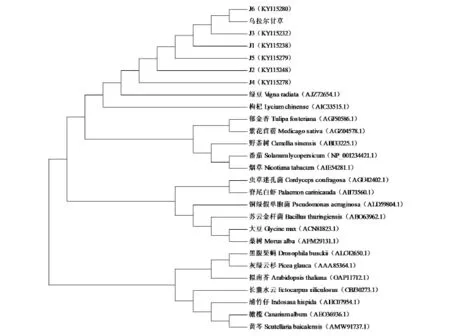
圖3 CHI系統進化樹圖
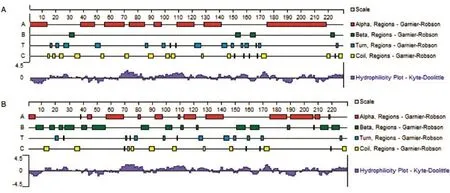
圖4 脹果甘草CHI二級結構預測
用TMHMM Serber v.2.0對脹果甘草CHI氨基酸序列的跨膜結構域進行預測,結果如圖7。J3、J2序列均不存在跨膜結構域,這也與它們的親水性較高相符。由此可推斷脹果甘草查爾酮異構酶無跨膜區,存在于膜外。
3 討論
近年來因過度采挖,已造成野生甘草瀕臨滅絕的現象[13]。目前市售甘草藥材多為栽培甘草,因此對栽培甘草的合理利用和有效開發尤為重要。本課題組前期研究顯示栽培甘草中甘草苷的含量普遍偏低,難以達到藥典標準。因此,對甘草苷生物合成途徑中的關鍵酶開展相關研究,有可能揭示甘草苷生物合成的分子機制,從而提高栽培甘草中甘草苷的累積水平,是中藥現代化的具體表現,也是保護和發展中國傳統道地藥材的基礎性工作。
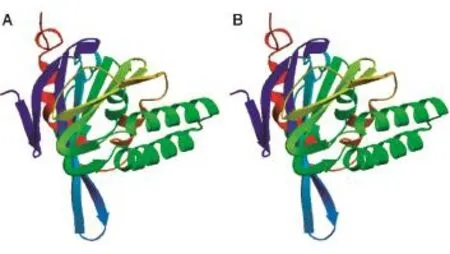
圖5 脹果甘草CHI三級結構預測
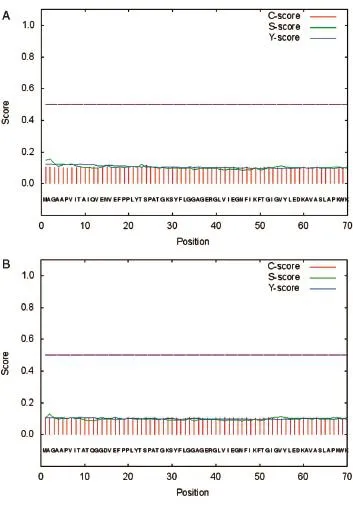
圖6 脹果甘草CHI信號肽預測

圖7 脹果甘草跨膜結構域預測
我們用RT-PCR方法克隆了脹果甘草CHI序列,獲得6種單倍型,比對分析后發現其cDNA序列雖不完全相同,但是其蛋白的理化性質、二級及三級結構等差異較小,且均包含一個查爾酮3超家族結構域,無信號肽和跨膜區。由此推斷cDNA序列單核苷酸多態性對其編碼酶的功能影響較小。分析顯示CHI在不同物種間同源性相對較低,因此這可能給同源克隆帶來一定的難度。
CHI廣泛存在于多種植物中,它是黃酮代謝途徑中的關鍵酶,其表達量的多少直接影響著金銀花顏色的形成[9];并在葡萄果皮、種子和果肉發育過程中有利于花青素的積累,對葡萄的質量和營養價值有重要意義[14]。除此之外,Verhoeyen等通過異位表達查爾酮異構酶,得到了總黃酮醇含量增加48倍的番茄[15];Lukaszewicz等將查爾酮合成酶基因導入馬鈴薯塊莖,基因的過表達引起了黃酮類化合物的增加,改善了馬鈴薯的抗氧化能力[16]。本研究克隆得到6種脹果甘草CHI單倍型,將有利于進一步探討甘草中黃酮類物質生物合成的分子調控機制以及提高甘草質量。
[1]中華人民共和國藥典Ⅰ部[M].北京:化學工業出版社, 2015:86-87.
[2]向誠,喬雪,葉敏,等.利用數據庫對甘草屬植物化學成分的分類和分布分析[J].藥學學報,2012,47(8):1023-1030.
[3]馬君義.國產四種甘草次生代謝產物的研究[D].蘭州:西北師范大學,2004.
[4]沈湛云,劉春生,王學勇.甘草β-香樹酯醇合成酶的編碼區克隆與序列分析[J].中國中藥雜志,2009,19:2438-2440.
[5]劉穎,許巧仙,王學勇,等.甘草3-羥基-3-甲基戊二酰輔酶A還原酶基因多態性與甘草酸含量的相關性分析[J].中國中藥雜志,2012,37(24):3789-3792.
[6]王青,苗文娟,向誠,等.烏拉爾甘草中黃酮類化學成分的研究[J].中草藥,2014,45(1):31-36.
[7]Jez J M,Bowman M E,Dixon R A,et al.Structure and mechanism of the evolutionarily unique plant enzyme chalcone isomerase[J].Nat Struct Biol,2000,7 (9):786-791.
[8]Shimada N,Aoki T,Sato S,et al.A cluster of genes encodes the two types of chalcone isomerase involved in the biosynthesis of general flavonoids and legumespecific 5-deoxy(iso)flavonoids in Lotus japonicus[J]. Plant Physiol,2003,131(3):941-951.
[9]喬中全.金銀花chs、fls、chi基因全長克隆及序列分析[D].長沙:中南林業科技大學,2012.
[10]瞿文全.水母雪蓮(S.Medusa Maxim)類黃酮合成及抗逆相關基因克隆與查耳酮異構酶(CHI)基因的原核表達[D].重慶:西南農業大學,2003.
[11]吳彥慶,趙大球,王靜,等.芍藥查爾酮異構酶基因(CHI)克隆、密碼子偏好性分析以及蛋白結構功能預測[J].華北農學報,2016,31(2):71-80.
[12]李建華.烏拉爾甘草黃酮異戊烯基轉移酶研究[D].北京:北京協和醫學院,2014.
[13]黃明進,王文全,魏勝利.我國甘草藥用植物資源調查及質量評價研究[J].中國中藥雜志,2010,35(8):947-952.
[14]周軍,姚泉洪,彭日荷,等.巨峰葡萄查爾酮異構酶基因克隆及表達分析[J].西北植物學報,2009,29(9):1723-1729.
[15]Verhoeyen M E,Bovy A,Collins G,et al.Increasing antioxidant levels in tomatoes through modification of the flavonoid biosynthetic pathway[J].J Exp Botany, 2002,53(377):2099-2106.
[16]Lukaszewicz M,Matysiak-Kata I,Skala J,et al.Antioxidant capacity manipulation in transgenic potato tuber by changes in phenolic compounds content.[J].J Agric Food Chem,2004,52(6):1526-1533.
Cloning and Sequence Analysis of Chalcone Isomerase Gene from Glycyrrhiza inflata Bat.
YIN Yan-Chao,ZHANG Xiao-Dong,MA Yong-Sheng, ZHOU Shan,GAO Zhi-Qiang,HU Ting,LIU Ying*
School of Chinese Pharmacy,Beijing University of Chinese Medicine,Beijing 100102,China
*Corresponding author,E-mail:liuyliwd@.sina.com
Objective:To clone and analyze the chalcone isomerase(CHI)gene fromGlycyrrhiza inflataBat..Methods:Specific primers were designed based on reportedCHIcDNA sequence ofG.uralensis,RT-PCR technique was used to amplifyCHIcDNA sequences ofG.inflata,and which were analyzed by bioinformatics.Results:TwentyCHIcDNA sequences ofG.inflatawith the length of 690 bp were obtained encoding 229 amino acid residues.Twenty-two variable sites were found in these 20 cDNA sequences and 6 haplotypes were determined,with a similarity of 99.54%.Physicochemical property ananlysis shows that theseCHIcoding protiens are all stably hydrophilic protein,with a molecular weigh of 24.5 kD and a isoelectric point between 5.3~6.2.It has no signal peptides or trans membrane domains.Its secondary structure mainly consists of alpha helix and random coil.Also chalcone super family domain is included in the conserved domain.Homology analysis indicates that the homology ofCHIamong different species is low.Conclusion:CHI cDNA sequeces were successfully cloned fromG.inflata.We hope this work will lay a foundation for further researches on the molecular mechanisms of flavonoid biosynthesis in different licorice origins and licorice molecular breeding.
Q943.2
A
1009-0002(2017)03-0274-07
10.3969/j.issn.1009-0002.2017.03.007
2016-11-25
北京中醫藥大學杰出青年人才項目(2016JYBXJQ002)
尹彥超(1993-),男,碩士研究生,(E-mail)yinyanc_pharm@sina.com
劉穎,(E-mail)liuyliwd@.sina.com

