基于異質網絡的長非編碼RNA和蛋白質相互作用的預測算法研究
鄭肖雄 朱文琰
(復旦大學計算機科學技術學院智能信息處理重點實驗室 上海 200433)
基于異質網絡的長非編碼RNA和蛋白質相互作用的預測算法研究
鄭肖雄 朱文琰
(復旦大學計算機科學技術學院智能信息處理重點實驗室 上海 200433)
長非編碼RNA在生物過程中扮演著非常重要的角色,長非編碼RNA可以與多種蛋白質結合發揮其生物功能,預測長非編碼RNA和蛋白質的相互作用也成為了研究長非編碼RNA功能的途徑之一。由于長非編碼RNA的低保守性,通過提取特征和用機器學習算法預測它和蛋白質之間的相互作用將會不太合適。LPHeteSim算法是一種基于對稱路徑隨機游走的方法,它可以衡量異質長非編碼RNA和蛋白質相互作用網絡中兩者的相關性。在導質網絡中,LPHeteSim算法可以有效地預測兩者的相互作用,實驗結果驗證了算法的有效性。
長非編碼RNA和蛋白質相互作用 隨機游走 異質網絡
0 引 言
長非編碼RNA(Long Noncoding RNA) 是指一類長度大于200個核苷酸、不編碼蛋白質的非編碼RNA[1]。在人類轉錄物組中,只有小部分(約1%)的RNA編碼蛋白質,其他的RNA都是不編碼蛋白質的非編碼RNA,其中的大多數屬于長非編碼RNA[2]。越來越多的證據表明長非編碼RNA在多種層面上(表觀遺傳調控、轉錄調控以及轉錄后調控等)調控基因的表達水平[3]。例如,長非編碼RNA參與了X染色體沉默[4],基因組印記以及染色質修飾、轉錄激活、轉錄干擾和核內運輸等多種重要的生物過程。此外,長非編碼RNA還參與了很多疾病的形成,Yang等人[13]從網絡的角度系統的研究了長非編碼RNA和疾病的關系,發現很多長非編碼RNA都和疾病都有聯系。長非編碼RNA功能的多樣性和復雜性是由于和多個蛋白質相互作用[5],它會通過與蛋白質結合實現自己的功能,從而對多個細胞過程進行調控。雖然近年來關于長非編碼RNA的研究進展迅猛,但是絕大部分的長非編碼RNA的功能仍然是不清楚的。
近年來,由于交聯免疫共沉淀生物技術和高通量測序技術的發展,越來越多關于長非編碼RNA和蛋白質的相互作用被發現。然而,通過實驗發現兩者相互作用仍然占很小比例。越來越多研究者通過計算的方法預測長非編碼RNA和蛋白質相互作用。2011年,Bellucci等人[6]提出了CatRAPID方法,他們在Xist網絡中預測RNA和蛋白質交互。同年,Pancaldi等人[7]用隨機森林和支持向量機的機器學習算法,從RNA和蛋白質的物理特性,二級結構等抽取特征,預測RNA和蛋白質的交互。Muppirala等人[8]從蛋白質和RNA序列中抽取特征,使用同樣的算法預測長非編碼RNA和蛋白質的相互作用。2013年,Wang等人[9]用樸素貝葉斯和擴展的樸素貝葉斯分類器進行預測。同年,Lu等人[10]提出了lncPro算法,他們將RNA和蛋白質編碼成數字向量,再用矩陣乘法對RNA-蛋白質對打分的方式進行預測。
以上算法都是基于RNA和蛋白質自身的特性預測兩者的相互作用,然而,由于長非編碼RNA的低保守性[11],基于長非編碼RNA本身的特性預測長非編碼RNA和蛋白質的相互作用可能會遇到很多困難。一些研究者采用了通過相互作用網絡的拓撲結構預測兩者的相互作用,他們通過構建長非編碼RNA和蛋白質的相互作用網絡,用網絡中的隱含信息預測新的交互。2015年Li等人[12]用異種網絡隨機游走方法預測長非編碼RNA和蛋白質交互。他們用長非編碼RNA的表達特征相似性構建長非編碼RNA相似度網絡、STRING數據庫構建蛋白質相互作用網路和NPInter數據庫構建蛋白質相互作用網絡,并提出了LPIHN方法,一種在異質網絡進行重啟型隨機游走的策略,取得了很好的效果。同年,Yang等人[13]采取了基于路徑的雙向隨機游走的算法(HeteSim),預測長非編碼RNA和蛋白質相互作用。然而他們在計算蛋白質之間的相似度時,并沒有考慮到蛋白質之間相似度的強弱關系,本文將在此基礎上采用基于HeteSim的方法,并通過融合不同的蛋白質相似度網絡,構造出更加可靠的蛋白質相似度網絡,預測長非編碼RNA和蛋白質的相互作用。
1 基于LPHeteSim算法預測
1.1 HeteSim 算法
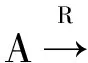
異質網絡中不同的結點之間的邊都包含著不同的信息,通過不同的相關路徑得到的相似度,其背后隱含的意義是各不相同的。例如,在異質長非編碼RNA和蛋白質相互作用網絡中,長非編碼RNA可以通過“長非編碼RNA-蛋白質-蛋白質”(LPP)和“長非編碼RNA-長非編碼RNA-蛋白質”(LLP)兩種路徑連接蛋白質。然而,這兩種路徑背后表達的意義是不一樣的,前者聯合了長非編碼RNA-蛋白質相互作用網絡和蛋白質-蛋白質相似度網絡,后者聯合了長非編碼RNA-長非編碼RNA相似度網絡和長非編碼RNA-蛋白質相互作用網絡。LPP是通過利用蛋白質相似度網絡得到新的長非編碼RNA和蛋白質相互作用,LLP則是利用了長非編碼RNA相似度網絡得到新的長非編碼RNA和蛋白質相互作用。
由于異質網絡中的路徑含有隱藏語義的,這使得對象之間的關系依賴于給定的相關路徑,HeteSim就是在此思想下提出來的。
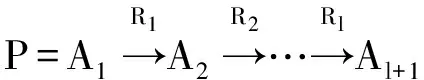
HeteSim(o1,ol|R1°R2°…°Rl-1°Rt)=
HeteSim(Oi(o1|R1),Ij(ol|Rl)|R2°…°Rl-1)
(1)
其中,O(o1|R1)是對象o1基于關系R1的出度鄰居集合,O(ol|Rl)對象ol基于關系Rl的入度鄰居集合。Oi(o1|R1)/Ij(ol|Rl)是O(o1|R1)/O(ol|Rl)中的i/j個對象。
在HeteSim中,如果對象o1和ol在關系I中相同,稱之為自我相關的,本文將定義這種關系為自相關關系(I關系)。
定義2(自相關HeteSim)s與t是相同類型時,基于自相關關系下做相似性度量時,其相似度分數定義如下:
HeteSim(s,t|I)=δ(s,t)
(2)
如果s和t相同時,δ(s,ol)=1,否則δ(s,t)=0。
1.2LPHeteSim算法
原始的自相關HeteSim分數定義s與t相同時δ(s,t)=1,顯然,此種定義不適合長非編碼RNA-蛋白質異質網絡,在Yang等人的論文中對其進行了如下改進:
定義3 如果源對象o1和目標對象ol有相互作用時,在其自相關關系I下做相似性度量時,自相關關系分數定義如下:
HeteSim(o1,ol|I)=δ(o1,ol)
(3)
如果x和y有相互作用,δ(o1,ol)=1,否則δ(o1,ol)=0。
然而,在衡量蛋白質之間的相似性時,直接將其相似度歸為1或者0將影響預測結果的準確性。實際上,現在有很多生物數據庫都提供蛋白質與蛋白質之間的相似度,他們通過生物實驗、計算方法對其相似度進行打分,例如STRING數據庫等,本文將直接采用這些數據庫的打分。所以,本文對HeteSim進行了改進,提出了LPHeteSim算法,該算法的自相關性定義如下:
定義4 源對象是長非編碼RNA(l),目標對象時蛋白質(p),其自相關關系分數定義如下:
LPHeteSim(l,p|I)=δ(l,p)
(4)
如果l和p有相互作用時,δ(l,p)=1,否則δ(l,p)=0。
源對象是蛋白質(p1),目標對象是蛋白質(p2),其自相關關系分數定義如下:
LPHeteSim(p1,p2|I)=δ(p1,p2)
(5)
δ(p1,p2)=p(p1,p2)
(6)
其中,p(p1,p2)是蛋白質p1和蛋白質p2的相似度。
對于基于路徑的雙向隨機游走方法,計算兩個結點的相似度,最重要的是路徑選擇,在Yang的論文中采用的“長非編碼RNA-蛋白質-蛋白質”(LPP)路徑要明顯好于其他路徑,在此,本文也將會采用LPP路徑驗證LPHeteSim算法的有效性。
2 基于LPHeteSim-SNF算法的預測
2.1 相似度網絡融合算法
在計算蛋白質之間的相似度時,有多種計算方式,例如計算蛋白質之間的基因本體(GeneOntology)相似度、域相似度和序列相似度等等,由此,構成不同的蛋白質相似度矩陣。然而,不同的計算方式得到的相似度都有可能含有噪音,最好的方式是通過融合多個相似度矩陣,取每個相似度網絡中的可靠的相似度,去除相似度網絡中不可靠的相似度,即噪音。在此本文整合四種計算蛋白質相似度的方法,對不同度量方式的蛋白質矩陣進行融合,得到一個更加穩健的相似度矩陣。本文采用了由Wang等人[15]在2014年提出的相似網絡融合算法。
假設有n種蛋白質和m種度量蛋白質相似度的方式。蛋白質相似網絡記作圖G=(V,E),V代表蛋白質集合{x1,x2,…,xn},即頂點,E代表蛋白質之間的權重,即邊,相似度矩陣記為W,W(xi,xj)代表蛋白質xi和蛋白質xj之間的相似度。
為了計算多個度量下的蛋白質相似度融合矩陣在頂點集V上定義一個全稀疏核P=D-1W,D是一個對角矩陣,由蛋白質相似度矩陣W得到,并對其進行正則化,D(i,i)=∑jW(i,j),所以∑jP(i,j)=1。然而,正則化之后可能會引入數值不穩定性,因為它涉及到W的對角項上的自我相似性,更好的一種正則化形式如下所示:
(7)
用Ni表示xi在圖G中的鄰居結點,在圖G中,用KNN計算其局部相關性,設置非鄰居結點值為0。假設鄰居結點的相似性比非鄰居結點的相似性可靠,之后再通過網絡將相似性傳播給其他結點:
(8)
在矩陣P中包含著相似性網絡的全部信息,矩陣S中攜帶著相對重要的信息。這里將把矩陣P作為初始狀態,然后對其進行迭代,矩陣S將作為融合過程中的核矩陣,用來獲得矩陣的局部結構性和增強計算效率。
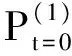
(9)
(10)
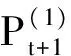
(11)
因為矩陣S是P的KNN圖,所以此迭代的過程中將慢慢降低矩陣中噪音的影響。從迭代步驟中看出,相似度僅會通過結點之間共同的鄰居傳播,這會讓相似度矩陣更加穩固。而且不同度量下的相似度矩陣都會從其他度量下的相似度矩陣得到信息補充,這樣就達到了相互融合的目的。對于m>2時,上述的迭代步驟將演變成:
(12)
2.2LPHeteSim-SNF算法
本文采用了四種度量蛋白質之間相似度的數據集,其中三種是通過蛋白質本身的特性對其相似度打分(基因本體相似性、域相似性、序列相似性),第四種是采用公共數據庫,即String數據庫中的蛋白質相似度分數。
1) 基于基因本體的蛋白質相似度計算
基因本體[16]是一個用規范化的基因和基因產物特性的術語描繪或詞義解釋的數據庫,其中的術語主要涉及到生物學的三個方面:細胞組件、分子功能和生物過程。基因本體是一個有向無環圖,它包含三種關系:分別是is_a、part_of和regulates。為了衡量蛋白質pi和蛋白質pj的基因本體相似度,這里采用Jaccard值[17]計算它們之間的相似度。蛋白質pi對應的基因本體術語集合為ti,蛋白質pj對應的基因本體術語集合為tj。則兩者的相似度如下所示:
(13)
其中,分子是兩個蛋白質對應的基因本體集合的交集,分母是兩者的并集。
2) 基于蛋白質域的相似度計算
蛋白質通常由一個或多個功能區域組成,被稱為蛋白質域。結構域的不同組合方式嘗試的蛋白質也不盡相同。Pfam數據庫[18]中搜集了大量的蛋白質家族,它依賴于由多序列比對和隱馬爾科夫模型產生的結果。
具有相同域的蛋白質之間的相似度會很大,可以將每個蛋白質表示為一個二值向量,如果蛋白質中存在這個域,則向量中對應該域的值為1,否則為0。最后,用廣義的Jaccard值計算它們之間的相似度。
(14)
其中,‖·‖是向量的模,xi和xj分別對應蛋白質pi和蛋白質pj的域向量,SomainSim(pi,pj)為二者的蛋白質域相似度。
3) 基于蛋白質序列的相似度計算
同樣通過序列的方式計算蛋白質之間的相似度,蛋白質序列數據可以從UniProt數據庫[19]中獲得。Uniprot數據庫是一個可供免費使用的蛋白質序列與功能信息數據庫,它包含了大量蛋白質的詳細信息。
本文采用規范化過的Smith-Waterman方法[20]計算蛋白質之間的序列相似度。
(15)
其中sw(pi,pj)是蛋白質pi和蛋白質pj的Smith-Waterman得分。為了將規范化之后的蛋白質相似度得分應用于所有的蛋白質對中,將計算之后的得分平均化:
(16)
為此,由上式可以得到蛋白質pi和蛋白質pj之間的序列相似度。
4) 基于STRING的蛋白質相似度計算
STRING數據庫[21]是一個包含大量蛋白質相互作用的數據庫,它覆蓋了超過2 000多種生物,不僅整合了已被實驗驗證的蛋白質相互作用,還包括通過計算方法得到的蛋白質相互作用。
在由STRING數據庫中得到的得分中,最高分數為999,為了將得分控制在[0,1]內,對其進行如下操作:
(17)
因此,可以得到蛋白質pi和蛋白質pj之間的STRING相似度。
在得到四種蛋白質相似度網絡,用相似度網絡融合算法(SNF)進行融合。在聯合長非編碼RNA和蛋白質相互作用網絡,構建異質長非編碼RNA和蛋白質相互作用網絡。在此異質網絡中,執行LPHeteSim算法,預測新的長非編碼RNA和蛋白質相互作用。
3 實驗分析
本文實驗從NPInter(V2.0)數據庫[22]中抽取所有非編碼RNA和蛋白質相互作用的數據,再從NONCODE(V4.0)數據庫[23]中抽取人類的長非編碼RNA數據,如果交互數據中的非編碼RNA是長非編碼RNA,抽取它與蛋白質的交互數據。因此,可以構建出長非編碼RNA和蛋白質相互作用網絡。對于在長非編碼RNA和蛋白質相互作用網絡中的蛋白質,分別從GO數據庫、Uniprot數據庫、Pfam數據庫中獲得它們的GO信息、序列信息和域信息,再分別計算出蛋白質之間的GO相似度、序列相似度以及域相似度。本文同樣從STRING(v10.0)數據庫中獲取由STRING數據庫計算的蛋白質之間相互作用的得分,并計算出蛋白質之間的STRING相似度得分。為此,經過上述操作,可以獲得四種蛋白質相似度網絡。同樣對數據集進行了預處理,去除只和一個蛋白質相互作用的長非編碼RNA,因為無法采用留一交叉驗證進行驗證。同樣丟棄和其他所有蛋白質的相似度都為0的蛋白質,因為,它們只會給實驗帶來噪音。最后,將得到的長非編碼RNA的數量、蛋白質的數量以及兩者的相互作用數量統計如表1所示。
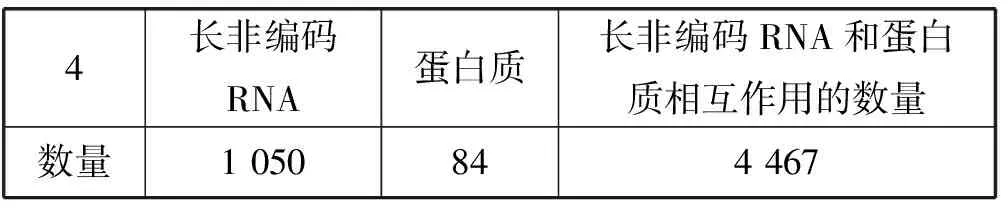
表1 長非編碼RNA和蛋白質以及它們相互作用的數量
本文采用留一交叉驗證驗證我們的算法有效性。對于每種實驗結果,分別得到它們的ROC曲線以及ROC曲線下的面積(AUC)值衡量算法的有效性。實驗中所采用的正樣本即是已知的長非編碼RNA和蛋白質相互作用,負樣本即是未被報道的長非編碼RNA和蛋白質相互作用。
先基于四個蛋白質相似度網絡測試LPHeteSim算法,對比算法為Yang等人所采用的HeteSim算法。
LPHeteSim和HeteSim算法在基因本體(Go)、蛋白質域、蛋白質序列、STRING數據庫四個數據集上ROC曲線如圖1、圖2、圖3和圖4所示。實線代表LPHeteSim算法在基于此蛋白質相似度網絡的ROC曲線,虛線代表HeteSim算法的ROC曲線。LPHeteSim的ROC曲線顯然比HeteSim的高,為此可以說明算法改進的有效性。然而,兩者都會在其中一點匯聚,因為LPHeteSim可以提高預測的精確度,并不會增多預測數量。
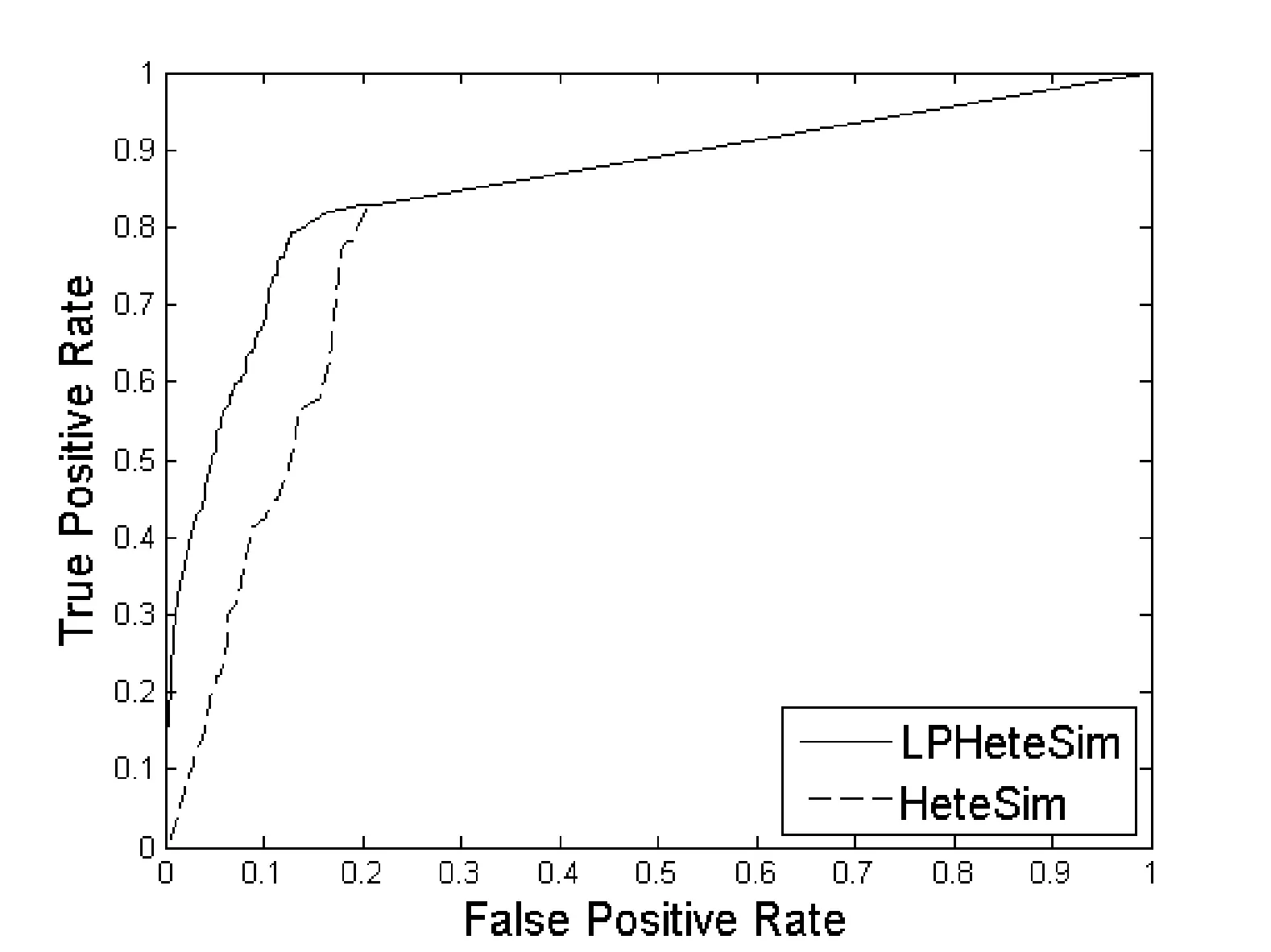
圖1 基于蛋白質基因本體相似度的LPHeteSim和HeteSim的ROC曲線
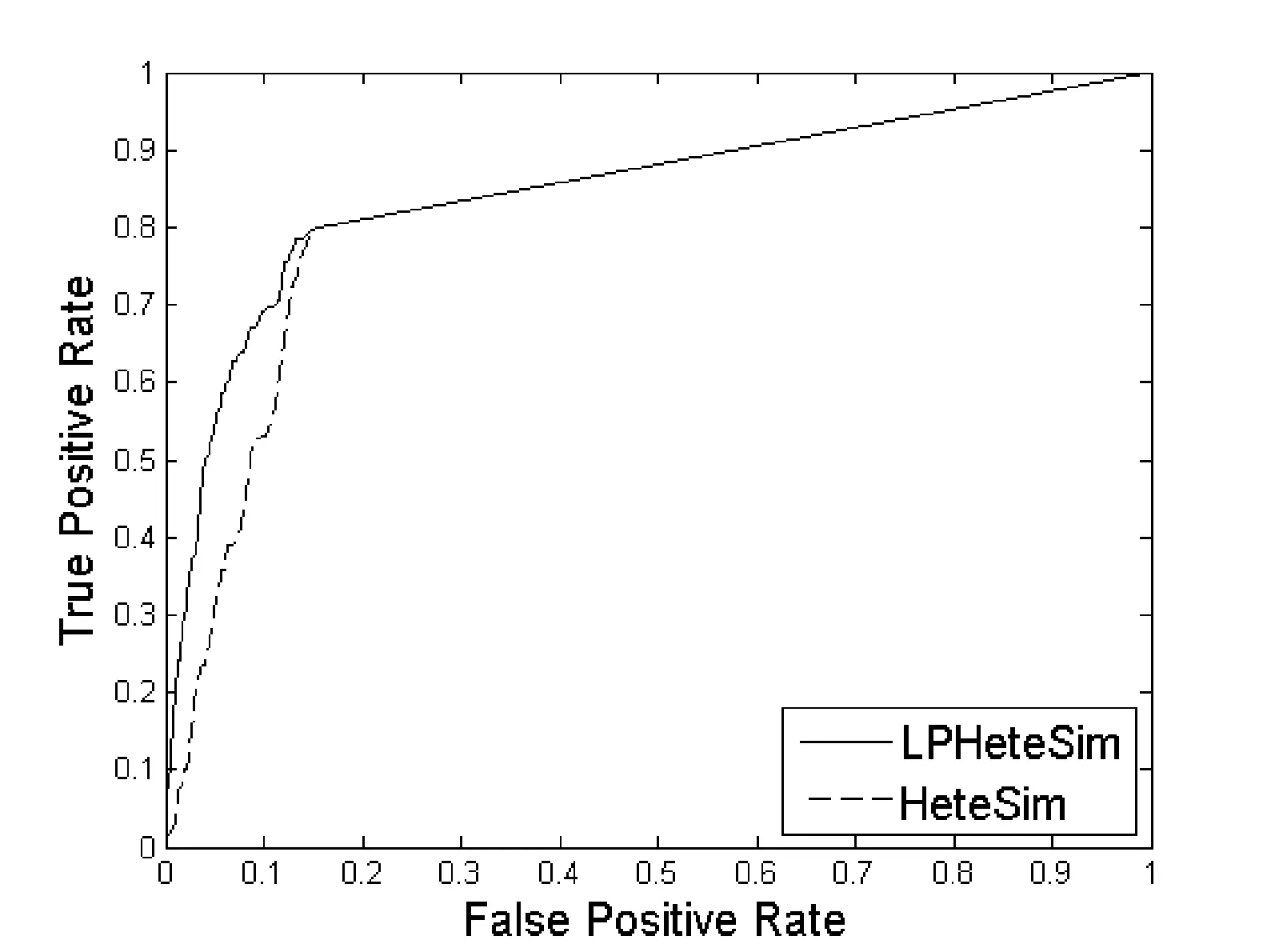
圖2 基于蛋白質域的LPHeteSim和HeteSim的ROC曲線
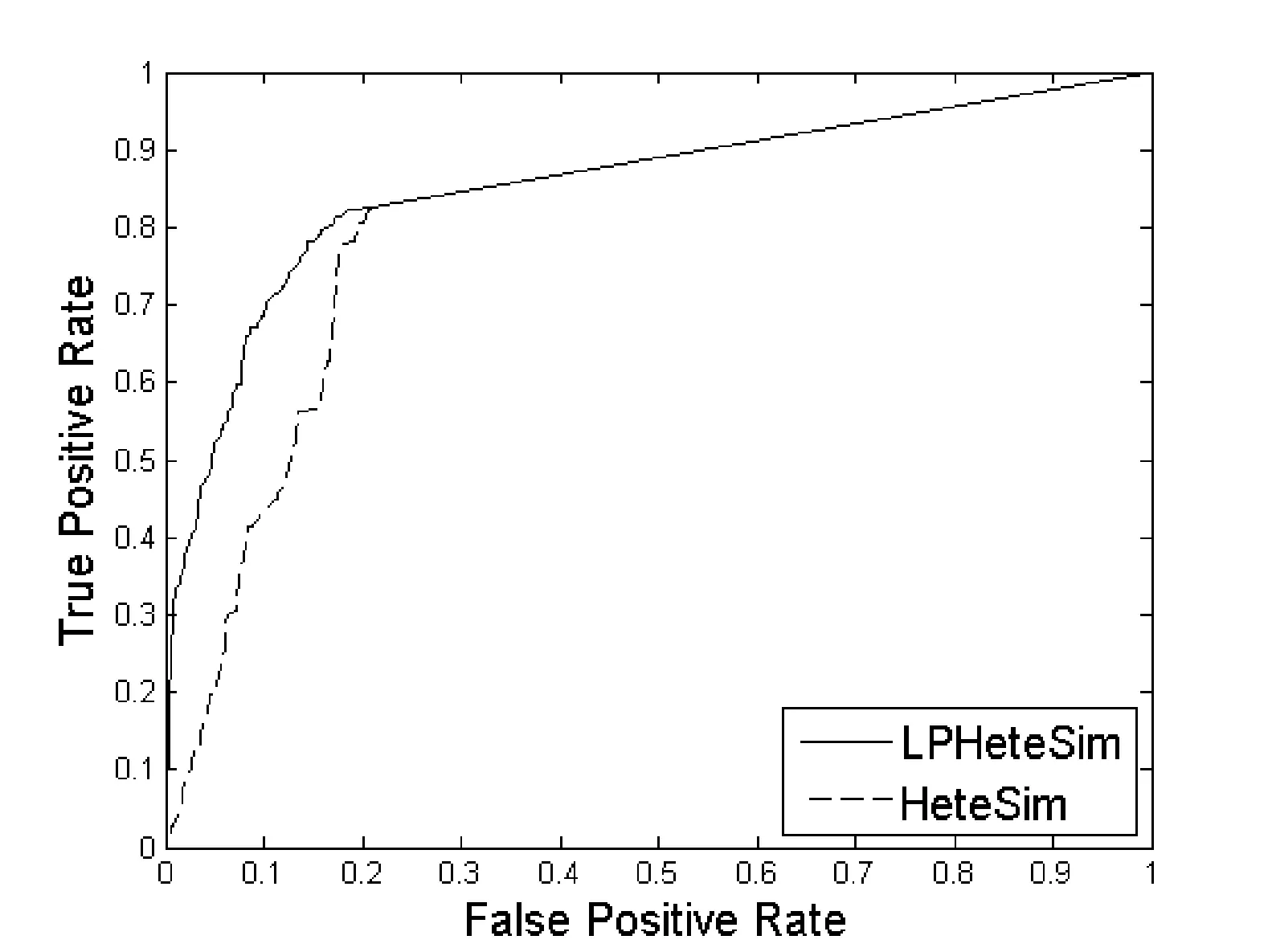
圖3 基于蛋白質序列相似度的LPHeteSim和HeteSim的ROC曲線
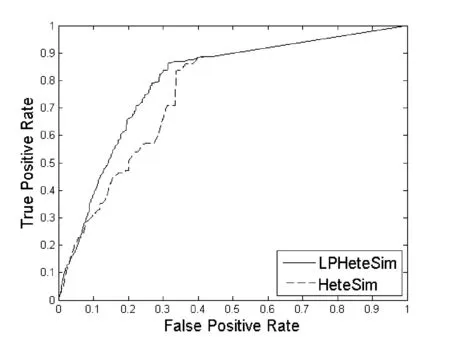
圖4 基于STRING數據庫的LPHeteSim和HeteSim的ROC曲線
LPHeteSim和HeteSim算法的AUC值如表2所示, LPHeteSim算法在各個蛋白質相似度網絡上的表現均要比HeteSim算法好很多,它們的AUC值分別為0.858 4、0.848 9、0.856 5和0.797 2。均要比HeteSim算法的預測效果好,為此,驗證了本文的算法有效性。
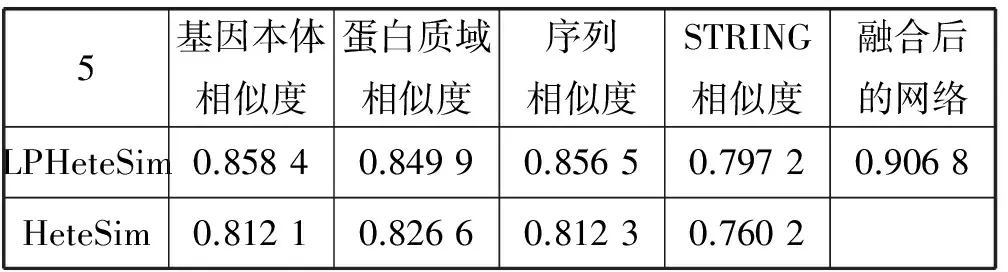
表2 LPHeteSim和HeteSim在各個數據集上的AUC值
然后用SNF算法融合四個蛋白質相似度網絡,并在基于融合之后的相似度網絡上執行LPHeteSim算法,預測新的長非編碼RNA和蛋白質相互作用。預測結果的ROC曲線如圖5所示。
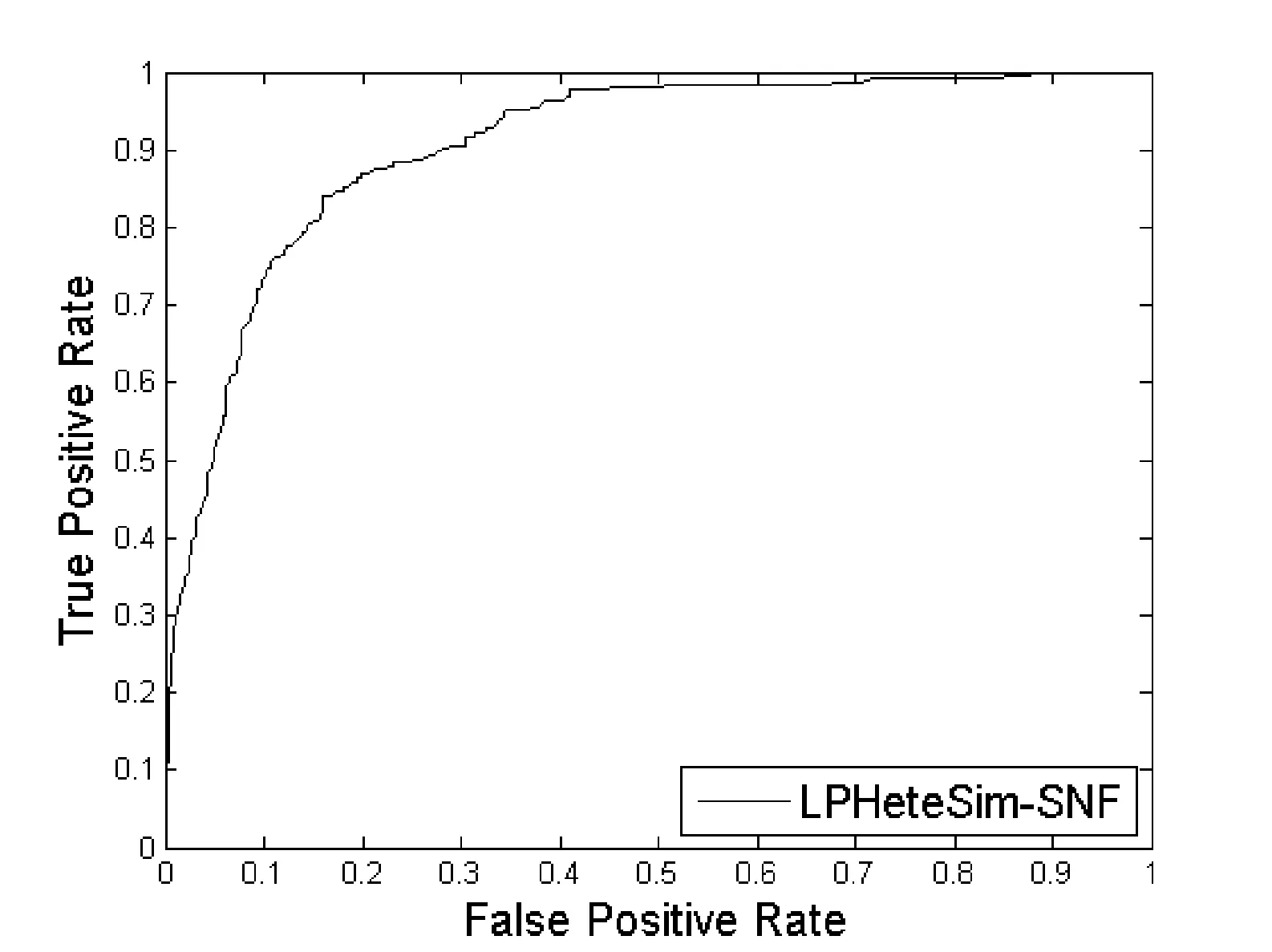
圖5 基于融合蛋白質相似度網絡的LPHeteSim的ROC曲線
可以從ROC曲線和AUC值看出,融合之后的AUC值為0.906 8,明顯比在每個蛋白質相似度網絡的預測效果高。因此,相似度網絡融合算法可以減少每個矩陣中的噪音,可以有效地提高預測算法的準確性。
4 結 語
本文在HeteSim算法的基礎上,通過改進自相關關系相似度的計算方式,提出了LPHeteSim算法。在單個蛋白質相似度網絡上的實驗表明,改進之后的算法的有效性要好于未改進的HeteSim算法。
為了進一步提高預測準確性,本文采用融合蛋白質相似度網絡的方法,構造出置信度更高的蛋白質相似度矩陣。實驗結果表明,在融合之后的蛋白質相似度網絡執行預測算法,要好于在單個蛋白質相似度網絡上的預測結果。因此,結合相似度融合算法和LPHeteSim算法,可以有效地提高預測新的長非編碼RNA和蛋白質相互作用的有效性。
[1] Bonasio R,Shiekhattar R.Regulation of transcription by long noncoding RNAs[J].Annual Review of Genetics,2014,48:433.
[2] International Human Genome Sequencing Consortium.Finishing the euchromatic sequence of the human genome[J].Nature,2004,431(7011):931-945.
[3] Geisler S,Coller J.RNA in unexpected places:long non-coding RNA functions in diverse cellular contexts[J].Nature reviews Molecular cell biology,2013,14(11):699-712.
[4] Engreitz J M,Pandya-Jones A,McDonel P,et al.The Xist lncRNA exploits three-dimensional genome architecture to spread across the X chromosome[J].Science,2013,341(6147):1237973.
[5] Zhu J J,Fu H J,Wu Y G,et al.Function of lncRNAs and approaches to lncRNA-protein interactions[J].Science China Life Sciences,2013,56(10):876-885.
[6] Bellucci M,Agostini F,Masin M,et al.Predicting protein associations with long noncoding RNAs[J].Nature Methods,2011,8(6):444-445.
[7] Pancaldi V,B?hler J.In silico characterization and prediction of global protein-mRNA interactions in yeast[J].Nucleic Acids Research,2011:gkr160.
[8] Muppirala U K,Honavar V G,Dobbs D.Predicting RNA-protein interactions using only sequence information[J].BMC Bioinformatics,2011,12(1):1.
[9] Wang Y,Chen X,Liu Z P,et al.De novo prediction of RNA-protein interactions from sequence information[J].Molecular BioSystems,2013,9(1):133-142.
[10] Lu Q,Ren S,Lu M,et al.Computational prediction of associations between long non-coding RNAs and proteins[J].BMC Genomics,2013,14(1):1.
[11] Pang K C,Frith M C,Mattick J S.Rapid evolution of noncoding RNAs:lack of conservation does not mean lack of function[J].Trends in Genetics,2006,22(1):1-5.
[12] Li A,Ge M,Zhang Y,et al.Predicting Long Noncoding RNA and Protein Interactions Using Heterogeneous Network Model[J].BioMed Research International,2015,2015.
[13] Yang J,Li A,Ge M,et al.Prediction of interactions between lncRNA and protein by using relevance search in a heterogeneous lncRNA-protein network[C]//Control Conference (CCC),2015 34th Chinese.IEEE,2015:8540-8544.
[14] Shi C,Kong X,Huang Y,et al.Hetesim:A general framework for relevance measure in heterogeneous networks[J].Knowledge and Data Engineering,IEEE Transactions on,2014,26(10):2479-2492.
[15] Wang B,Mezlini A M,Demir F,et al.Similarity network fusion for aggregating data types on a genomic scale[J].Nature Methods,2014,11(3):333-337.
[16] Ashburner M,Ball C A,Blake J A,et al.Gene Ontology:tool for the unification of biology[J].Nature Genetics,2000,25(1):25-29.
[17] Jcquart P.Nouvelles recherches sur la distribution florale[J].Bull.Soc.Vand.Sci.Nat,1908(0):44.
[18] Finn R D.Pfam:the protein families database[J].Encyclopedia of Genetics, Genomics, Proteomics and Bioinformatics,2012.
[19] Apweiler R,Martin M J,O’Donovan C,et al.Update on activities at the Universal Protein Resource (UniProt) in 2013[J].Nucleic acids research,2013,41(D1):D43-D47.
[20] Smith T F,Waterman M S.Identification of common molecular subsequences[J].Journal of Molecular Biology,1981,147(1):195-197.
[21] Szklarczyk D,Franceschini A,Wyder S,et al.STRING v10:protein-protein interaction networks,integrated over the tree of life[J].Nucleic Acids Research,2014:gku1003.
[22] Yuan J,Wu W,Xie C,et al.NPInter v2. 0:an updated database of ncRNA interactions[J].Nucleic Acids Research,2014,42(D1):D104-D108.
[23] Xie C,Yuan J,Li H,et al.NONCODEv4:exploring the world of long non-coding RNA genes[J].Nucleic Acids Research,2014,42(D1):D98-D103.
RESEARCH ON PREDICTION ALGORITHM FOR LNCRNA-PROTEIN INTERACTIONSBASED ON HETEROGENEOUS NETWORK
Zheng Xiaoxiong Zhu Wenyan
(ShanghaiKeyLabofIntelligentInformationProcessing,SchoolofComputerScience,FudanUniversity,Shanghai200433,China)
Long noncoding RNA (lncRNA) plays a key role in biological and pathological processes. LncRNA can have interaction with multiple proteins, therefore predicting lncRNA-protein interaction comes to be one of the ways to study the functions of lncRNA. Because of the low conservation of lncRNA, the methods of predicting lncRNA-protein interaction by using machine learning algorithms with extracting the features of lncRNA and protein may be not fit enough. LPHeteSim algorithm is a method based on pair-wise random walk which can measure the relevance between lncRNA and protein in the lncRNA-protein heterogeneous network. In order to decrease the noise of protein similarity networks, we use similarity network fusion (SNF) algorithm to fuse the protein similarity networks under different metric together, and reach a better result.
LncRNA-protein interaction Randow walk Heterogeneous network
2016-03-14。鄭肖雄,碩士,主研領域:生物信息。朱文琰,碩士。
TP3
A
10.3969/j.issn.1000-386x.2017.03.041

