基于GBrowse的多源長(zhǎng)非編碼RNA數(shù)據(jù)可視化系統(tǒng)①
孫 磊, 陳 璇, 唐 紅, 魏李婷, 姬嵐洋, 施勝飛, 楊曉華
?
基于GBrowse的多源長(zhǎng)非編碼RNA數(shù)據(jù)可視化系統(tǒng)①
孫 磊, 陳 璇, 唐 紅, 魏李婷, 姬嵐洋, 施勝飛, 楊曉華
(揚(yáng)州大學(xué)信息工程學(xué)院, 揚(yáng)州 225127)
針對(duì)長(zhǎng)非編碼RNA(long non-coding RNA, lncRNA)數(shù)據(jù)類型多樣帶來(lái)的有用信息提取困難的問(wèn)題, 提出基于基因組瀏覽器GBrowse(Generic Genome Browser)的多源lncRNA數(shù)據(jù)可視化系統(tǒng). 該系統(tǒng)主要包括網(wǎng)頁(yè)服務(wù)器和lncRNA數(shù)據(jù)存儲(chǔ). 其中, 網(wǎng)頁(yè)服務(wù)器主要由HTTP服務(wù)和GBrowse網(wǎng)頁(yè)組件構(gòu)成, 支持純文本、MySQL、SQLite等多種數(shù)據(jù)存儲(chǔ)方式. 系統(tǒng)實(shí)現(xiàn)流程包括GBrowse安裝與配置、多源lncRNA數(shù)據(jù)的收集、數(shù)據(jù)預(yù)處理、數(shù)據(jù)存儲(chǔ)、數(shù)據(jù)訪問(wèn)及可視化配置. 原型系統(tǒng)收集了六種人類lncRNA數(shù)據(jù), 包括人類基因注釋、基因組序列、組蛋白修飾H3K4me3信號(hào)及其位點(diǎn)、轉(zhuǎn)錄因子CTCF綁定位點(diǎn)信號(hào)及其位點(diǎn)的數(shù)據(jù), 并對(duì)數(shù)據(jù)進(jìn)行了預(yù)處理. 通過(guò)MySQL、SQLite等建立了lncRNA數(shù)據(jù)庫(kù), 對(duì)數(shù)據(jù)的訪問(wèn)方式和可視化參數(shù)進(jìn)行配置. 實(shí)驗(yàn)結(jié)果表明, 多源lncRNA數(shù)據(jù)在GBrowse框架下能夠得到整合與可視化, 并在基因組空間同時(shí)呈現(xiàn), 這使得研究者能夠以更加直觀的方式觀測(cè)數(shù)據(jù), 進(jìn)而建立新的科學(xué)假說(shuō).
長(zhǎng)非編碼RNA; 基因組瀏覽器; 數(shù)據(jù)庫(kù); 可視化
長(zhǎng)非編碼RNA(long noncoding RNA, lncRNA)是一類具有重要生物學(xué)功能的非編碼RNA. 研究表明lncRNA參與胚胎干細(xì)胞凋零、細(xì)胞循環(huán)調(diào)控等細(xì)胞過(guò)程[1,2]. 近年來(lái), 隨著高通量測(cè)序技術(shù)的發(fā)展和應(yīng)用(如RNA-Seq), 成千上萬(wàn)的功能性lncRNA被發(fā)現(xiàn), 同時(shí)也產(chǎn)生了大量用于分析lncRNA功能和機(jī)制的生物數(shù)據(jù). lncRNA數(shù)據(jù)來(lái)源廣泛, 主要包括與lncRNA直接相關(guān)的基因注釋、序列、組蛋白修飾、轉(zhuǎn)錄因子綁定位點(diǎn)等數(shù)據(jù)和信息, 以及蛋白質(zhì)編碼RNA數(shù)據(jù)、物種間序列比對(duì)、保守性分值等用于與lncRNA數(shù)據(jù)進(jìn)行比較分析的數(shù)據(jù). 如何有效分析這些多源lncRNA數(shù)據(jù)已成為lncRNA功能研究的重要挑戰(zhàn).
為了準(zhǔn)確推斷l(xiāng)ncRNA的功能和機(jī)制, 可首先對(duì)多源lncRNA數(shù)據(jù)進(jìn)行可視化, 后根據(jù)數(shù)據(jù)在基因組空間的關(guān)系設(shè)立假說(shuō)并建模, 再通過(guò)統(tǒng)計(jì)分析對(duì)lncRNA的功能機(jī)制進(jìn)行推斷. 其中, lncRNA數(shù)據(jù)可視化是關(guān)鍵步驟. 基于網(wǎng)頁(yè)技術(shù)的基因組瀏覽器為包括lncRNA數(shù)據(jù)在內(nèi)的基因數(shù)據(jù)的可視化和交互操作提供了有效方法. 當(dāng)前流行的基因組瀏覽器是加州大學(xué)圣克魯茲分校基因組瀏覽器(UCSC genome browser)[3], 但由于其服務(wù)器遠(yuǎn)在美國(guó), 因此數(shù)據(jù)上傳和下載可能會(huì)受網(wǎng)絡(luò)連接狀況和帶寬限制等因素的影響. 另一方面, 類似UCSC基因組瀏覽器的公共瀏覽器在免費(fèi)使用情況下并不能提供完善的服務(wù)(如數(shù)據(jù)共享等). 因此, 當(dāng)研究者的lncRNA數(shù)據(jù)量特別大或需要高級(jí)訪問(wèn)服務(wù)時(shí), 公共基因組數(shù)據(jù)瀏覽器可能無(wú)法滿足研究需要. 相較而言, 可在本地建立諸如UCSC基因組瀏覽器、GBrowse[4]、JBrowse[5]等瀏覽器. 在本地私有網(wǎng)絡(luò)環(huán)境下, 數(shù)據(jù)的傳輸速率將大大提高. 研究者還可根據(jù)需要設(shè)置相應(yīng)的服務(wù)選項(xiàng), 以增加數(shù)據(jù)整合與可視化的靈活性. GBrowse是一種開(kāi)放源代碼的通用基因組瀏覽器(Generic Genome Browser), 它為用戶提供了豐富的生物數(shù)據(jù)存儲(chǔ)、交互式管理以及可視化方法. GBrowse憑借其存儲(chǔ)、管理、可視化數(shù)據(jù)方面的諸多優(yōu)點(diǎn), 已廣泛應(yīng)用于如植物lncRNA數(shù)據(jù)庫(kù)PLncDB[6]、家禽lncRNA數(shù)據(jù)庫(kù)ALDB[7]、深度測(cè)序信號(hào)可視化VING[8]、轉(zhuǎn)錄起始位點(diǎn)的識(shí)別[9]等研究. 針對(duì)lncRNA功能研究過(guò)程中由于lncRNA數(shù)據(jù)量不斷增加且類型眾多帶來(lái)的有用信息提取困難的問(wèn)題, 本文提出了基于GBrowse的多源lncRNA數(shù)據(jù)可視化系統(tǒng). 實(shí)驗(yàn)以人類lncRNA數(shù)據(jù)的可視化為例, 詳細(xì)介紹該系統(tǒng)的實(shí)現(xiàn)流程. 在此基礎(chǔ)上, 將研究討論轉(zhuǎn)錄因子CTCF、表觀遺傳信息與lncRNA基因之間的相互關(guān)系.
1 系統(tǒng)概述
基于GBrowse的多源lncRNA數(shù)據(jù)可視化系統(tǒng)主要由網(wǎng)頁(yè)服務(wù)器和lncRNA數(shù)據(jù)存儲(chǔ)構(gòu)成(如圖1所示). 其中, lncRNA數(shù)據(jù)可根據(jù)需要存儲(chǔ)于各種類型的數(shù)據(jù)庫(kù), 如Berkeleydb、SQLite、MySQL、Oracle、PostgreSQL, 以及GFF格式文本. 網(wǎng)頁(yè)服務(wù)器除了包括常用的HTTP服務(wù)進(jìn)程之外, 最重要的是包含了GBrowse網(wǎng)頁(yè)組件. GBrowse組件中有豐富的數(shù)據(jù)訪問(wèn)接口, 提供對(duì)以上多種類型數(shù)據(jù)庫(kù)的訪問(wèn).

圖1 系統(tǒng)架構(gòu)圖
2 系統(tǒng)實(shí)現(xiàn)流程
基于GBrowse的多源lncRNA數(shù)據(jù)可視化系統(tǒng)的實(shí)現(xiàn)流程主要包括“GBrowse安裝與配置”、“多源lncRNA數(shù)據(jù)的收集”、“數(shù)據(jù)預(yù)處理”、“數(shù)據(jù)存儲(chǔ)”和“數(shù)據(jù)訪問(wèn)及可視化配置”五個(gè)步驟(如圖2所示). 本節(jié)將以人類lncRNA數(shù)據(jù)的可視化為例, 詳細(xì)介紹系統(tǒng)的實(shí)現(xiàn)流程.

圖2 系統(tǒng)實(shí)現(xiàn)流程
2.1 GBrowse安裝與配置
GBrowse可安裝在Linux等類UNIX操作系統(tǒng)上. 本文采用Ubuntu 12.04 Linux操作系統(tǒng), 通過(guò)Ubuntu軟件中心安裝了版本號(hào)為2.42的GBrowse軟件. 另外, 對(duì)于GBrowse及網(wǎng)頁(yè)服務(wù)器運(yùn)行過(guò)程中所依賴的其他軟件(如Apache2、Perl、MySQL、SQLite等)的安裝, 可參考文檔: http://gmod.org/wiki/GBrowse_2.0_Install_ HOWTO.
2.2 多源lncRNA數(shù)據(jù)的收集
為了幫助推斷人類lncRNA的調(diào)控機(jī)制, 從公共數(shù)據(jù)庫(kù)收集了包括人類基因注釋、人類基因組序列、轉(zhuǎn)錄因子綁定位點(diǎn)和組蛋白修飾在內(nèi)的多種來(lái)源的lncRNA數(shù)據(jù)(如表1所示). 其中, 人類基因注釋數(shù)據(jù)(編號(hào): D1) 下載自GENCODE[10], D1數(shù)據(jù)包含了人類基因的位置、結(jié)構(gòu)、ID號(hào)、數(shù)據(jù)源等信息, 數(shù)據(jù)格式為GFF3 (Generic Feature Format Version 3). 為了獲取與lncRNA相關(guān)的基因組序列信息, 從UCSC基因組瀏覽器下載了人類基因組序列數(shù)據(jù)(編號(hào): D2). 已有研究表明增強(qiáng)子可通過(guò)lncRNA與基因啟動(dòng)子作用以影響基因轉(zhuǎn)錄, 而蛋白質(zhì)CTCF與靶順序因子的結(jié)合可阻斷增強(qiáng)子和啟動(dòng)子的相互作用. 為了研究CTCF與lncRNA之間的關(guān)系, 從ENCODE項(xiàng)目網(wǎng)站(https://www.encodeproject.org/)下載了利用ChIP-Seq技術(shù)獲得的轉(zhuǎn)錄因子CTCF的綁定位點(diǎn)信息, 該信息包含了CTCF綁定位點(diǎn)的信號(hào)(編號(hào): D3)及預(yù)測(cè)出的最佳信號(hào)峰值區(qū)域(編號(hào): D4). 另外, 由于三甲基化組蛋白H3賴氨酸(H3K4me3)與基因轉(zhuǎn)錄起始位點(diǎn)有關(guān), 因此還下載了利用ChIP-Seq技術(shù)獲得的人類骨骼肌細(xì)胞基因的H3K4me3位置信息(包含了H3K4me3的信號(hào)D5和峰值信號(hào)區(qū)域D6). 其中, bigWig格式數(shù)據(jù)提供了通過(guò)測(cè)序方法獲得的信號(hào)強(qiáng)度信息, narrowPeak (BED6+4)和broadPeak (BED6+3) 格式數(shù)據(jù)提供了預(yù)測(cè)出的最佳目標(biāo)區(qū)域信息.
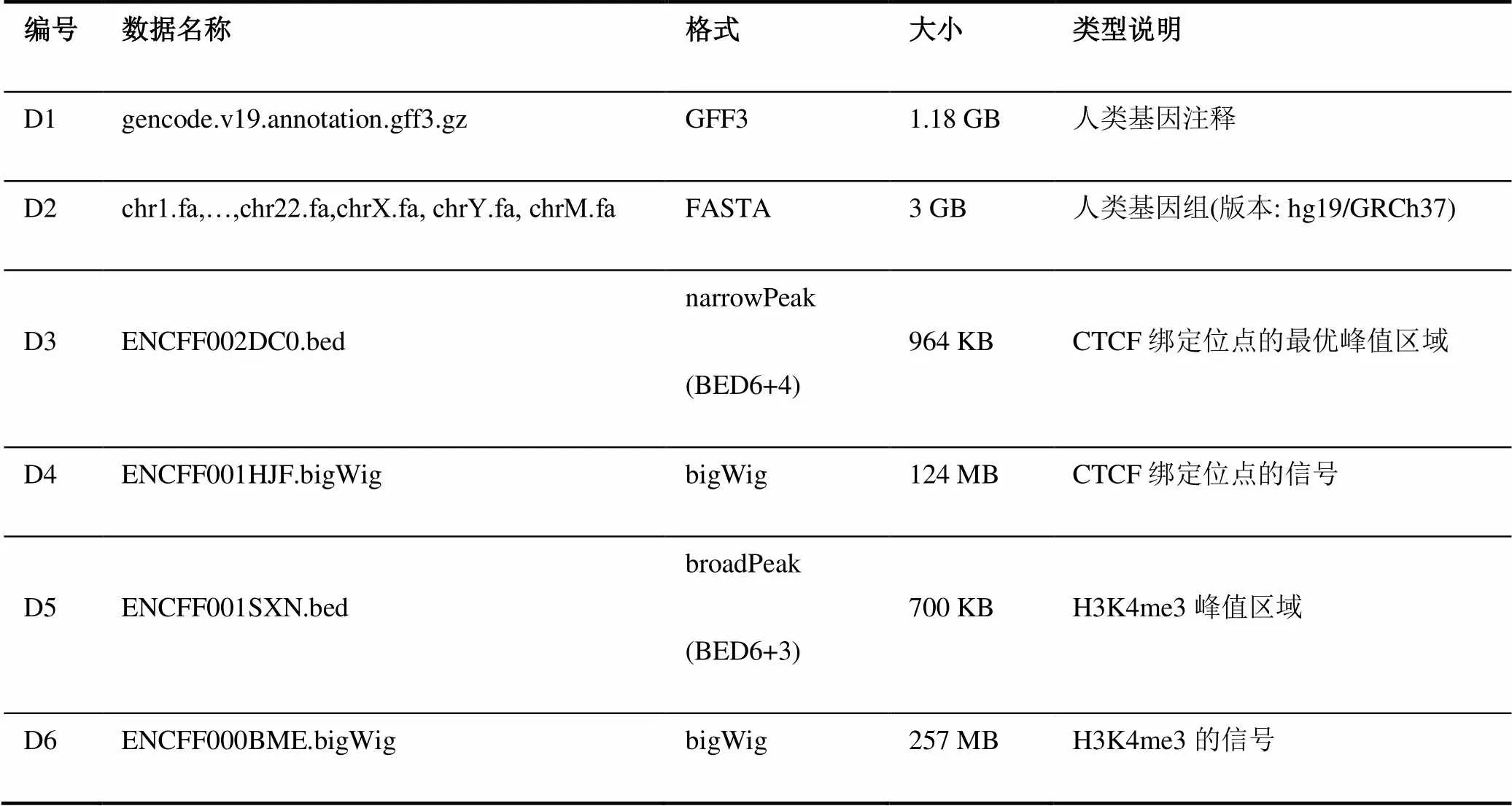
表1 多源lncRNA數(shù)據(jù)信息
2.3數(shù)據(jù)預(yù)處理
為了達(dá)到有效組織和整合lncRNA數(shù)據(jù)的目的, 須要對(duì)多源lncRNA數(shù)據(jù)進(jìn)行預(yù)處理, 本實(shí)驗(yàn)需要預(yù)處理的數(shù)據(jù)包括D1、D3、D5. 由于D1數(shù)據(jù)包含了人類編碼和非編碼基因的注釋信息, 因此通過(guò)腳本程序提取了其中l(wèi)ncRNA基因的注釋信息, 并命名為gencode.v19.lncRNAs.gff3(編號(hào): D1-1, 大小: 44M). 為了便于GBrowse處理, narrowPeak格式的D3和broadPeak格式的D5都轉(zhuǎn)換成了BED6格式, 并分別命名為D3-1和D5-1.
2.4 數(shù)據(jù)存儲(chǔ)
對(duì)于數(shù)據(jù)存儲(chǔ), GBrowse支持多種數(shù)據(jù)庫(kù)后端(backend), 如Berkeleydb、SQLite、MySQL、Chado、BioSQL等. 為了便于GBrowse快速顯示數(shù)據(jù), 根據(jù)已收集數(shù)據(jù)的類型和大小設(shè)計(jì)了如下的數(shù)據(jù)存儲(chǔ)方案: 由于D1數(shù)據(jù)(如表1所示)包含了lncRNA的基因位置、結(jié)構(gòu)、名稱、數(shù)據(jù)源等信息, 內(nèi)存訪問(wèn)比較緩慢, 因此為其建立了MySQL數(shù)據(jù)庫(kù)(名稱: “hg19”), 以提高D1-1的訪問(wèn)速率. 另外, 由于D2數(shù)據(jù)規(guī)模較大, 因此也將其導(dǎo)入“hg19”數(shù)據(jù)庫(kù). 其次, 建立了兩個(gè)SQLite數(shù)據(jù)庫(kù), 分別存儲(chǔ)D3-1和D5-1數(shù)據(jù). 對(duì)于二進(jìn)制格式的bigWig數(shù)據(jù), 由于可通過(guò)GBrowse中的Perl模塊bigWig.pm進(jìn)行讀取, 因此無(wú)需對(duì)D4和D6數(shù)據(jù)建庫(kù).
2.5 數(shù)據(jù)訪問(wèn)與可視化配置
數(shù)據(jù)存儲(chǔ)完成之后, 在GBrowse配置文件目錄下建立了用于配置數(shù)據(jù)訪問(wèn)和可視化方法的文件hg19.conf, 同時(shí)在GBrowse.conf文件末尾添加關(guān)于hg19.conf的段落(section). 通過(guò)設(shè)置hg19.conf中的參數(shù)對(duì)數(shù)據(jù)訪問(wèn)接口和可視化方法進(jìn)行配置(如表2所示), 以實(shí)現(xiàn)對(duì)已存儲(chǔ)數(shù)據(jù)的顯示, 并優(yōu)化數(shù)據(jù)的可視化效果. 表2中的訪問(wèn)接口是指與各數(shù)據(jù)相對(duì)應(yīng)的Perl適配模塊(adaptor). 不同數(shù)據(jù)要設(shè)置成合適的形狀才可以得到正確顯示, 而各數(shù)據(jù)軌道(Track)應(yīng)設(shè)置成容易區(qū)分和觀察的形狀和顏色. 參數(shù)說(shuō)明和配置方法可參考文檔: http://cloud.gmod.org/gbrowse2/tutorial/ tutorial.html.

表2 數(shù)據(jù)訪問(wèn)及可視化的主要配置參數(shù)
3 結(jié)果與分析
通過(guò)以上實(shí)現(xiàn)流程, 建立了一個(gè)人類lncRNA數(shù)據(jù)可視化的原型系統(tǒng) (名稱: HlncRNAdb-demo, 訪問(wèn): http://bioinf.yzu.edu.cn:40/cgi-bin/gb2/gbrowse/hg19/), 該系統(tǒng)為研究者提供了直觀的人類lncRNA數(shù)據(jù)可視化(如圖3和圖4所示).

圖3 chr18:77721985-78016680范圍內(nèi)的lncRNA數(shù)據(jù)顯示
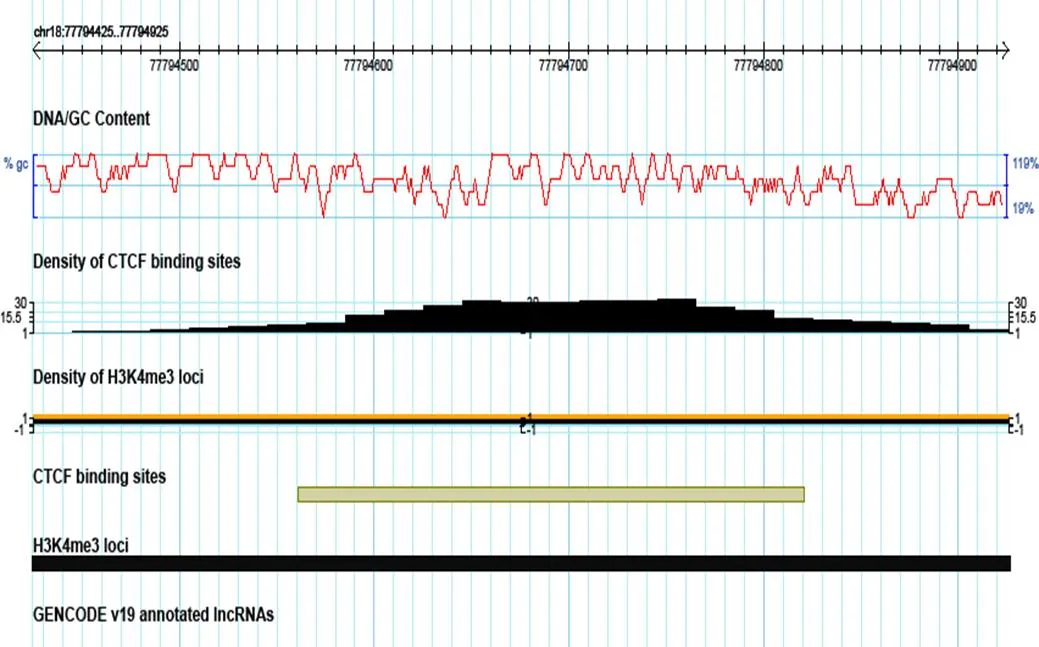
圖4 chr18:777994425-7794925范圍內(nèi)的lncRNA數(shù)據(jù)顯示
3.1 lncRNA數(shù)據(jù)的可視化
HlncRNAdb-demo通過(guò)GBrowse成功整合了包括人類lncRNA基因注釋(GENCODE v19 annotated lncRNAs)、人類基因組序列/GC含量(DNA/GC Content)、組蛋白修飾H3K4me3信號(hào)(Density of H3K4me3 loci)及其預(yù)測(cè)位點(diǎn)(H3K4me3 loci)、轉(zhuǎn)錄因CTCF綁定位點(diǎn)信號(hào)(Density of CTCF binding sites)及其預(yù)測(cè)位點(diǎn)(CTCF binding sites)在內(nèi)的多源lncRNA數(shù)據(jù), 數(shù)據(jù)格式包含GFF3、FASTA、BED6、bigWig等.
多源lncRNA數(shù)據(jù)在基因組空間得到整合, 并以各自的形狀和顏色加以顯示, 從而區(qū)別于其他軌跡. 圖3展示了在比例縮小(zoom out)情況下對(duì)基因組chr18:77721985-78016680(295 kilo base pairs/295kbp)范圍內(nèi)的整合數(shù)據(jù)進(jìn)行可視化的概況, 而圖4是將比例放大(zoom in)后對(duì)chr18:777994425-7794925(500bp)范圍內(nèi)的整合數(shù)據(jù)進(jìn)行顯示的概況. 圖中紅色波浪狀曲線顯示的是“DNA/GC Content”軌道, 紅色曲線實(shí)際上是將區(qū)域內(nèi)計(jì)算得到的序列GC含量通過(guò)圖形化顯示后的結(jié)果. 如果放大倍數(shù)足夠, 便能夠看到基因組序列的堿基構(gòu)成. H3K4me3信號(hào)的分布及預(yù)測(cè)的峰值區(qū)域分別如圖中的“Density of H3K4me3 loci”和“H3K4me3 loci”軌道所示. 類似地, CTCF綁定位點(diǎn)信號(hào)的分布及預(yù)測(cè)區(qū)域分別如圖中的“Density of CTCF binding sites”和“CTCF binding sites”所示. 其中, “H3K4me3 loci”和“CTCF binding sites”均采用graded_segments形狀進(jìn)行顯示, 其中的顏色灰度會(huì)根據(jù)原BED數(shù)據(jù)文件中的分值進(jìn)行顯示. 圖3中最下方的軌道“GENCODE v19 annotated lncRNAs”顯示的是GENCODE 發(fā)布的v19版的lncRNA的結(jié)構(gòu)和位置信息. 綜上, 研究者能夠在同一空間范圍內(nèi)對(duì)多源lncRNA數(shù)據(jù)進(jìn)行觀測(cè)和比較. 通過(guò)鼠標(biāo)拖放可選取觀察范圍, 或放大或縮小. 對(duì)于每個(gè)軌道中的標(biāo)記對(duì)象, 可通過(guò)鼠標(biāo)點(diǎn)擊獲取結(jié)構(gòu)化的詳細(xì)數(shù)據(jù)/信息表, 此表可幫助研究者查看目標(biāo)圖形的數(shù)據(jù)詳情. 另外, 在GBrowse界面中, 選定范圍內(nèi)各種數(shù)據(jù)的特征和相對(duì)關(guān)系一目了然, 起到了數(shù)據(jù)顯微鏡的作用.
3.2 可視化數(shù)據(jù)的分析
借助基于GBrowse的多源lncRNA數(shù)據(jù)可視化系統(tǒng), 研究者可在基因組空間中清晰地觀測(cè)多源lncRNA數(shù)據(jù), 這可以幫助驗(yàn)證已有的假說(shuō)、推論或建立新的科學(xué)假說(shuō)或模型. 如圖3所示, CTCF和H3K4me3 信號(hào)出現(xiàn)在lncRNA基因上游啟動(dòng)子附近, 說(shuō)明CTCF和H3K4me3可能與該基因的表達(dá)調(diào)控有關(guān)聯(lián), 此數(shù)據(jù)顯示反映出的特征與當(dāng)前流行的研究觀點(diǎn)保持了一致. 又如圖3, 可以觀測(cè)到許多CTCF信號(hào)的出現(xiàn)位點(diǎn)都會(huì)伴隨有H3K4me3信號(hào)的出現(xiàn), 而其中的本質(zhì)原因值得進(jìn)一步探討, 比如可以建立如下假說(shuō): CTCF能夠識(shí)別H3K4me3位點(diǎn), 然后綁定到H3K4me3區(qū)域, 進(jìn)而對(duì)基因產(chǎn)生調(diào)控作用. 當(dāng)然, 假說(shuō)的驗(yàn)證需要依據(jù)后期更多的實(shí)驗(yàn)和分析. 由此可見(jiàn), 基于GBrowse的多源lncRNA數(shù)據(jù)可視化系統(tǒng)能夠幫助研究者獲得更多的關(guān)于lncRNA的研究信息和思路.
4 結(jié)語(yǔ)
本文提出了基于GBrowse的多源lncRNA數(shù)據(jù)可視化系統(tǒng), 并介紹了系統(tǒng)的實(shí)現(xiàn)流程. 實(shí)驗(yàn)建立了人類lncRNA數(shù)據(jù)的可視化系統(tǒng)原型HlncRNAdb-demo. 實(shí)驗(yàn)結(jié)果表明該系統(tǒng)能夠?qū)崿F(xiàn)在同一基因組空間上對(duì)多源lncRNA數(shù)據(jù)進(jìn)行整合與可視化, 便于研究者從中獲取信息, 進(jìn)而助其進(jìn)行理論驗(yàn)證或建立新的科學(xué)假說(shuō). 對(duì)于本文的后續(xù)工作, 有如下計(jì)劃和建議: ①可根據(jù)研究需要收集和整合其它lncRNA數(shù)據(jù), 以增加lncRNA功能研究的信息; ②采用新的方法以提高數(shù)據(jù)訪問(wèn)的速率, 比如可采用FastCGI; ③在網(wǎng)頁(yè)中添加對(duì)lncRNA二級(jí)結(jié)構(gòu)進(jìn)行可視化的功能, 能在二維或三維空間整合和觀測(cè)lncRNA數(shù)據(jù). 綜上, 多源lncRNA數(shù)據(jù)在GBrowse框架下得到有效整合與可視化, 能夠推動(dòng)lncRNA功能研究的發(fā)展.
1 Loewer S, Cabili MN, Guttman M, Loh YH, Thomas K, Park IH, Garber M, Curran M, Onder T, Agarwal S, Manos PD, Datta S, Lander ES, Schlaeger TM, Daley GQ, Rinn JL. Large intergenic non-coding RNA-RoR modulates reprogramming of human induced pluripotent stem cells. Nature Genetics, 2010, 42(12): 1113–1117.
2 Hung T, Wang Y, Lin MF, Koegel AK, Kotake Y, Grant GD, Horlings HM, Shah N, Umbricht C, Wang P, Wang Y, Kong B, Langerod A, Borresen-Dale AL, Kim SK, van de Vijver M, Sukumar S, Whitfield ML, Kellis M, Xiong Y, Wong DJ, Chang HY. Extensive and coordinated transcription of noncoding RNAs within cell-cycle promoters. Nat Genet, 2011, 43(7): 621–629.
3 Kent WJ, Sugnet CW, Furey TS, Roskin KM, Pringle TH, Zahler AM, Haussler D. The human genome browser at UCSC. Genome Research, 2002, 12(6): 996–1006.
4 Stein LD, Mungall C, Shu S, Caudy M, Mangone M, Day A, Nickerson E, Stajich JE, Harris TW, Arva A, Lewis S. The generic genome browser: A building block for a model organism system database. Genome Research, 2002, 12(10): 1599–1610.
5 Skinner ME, Uzilov AV, Stein LD, Mungall CJ, Holmes IH. JBrowse: A next-generation genome browser. Genome Research, 2009, 19(9): 1630–1638.
6 Jin J, Liu J, Wang H, Wong L, Chua NH. PLncDB: Plant long non-coding RNA database. Bioinformatics, 2013, 29(8): 1068–1071.
7 Li A, Zhang J, Zhou Z, Wang L, Liu Y, Liu Y. ALDB: A domestic-animal long noncoding RNA database. PLoS ONE, 2015, 10(4): e0124003.
8 Descrimes M, Zouari YB, Wery M, Legendre R, Gautheret D, Morillon A. VING: A software for visualization of deep sequencing signals. BMC Research Notes, 2015, 8: 419.
9 Cumbie JS, Ivanchenko MG, Megraw M. NanoCAGE-XL and CapFilter: An approach to genome wide identification of high confidence transcription start sites. BMC Genomics, 2015, 16(1): 597.
10 Harrow J, Frankish A, Gonzalez JM, Tapanari E, Diekhans M, Kokocinski F, Aken BL, Barrell D, Zadissa A, Searle S. GENCODE: The reference human genome annotation for the ENCODE project. Genome Research, 2012, 22(9): 1760–1774.
Visualization System of Multi-Source Long Non-Coding RNA Data Based on GBrowse
SUN Lei, CHEN Xuan, TANG Hong, WEI Li-Ting, JI Lan-Yang, SHI Sheng-Fei, YANG Xiao-Hua
(School of Information Engineering, Yangzhou University, Yangzhou 225127, China)
In consideration of the problem that useful information cannot be easily extracted from various types of long noncoding RNA (lncRNA) data, this paper proposes a visualization system of multi-source lncRNA data based on generic genome browser (GBrowse). The system mainly includes a web server including HTTP service and GBrowse components, and lncRNA data storage which supports flat files, MySQL, SQLite and other types of databases. The main steps of constructing the system include GBrowse installation and configuration, multi-source lncRNA data collection, preprocessing, storage, and access and visualization configuration. A demo system is constructed by firstly collecting six sets of human lncRNA data, including human gene annotation, genome sequence, histone modification H3K4me3 signals and their loci predicted, signals of transcription factor CTCF binding sites and their loci predicted. After preprocessing, these data are stored by databases such as MySQL, SQLite and so on, and data access and visualization methods are also configured. The experiment results demonstrate that multi-source lncRNA data can be integrated and visualized within the GBrowse framework, and be showed in the genome spatial space simultaneously, which can make researchers observe the lncRNA data more intuitively, thereby helps to produce novel scientific hypothesis.
long non-coding RNA; genome browser; database; visualization
國(guó)家自然科學(xué)基金(61301220);揚(yáng)州大學(xué)大學(xué)生學(xué)術(shù)科技創(chuàng)新基金(x2015423, x2015444)
2016-06-23;
2016-07-25
[10.15888/j.cnki.csa.005633]

