木薯PHOR1基因的序列分析及其乙烯和茉莉酸甲酯誘導表達特性
叢漢卿+齊堯堯+朱文麗
摘要:為研究木薯中PHOR1基因如何被逆境信號調控,根據楊樹2個PHOR1同源基因搜索木薯基因組數據庫,得到2個高度同源的木薯PHOR1基因,即MePHOR1_1和MePHOR1_2。序列分析表明,它們都具有E3泛素連接酶家族成員特征:含有U-box和armadillo重復序列。此外其啟動子區具有乙烯響應元件和茉莉酸響應元件。以木薯品種華南八號懸浮培養細胞為材料,利用qRT-PCR檢測MePHOR1_1和MePHOR1_2基因在乙烯利和茉莉酸甲酯處理后的表達特性。結果顯示:在乙烯和茉莉酸甲酯分別處理后的細胞懸浮液中,2個PHOR1基因的表達變化呈現相似趨勢,即乙烯處理后,2個基因的表達均出現先下降后緩慢上升的趨勢;而對茉莉酸信號則呈現先上升后下降又上升再下降的波動性趨勢,故推測2個PHOR1基因可被乙烯和茉莉酸信號調控,并可在后續的根生長和淀粉積累等一系列生理生化過程中發揮作用。
關鍵詞:木薯;PHOR1基因;乙烯;茉莉酸;表達分析
中圖分類號:S533.01 文獻標志碼:A
文章編號:1002-1302(2016)11-0029-05
木薯(Manihot esculenta Crantz)屬于大戟科(Euphorbiaceae)木薯屬(Manihot P.Miller),原產于熱帶南美洲,是世界三大薯類作物之一,是第六大糧食作物,是重要的能源和淀粉作物。木薯的根作為其主要經濟部位,研究其生長發育和淀粉積累具有重要意義。
Amador等在研究馬鈴薯(Solanum tuberosum)短日照條件下在上調表達的調控因子中首次獲得了PHOR1基因,并通過煙草轉基因試驗證明PHOR1對GA信號途徑具有正調控作用[1]。PHOR1編碼1個Cys-Pro-Ile motif(CPI)結構域和7個armadillo(ARM)重復序列的蛋白[2]。此CPI結構域具有典型的U-box序列特征,U-box結構一般存在于E3泛素連接酶中[3],可促進特異目標蛋白的泛素化[4],缺失試驗表明,CPI是一種可被GA抑制的細胞質內滯留信號。而ARM重復序列是PHOR1的核定位信號,其核定位功能可為CPI所逆轉。目前,對PHOR1基因的研究已有一定基礎,但在木薯中的表達特性和基因功能還未被研究。
茉莉酸(jasmonic acid,簡稱JA)和乙烯(ethylene,簡稱ET)是植物逆境響應的信號分子,在涉及植物對生物性和非生物性逆境脅迫和植物生長發育過程中的基因調控上發揮著重要作用。JA作為一種逆境脅迫轉導信號,具有誘導植物防御基因的表達、調控植物對機械傷害、鹽害及紫外線照射等非生物脅迫的反應的功能[5-6]。而ET是另外一種植物生長調節物質,可影響植物多個生長發育過程,對植物自身來說,乙烯在種子萌發、開花、葉片伸展、根的生長、果實成熟及器官衰老等方面發揮著作用;對植物與外界環境的關系來說,乙烯主要參與植物面對外界生物與非生物脅迫的反應過程。很多涉及上述相關過程的基因序列上都有乙烯響應元件[7-8]。
楊樹中的PHOR1同源基因被發現具有調控芽和根生長、淀粉積累、木質部形成的功能[9]。根據楊樹PHOR1同源基因PtPHOR1_1(POPTR_0007s03730)和PtPHOR1_2(POPTR_0005s05880)的序列在JGI的木薯基因組數據庫 v6.1[10]中搜索獲得2個高度同源序列:Manes.12G152200和Manes.13G071800,分別命名為MePHOR1_1和MePHOR1_2。生物信息學分析發現,其具有典型的U-box結構和ARM重復序列,且MePHOR1_1啟動子區具有1個乙烯響應元件和1個茉莉酸響應元件,MePHOR1_2具有2個茉莉酸響應元件。同時利用木薯懸浮培養細胞對其ET和JA甲酯誘導表達特性分析中也發現,PHOR1基因可受ET和JA信號調控,并呈現不同的表達趨勢。本研究旨在通過序列分析和表達分析,確定ET和JA信號是否能對PHOR1基因表達造成影響,并初步探索表達變化特性及其機制,為進一步研究逆境信號通過PHOR1基因在木薯塊根發育和淀粉積累過程中所發揮的作用提供參考。
1 材料與方法
1.1 材料與試劑
木薯(Manihot esculenta Crantz)品種華南八號(SC8)懸浮培養細胞,該細胞通過葉片愈傷組織建立,保存于中國熱帶農業科學院熱帶作物品種資源研究所。Axygen總RNA小量制備試劑盒(Axygen);PrimeScriptTM Reverse Transcriptase反轉錄試劑盒(TaKaRa);SYBR Premix Ex TaqTM Ⅱ(Pefect Real Time)實時定量試劑盒(TaKaRa);引物由Invitrogen(上海)貿易有限公司合成。
1.2 方法
1.2.1 材料處理
吸取處于指數增長期的懸浮培養細胞 1 mL 于離心管中并置于搖床25 ℃穩定0.5 h,分為3組,其中2組分別以濃度為400 μL/L的乙烯利(ethephon)(Solarbio)、茉莉酸甲酯(methyl jasmonate,簡稱MeJA)(Sigma-Aldrich)處理,另一組不進行任何處理作為對照。選取處理后0.5、1.0、2.0、4.0、8.0 h共5個時間點的樣品,微離心后棄上清液氮速凍-80 ℃冰箱保存。此步驟重復3次,獲得3批生物學重復。
1.2.2 PHOR1基因及啟動子區序列分析
使用JGI的木薯基因組數據庫(http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Mesculenta_er)獲得MePHOR1_1和MePHOR1_2的CDS序列(coding sequence)和DNA序列(genomic sequence)[10]。用NetGene2 V2.4(http://www.cbs.dtu.dk/services/NetGene2/)分析基因結構[11-12]。用GSDS(http://gsds.cbi.pku.edu.cn)分析內含子相位[13]。用TSSP(http://linux1.softberry.com/berry.phtml?topic=tssp&group=programs&subgroup=promoter)分析轉錄起始位點[14],用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析啟動子區序列,尋找順勢作用元件位點[15]。
1.2.3 PHOR1蛋白分析
使用Blast工具對預測的氨基酸序列進行比對分析,用PortParam(http://web.expasy.org/protparam/)預測其蛋白分子量和等電點[16],使用InterPro(http://www.ebi.ac.uk/interpro/)預測結構域[17]。利用MEME(http://meme-suite.org/tools/meme)[18]和MAST(http://meme-suite.org/tools/mast)[19]分析木薯PHOR1蛋白的基序,基序數目設為10,E-values為默認的1.0。用TMHMM Server在線工具(http://www.cbs.dtu.dk/services/TMHMM/)進行跨膜區預測[20],使用SubLoc在線工具(http://www.bioinfo.tsinghua.edu.cn/SubLoc/)進行蛋白亞細胞定位預測[21]。使用PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)預測二級結構[22]。利用Swiss-model(http://swissmodel.expasy.org/)預測三維結構[23]。使用PFP(http://kiharalab.org/web/pfp.php)預測蛋白功能[24]。
1.2.4 序列比對和進化樹分析
鑒于目前只有少數作物對PHOR1進行了研究,目前我們只找到了除木薯外的已報道的6種植物中的PHOR1基因。來自木薯的2個PHOR1基因MePHOR1_1和MePHOR1_2、楊樹(Populus trichocarpa)的PHOR1基因PtPHOR1_1(POPTR_0007s03730)和PtPHOR1_2(POPTR_0005s05880)[9]、馬鈴薯(Solanum tuberosum)StPHOR1(AJ306423)[1]、番茄(Lycopersicon esculentum)的LePHOR1(Solyc04g071030)[25]、擬南芥(Arabidopsis thaliana)HIM1/HIM2/HIM3[3]、龍眼(Dimocarpus longan L.)DlPHOR1[26]、白羽扇豆(Lupinus albus)中具有完整CDS的3條PHOR1同源contigs(LAGI01_34089、LAGI01_33328、LAGI01_27558)[27]。使用MEGA 6對其氨基酸進行序列比對,并構建鄰接(neighbor-joining,N-J)聚類進化樹。使用bootstrap方法1 000次重復對進化樹進行驗證。
1.2.5 PHOR1誘導表達的qRT-PCR檢測
提取RNA后進行cDNA反轉錄,利用qRT-PCR檢測木薯PHOR1基因在懸浮培養細胞中受乙烯利和茉莉酸甲酯誘導表達情況,所有的qRT-PCR反應均來自同一批反轉錄的cDNA產物,18S rRNA為內參。所用PHOR1基因及18S rRNA的引物為Primer premier 5.0軟件設計(表1)。
2 結果與分析
2.1 基因結構分析
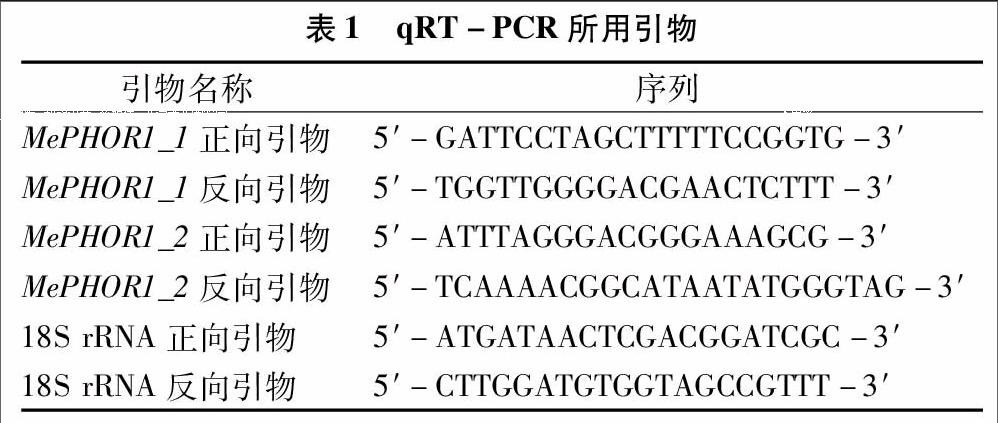
基因結構分析發現,MePHOR1_1和MePHOR1_2基因僅有1個外顯子,都沒有內含子。轉錄起始位點預測顯示:MePHOR1_1基因的轉錄起始位點位于起始密碼子上游 857 bp 處,而MePHOR1_2基因的轉錄起始位點位于起始密碼子上游1 282 bp處。順勢作用元件位點分析發現:MePHOR1_1基因啟動子區域包含典型啟動子調控元件 CAAT-box 和 TATA-box及多個光響應順式元件、1個乙烯響應元件和1個茉莉酸響應元件;此外還包含真菌誘導子響應元件、MYB結合位點以及參與細胞周期和胚乳表達響應元件。而MePHOR1_2除具有典型啟動子調控元件CAAT-box和 TATA-box外,也存在多個光響應順式元件和2個茉莉酸響應元件,但不具有乙烯響應元件;另外還包含厭氧誘導的響應元件、真菌誘導子響應元件、MYB結合位點以及生長素響應元件(表2)。
2.2 蛋白分析
MePHOR1_1有418個氨基酸,分子量為46 610.3 u,理論等電點為8.28。MePHOR1_2有418個氨基酸,分子量為 46 795.6 u,理論等電點為7.47。跨膜區分析顯示此蛋白可能無跨膜結構。亞細胞定位預測表明,這2個蛋白有可能定位在核上。二者具有相同基序。符合E-value<1的基序有6個,二級結構預測MePHOR1_1和MePHOR1_2有22個螺旋結構,可參考其三維預測結構(圖1)。PFP蛋白功能預測顯示:MePHOR1_1和MePHOR1_2基因最符合Gene Ontology(GO)中的GO:0005515條目,具有蛋白結合功能。
2.3 PHOR1基因進化樹分析
從進化樹可以看出,同一科植物的PHOR1基因基本聚為一類,木薯作為大戟科植物,2個PHOR1處于一個單獨分支上,且在親緣關系上較為接近楊樹和龍眼(圖2);楊樹與龍眼作為木本植物處于同一分支,且龍眼的DlPHOR1與楊樹PtPHOR1_1的親緣關系要近于楊樹自身的PtPHOR1_2;此外擬南芥的3個基因也處于同一分支;馬鈴薯和番茄同為茄科植物,處于同一分支;與此親緣關系較遠的白羽扇豆位于一個距離較遠的獨立分支上。
2.4 茉莉酸甲酯和乙烯誘導木薯PHOR1表達水平變化
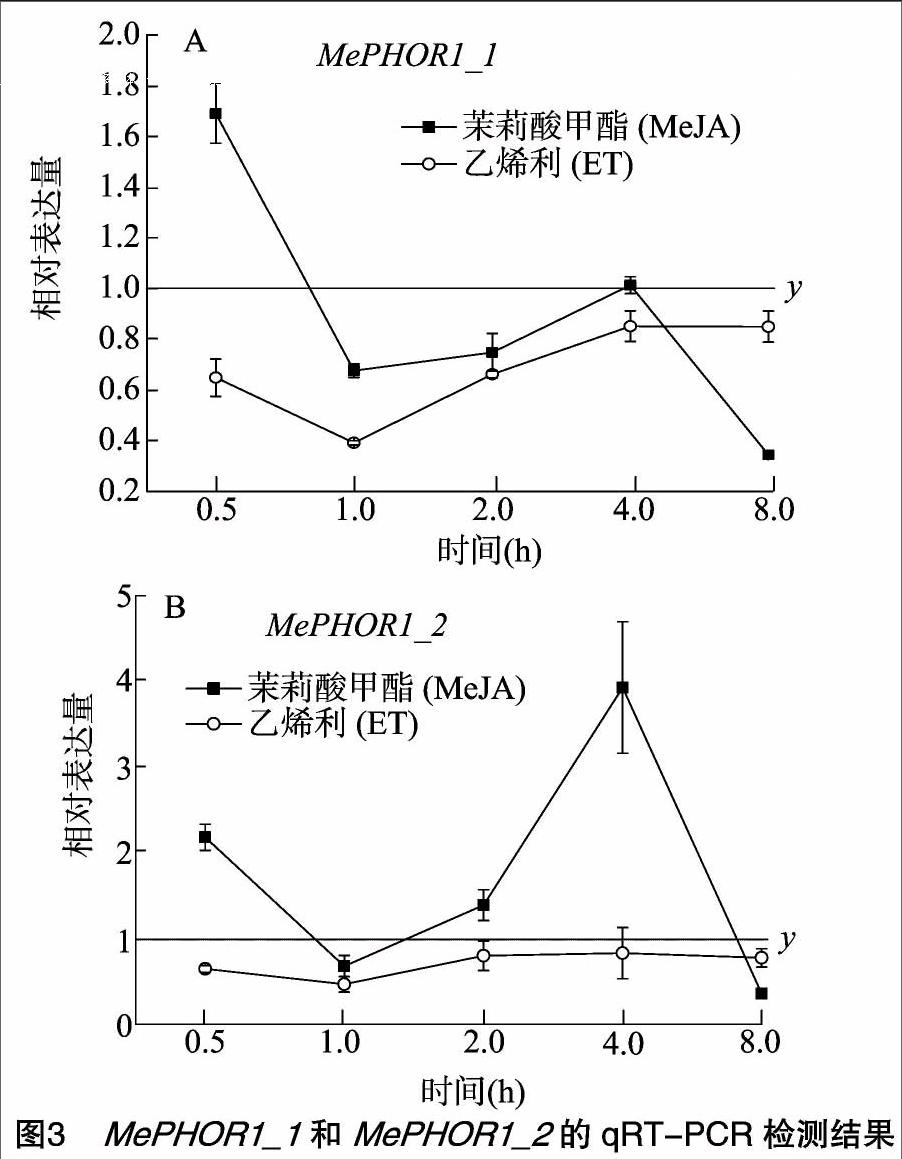
為排除PHOR1基因本底變化造成的影響,將茉莉酸甲酯和乙烯誘導后的qRT-PCR表達量值除以相應對照的表達量值,值1.0為本底表達水平。最終獲得其在2種處理下的表達趨勢,結果顯示兩者呈現不同趨勢:MePHOR1_1在茉莉酸甲酯的響應整體呈現波浪狀,處理0.5 h后表達量提升至1.7左右,又在1 h快速下降至0.7水平,后在4 h時,逐漸恢復到初始水平,但又在8 h出現劇烈下降至最低水平。而乙烯信號對其的影響呈現先在1 h下降至最低點后又逐漸上升的趨勢(圖3-A)。茉莉酸甲酯誘導后,MePHOR1_2的趨勢與MePHOR1_1較為類似,也呈現波浪狀,但表達水平有較大差異,處理0.5 h后表達量提升至2.0以上,又在 1 h 快速下降至初始水平以下,但在4 h時提升到4.0,后又在8 h出現劇烈下降。而乙烯信號對其沒有非常明顯的影響,趨勢線較為平直得維持在略低于初始水平的狀態下(圖3-B)。
3 結論與討論
植物懸浮培養細胞是分子生物學實驗中研究信號傳遞和基因響應的常用方法。加入外源信號物質刺激后,信號物質可快速、均勻地分布在每個細胞周圍,操作簡易,為試驗結果的一致性和重復性提供了保障。本研究選用的懸浮培養細胞大小接近、生長迅速且生長周期基本一致。本試驗中,PHOR1基因在逆境信號物質施加后0.5 h內即作出了響應,并且在整個過程中表達量最高可達初始水平的4倍,表明本試驗所選用的材料適用于基因表達分析研究。
如同楊樹PtPHOR1[9],木薯PHOR1基因也是一類較為保守的基因,MePHOR1_1和MePHOR1_2兩者具有很高的序列相似性。在蛋白結構上具有和已知的植物PHOR1基因類似的U-box結構和ARM重復序列。進化樹分析中,與楊樹的PtPHOR1和龍眼的DlPHOR1基因具有較近的親緣關系,可以推斷MePHOR1是一類典型的PHOR1基因。同時啟動子區分析發現MePHOR1具有乙烯響應因子ERE及茉莉酸響應元件CGTCA-motif和TGACG-motif,初步解釋了乙烯和茉莉酸信號可影響PHOR1基因的表達。盡管MePHOR1_1有1個乙烯響應元件,但乙烯響應的變化趨勢卻是下調的,可能有其他調控機制抑制了乙烯信號的順勢調控,具體原因還須進一步探索。MePHOR1_2沒有乙烯響應元件,其ET處理后的變化曲線盡管略低于本底水平1.0,但趨勢較為平緩,從一個角度解釋了MePHOR1_2為何對乙烯信號不敏感。此外,MePHOR1_2在0.5 h和4.0 h對JA響應的表達量要明顯高于MePHOR1_1,推測與其具有2個茉莉酸響應元件有一定關系。
在乙烯和茉莉酸處理后0.5 h內,PHOR1呈現出不同的響應趨勢,ET導致下調而JA導致上調,表明雖同為逆境信號物質,卻對PHOR1具有不同的調控作用。植物中茉莉酸和乙烯與其他信號轉導通路間的激素的交叉相互作用在整個生活周期內、不同組織器官間、不同類型細胞中一直存在。不同激素信號的整合共同介導了植物的生長發育、對環境的反應及衰老死亡。此外,2個PHOR1基因的啟動子區還有各自獨有的調控元件,如生長素響應、MYB結合位點等。故此推測,2種不同逆境信號施加后與其他不同激素的綜合作用,產生了迥異的調控模式,最終造成PHOR1基因不同的表達特性。
楊樹中PHOR1基因受到抑制后,其莖部和根部的淀粉積累出現了上升[9],馬鈴薯中,PHOR1的下調可以促進塊莖的生長[1]。PHOR1具有抑制根和芽生長的功能,眾所周知,當不利環境來臨時,植物開始大量合成貯藏營養物質,并伴隨著生長的停滯[28]。因此,PHOR1是一種當不利環境來臨時植物將碳水化合物從用于生長轉為用于貯藏的調控機制的一部分。PHOR1調控淀粉代謝的方式可能與GA信號途徑有關[29]。PHOR1基因可能在根的發育過程中起一定的控制作用,楊樹PHOR1基因的過量表達會顯著減少生根[9]。有研究表明,GA會嚴重影響生長素的運輸[30-32]。因此,PHOR1作為GA信號的一部分,可能會干擾生長素的運輸和其感知定位生長素濃度信息的能力,這對根的形成至關重要。總之,PHOR1基因即可受光誘導,又可與GA信號互相影響,說明其是鏈接光信號途徑和GA途徑的一個橋梁[1]。由此推測:隨著季節更替,日照時間發生變化,這一光信號將影響到植物PHOR1基因的表達,進而通過調節GA影響植株芽和根的生長,并促進淀粉積累,以迎接即將到來的秋冬季節的低溫不利環境。
木薯作為重要的糧食作物及工業乙醇和淀粉來源[33-34],根作為其主要經濟部位,其生長發育和淀粉的積累具有重要的科研意義和經濟價值,PHOR1基因作為可以調控此二者的重要基因,研究逆境信號如何對其造成影響并探索相關機制,可以進一步為木薯產量的提高和質量的提升奠定理論基礎。目前我們已知木薯的PHOR1基因可受到乙烯和茉莉酸信號影響,并初步確定其表達特性,但具體的信號轉導路徑及此蛋白的具體功能,還需要進一步驗證和探索。
參考文獻:
[1]Amador V,Monte E,Martínez-García J L,et al. Gibberellins signal nuclear import of PHOR1,a photoperiod-responsive protein with homology to Drosophila armadillo[J]. Cell,2001,106(3):343-354.
[2]Olszewski N,Sun T P,Gubler F. Gibberellin signaling:biosynthesis,catabolism,and response pathways[J]. Plant Cell,2002,14:S61-S80.
[3]Monte E,Amador V,Russo E,et al. PHOR1,a U-Box GA signaling component with a role in proteasome degradation? [J]. Journal of Plant Growth Regulation,2003,22:152-162.
[4]Pringa E,Martinez-Noel G,Muller U,et al. Interaction of the RING finger-related U-box motif of a nuclear dot protein with ubiquitin-conjugating enzymes[J]. Journal of Biological Chemistry,2001,276:19617-19623.
[5]Conconi A,Smerdon M J,Howe G A,et al. The octadecanoid signalling pathway in plants mediates a response to ultraviolet radiation[J]. Nature,1991,383(6603):826-829.
[6]Turner J G,Ellis C,Devoto A. The jasmonate signal pathway[J]. Plant Cell,2002,14(Suppl1):S153-S164.
[7]Bleecker B,Kende H. Ethylene:a gaseous signal molecule in plants[J]. Annual Review of Cell and Developmental Biology,2000,16:1-18.
[8]Johnson P R,Ecker J R. The ethylene gas signal transduction pathway:a molecular perspective[J]. Annual Review of Genetics,1998,32:227-254.
[9]Zawaski C,Ma C,Strauss S H,et al. PHOTOPERIOD RESPONSE 1 (PHOR1) -like genes regulate shoot/root growth,starch accumulation,and wood formation in Populus[J]. Journal of Experimental Botany,2012,63(15):5623-5634.
[10]Prochnik S,Marri P R,Desany B,et al. The cassava genome:current progress,future directions[J]. Tropical Plant Biology,2012,5:88-94.
[11]Brunak S,Engelbrecht J,Knudsen S. Prediction of human mRNA donor and acceptor sites from the DNA sequence[J]. Journal of Molecular Biology,1991,220:49-65.
[12]Hebsgaard S M,Korning P G,Tolstrup N,et al. Splice site prediction in Arabidopsis thaliana pre-mRNA by combining local and global sequence information[J]. Nucleic Acids Research,2010,24(17):3439-3452.
[13]Hu B,Jin J P,Guo A Y,et al. GSDS 2.0:an upgraded gene feature visualization server[J]. Bioinformatics,2015,31(8):1296-1297.
[14]Solovyev V V,Shahmuradov I A. PromH:Promoters identification using orthologous genomic sequences[J]. Nucleic Acids Research,2003,31(13):3540-3545.
[15]Lescot M,Déhais P,Moreau Y,et al. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.
[16]Gasteiger E,Gattiker A,Hoogland C,et al. ExPASy:the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research,2003,31(13):3784-3788.
[17]Hunter S,Apweiler R,Attwood T K,et al. InterPro:the integrative protein signature database[J]. Nucleic Acids Research,2009,37(Database issue):D211-D215.
[18]Bailey T L,Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J]. Intell Syst Mol Biol,1994,2:28-36.
[19]Bailey T L,Gribskov M. Combining evidence using p-values:application to sequence homology searches[J]. Bioinformatics,1998,14(1):48-54.
[20]Krogh A,Larsson B,Von Heijne G,et al. Predicting transmembrane protein topology with a hidden Markov model:application to complete genomes[J]. Journal of Molecular Biology,2001,305:567-580.
[21]Hua S J,Sun Z R. Support vector machine approach for protein subcellular localization prediction[J]. Bioinformatics,2015,17(8):721-728.
[22]Jones D T. Protein secondary structure prediction based on position-specific scoring matrices[J]. Journal of Molecular Biology,1999,292:195-202.
[23]Arnold K,Bordoli L,Kopp J,et al. The SWISS-MODEL Workspace:a web-based environment for protein structure homology modelling[J]. Bioinformatics,2006,22:195-201.
[24]Khan I K,Wei Q,Chitale M,et al. PFP/ESG:automated protein function prediction servers enhanced with Gene Ontology visualization tool[J]. Bioinformatics,2015,31(2):271-272.
[25]Martinelli F,Uratsu S L,Reagan R L,et al. Gene regulation in parthenocarpic tomato fruit[J]. Journal of Experimental Botany,2009,60:3873-3890.
[26]Jia T,Wei D,Meng S,et al. Identification of regulatory genes implicated in continuous flowering of longan (Dimocarpus longan L.) [J]. PLoS One,2014,9(12):e114568.[HJ1.76mm]
[27]ORourke J A,Yang S S,Miller S S,et al. An RNA-Seq transcriptome analysis of orthophosphate-deficient white lupin reveals novel insights into phosphorus acclimation in plants[J]. Plant Physiology,2013,161:705-724.
[28]Hermans C,Hammond J P,White P J,et al. How do plants respond to nutrient shortage by biomass allocation? [J]. Trends in Plant Science,2006,11:610-617.
[29]Thomas S G,Sun T P. Update on gibberellin signaling:a tale of the tall and the short[J]. Plant Physiology,2004,135:668-676.
[30]Bjorklund S,Antti H,Uddestrand I,et al. Cross-talk between gibberellin and auxin in development of Populus wood:gibberellin stimulates polar auxin transport and has a common transcriptome with auxin[J]. The Plant Journal,2007,52:499-511.
[31]Gou J Q,Strauss S H,Tsai C J,et al. Gibberellins regulate lateral root formation in Populus through interactions with auxin and other hormones[J]. Plant Cell,2010,22:623-39.
[32]Mauriat M,Sandberg L G,Moritz T. Proper gibberellin localization in vascular tissue is required to control auxin-dependent leaf development and bud outgrowth in hybrid aspen[J]. The Plant Journal,2011,67:805-816.
[33]曾文丹,羅興錄. 2個淀粉含量不同木薯品種SS[QX(Y15]Ⅱ[QX)]基因序列及不同生育期淀粉含量比較[J]. [JP3]江蘇農業科學,2015,43(5):35-38.
[34]安飛飛,簡純平,楊搖龍,等. 木薯幼苗葉綠素含量及光合特性對鹽脅迫的響應[J]. 江蘇農業學報,2015,31(3):500-504.

