基于質譜分析的代謝組學研究進展
任向楠,梁瓊麟
(清華大學 化學系,清華大學分析中心,北京 100084)
基于質譜分析的代謝組學研究進展
任向楠,梁瓊麟*
(清華大學 化學系,清華大學分析中心,北京 100084)
質譜分析技術是代謝組學研究的重要技術之一。該文通過近5年來的文獻分析,對基于質譜分析的代謝組學研究方法的新進展,包括樣品前處理方法、分析檢測方法、數據處理方法等,以及近年來代謝組學在疾病診斷、藥物研發、營養學、毒理學、運動醫學等領域的應用進展,進行了較全面的綜述,并對未來的發展趨勢進行了展望。
質譜分析;代謝組學;研究進展;綜述
代謝組學(Metabolomics,Metabonomics)是指生物體系因生物刺激、病理生理擾動或遺傳信息改變等引起的總體、動態的代謝變化[1]。代謝組學作為系統生物學最下游的“組學”,是整體性研究生命體系功能變化的重要學科分支。代謝組學的研究對象為復雜的生物體系,種類非常多,常見的生物樣本包括生物體液(尿液、血清、血漿、唾液、膽汁、腦脊液、羊水、眼淚、胰液、腸液等)、動物或人體組織(腫瘤、肝臟、腦、脂肪組織等)和細胞[2]。代謝組學常用的研究手段包括質譜和核磁兩大類,本文重點綜述了2010年以來基于質譜(MS)分析的代謝組學在醫學領域的研究進展,主要包括疾病診斷、藥物研發、營養學、毒理學、運動醫學等方面。
1 代謝組學分析方法的新進展
代謝組學又分為靶向性代謝組學(Targeted metabolomics)和非靶向性代謝組學(Untargeted metabolomic)。靶向性代謝分析主要是定量特定的代謝物,典型的是關注一種或幾種相關的代謝通路。常用于研究藥物代謝的藥代動力學,衡量某種療法或基因修飾的效果。靶向性代謝分析的流程是代謝物標準品的液相色譜-質譜聯用分析(LC-MS),選擇性離子監測,分析方法優化和標準曲線制作,樣品前處理,樣品提取物LC-MS分析,通過對比樣品和標準品進行數據分析,定量特定的代謝物[3-6]。非靶向性代謝組學范圍寬泛,目標是從生物樣本中同時分析盡可能多的代謝物。而非靶向性代謝組學的步驟是樣品前處理,代謝物提取,LC-MS全掃描檢測,數據預處理,統計分析及差異代謝物結構鑒定[3-4,7-9]。
1.1 樣品前處理
樣品前處理直接關系到信息獲取的準確性,非靶向性代謝組學的樣品前處理要求盡量不破壞代謝組分,以獲得盡可能全的代謝信息。靶向性代謝組學則更關注提取的效率,用回收率、精密度、準確性等表征。本實驗室用甲醇-乙腈(1∶1)去除血漿中的蛋白,建立了大鼠血漿的高效液相色譜-時間飛行質譜聯用(HPLC-TOF-MS)的分析方法,同時定量了大鼠服用知母后的21種藥物代謝物,用精確質量測定了代謝物的基本組成并確認其結構,其中包括同分異構體的代謝物[10]。成玉等[11]優化了去除血清蛋白的方法,發現甲醇-乙腈混合除蛋白檢測的離子峰多于純甲醇和純乙腈,這與本實驗室的研究結果相一致,另外一方面通過對比方法的重現性,發現甲醇-乙腈(1∶9)去除血清中的蛋白優于甲醇-乙腈(1∶1)。Want 等[12]和Sarafian 等[13]處理膽汁酸的樣本時,均采用甲醇除蛋白法。非靶向分析實體組織代謝物研究方面,多篇文獻均采用甲醇-水(1∶1)提取水溶性代謝物,二氯甲烷-甲醇(3∶1)提取有機類代謝物[14-18]。在液質聯用(LC-MS)分析糞便代謝物方面,Su 等[19]處理糞便樣本時,先用水提取,再用相同體積的乙腈提取,合并上清液、吹干,用同位素丹磺酰氯衍生化,數據結合HMDB數據庫,鑒定出3位女性7 d糞便中的39種代謝物;Tian 等[20]則采用甲醇渦旋提取處理糞便樣本,用液質聯用分析出對照組和給藥組的16個代謝標志物。氣相色譜-質譜聯用(GC-MS)能直接從標準譜庫中比對化合物信息,易于定性,但其不適用于難揮發和高極性代謝物分析,前處理需衍生化,相對處理復雜,常用的衍生化方法有硅烷化、酰化、烷基化[21-22]。Sulek 等[23]用氣質聯用分析頭發代謝物時,分別用氫氧化鈉水解、酸中和、冷凍干燥、甲醇水混合物再提、蒸干、氯甲酸甲酯衍生、棄去水層的步驟處理頭發樣本。Tsutsui 等[24]用液質聯用分析頭發代謝物時,用甲醇鹽酸混合物超聲提取頭發樣本,前處理方面較氣質聯用簡單。本實驗室建立了從細胞中提取線粒體及其樣本制備的方法,并將代謝組學的應用推廣到亞細胞層面[25]。各類樣本的靶向分析和非靶向分析的前處理方法詳見表1。
1.2 質譜分析技術
質譜(MS)分析主要包括液相色譜-質譜聯用(LC-MS)和氣相色譜-質譜聯用(GC-MS),液相色譜又分為超高效液相色譜(UPLC)和高效液相色譜(HPLC)。非靶向性代謝組學的分析,色譜可選擇與飛行時間(TOF)、靜電軌道阱(Orbitrap)、離子阱飛行時間(IT-TOF)、四極桿-飛行時間(Q-TOF)等質譜聯用;靶向性代謝組學的分析,色譜可與三重四極桿(QQQ)或四極桿離子阱(Q-Trap)等質譜串聯,用多反應監測器(MRM)進行檢測。大氣固體分析探針(Atmospheric solids analysis probe,ASAP)結合質譜技術(ASAP-MS)采用毛細管吸取的方式進樣,操作簡單,可用于較復雜物質的離子檢測,例如鼠的尿液以及狗和鼠的膽汁,能有效地將樣品來源歸類,但其產生的數據不如液質和氣質全面,可以用于樣本篩選方面[26]。近年來逐漸發展了高集成和高通量的微流控芯片與質譜聯用的方法,林金明等[27]在這方面做了很多工作,將微流控芯片與電噴霧質譜聯用技術相結合用于細胞分析及藥物動力學的研究,使細胞藥物代謝研究和細胞間信號傳遞的研究深化到分子水平。

表1 生物樣本的質譜代謝組學研究案例Table 1 The cases of metabolomics based on mass spectrometry of biological samples
(續表1)

代謝組學類型樣本前處理方法分析方法數據處理方法參考文獻非靶向分析(尿液代謝物)尿液稀釋UPLC-MSPCA,ROCOPLS-DA相關性分析[2,14,33-34]非靶向分析(尿液代謝物)尿液衍生化處理GC-MSPCA[21]非靶向分析(尿液代謝物)尿液毛細管吸取ASAP-MSPCA[26]非靶向分析(尿液代謝物)尿液蛋白沉淀、稀釋CE-ESI-MS-TOFPCAOPLS-DA[35]非靶向性分析肝臟組織甲醇-水(1∶1)水提,二氯甲烷-甲醇(3∶1)提取,甲醇-水(1∶1)復溶UPLC-MSHSST3柱PCA[16]非靶向性分析動脈斑塊組織甲醇-水(1∶1)水提,二氯甲烷-甲醇(3∶1)提取有機物UPLC-MS水提物-HILIC柱有機物-反相柱PCAOPLS-DA變異系數文氏圖代謝通路分析[17-18]非靶向性分析動物、人體組織甲醇-水(1∶1)水提,二氯甲烷-甲醇(3∶1)提取,甲醇-水(1∶1)復溶UPLC-MS水提物-HILIC柱有機物-C18柱HSST3柱PCAPLS-DA相關性分析變異系數文氏圖代謝通路分析[14-15]非靶向分析(膽汁代謝物)膽汁毛細管吸取ASAP-MSPCA[26]非靶向分析(細胞代謝物)細胞細胞制備后用生物樣品均質器提取代謝物干冰甲醇提取聲波降解法UPLC-MSHILIC柱PCA相關性分析代謝通路分析比較代謝物分析[14,36-37]非靶向分析(腦脊液代謝物)腦脊液甲醇提取UPLC-MSHPLC-MS相關性分析PCA[14]非靶向分析(唾液代謝物)唾液除蛋白(氣質需衍生化)UPLC-MSGC-MS相關性分析PCA[38-39]非靶向分析(羊水代謝物)羊水丙酮除蛋白并衍生化GC-MSPCAPLS-DAOPLS-DA代謝通路分析[22,40]非靶向分析(眼淚代謝物)眼淚80%冷甲醇提取HPLC-MSIDALC-MS/MSPCA代謝富集分析代謝通路分析[41-42]非靶向分析(糞便代謝物)糞便水提取,乙腈提取,合并上清液,同位素丹磺酰氯衍生化;甲醇提取UPLC-MSHPLC-QTOF-MSPCAPLS-DAOPLS-DA[19-20]非靶向分析(頭發代謝物)頭發氫氧化鈉水解、酸中和、冷凍干燥、甲醇水混合物再提、蒸干、衍生化、棄去水層;甲醇鹽酸混合物提取、超聲、過濾GC-MSUPLC-ESI-TOFMSROC曲線T檢驗PCAOPLS-DA[23-24]
1.3 數據處理
代謝組學常用的數據處理軟件有儀器公司的軟件(例如Waters的MarkerLynx,Thermo Fisher的SIEVE,Agilent的MassHunter,Applied Biosystems的Marker View等)和一些公開免費的數據分析網站(例如XCMS[2,34,36,43-46],MzMATCH[36,47],MZmine[8,45-46],Metalign[45-46],Metaboanalyst[22,41,48],MeltDB[46,48],metaP-Server[48-49]等),還有專門的數據處理軟件(SIMCA-P,Matlab,SAS等)。代謝組學常用的數據庫主要有一般化學數據庫(如PubChem)、代謝通路數據庫(如BioCyc)、代謝物數據庫(HMDB)、質譜數據庫(如NIST)等[50]。Xia等[48]制定了Metaboanalyst軟件處理代謝組學數據的標準,在格式化和上傳數據、處理和標準化數據、鑒定有顯著性的物質、利用代謝富集分析和代謝通路分析來闡明可能的生物機理方面提供了一系列流程,并且對比了Metaboanalyst,MeltDB,metaP-Server,SIMCA-P,SAS軟件在處理代謝組學數據時的優缺點,發現Metaboanalyst網站在處理數據時應用較廣,可以進行單變量分析、多變量分析、聚類分析、富集分析和通路分析。MeltDB網站則可以結合其它組學的數據。XCMS網站可以結合其它物質的數據庫。各個代謝組學軟件或網站的特點詳見表2。
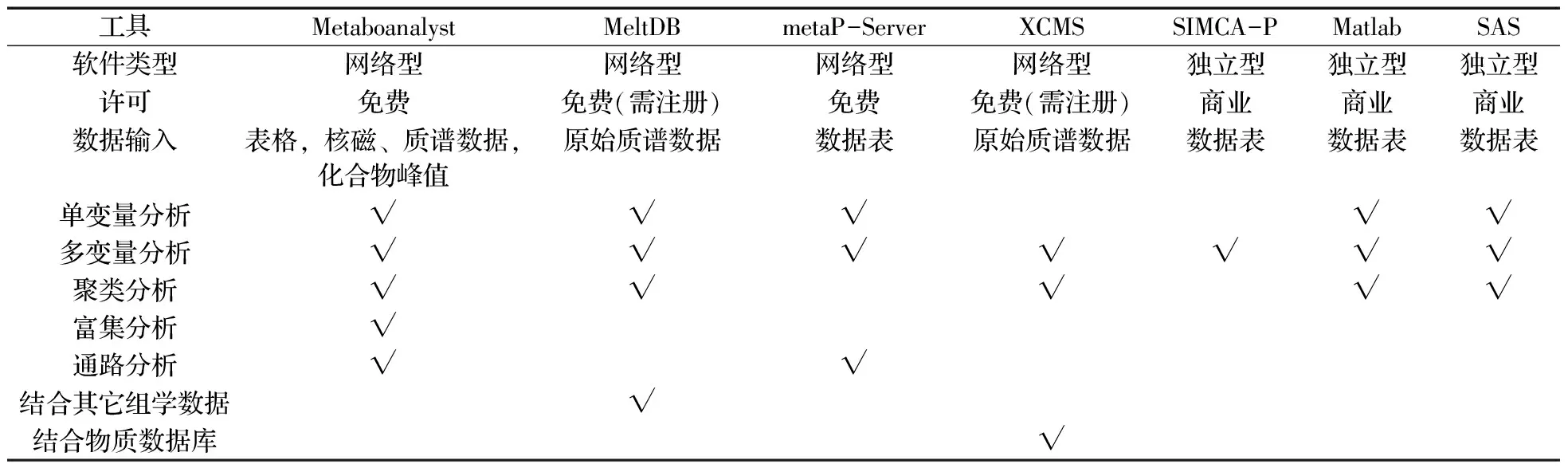
表2 代謝組學常用分析軟件的特點Table 2 The characteristics of analysis software of metabolomics research
“√” means that the specific software can analyze the statistic analysis in column(“√”代表某種軟件下可以進行縱列的統計分析)
2 基于質譜分析的代謝組學應用研究進展
基于質譜分析的代謝組學在醫學領域的應用主要包括疾病診斷、藥物研發、營養學、毒理學、運動醫學等領域,表3總結了這幾大領域主要關注的問題。
2.1 疾病診斷領域
傳統的疾病診斷方法是檢測單一的一個或幾個指標,生物體的作用是一個協同的整體,評價單一的指標具有片面性。代謝組學是從整體上評價機體的代謝變化,較為全面。在實驗設計上有橫斷面設計,比較疾病組和正常組,也有縱向設計,干預前后或不同時間點的代謝研究。Vorkas 等[17-18]利用UPLC-MS法對腹主動脈瘤、頸動脈狹窄性斑塊、股動脈狹窄性斑塊病人外科手術取的組織進行了非靶向性分析,發現這幾類患者組織的代謝物均能得以區分,發現動脈粥樣硬化相關的代謝包括膽固醇代謝、嘌呤代謝、嘧啶代謝、神經酰胺代謝。Roy 等[8]利用UPLC-MS分析了非靶向性的血漿代謝物和靶向性的代謝物,定量了8種氨基酸和8種酰基肉毒堿,被認為是發生心血管疾病風險的生物標志物。Fattuoni 等[22]研究了孕婦穿刺的羊水代謝物,有助于監測病毒性感染孕婦和胎兒的早期診斷。Musharraf 等[51]用GC-MS法研究了急性骨髓性白血病、急性淋巴細胞白血病、再生障礙性貧血和對照組病人的血清樣本,發現了27個顯著不同的代謝物,患者的脂肪酸代謝失調,這有助于早期診斷急性白血病。Ciavardelli 等[52]用代謝組學的方法研究了阿爾茨海默氏癥患者的代謝物,與對照組及輕度認知功能的患者相比,阿爾茨海默氏患者的一部分血漿酰基肉堿含量較低,前額葉灰質體積較少,并且存在認知功能障礙。Yokoi 等[53]用GC-MS法研究了大鼠模型的糖尿病診斷,發現自發型糖尿病大鼠和正常大鼠相比,天冬酰胺、谷氨酰胺、甘油、犬尿氨酸、甘露糖、牛磺酸、蘇氨酸和色氨酸有顯著性差異,自發型糖尿病大鼠在12周齡以后色氨酸代謝途徑的代謝產物(色氨酸和犬尿氨酸)明顯降低,色氨酸及其代謝產物是糖尿病前期潛在的生物標志物,色氨酸的代謝可能是糖尿病治療干預的一個潛在靶點。Zhu 等[54]利用代謝組學研究了二型糖尿病和糖尿病腎病患者的磷脂代謝物,發現了7大類磷脂中的18種顯著的代謝標志物,其中3種是二型糖尿病的代謝標志物,8種是糖尿病腎病的代謝標志物,7種是二者共同的代謝標志物。有2個標志物,PI C18∶0/22∶6和SM dC18∶0/20∶2可用于區分健康人、二型糖尿病和糖尿病腎病人。Huang 等[55]利用代謝組學研究了早期二型糖尿病腎病的代謝標志物,發現了與其相關的6個臨床指標、40種代謝物和5個基因。Cordeiro 等[72]利用電噴霧電離質譜(ESI-MS)研究了臨床病人卵泡液中的脂質代謝,發現了對照組的代謝物中有磷脂酰甘油磷酸鹽、磷脂酰膽堿、磷脂酰絲氨酸、磷脂酰肌醇二磷酸,子宮內膜異位癥病人的代謝物中鞘脂類、磷脂酰膽堿含量高,子宮肌瘤病人的代謝物有較高豐度的鞘脂類、磷脂酰膽堿,有助于潛在生物標志物的發現。Zhao 等[73]利用代謝組學方法研究了多囊卵巢綜合征患者的卵泡液代謝物變化,認為線粒體功能、氧化還原電勢、卵丘細胞的氧化應激是促進卵母細胞受精前生長的必要因素,多囊卵巢綜合征患者可能存在線粒體功能紊亂、氧化還原電位不平衡和卵丘細胞的氧化應激增加,從而為多囊卵巢綜合征發病機理的研究提供了依據。
2.2 藥物研究領域
代謝組學在藥物領域的研究主要有藥物品質評價、藥物效果及作用機制、藥物吸收代謝、藥物生物轉化代謝、藥物活性成分研究等。本實驗室前期在中藥藥效評價和藥代動力學方面做了一些工作。Liang等[56]利用代謝組學評價了中藥復方對大鼠心肌梗塞的作用,發現治療效果優于人參和單參單方。由于中藥復方在心肌能量新陳代謝方面的潛在藥效,尿液中三羧酸循環及磷酸戊糖途徑的改變表明其可能減少心肌梗塞中心臟的損傷。Xie等[10]研究了大鼠服用知母后的藥代動力學,定量了21種代謝中間產物,涉及的代謝通路有水解作用、甲基化作用、去糖基化、糖脂化作用、糖苷結合、硫酸化、脫水作用和異構化反應,揭示了知母藥片抗糖尿病的主要多酚組分。Huang等[57]用HPLC-ESI-MS方法研究了異丙嗪和咖啡因聯合治療運動病和抵抗異丙嗪的功效,發現了10多個藥物代謝中間產物。Qin等[58]結合血漿代謝組學和脂質組學在大鼠模型上研究了中藥清開靈的代謝,結果發現清開靈1 h有15種差異顯著的代謝物(p<0.05),2 h有19種差異顯著的代謝物(p<0.05),其中與退熱功效有關的包括氨基酸、磷脂、溶血磷脂酰膽堿,涉及的代謝通路有氨基酸代謝和脂質代謝。Li等[59]用UPLC-MS代謝組學方法研究了人參皂苷Rg1和Rg2對阿爾茨海默氏癥患者的記憶力下降的減緩效果,研究結果顯示,在阿爾茨海默氏癥大鼠模型發現了11個潛在的生物標志物,與溶血磷脂酰膽堿、次黃嘌呤和鞘脂代謝有關,在給予人參皂苷Rg1和Rg2后這些代謝物水平有恢復,通過腦代謝通路平衡而改變認知功能和形態學變化。Tian等[20]利用大鼠糞便代謝物研究了太子參、丹參、沙參、苦參4味藥材對于動脈粥樣硬化的治療效果,在大鼠糞便中發現了16個潛在的生物標志物,其中5個在服用混合中藥后發生改變,涉及相關的代謝通路有脂代謝、膽固醇代謝、能量周期和炎癥反應。劉琦等[60]利用中醫方證代謝組學研究了男仕膠囊對腎陽虛證大鼠的治療效果,研究確定了11個與腎陽虛證大鼠模型相關的代謝標記物,涉及類固醇激素的生物合成、色氨酸代謝和酪氨酸代謝等途徑,并發現了男仕膠囊治療腎陽虛證的8種藥效物質基礎。秦昆明等[74]綜述了常見中藥材的藥效作用機制和常見中藥復方的配伍規律及作用機制。吳昱錚等[75]綜述了典型中藥材對于疾病的代謝組學研究。張愛華等[76]綜述了代謝組學在中藥藥理、中醫證候、中醫方證、中藥安全性評價幾個方面的應用。
2.3 營養學領域
代謝組學在營養學領域的研究主要包括活性成分的功能研究、膳食模式的評價、營養素需要量的研究、營養代謝的研究、營養流行病學研究等。Liu等[61]利用代謝組學的手段研究了不同季節綠茶的代謝表型;Lee等[62]研究了綠茶對大鼠高脂血癥的預防效果,分析了大鼠血清和肝臟代謝組學,發現綠茶能減緩由于高脂飼料誘導的線粒體β氧化異常;Perez-Cornago等[63]利用代謝組學手段評價了對超重、肥胖人群能量控制飲食干預后代謝的變化,發現總飽和脂肪酸、棕櫚酸、硬脂酸、單不飽和脂肪酸降低;Martin等[64]研究了不同喂養模式嬰兒的代謝特點,發現用配方奶粉喂養嬰兒,在其糞便中會發現由蛋白衍生成的短鏈脂肪酸和氨基酸含量較高,尿液代謝組學揭示了配方奶粉中膳食蛋白質的細菌繁殖過程和蛋白含量增加刺激宿主蛋白質代謝之間的關系。Zhao等[65]利用代謝組學和脂質組學研究了中藥復方對糖尿病腎病的療效,發現中藥復方可以保護糖尿病腎病患者的腎臟,發現服用中藥復方的糖尿病腎病患者能顯著抑制有機毒物的升高及磷脂的減少。
2.4 毒理學領域
由于中毒或代謝失常而引起機體功能紊亂會反映在血液、尿液或組織代謝物中,代謝組學在毒理學方面可以應用在毒性生物標志物篩選、食品毒品毒理和食品安全研究等。Shi等[66]利用代謝組學手段研究了丙烯酰胺的慢性暴露毒性,發現了3種新的生物標志物,認為丙烯酰胺會使脂類和氨基酸代謝紊亂、誘發氧化應激。Yu等[67]用代謝組學研究了全氟辛酸的暴露會引起個體代謝通路的改變,主要影響氨基酸、脂類、碳水化合物和能量代謝,在腦中和肝臟中分別發現有10和18種生物標志物。在大腦中,全氟辛酸會影響神經遞質的濃度,包括5-羥色胺、多巴胺、去甲腎上腺素、谷氨酸鹽,在肝臟中,因全氟辛酸引發肝中毒,脂類代謝方面出現β脂質氧化、飽和及不飽和脂肪酸的生物合成,花生四烯酸代謝的改變提示了全氟辛酸可能引起肝臟的炎癥反應。Qi等[68]在大鼠模型上利用代謝組學方法評價了轉基因大米的安全性,結合腸道菌群、血液指標和尿液代謝物綜合分析,發現與食用非轉基因大米的大鼠相比,服用轉基因大米的大鼠的代謝物存在顯著性差異,但與對照組相比差異不顯著。
2.5 運動醫學領域
代謝組學在運動領域常用的研究方向有競技運動訓練方面的問題、運動營養問題、急性慢性運動對代謝的影響、興奮劑問題、運動對慢性疾病的影響等。Wang等[69]研究了不同運動強度下的尿液代謝變化,結果發現隨著運動強度和運動量的增加,乳酸、丙氨酸、三甲胺、丙二酸、牛磺酸、甘氨酸水平下降,隨后隨著運動量下降,代謝物無顯著變化。通過全面評估不同運動形式、不同運動強度、不同運動時間以及不同運動頻率對機體所產生的代謝物變化來指導科學合理的運動訓練,可以對運動訓練方案的設計、運動訓練過程的監控和運動效果評價等提供更全面的證據。Mukherjee等[70]用DNA芯片技術和代謝組學的方法研究了老齡化的代謝紊亂問題,選取運動員和非運動員兩組,給予不同的運動強度,發現每周連續5 h以上高強度運動員組的基因、能量代謝、脂類代謝、胰島素信號、心血管功能與非運動員組相比均有顯著差異。Yan等[71]用代謝組學手段監測了力量型訓練下運動員生理狀態的變化,發現不同訓練年限的運動員、不同訓練時間的代謝物能被顯著區分,顯著不同的代謝物有丙氨酸、乳酸、焦谷氨酸、半胱氨酸、谷氨酸、檸檬酸、游離脂肪酸、纈氨酸、苯丙氨酸、酪氨酸、谷氨酸鹽、苯基丙氨酸、酪氨酸等,涉及到的代謝有糖代謝、氧化應激、能量代謝、脂代謝、氨基酸代謝。
3 展 望
靶標代謝組學和非靶向代謝組學(發現代謝組學)已成為代謝組學應用的兩種主要模式。靶標分析具有定量準確、刻畫精細、先驗知識充分利用的特點,但往往只關注于其中的一點而漏掉其它重要的信息,不具有整體性,過于依賴基于先驗知識所預設的目標分析物的合理性。代謝指紋譜屬于無歧視的整體表征方法,不要求預先設定目標分析物,可以發現未知生物標志物,但先驗知識利用率低,分析方法未針對特定的目標分析物進行優化,難以做到準確的定量分析,且生物樣本中常見的高豐度以及質譜離子化效率高的代謝物往往容易檢查并可能對某些低豐度、質譜信號響應性差的代謝物檢測造成干擾,因此代謝指紋譜的無歧視分析在實際上難以做到,某些重要的低豐度特征信息可能丟失,所發現潛在代謝標志物的針對性較差。因此,對于生物復雜系統的表征需要將代謝指紋譜分析(非靶向代謝組學)與多靶標代謝物定量分析(靶向代謝組學)相結合,整體表征與局部表征相結合,定性與定量分析相結合,代謝分析與臨床生化分析相結合,構建多學科參與、多種分析方法整合、大數據分析的臨床定量代謝組學平臺[77]。定量代謝組學平臺既具有全局的視野和對代謝輪廓譜的整體把握,又能實現對特定的代謝途徑和生物標志物的精準分析,在多因素復雜疾病和生物復雜系統的研究中具有廣闊的應用前景。在分析方法的發展方面,如何使得代謝指紋譜(輪廓譜)測定的代謝物更加全面,如何使得靶標代謝物分析更加精準,特別是低豐度、強極性等難檢出化合物的分離分析等,仍然值得重點關注。在代謝組學分析方法的標準化方面,目前Nature Protocol已陸續發表了尿液、腦脊液以及大部分實體組織和細胞樣本代謝組學分析的操作規程[14-15,21,33,37],但一些特殊的樣本,如精液、卵泡液、羊水、眼淚、糞便、頭發等樣本,目前還缺乏公認度較高的標準操作規程。對于細胞代謝物的亞細胞代謝組學是將宏觀與微觀相結合的一個新視角。在數據分析方面,包括非監督分析和有監督分析方法,近年來人工神經網絡等一些非線性分析方法在代謝組學中的應用值得關注。如何將代謝組學數據與其它組學技術相結合是一大難點,大數據的挖掘將有助于發現新的有價值信息。隨著科學技術的發展,大數據時代的到來,將各種組學技術相整合,必將促進分析測試技術的大發展。
[1] Nicholson J K,Lindon J C.Nature,2008,455:1054.
[2] Gika H G,Zisi C,Theodoridis G,Wilson I D.J.Chromatogr.B,2016,1:15-25.
[3] Klepacki J,Klawitter J,Karimpour-Fard A,Thurman J,Ingle G,Patel D,Christians U.Clin.Biochem.,2016,49(13/14):955-961.
[4] Patti G J,Yanes O,Siuzdak G.Nat.Rev.Mol.CellBiol.,2012,13(4):263-269.
[5] Millan L,Sampedro M C,Sanchez A,Delporte C,Van Antwerpen P,Goicolea M A,Barrio R J.J.Chromatogr.A,2016,8:67-77.
[6] Zhao L J,Zhao A,Chen T L,Chen W L,Liu J W,Wei R M,Su J,Tang X L,Liu K,Zhang R,Guo X X,Panee J,Qiu M F,Jia W.J.ProteomeRes.,2016,15(7):2327-2336.
[7] Ranninger C,Schmidt L E,Rurik M,Limonciel A,Jennings P,Kohlbacher O,Huber C G.Anal.Chim.Acta,2016,930:13-22.
[8] Roy C,Tremblay P Y,Bienvenu J F,Ayotte P.J.Chromatogr.B,2016,14:40-49.
[9] Zhao C X,Xu G W.J.Anal.Sci.(趙春霞,許國旺.分析科學學報),2014,30(5):761-766.
[10] Xie Y Y,Wang X M,Wang S H,Wang Y M,Tian H F,Yuan Y S,Li H Y,Liang Q L,Luo G A.J.Chromatogr.B,2015,1026:134-144.
[11] Cheng Y,Liu Y M,Huang F J,Chen T L,Zheng X J,Zhao A H,He P G,Jia W.Chem.J.Chin.Univ.(成玉,劉玉敏,黃鳳杰,陳天璐,鄭曉皎,趙愛華,何品剛,賈偉.高等學校化學學報),2013,34(1):77-83.
[12] Want E J,Coen M,Masson P,Keun H C,Pearce J T,Reily M D,Robertson D G,Rohde C M,Holmes E,Lindon J C,Plum R S,Nicholson J K.Anal.Chem.,2010,82(12):5282-5289.
[13] Sarafian M H,Lewis M R,Pechlivanis A,Ralphs S,McPhail M J,Patel V C,Dumas M E,Holmes E,Nicholson J K.Anal.Chem.,2015,87(19):9662-9670.
[14] Yuan M,Breitkopf S B,Yang X,Asara J M.Nat.Protoc.,2012,7(5):872-881.
[15] Want E J,Masson P,Michopoulos F,Wilson I D,Theodoridis G,Plumb R S,Shockcor J,Loftus N,Holmes E,Nicholson J K.Nat.Protoc.,2013,8(1):17-32.
[16] Masson P,Alves A C,Ebbels T M,Nicholson J K,Want E J.Anal.Chem.,2010,82(18):7779-7786.
[17] Vorkas P A,Isaac G,Anwar M A,Davies A H,Want E J,Nicholson J K,Holmes E.Anal.Chem.,2015,87(8):4184-4193.
[18] Vorkas P A,Shalhoub J,Isaac G,Want E J,Nicholson J K,Holmes E,Davies A H.J.ProteomeRes.,2015,14(3):1389-1399.
[19] Su X L,Wang N,Chen D Y,Li Y N,Lu Y F,Huan T,Xu W,Li L J.Anal.Chim.Acta,2016,903:100-109.
[20] Tian F,Gu L,Si A Y,Yao Q B,Zhang X W,Zhao J H,Hu D D.J.Chromatogr.B,2015,15:140-148.
[21] Chan E C,Pasikanti K K,Nicholson J K.Nat.Protoc.,2011,6(10):1483-1499.
[22] Fattuoni C,Palmas F,Noto A,Barberini L,Mussap M,Grapov D,Dessi A,Casu M,Casanova A,Furione M,Arossa A,Spinillo A,Baldanti F,Fanos V,Zavattoni M.Clin.Chim.Acta,2016,460:23-32.
[23] Sulek K,Han T L,Villas-Boas S G,Wishart D S,Soh S E,Kwek K,Gluckman P D,Chong Y S,Kenny L C,Baker P N.Theranostics,2014,4(9):953-959.
[24] Tsutsui H,Maeda T,Min J Z,Inagaki S,Higashi T,Kagawa Y,Toyo’oka T.Clin.Chim.Acta,2011,412(11/12):861-872.
[25] Wang H,Liang X P,Luo G A,Ding M Y,Liang Q L.Mol.Biosyst.,2016,12(7):2257-2264.
[26] Twohig M,Shockcor J P,Wilson I D,Nicholson J K,Plumb R S.J.ProteomeRes.,2010,9(7):3590-3597.
[27] Li H F,Zhang Y,Lin J M.Sci.Sin.:Chim.(李海芳,張英,林金明.中國科學),2014,44(5):777-783.
[28] Kadar H,Dubus J,Dutot J,Hedjazi L,Srinivasa S,Fitch K V,Grinspoon S K,Nicholson J K,Dumas M E,Gauguier D.ArchBiochem.Biophys.,2016,597:12-20.
[29] Sen A,Knappy C,Lewis M R,Plumb R S,Wilson I D,Nicholson J K,Smith N W.J.Chromatogr.A,2016,3:141-155.
[30] Benton H P,Want E,Keun H C,Amberg A,Plumb R S,Goldfain-Blanc F,Walther B,Reily M D,Lindon J C,Holmes E,Nicholson J K,Ebbels T M.Anal.Chem.,2012,84(5):2424-2432.
[31] Hou W E,Zhong D M,Zhang P T,Li Y M,Lin M N,Liu G H,Yao M C,Liao Q F,Xie Z Y.J.Chromatogr.A,2016,15:207-217.
[32] Dunn W B,Broadhurst D,Begley P,Zelena E,Francis-McIntyre S,Anderson N,Brown M,Knowles J D,Halsall A,Haselden J N,Nicholls A W,Wilson I D,Kell D B,Goodacre R.Nat.Protoc.,2011,6(7):1060-1083.
[33] Want E J,Wilson I D,Gika H,Theodoridis G,Plumb R S,Shockcor J,Holmes E,Nicholson J K.Nat.Protoc.,2010,5(6):1005-1018.
[34] Wang Y,Caldwell R,Cowan D A,Legido-Quigley C.Anal.Chem.,2016,88(4):2243-2249.
[35] Garcia-Perez I,Lindon J C,Minet E.Bioanalysis,2014,6(20):2733-2749.
[36] Stipetic L H,Dalby M J,Davies R L,Morton F R,Ramage G,Burgess K E.Metabolomics,2016,12:75.
[37] Martano G,Delmotte N,Kiefer P,Christen P,Kentner D,Bumann D,Vorholt J A.Nat.Protoc.,2015,10(1):1-11.[38] Halama A,Kulinski M,Kader S A,Satheesh N J,Abou-Samra A B,Suhre K,Mohammad R M.J.Transl.Med.,2016,14(1):140.
[39] Martel J,Wu C Y,Hung C Y,Wong T Y,Cheng A J,Cheng M L,Shiao M S,Young J D.Nanoscale,2016,8(10):5537-5545.
[40] Dudzik D,Revello R,Barbas C,Bartha J L.J.ProteomeRes.,2015,14(3):1432-1444.
[41] Karamichos D,Zieske J D,Sejersen H,Sarker-Nag A,Asara J M,Hjortdal J.Exp.EyeRes.,2015,132:1-8.
[42] Chen L Y,Zhou L,Chan E C,Neo J,Beuerman R W.J.ProteomeRes.,2011,10(10):4876-4882.
[43] Telu K H,Yan X,Wallace W E,Stein S E,Simon-Manso Y.RapidCommun.MassSpectrom.,2016,30(5):581-593.[44] Mahieu N G,Genenbacher J L,Patti G J.Curr.Opin.Chem.Biol.,2016,30:87-93.
[45] Chang H Y,Chen C T,Lih T M,Lynn K S,Juo C G,Hsu W L,Sung T Y.PLoSOne,2016,11(1):e0146112.
[46] Niu W,Knight E,Xia Q,McGarvey B D.J.Chromatogr.A,2014,29:199-206.
[47] Daly R,Rogers S,Wandy J,Jankevics A,Burgess K E,Breitling R.Bioinformatics,2014,30(19):2764-2771.
[48] Xia J,Wishart D S.Nat.Protoc.,2011,6(6):743-760.
[49] Kastenmuller G,Romisch-Margl W,Wagele B,Altmaier E,Suhre K.J.Biomed.Biotechnol.,2011,1-7.
[50] Kong H W,Dai W D,Xu G W.Chin.J.Chromatogr.(孔宏偉,戴偉東,許國旺.色譜),2014,32(10):1052-1057.
[51] Musharraf S G,Siddiqui A J,Shamsi T,Naz A.Hematol.Oncol.,2016,1-7.
[52] Ciavardelli D,Piras F,Consalvo A,Rossi C,Zucchelli M,Di Ilio C,Frazzini V,Caltagirone C,Spalletta G,Sensi S L.Neurobiol.Aging,2016,43:1-12.
[53] Yokoi N,Beppu M,Yoshida E,Hoshikawa R,Hidaka S,Matsubara T,Shinohara M,Irino Y,Hatano N,Seino S.Metabolomics,2015,11(5):1277-1286.
[54] Zhu C,Liang Q L,Hu P,Wang Y M,Luo G A.Talanta,2011,85(4):1711-1720.
[55] Huang M,Liang Q L,Li P,Xia J F,Wang Y,Hu P,Jiang Z T,He Y X,Pang L Q,Han L D,Wang Y M,Luo G A.Mol.Biosyst.,2013,9(8):2134-2141.
[56] Liang X P,Chen X,Liang Q L,Zhang H Y,Hu P,Wang Y M,Luo G A.J.ProteomeRes.,2011,10(2):790-799.
[57] Huang M,Gao J Y,Zhai Z G,Liang Q L,Wang Y M,Bai Y Q,Luo G A.J.Pharm.Biomed.Anal.,2012,62:119-128.
[58] Qin L M,Zhang Z X,Guo M X,Zhang Q Q,Wang Q,Lu Z W,Zhao H Z,Liu Y H,Fu S,Wang M L,Gao X Y.Chem.Biol.Interact,2016,254:24-33.
[59] Li N J,Liu Y,Li W,Zhou L,Li Q,Wang X Q,He P.J.GinsengRes.,2016,40(1):9-17.
[60] Liu Q,Zhao H W,Zhang A H,Sun H,Zhou X H,Nan Y,Kong L,Guan Y,Zou S Y,Ma Z H,Wang X J.J.Chin.Mater.Med.(劉琦,趙宏偉,張愛華,孫暉,周曉航,南洋,孔玲,關瑜,鄒士玉,馬忠華,王喜軍.中國中藥雜志),2016,41(15):2901-2914.
[61] Liu J W,Zhang Q F,Liu M Y,Ma L F,Shi Y Z,Ruan J Y.J.Agric.FoodChem.,2016,64(16):3302-3309.
[62] Lee L S,Choi J H,Sung M J,Hur J Y,Hur H J,Park J D,Kim Y C,Gu E J,Min B,Kim H J.Mol.Nutr.FoodRes.,2015,59(4):784-794.
[63] Perez-Cornago A,Brennan L,Ibero-Baraibar I,Hermsdorff H H,O’Gorman A,Zulet M A,Martinez J A.J.Physiol.Biochem.,2014,70(2):593-602.
[64] Martin F P,Moco S,Montoliu I,Collino S,Da Silva L,Rezzi S,Prieto R,Kussmann M,Inostroza J,Steenhout P.Pediatr.Res.,2014,75(4):535-543.
[65] Zhao T,Zhang H J,Zhang X L,Zhao T T,Lan H Y,Liang Q L,Luo G A,Li P.J.Ethnopharmacol.,2015,166:31-41.
[66] Shi H D,Hu L Y,Chen S,Bao W,Yang S,Zhao X J,Sun C H.Xenobiotica,2016,27:1-11.
[67] Yu N Y,Wei S,Li M Y,Yang J P,Li K,Jin L,Xie Y W,Giesy J P,Zhang X W,Yu H X.Sci.Rep.,2016,6:1-10.
[68] Qi X Z,Chen S Y,Sheng Y,Guo M Z,Liu Y F,He X Y,Huang K L,Xu W T.FoodChem.Toxicol.,2015,76:1-10.
[69] Wang F Q,Han J,He Q,Geng Z F,Deng Z W,Qiao D C.J.Anal.MethodsChem.,2015,1-9.
[70] Mukherjee K,Edgett B A,Burrows H W,Castro C,Griffin J L,Schwertani A G,Gurd B J,Funk C D.PLoSOne,2014,9(3):e92031.
[71] Yan B,A J,Wang G J,Lu H L,Huang X P,Liu Y,Zha W B,Hao H P,Zhang Y,Liu L S,Gu S H,Huang Q,Zheng Y T,Sun J G.J.Appl.Physiol.,2009,106(2):531-538.
[72] Cordeiro F B,Cataldi T R,Perkel K J,do Vale Teixeira da Costa L,Rochetti R C,Stevanato J,Eberlin M N,Zylbersztejn D S,Cedenho A P,Turco E G.J.Assist.Reprod.Genet.,2015,32(12):1817-1825.
[73] Zhao H C,Zhao Y,Li T J,Li M,Li J S,Li R,Liu P,Yu Y,Qiao J.FreeRadic.Biol.Med.,2015,86:295-307.
[74] Qin K M,Wang B,Chen L W,Zhang M S,Yang G M,Su Y C,Cai B C.Chin.Mater.Med.(秦昆明,王彬,陳林偉,章茂森,楊光明,束雅春,蔡寶昌.中國中藥雜志),2014,39(16):3010-3017.
[75] Wu Y Z,Wang G J,Hao H P.Chin.Pharm.Univ.(吳昱錚,王廣基,郝海平.中國藥科大學學報),2014,45(2):129-135.
[76] Zhang A H,Wang X J.Mod.Tradit.Chin.Med.Mater.Med.(張愛華,王喜軍.世界科學技術_中醫藥現代化),2013,15(4):643-647.
[77] Luo G A,Wang Y M,Liang Q L,Liu Q F.SystemsBiologyforTraditionalChineseMedicine.New York:A John Wiley & Sons,2012:191-194.
Advance in Metabolomics Based on Mass Spectrometry
REN Xiang-nan,LIANG Qiong-lin*
(Department of Chemistry,Analysis Center,Tsinghua University,Beijing 100084,China)
Mass spectrometry(MS) is one of the most important analytical technologies for metabolomics.In recent years,metabolomics based on MS has been widely used in disease diagnosis,drug effect research,nutrition,toxicology,sports medicine,etc.This paper reviews the sample pretreatment,analytical tools,data processing method and main application fields in the past five years.The development tendency of metabolomics in future is also proposed.
mass spectrometry(MS);metabolomics;advance;review
2016-09-30;
2016-11-28
國家自然科學基金項目(U1333132,81230079);科技部國際科技合作項目(2011DFA31860);國家科技重大專項(2014ZX09304307001,2014ZX09201022-004,2014ZX09201022-005)
10.3969/j.issn.1004-4957.2017.02.002
O657.63;O629.73
A
1004-4957(2017)02-0161-09
*通訊作者:梁瓊麟,研究方向:生命分析化學,Tel:010-62772263,E-mail:liangql@tsinghua.edu.cn

